蛋白质是生命活动的直接执行者,要深入认识复杂的生命活动必然要在整体、动态以及作用网络的水平上开展蛋白质组学研究。因此,以细胞内全部蛋白质及其活动方式为研究对象的蛋白组学成为后基因时代生命科学的重要研究内容[ 1 ]。传统的蛋白组学研究方法主要以双向凝胶电泳技术为主[ 2 ],但是它在膜蛋白样品溶解性、低丰度蛋白质检测、极酸极碱蛋白质及低分子量和高分子量蛋白质分离等方面仍存在着很大的局限性[ 3 ]。稳定核素标记和高分辨率质谱(mass spectrometry,MS)[ 4 ]的发展使人们可以利用蛋白定量的方法获取细胞内某些特异的或互补的生物学信息[ 5 ]。细胞培养稳定核素标记(stable isotope labeling by amino acids in cell culture,SILAC)是一种新兴的基本无毒副作用的细胞培养标记方式,该技术结合高分辨率质谱开展的蛋白定量已经成为定量蛋白组学研究的重要工具[ 6 ]。本文就SILAC技术的发展历史、方法原理以及应用等方面作一综述。
1 SILAC技术的发展历史
稳定核素标记技术的出现依赖于质谱技术的发展及其在小分子化合物定量方面的应用。2000年左右,Chait等[ 7 ]以及随后的Lahm等[ 8 ]首次运用含有13C和15N的培养基标记微生物进行蛋白相对定量。2002年,Ong实验室改进了Chen等[ 9 ]提出的氨基酸核素标记(amino acid-coded mass tagging,AACT)技术,建立了SILAC法[ 10,11 ]。在之后的发展中,SILAC技术的适用对象逐步扩大,从最初的哺乳动物细胞标记发展到酵母细胞[ 12 ]、植物细胞[ 13 ]、整体哺乳动物[ 14 ]、组织样品[ 15 ]、线虫[ 16 ]等众多研究领域。近年来,SILAC技术结合质谱、液质联用(high performance liquid chromatography-mass spectrometry,HPLC-MS)已经成为生物医学定量蛋白质的有效方法之一,无论在功能蛋白研究[ 17 ]还是整体蛋白质组的差异检测[ 18 ]均有着广泛的应用。
2 SILAC技术原理
SILAC技术是利用含有稳定核素的氨基酸进行代谢标记的蛋白质相对定量技术,其主要实验流程参见图 1。
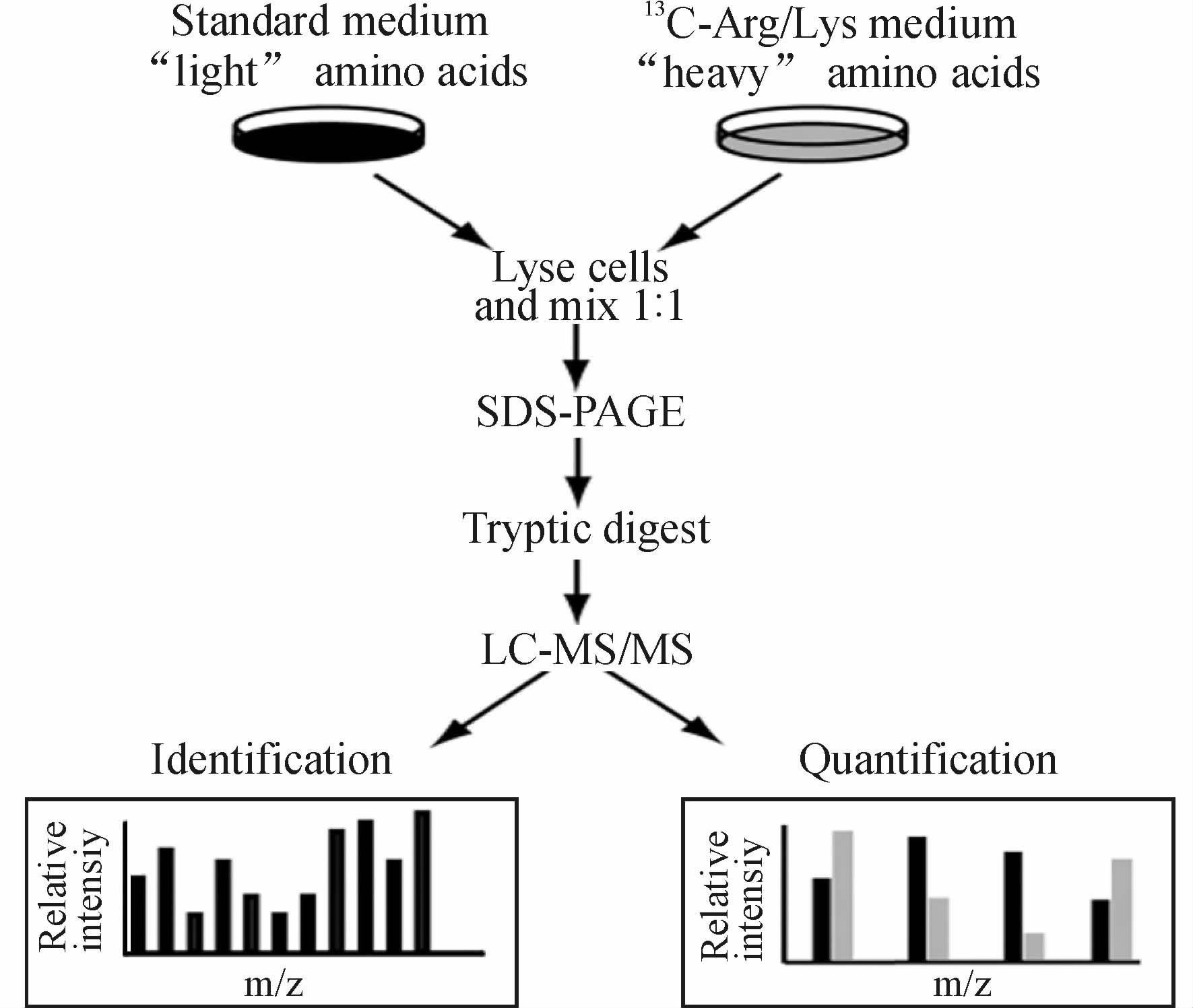
|
图 1 细胞稳定核素标记技术(SILAC)实验流程图[ 19 ] Fig 1 The workflow of stable isotope labeling of amino acids in cell culture(SILAC)[ 19 ] SDS-PAGE: Sodium dodecyl sulfate-polyacrylamide gel electrophoresis; LC: Liquid chromatography; MS: Mass spectrometry |
实验过程中,需要在实验组和对照组细胞的培养基中分别加入含有重型稳定核素(如13C、15N)或轻型稳定核素(如12C、14N)的必需氨基酸,大约经过5~10次传代后,测定重型核素的标记效率,确保其在95%以上。稳定核素标记可以造成质谱中蛋白碎片离子质量的偏移,以此区分序列相同而质量不同的肽段。通常使用的重型氨基酸有2H3-亮氨酸、13C6-精氨酸、13C615N4-精氨酸和13C6-赖氨酸等,与轻型氨基酸相比,它们能使碎片质量分别发生3、6、10、6的偏移。在细胞标记完成后,根据样品处理同一性原则,将实验组和对照组的细胞或者细胞蛋白等比例混合,经过SDS-PAGE或者亲和纯化等方式分离目的蛋白后酶解。酶解通常在胶内进行,常用的酶包括胰酶、Lys-C、Arg-C等,它们的切割位点为Arg或Lys的C端,因此保证酶解的片段均含有重型或轻型氨基酸,通过比较同一氨基酸序列轻、重稳定核素的丰度比即可完成蛋白质的相对定量[ 20,21 ]。
SILAC技术与传统的体内外标记方式相比,具有标记效率高、无细胞毒性、无需样品预处理、适于低丰度蛋白的鉴定等优点。重型核素标记细胞与轻型核素标记细胞的蛋白样品可以在实验的早期混合,避免了酶解消化以及各步分离纯化操作所产生的实验误差。SILAC技术的局限性主要体现在不能直接分析临床组织细胞样本、原代培养细胞标记效率低,细胞中存在精氨酸-脯氨酸转换等问题[ 22,23,24 ]。 3 SILAC技术的应用进展 3.1 SILAC研究蛋白质与其他分子的相互作用 3.1.1 蛋白质与DNA的相互作用
DNA位点特异性结合蛋白的性质是研究基因的表达调控的重要内容[ 25 ]。结合适当的亲和富集方式,SILAC技术可以在确定DNA位点特异性结合蛋白方面发挥独特的优势。Spruijt等[ 26 ]利用SILAC技术和DNA亲和富集,展示了完整的用于研究转录因子结合位点、单核苷酸多态性、甲基化CpG岛等功能性DNA元件的实验流程。Kappei等[ 27 ]采用DNA生物素亲和纯化以及SILAC质谱鉴定技术筛选出与端粒双链重复序列作用的蛋白HOT1。HOT1是端粒-端粒酶相互作用过程中第一个与端粒DNA结合的蛋白。它通过同源异型结构域识别端粒DNA序列,同时与有活性的端粒酶复合物以及卡哈尔体(CBs)相互作用,完成端粒酶的招募,从而正性调节端粒的伸长。这项发现促进了人们对端粒酶的招募和选择机制的理解,有助于解决端粒酶功能障碍等问题。 3.1.2 蛋白质与RNA的相互作用
与RNA生成和作用的蛋白质以及蛋白质相互作用因子是转录组学、RNA沉默等研究的热点问题[ 28 ]。Lee等[ 29 ]在研究蛋白质与EB病毒转录的小RNA分子EBER1相互作用时,通过噬菌体衣壳蛋白MS2、MBP与EBER1的亲和性,分离纯化EBER1-蛋白质复合物,发现了与EBER1相互作用的蛋白-异质核糖核蛋白D(AUF1/hnRNP D)。紫外交联显示AUF1的4个亚型无论在体内还是体外都能够与EBER1结合,且以p40亚型结合能力最强。在被EB病毒感染的细胞中,电泳迁移数据说明EBER1能够与A/U富集元件ARE竞争结合AUF1 p40蛋白亚型,扰乱含有ARE元件的RNA与AUF1的动态平衡,破坏细胞的正常生理活动。该文验证了MS2发卡适体捕获EBER1-核糖核酸蛋白质复合物(RNPs)的可行性,首次发现AUF1蛋白能与EBER1相互作用,有助于人们理解EBER1表达与细胞癌症表型之间的关联。 3.1.3 蛋白质与蛋白酶体之间的相互作用
研究蛋白质相互作用的方法有酵母双杂交、串联亲和纯化、免疫共沉淀、蛋白质芯片、等离子表面共振(SPR)以及噬菌体展示等多种技术[ 30 ],而SILAC技术结合这些传统方法可以使定性信息转化为定量信息。Kaake等[ 31 ]利用体内串联亲和纯化交联蛋白质复合体的定量分析(QTAX)策略以及13C15N-Lys/Arg稳定核素标记技术鉴定了酵母菌细胞周期特异性蛋白与26S蛋白酶体的相互作用网络。数据基于SILAC比率的显著性、细胞周期特异性以及蛋白功能等多个指标进行矢量聚类,最终确定了20组功能显著的蛋白质-蛋白质相互作用(PIPS),其中3组PIPS的功能与细胞周期密切相关。20组PIPS整理后得到36个与细胞功能相关的蛋白子集、9个组成蛋白复合物的子集以及26个信号通路相关的蛋白子集。该文对蛋白质-26S蛋白酶体作用网络进行了大规模分析,为人们研究细胞周期调节过程中蛋白酶体的动态变化提供了重要信息。 3.1.4 蛋白质与小分子药物的相互作用
经过条件摸索和改进之后,结合亲和富集和质谱的SILAC技术成为一种适合于药物靶点发现的蛋白定量方法,该方法无需考虑靶蛋白的丰度以及药物-靶蛋白结合力的强弱,可以直接通过与小分子作用的蛋白丰度比值确定靶点[ 32,33 ]。Daub等[ 34 ]及Breitkopf等[ 35 ]在人类慢性髓系白血病细胞K562中转入Bcr-Abl融合基因,并利用重型核素标记,用以研究伊马替尼处理后细胞内蛋白与连有蛋白激酶抑制剂的树脂亲和力的改变,同时定量分析树脂保留蛋白磷酸化程度的变化情况。实验将VI16832、purvalanol B、bisindolylmaleimide X、AX14596和SU6668的药物分子-微珠按一定比例混合成非选择性激酶抑制剂混合物,通过强阳离子交换-TiO2层析富集磷酸化蛋白。结果显示,伊马替尼可以和Bcr-Abl蛋白激酶的酪氨酸、丝氨酸、苏氨酸等磷酸化位点结合,抑制Bcr-Abl蛋白激酶与癌蛋白的相互作用。在伊马替尼处理的细胞组中,Syk、Btk、Fer以及FAK激酶磷酸化位点的SILAC比率比对照组降低了约3倍,揭示伊马替尼除了抑制已知的Bcr-Abl、Syk激酶等靶点外,还会影响Btk、Fer、FAK激酶以及下游的黄素腺嘌呤二核苷酸合成酶等蛋白的磷酸化,这些蛋白可能成为Bcr-Abl激酶介导的肿瘤发生过程中的潜在药物靶点。
在研究蛋白质与小分子的相互作用时,采用不同的亲和纯化方式可以分离提取特异性作用蛋白。除此之外,SILAC技术可以直接用于反映不同的实验条件对蛋白表达量的影响,例如:不同的细胞品系[ 36 ]、不同的细胞培养条件[ 37 ]、药物处理[ 38,39 ]、RNA干扰[ 40 ]、基因敲除[ 41 ]等造成的蛋白表达差异。 3.2 SILAC研究蛋白的翻译后修饰过程 3.2.1 磷酸化修饰
磷酸化修饰是活细胞中最基本的控制细胞信号传导的机制。磷酸化蛋白富集纯化常用的方式有:强阳离子交换(SCX)、TiO2亲和层析、固定化金属亲和层析(IMAC)以及抗磷酸化酪氨酸抗体亲和纯化等[ 42,43 ]。Zanivan等[ 44 ]运用哺乳动物整体SILAC标记技术,结合高分辨率质谱对不同期皮肤癌的蛋白组以及磷酸化差异进行分析。基于GO分析得到了与细胞黏附、蛋白激酶活性以及转录因子活性相关的蛋白质和磷酸化位点。实验总结了与皮肤癌各个阶段相关的MAPK、CDK以及GSK3 等特异性蛋白激酶的变化情况。文中尤其强调了PAK4-PKC通路可以在鳞状细胞癌中提高SRC激酶的活性,进而改变鳞状细胞癌的侵袭力,这一发现可以为皮肤鳞状细胞癌的抗癌研究提供新的靶点。文中大规模分析了不同癌症时期的蛋白组及磷酸化水平,得到的蛋白质组功能以及定量信息为人们研究肿瘤进程提供了有用线索。 3.2.2 甲基化修饰
组蛋白的翻译后修饰在染色体的稳定以及基因转录调控中发挥着重要作用,SILAC技术结合LC-MS可以根据肽段的质量和保留时间,较好地区分单甲基化以及二、三甲基化等多种甲基化形式[ 45,46 ]。Jung等[ 47 ]利用重型赖氨酸Lys-8标记Suz12缺失的小鼠胚胎干细胞,在LTQ-Orbitrap质谱仪中结合碰撞诱导解离(CID)和电子转移解离裂解(ETD)对H3.2和H3.3蛋白进行分析。质谱数据对于大部分已知的H3.2、H3.3甲基化修饰的准确反映,揭示了此方法在甲基化研究方面的高灵敏度和特异性。实验首次发现H3K27ac/K36ac在哺乳动物胚胎干细胞中同时出现,作者推测两者的协同作用可能会加强转录激活。在Suz12缺失组中,作者发现Lys-27位点存在相互拮抗的甲基化/乙酰化修饰,且液相色谱峰显示K27me2/K36me2的亚型在甲基化位点中占有很大比例。实验中,作者比较了SILAC质谱定量和蛋白质印迹检测组蛋白甲基化的优劣,在质谱定量中ETD、CID相结合能够增加肽段的检全率,同时利用反相高效液相色谱法分离相对分子质量相同的蛋白亚型能够增强实验的准确性,而蛋白质印迹在检测蛋白甲基化、乙酰化等多个翻译后修饰共存时存在较大的局限,这一比较结果促进了SILAC质谱定量在组蛋白研究中更广泛的应用。 3.2.3 糖基化修饰
生物体内50%以上的蛋白质存在糖基化修饰,涉及到细胞免疫、蛋白翻译调控、蛋白降解等众多生物过程,SILAC技术结合质谱或LC-MS不仅可以研究糖基化蛋白的变化,还可以用于确定蛋白的糖基化位点[ 48 ]。Boersema等[ 49 ]利用super-SILAC实验对人类乳腺癌不同阶段的血液样本中N-糖基化的分泌蛋白组进行了定量研究。通过过滤辅助样品制备(FASP)的糖肽富集方法和质谱分析发现了1 398个N-糖基化肽段。数据集群分析发现,在晚期癌症阶段丰度较低的蛋白主要与细胞黏附功能相关,丰度较高的糖基化蛋白主要包括四旋蛋白(TSPAN)超家族以及与生长因子信号通路相关的蛋白。同一阶段的乳腺癌细胞中糖基化蛋白分布相似,但雌激素受体、孕激素受体以及原癌基因erbB-2编码的膜受体会出现细胞系间的差异,这有可能促进人们发现新的与癌症相关的糖基化生物标记物。 3.3 SILAC用于寻找新的生物标记物
SILAC技术可以根据细胞中蛋白表达量或者磷酸化、甲基化、糖基化程度的不同,确定生物标记物,推动疾病的早期诊断和治疗[ 50 ]。Orsburn[ 51 ]详细描述了分析磷酸化生物标记物的操作流程,为人们分析磷酸化蛋白以及寻找生物标记物提供了参考。Sun等[ 52 ]利用SILAC技术分析了人类正常细胞系HL-7702与肝癌细胞系HepG2和SK-HEP-1之间蛋白组的差异,鉴定出116种蛋白可能是潜在的肝癌生物标记物。在某些AFP缺陷的肝癌细胞(SK-HEP-1 和 Bel-7402)以及大约半数的血清AFP低表达的肝癌组织中,谷氨酰胺转移酶2(TGM2)发生过表达。通过ELISA检测发现,肝癌患者血清中TGM2水平显著偏高。据此,他们认为对于血清中AFP水平正常的肝癌患者来说,TGM2可以作为一种新型的组织/血清肝癌生物标记物的参考指标用于肝癌的早期诊断。
4 展 望
SILAC技术既可以用于宏观蛋白质组分析,能确定实验条件引起的整个蛋白质组的变化,又可以确定小分子物质的作用机制和结合位点,该方法尤其适合于多靶点药物的分析,用于寻找各个靶点之间的协同或拮抗作用,推动药物的研发和机制研究。但其产生的质谱数据转化为蛋白定量数据需要多个算法步骤,由标记效率引起的误差也需要相应的算法校正,而现今最常用的软件MaxQuant并不能适用于所有蛋白定量数据的分析,因此建立系统的SILAC质谱数据定量分析方法和软件工具是进一步提高SILAC定量的准确性与可靠性的重要保证。在质谱分析时,基于多肽分子量的质谱数据纷繁复杂,如何利用这些数据进行良好的生物信息学分析,如何正确的集群归类得到想要的蛋白质信息,是SILAC技术发展迫切需要解决的问题。蛋白质的复杂性和多样性使得单方面的质谱数据可能无法揭示其规律,因此应当将多种蛋白分析方法互补结合,从而建立高效、灵敏的大规模蛋白质组学研究平台。
5 利益冲突
所有作者声明本文不涉及任何利益冲突。
| [1] | Bruce C, Stone K, Gulcicek E, Williams K.Proteomics and the analysis of proteomic data:2013 overview of current protein-profiling technologies[J].Curr Protoc Bioinformatics, 2013, Chapter 13: Unit 13.21. |
| [2] | Kurien B T, Scofield R H.A brief review of other notable protein detection methods on acrylamide gels[J].Methods Mol Biol, 2012, 869:617-620. |
| [3] | 冯海燕, 景志忠, 房永祥, 莫斯科.双向凝胶电泳技术及其应用[J].生物技术通报, 2009(1): 59-63. |
| [4] | Bantscheff M, Lemeer S, Savitski M M, Kuster B.Qua-ntitative mass spectrometry in proteomics:critical review update from 2007 to the present[J].Anal Bioanal Chem, 2012, 404:939-965. |
| [5] | Lanucara F, Eyers C E.Mass spectrometric-based quantitative proteomics using SILAC[J].Methods Enzymol, 2011, 500:133-150. |
| [6] | Washburn M P.Driving biochemical discovery with qua-ntitative proteomics[J].Trends Biochem Sci, 2011, 36:170-177. |
| [7] | Oda Y, Huang K, Cross F R, Cowburn D, Chait B T.Accurate quantitation of protein expression and site-specific phosphorylation[J].Proc Natl Acad Sci USA, 1999, 96:6591-6596. |
| [8] | Lahm H W, Langen H.Mass spectrometry: a tool for the identification of proteins separated by gels[J].Electrophoresis, 2000, 21:2105-2114. |
| [9] | Chen X, Smith L M, Bradbury E M.Site-specific mass tagging with stable isotopes in proteins for accurate and efficient protein identification[J].Anal Chem, 2000, 72:1134-1143. |
| [10] | Ong S E, Blagoev B, Kratchmarova I, Kristensen D B, Steen H, Pandey A, et al.Stable isotope labeling by amino acids in cell culture, SILAC, as a simple and accurate approach to expression proteomics[J].Mol Cell Proteomics, 2002, 1: 376-386. |
| [11] | Schneider L V, Hall M P.Stable isotope methods for high-precision proteomics[J].Drug Discov Today, 2005, 10: 353-363. |
| [12] | Gruhler A, Olsen J V, Mohammed S, Mortensen P, Faergeman N J, Mann M, et al.Quantitative phosphoproteomics applied to the yeast pheromone signaling pathway[J].Mol Cell Proteomics, 2005, 4:310-327. |
| [13] | Gruhler A, Schulze W X, Matthiesen R, Mann M, Jensen O N. Stable isotope labeling of Arabidopsis thaliana cells and quantitative proteomics by mass spectrometry[J]. Mol Cell Proteomics, 2005, 4:1697-1709. |
| [14] | KrÜger M, Moser M, Ussar S, Thievessen I, Luber C A, Forner F, et al.SILAC mouse for quantitative proteomics uncovers kindlin-3 as an essential factor for red blood cell function[J]. Cell, 2008, 134:353-364. |
| [15] | Geiger T, Cox J, Ostasiewicz P, Wisniewski J R, Mann M.Super-SILAC mix for quantitative proteomics of human tumor tissue[J]. Nat Methods, 2010, 7:383-385. |
| [16] | Fredens J, Engholm-Keller K, Giessing A, Pultz D, Larsen M R, Højrup P, et al.Quantitative proteomics by amino acid labeling in C. elegans[J]. Nat Methods, 2011, 8:845-847. |
| [17] | Somasekharan S P, Stoynov N, Rotblat B, Leprivier G, Galpin J D, Ahern C A, et al.Identification and quantification of newly synthesized proteins translationally regulated by YB-1 using a novel Click-SILAC approach[J]. J Proteomics, 2012, 77:e1-e10. |
| [18] | de Godoy L M, Olsen J V, Cox J, Nielsen M L, Hubner N C, Fröhlich F, et al.Comprehensive mass-spectrometry-based proteome quantification of haploid versus diploid yeast[J]. Nature, 2008, 455:1251-1254. |
| [19] | Wiseman R L, Chin K T, Haynes C M, Stanhill A, Xu C F, Roguev A, et al.Thioredoxin-related protein 32 is an arsenite-regulated thiol reductase of the proteasome 19 S Particle[J]. J Biol Chem, 2009, 284:15233-15245. |
| [20] | DeSouza L V, Siu K W.Mass spectrometry-based qua-ntification[J]. Clin Biochem, 2013, 46:421-431. |
| [21] | Harsha H C, Molina H, Pandey A.Quantitative proteomics using stable isotope labeling with amino acids in cell culture[J]. Nat Protoc, 2008, 3:505-516. |
| [22] | Ong S E, Foster L J, Mann M.Mass spectrometric-based approaches in quantitative proteomics[J]. Methods, 2003, 29:124-30. |
| [23] | Gruhler A, Olsen J V, Mohammed S, Mortensen P, Faergeman N J, Mann M, et al. Quantitative phosphoproteomics applied to the yeast pheromone signaling pathway[J]. Mol Cell Proteomics, 2005, 4:310-327. |
| [24] | Fröhlich F, Christiano R, Walther T C.Native SILAC: metabolic labeling of proteins in prototroph microorganisms based on lysine synthesis regulation[J].Mol Cell Proteomics, 2013, 12:1995-2005. |
| [25] | Le S, Chen H, Cong P, Lin J, Dröge P, Yan J.Mechanosensing of DNA bending in a single specific protein-DNA complex[J]. Sci Rep, 2013, 3:3508. |
| [26] | Spruijt C G, Baymaz H I, Vermeulen M.Identifying specific protein-DNA interactions using SILAC-based quantitative proteomics[J]. Methods Mol Biol, 2013, 977:137-157. |
| [27] | Kappei D, Butter F, Benda C, Scheibe M, Draškovič I, Stevense M, et al.HOT1 is a mammalian direct telomere repeat-binding protein contributing to telomerase recruitment[J]. EMBO J, 2013, 32:1681-1701. |
| [28] | 刘默芳, 王恩多.小RNA 与蛋白质的相互作用[J].生命科学, 2008, 20:178-182. |
| [29] | Lee N, Pimienta G, Steitz J A.AUF1/hnRNP D is a novel protein partner of the EBER1 noncoding RNA of Epstein-Barr virus[J].RNA, 2012, 18:2073-2082. |
| [30] | 沈瑶瑶, 严庆丰.蛋白质相互作用研究进展[J].生命科学, 2013, 25:269-274. |
| [31] | Kaake R M, Milenkovic ' T, Przulj N, Kaiser P, Huang L.Characterization of cell cycle specific protein interaction networks of the yeast 26S proteasome complex by the QTAX strategy[J]. J Proteome Res, 2010, 9:2016-2029. |
| [32] | Ong S E, Li X, Schenone M, Schreiber S L, Carr S A.Identifying cellular targets of small-molecule probes and drugs with biochemical enrichment and SILAC[J]. Methods Mol Biol, 2012, 803:129-140. |
| [33] | Ong S E, Schenone M, Margolin A A, Li X, Do K, Doud M K, et al.Identifying the proteins to which small-molecule probes and drugs bind in cells[J]. Proc Natl Acad Sci USA, 2009, 106:4617-4622. |
| [34] | Daub H, Olsen J V, Bairlein M, Gnad F, Oppermann F S, Körner R, et al.Kinase-selective enrichment enables quantitative phosphoproteomics of the kinome across the cell cycle[J].Mol Cell, 2008, 31:438-448. |
| [35] | Breitkopf S B, Oppermann F S, Keri G, Grammel M, Daub H.Proteomics analysis of cellular imatinib targets and their candidate downstream effectors[J].J Proteome Res, 2010, 9:6033-6043. |
| [36] | Lee H J, Na K, Kwon M S, Kim H, Kim K S, Paik Y K.Quantitative analysis of phosphopeptides in search of the disease biomarker from the hepatocellular carcinoma specimen[J]. Proteomics, 2009, 9:3395-3408. |
| [37] | Bennetzen M V, Larsen D H, Dinant C, Watanabe S, Bartek J, Lukas J, et al.Acetylation dynamics of human nuclear proteins during the ionizing radiation-induced DNA damage response[J]. Cell Cycle, 2013, 12:1688-1695. |
| [38] | Barbi de Moura M, Vincent G, Fayewicz S L, Bateman N W, Hood B L, Sun M, et al.Mitochondrial respiration——an important therapeutic target in melanoma[J]. PLoS One, 2012, 7:e40690. |
| [39] | Raj L, Ide T, Gurkar A U, Foley M, Schenone M, Li X, et al.Selective killing of cancer cells by a small molecule targeting the stress response to ROS[J].Nature, 2011, 475:231-234. |
| [40] | Bargaje R, Gupta S, Sarkeshik A, Park R, Xu T, Sarkar M, et al.Identification of novel targets for miR-29a using miRNA proteomics[J]. PLoS One, 2012, 7:e43243. |
| [41] | Choudhury N R, de Lima Alves F, de Andrés-Aguayo L, Graf T, Cáceres J F, Rappsilber J, et al.Tissue-specific control of brain-enriched miR-7 biogenesis[J]. Genes Dev, 2013, 27:24-38. |
| [42] | Washburn M P.Driving biochemical discovery with quantitative proteomics[J]. Trends Biochem Sci, 2011, 36:170-177. |
| [43] | Imamura H, Yachie N, Saito R, Ishihama Y, Tomita M.Towards the systematic discovery of signal transduction networks using phosphorylation dynamics data[J]. BMC Bioinformatics, 2010, 11:232. |
| [44] | Zanivan S, Meves A, Behrendt K, Schoof E M, Neilson L J, Cox J, et al.In vivo SILAC-based proteomics reveals phosphoproteome changes during mouse skin carcinogenesis[J] Cell Rep, 2013, 3:552-566. |
| [45] | Suganuma T, Workman J L.Crosstalk among histone modifications[J]. Cell, 2008, 135:604-607. |
| [46] | Guan X, Rastogi N, Parthun M R, Freitas M A.Discovery of histone modification crosstalk networks by stable isotope labeling of amino acids in cell culture mass spectrometry (SILAC MS)[J]. Mol Cell Proteomics, 2013, 12:2048-2059. |
| [47] | Jung H R, Pasini D, Helin K, Jensen O N.Quantitative mass spectrometry of histones H3.2 and H3.3 in Suz12-deficient mouse embryonic stem cells reveals distinct, dynamic post-translational modifications at Lys-27 and Lys-36[J]. Mol Cell Proteomics, 2010, 9:838-850. |
| [48] | 李 军, 杜 鑫, Hosseini Moghaddam S H, 陈玉银.蛋白质糖基化修饰研究进展[J].科技通报, 2009, 25:773-778. |
| [49] | Boersema P J, Geiger T, Wisniewski J R, Mann M.Quantification of the N-glycosylated secretome by super-SILAC during breast cancer progression and in human blood samples[J]. Mol Cell Proteomics, 2013, 12:158-171. |
| [50] | Fenselau C.A review of quantitative methods for proteomic studies[J]. J Chromatogr B Analyt Technol Biomed Life Sci, 2007, 855:14-20. |
| [51] | Orsburn B C.SILAC in biomarker discovery[J]. Methods Mol Biol, 2013, 1002:123-131. |
| [52] | Sun Y, Mi W, Cai J, Ying W, Liu F, Lu H, et al.Quantitative proteomic signature of liver cancer cells: tissue transglutaminase 2 could be a novel protein candidate of human hepatocellular carcinoma[J]. J Proteome Res, 2008, 7:3847-3859. |
 2014, Vol. 35
2014, Vol. 35


