2. 第二军医大学长征医院内分泌科, 上海 200003;
3. 第二军医大学长海医院普通外科, 上海 200433
2. Department of Endocrinology, Changzheng Hospital, Second Military Medical University, Shanghai 200003, China;
3. Department of General Surgery, Changhai Hospital, Second Military Medical University, Shanghai 200433, China
近年来,随着糖尿病发病率的逐年上升,胰岛β细胞基因功能研究日益成为人们关注的焦点[1]。基于Cre-loxP重组系统建立的条件基因敲除模型[2],为研究胰岛β细胞功能提供了良好的技术平台。在条件基因敲除模型的建立过程中,Cre工具鼠起着非常重要的作用。目前国内外研究胰岛β细胞的Cre工具鼠主要有受大鼠胰岛素基因启动子(RIP)调控的Cre (RIP-Cre)小鼠[1,3,4,5]、受转录因子胰十二指肠同源盒基因1(pancreatic duodenum homeobox 1,Pdx1)启动子或神经元素3(neurogenin3,Ngn3)启动子调控的胰腺特异性的Cre转基因小鼠[4 ,5,6,7,8,9,10],以及受雌激素类似物他莫昔芬(tamoxifen)诱导的Cre工具鼠(如MIP-CreER[11]、Pdx1-CreER[7]等)。在上述动物模型中,控制Cre表达的启动子均为外源性,片段较小,而且都是通过受精卵显微注射方法获得转基因小鼠。由于外源性DNA通过随机整合的方式整合至基因组中,整合的部位及拷贝数无法控制,导致Cre的表达水平及其重组效率难以控制。除此之外,这些Cre工具鼠还存在Cre表达的组织特异性不够理想的问题[5],同时有文献报道一些RIP-Cre小鼠表现出葡萄糖耐量异常和胰岛素分泌功能障碍[13]。这一系列问题的出现为B细胞功能研究带来了很大困扰,为此,本研究以此为出发点,通过构建受内源性胰岛素2(Ins2)基因严格调控、在胰岛β细胞中特异性表达Cre重组酶的基因敲入型打靶载体,以期为获得胰岛β细胞中基因特异性敲除小鼠模型打下重要基础,最终为胰岛β细胞研究提供理想的动物模型。
含Ins2基因组DNA的BAC菌株DH10B(克隆号:365C19)购自Cell Biolabs公司;EL350菌株购自Biovector Science Lab公司;pSK-IRES-Cre-pA质粒由第二军医大学基础部病理生理学教研室构建及保存;pBR322-2s、pSK-Ⅱ和pL451质粒由第二军医大学基础部病理生理学教研室保存。
LB培养基(胰蛋白胨10 g,酵母粉 5 g,NaCl 5 g)购自上海生工生物工程技术服务有限公司;限制性内切酶购自NEB公司;DNA Ladder、T4 DNA连接酶购自TaKaRa公司;DNA纯化试剂盒购自Qiagen公司;凝胶回收试剂盒购自Axygen公司;质粒小量抽提试剂盒购自Omega公司;PCR相关试剂购自北京艾德莱生物科技有限公司。
根据GenBank上已发表的Ins2基因序列设计引物,引物合成和质粒测序均由上海杰李生物技术有限公司完成。引物序列见表 1。
| 表1 寡核苷酸引物序列 Tab 1 Primer sequences of oligonucleotide |
收集培养 16 h 的含Ins2基因组DNA的BAC菌株(DH10B)单克隆菌液 1.5 mL,加入质粒小量抽提试剂溶液(溶液Ⅰ/Ⅱ/Ⅲ分别为 250、250 和 350 μL),13 000×g离心后取上清加入等体积的异丙醇,室温静置 10 min后16 000×g 4℃离心 10 min,弃上清后用 1 mL 70%乙醇洗 1次,室温干燥后溶于20 μL TE缓冲液中,稀释至100 ng/μL备用(注:用于电转时BAC需新鲜制备)。用PCR扩增鉴定BAC DNA,引物(RA5和RA3)序列详见表 1。
将EL350感受态细胞接种至 5 mL 不含抗生素的LB培养液中,32℃摇菌至D600=1.2时收集细菌后,17 000×g 0℃离心5 min,900 μL冰水重悬后转移至预冷的 1.5 mL 离心管中,5 000×g室温离心 20 s,弃上清,重复2次后重悬于 50 μL 冰水中备用。
取100 ng BAC DNA与EL350感受态细胞混匀,冰浴 15 min 后电击(BTX-ECM830电穿孔仪,电击条件:1.75 kV、171 ms、5 pulse),向其中加入 900 μL LB培养液培养 1 h 后取适量菌液涂于氯霉素平板上,32℃培养后挑单克隆菌落抽提质粒,通过PCR扩增进行鉴定,引物(RA5和RA3)序列详见表 1。
将正确导入BAC DNA的单克隆菌落在含氯霉素抗性的LB培养液中32℃培养至D600=1.2 时1:1000 转接至 100 mL LB培养液中继续培养,直至D600=0.5时转至42℃水浴摇床,180转/分15 min后立即置于冰水中使其迅速降温;冰浴 10 min 后收集细菌,冰水洗涤 3 次、15%甘油洗涤 1 次后用预冷的 800 μL 15%甘油重悬,-80℃分装保存。
以BAC DNA为模板,PCR扩增得到的RA5片段用BamHⅠ/XhoⅠ酶切、RA3片段用XhoⅠ/Hind Ⅲ酶切纯化后备用。
将RA5和RA3两段同源臂连接至BamHⅠ和Hind Ⅲ双酶切后的载体pBR322-2s上,用Hind Ⅲ进行单酶切鉴定并测序(质粒编号:#454)。
取XhoⅠ线性化的取获载体 10 ng 加入制备好的表达Red重组系统的BAC宿主菌中,电击转化后用氨苄青霉素抗性平板进行筛选培养,提取质粒pBR322-Ins2用Hind Ⅲ和EcoRⅠ分别进行单酶切鉴定并测序(质粒编号:#455)。
以BAC DNA为模板,常规PCR扩增Ins2基因的第3外显子(exon3),Hind Ⅲ/XbaⅠ双酶切后克隆至pSK-Ⅱ载体上,并用这2个酶切位点进行鉴定。将正确的质粒通过点突变在编码区和3′端非翻译区(untranslated region,UTR)之间改建一个EcoRⅤ酶切位点,点突变所用的引物Mutant见表 1,用XhoⅠ/EcoRⅤ双酶切进行鉴定;点突变成功的质粒用EcoRⅤ使其线性化后与Linker EcoRⅠ/BamHⅠ连接,挑取单克隆菌落进行培养,质粒抽提后用EcoRⅠ/XhoⅠ酶切鉴定。酶切正确的质粒用M13-21引物测序确保Linker连接方向正确(质粒编号:#453)。
pSK-IRES-Cre-pA质粒用EcoRⅠ/XhoⅠ酶切,电泳回收IRES-Cre-pA片段(约1.9 kb);pL451质粒用SalⅠ/BamHⅠ酶切后,电泳回收FRT-pGK-Neo-FRT片段(约2 kb),将这两个片段连接至pSK-Ins2E3质粒上,并用EcoRⅠ/BamHⅠ酶切鉴定,酶切正确的质粒用M13-21引物测序(质粒编号:#456)。
含有目的基因同时又表达Red重组系统的感受态细胞的制备方法同1.2.4。将构建正确的微型打靶载体用XhoⅠ/NotⅠ酶切后胶回收IRES-Cre-FRT-PGK-Neo-FRT片段,取约150 ng 胶回收产物与制备的感受态细胞混匀,冰浴 15 min 后电击转化,挑取单克隆菌落培养,质粒抽提后酶切鉴定并进行测序(质粒编号:#458)。
在本实验中我们选择具有Red同源重组系统的EL350,以抽提的BAC DNA为模板,用RA5和RA3引物常规PCR扩增微型打靶载体的5′和3′两个同源重组臂,大小分别为 270 bp和 488 bp。
将BAC DNA电转至EL350中后进行质粒抽提,用RA5和RA3 PCR引物扩增得到大小分别为 270 bp和488 bp 的条带。此时BAC宿主菌可直接通过温度诱导制备成表达Red重组系统的感受态细胞。
常规PCR扩增的目的片段越长就越难得到PCR产物,PCR产物越大产生突变的概率也越高。由于Red重组系统可以对长达 80 kb的DNA进行亚克隆,所以用其进行打靶载体构建时可以按需要改变同源区的长度。在本实验中克隆目的片段全长约 12 kb,预打靶载体构建示意图如图 1所示。为了保证重组过程中质粒的稳定性,本实验中将两端同源臂直接连接至选择具有负性筛选标记的低拷贝载体pBR322-2s上。
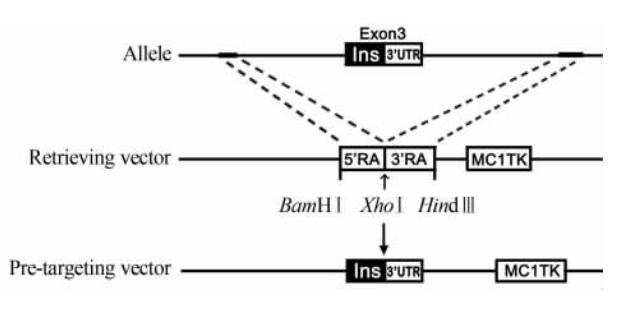 | 图1 预打靶载体构建示意图 Fig 1 Procedure for constructing pre-targeting vector Ins: Insulin 2; UTR: Untranslated region; RA: Retrieving arm; TK: Thymidine kinase |
将XhoⅠ线性化的取获载体电转至表达Red重组系统的BAC感受态细胞中,利用细菌内Red重组酶介导的缺口修复(gap-repair)的方法,载体两端的同源臂与BAC DNA上的同源序列发生交换,目的片段交换至取获载体上。克隆成功后的质粒大小约为 18 kb,酶切位点分析发现目的片段第231、4 870、6 309位处有HindⅢ酶切位点,同时取获载体在3′同源臂连接处也有1个HindⅢ酶切位点,因此酶切后理论上会有4.5、1.4 kb和2条 6 kb左右的条带,HindⅢ酶切后电泳图见图 2A。由于酶切后的2条 6 kb左右的条带相隔较近不能很清楚地看到,因此用EcoRⅠ(共4个酶切位点,分别位于第1 885、2 718、10 511、11 617位处)进一步酶切鉴定,电泳图如图 2B所示。条带的大小与理论相符,初步说明目的片段克隆成功。
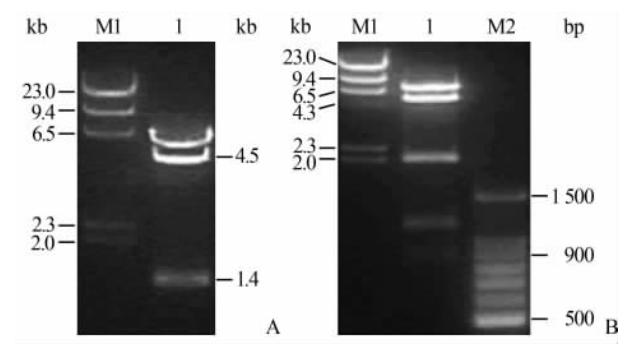 | 图2 质粒pBR322-Ins2酶切鉴定结果 Fig 2 Digestive identification of plasmid pBR322-Ins2 A: HindⅢ digestion of pre-targeting vector (M1: Marker; 1: HindⅢ digestion of pBR322-Ins2); B: EcoRⅠdigestion of pre-targeting vector (M1 and M2: Markers; 1: EcoRⅠdigestion of pBR322-Ins2) |
pSK-Ins2E3质粒构建示意图如图 3A所示。PCR扩增得到 405 bp大 小的Ins2基因第3外显子连接至pSK-Ⅱ载体上后,在第3外显子的编码区和3′UTR区之间改建一个EcoRⅤ酶切位点再与Linker连接。
微型打靶载体构建示意图见图 3A。此载体构建的目的就是将IRES-Cre-pA片段和Neo基因插入到Ins2基因第3外显子的3′UTR之前。两个片段和载体之间如果连接顺序正确,质粒上共有3个HindⅢ 酶切位点,除载体上的1个,另外2个分别位于插入片段的第413位和第2 177位,这样酶切理论上可以切出 420 bp和 1.8 kb左右的条带。实际酶切鉴定如图3B所示,条带大小与预期一致,说明载体初步构建成功。
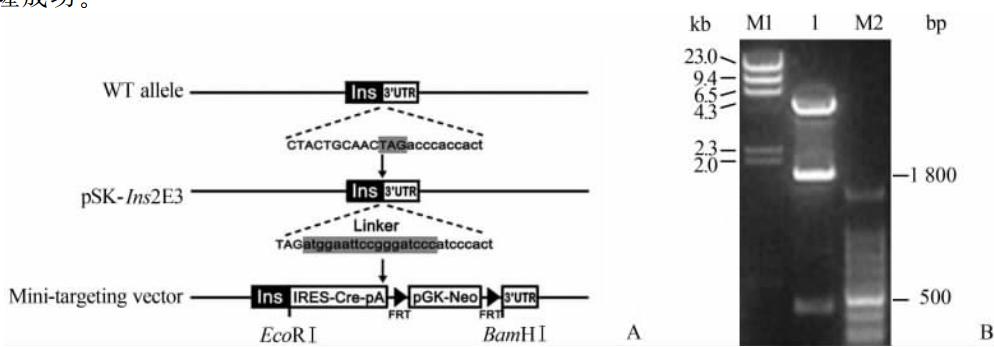 | 图3 微型打靶载体的构建Fig 3 Constructing a mini-targeting vector A: Diagram of the strategy to generate mini-targeting vector (WT: Wild type; Ins: Insulin 2; UTR: Untranslated region; Neo: Neomycin; pGK: Phosphoglycerine kinase); B: Identification of plasmid mini-targeting vector digested by HindⅢ. (M1 and M2: Markers; 1: HindⅢ digestion of mini-targeting vector) |
将酶切正确的质粒用M13-21引物测序,测序所得序列与预期一致,进一步表明载体构建成功。
载体构建的示意图如图 4所示。用XhoⅠ/NotⅠ酶切微型打靶载体后回收5′UTR-Ins2-Cre-Neo-3′UTR片段(约 4.3 kb),通过电转使片段两端与Ins基因组两端的同源序列发生同源重组。通过此次的同源重组,将IRES-Cre-pA-FRT-pGK- Neo-FRT片段插入到Ins2基因的第3外显子中翻译区和3′UTR之间。如果同源重组成功,在质粒的第1 885、2 718、3 342、5 346、14 417和第15 523位分别有EcoR Ⅰ酶切位点,理论上可以切出833 bp、624 bp、2 004 bp、9 071 bp和1 106 bp大小的条带,酶切结果见图 5。用EcoRⅤ和HindⅢ 分别单酶切,切出条带大小均与预期一致。利用引物MC1-R和RA3的反向引物分别对酶切正确的质粒进行测序,测序所得的序列通过与预期重组后基因组序列进行对比,发现实际序列一致无突变,表明载体构建成功。
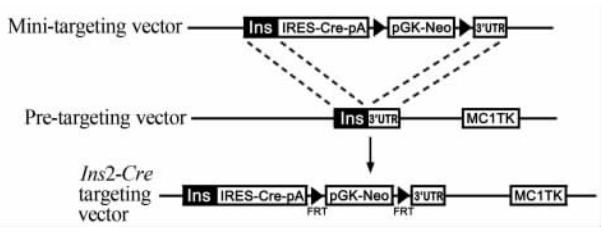 | 图4 Ins2-Cre打靶载体构建示意图 Fig 4 Diagram for generating Ins2-Cre targeting vectorpGK: Phosphoglycerine kinase; UTR: Untranslated region; Ins: Insulin 2; Neo: Neomycin |
图 5)。
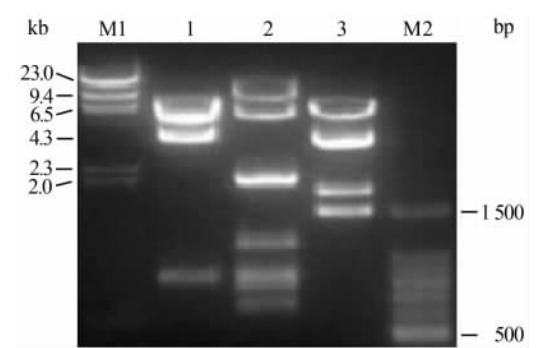 | 图5 Ins2-Cre打靶载体酶切鉴定结果Fig 5 Digestive identification of plasmid Ins2-Cre targeting vector M1: λ/HindⅢ DNA marker; 1-3: EcoRⅤ, EcoRⅠ and HindⅢ digestion of plasmid Ins2-Cre, respectively; M2: DNA ladder. Ins: Insulin 2 |
基因敲除小鼠模型的应用为基因功能研究提供了良好的实验手段。利用Cre-loxP位点特异性重组系统能够有效避免基因在发育过程中导致胚胎死亡的现象,因此在条件性基因敲除中得到广泛应用[14]。Cre-loxP系统介导的条件基因敲除模型包含2种小鼠,一种是目的基因两侧插入loxP位点小鼠,另外是受各种组织或细胞特异性启动子调控的Cre工具鼠。2种小鼠交配后Cre重组酶介导的重组能够有效切出2个loxP位点间的序列,从而达到基因敲除的目的。目前能够用于实现条件敲除、在不同组织中表达Cre重组酶的Cre小鼠世系已经有很多种[15],如用于胰腺组织基因特异性敲除的RIP-Cre、Pdx1-Cre和Ngn3-Cre小鼠,在启动子调控下的Cre重组酶在下丘脑不同部位均有不同程度的表达:RIP启动子调控下的Cre重组酶主要在下丘脑弓状核表达,Pdx1和Ngn3启动子调控下的Cre重组酶分别在下丘脑的腹内侧与核群等部位有活性[12]。理想动物模型的缺失给研究胰岛β细胞基因功能带来很大困扰。
转基因动物模型的建立主要有DNA显微注射和基因打靶两种方法。DNA显微注射具有外源基因随机整合、拷贝数无法控制且整合效率较低等缺点,因此目前转基因动物模型的建立常用基因打靶技术。IRES(内部核糖体进入位点) 是mRNA 5′UTR的一段特殊序列,允许核糖体直接在此序列处结合mRNA并起始翻译。在打靶载体的设计过程中,我们将编码Cre重组酶的DNA序列置于IRES序列之后,再定点整合于Ins2基因第3外显子3′UTR之前。利用IRES能独立启动不依赖5′帽子结构翻译这一特点,Cre与 Ins2的翻译各自独立,同时Cre的表达受到内源性Ins2基因的严格调控,实现了外源基因Cre与Ins2的共表达。本研究中打靶载体的构建是利用λ噬菌体Red系统介导的同源重组技术对靶基因组DNA进行直接修饰[16 ,17,18],这种基于BAC同源重组的方法减少了为克隆目的基因寻找合适的限制性酶切位点所花费的精力,同时也简化了传统打靶载体构建工作,具有高效、省时、灵活的特点。
Red重组系统构建法包含两种方式:一种是构建诱导表达型质粒,再将这种质粒导入到需要进行同源重组的细胞中,表达相应的重组系统;另一种是通过将突变失活的噬菌体有关序列整合到大肠杆菌的染色体中,构建具有同源重组功能的菌株,然后在使用时将需要进行同源重组的DNA导入到这些菌株中。比较而言后者重组效率较高,应用较广泛,目前常用的方法是将BAC载体转入到有重组酶的细菌(如EL250和EL350等)中。本实验构建的基因敲入型打靶载体是将修饰后Ins2基因第3外显子直接导入到含有BAC的EL350中,操作简单快捷。在构建的过程中我们首先通过PCR扩增Ins2基因两端的同源臂,借助λ噬菌体Red系统介导的同源重组反应从BAC中成功取获了Ins2基因组DNA序列,由于重组反应的整个过程都是在大肠杆菌细胞内部完成,因此碱基突变的频率也非常低。为了不影响Ins2基因的正常功能,在实验设计时选择在第3外显子的终止密码子和3′UTR之间插入IRES-Cre序列,这样既不影响Ins2基因的表达,又能独立翻译出Cre重组酶,保证了Cre重组酶表达的特异性。
本实验利用λ噬菌体Red重组酶系统构建了Ins2-Cre敲入型打靶载体,为获得胰岛β细胞中特异性表达Cre重组酶小鼠模型打下重要基础,同时为进一步构建B细胞基因特异性敲除小鼠,研究相关基因的生物学功能奠定了基础。
所有作者声明本文不涉及任何利益冲突。
| [1] | Xu Y, Wang L, He J, Bi Y, Li M, Wang T, et al;2010 China Noncommunicable Disease Surveillance Group.Prevalence and control of diabetes in Chinese adults[J].JAMA, 2013, 310:948-959. |
| [2] | Sauer B, Henderson N.Cre-stimulated recombination at loxP-containing DNA sequences placed into the mammalian genome[J].Nucleic Acids Res, 1989, 17:147-161. |
| [3] | Postic C, Shiota M, Niswender K D, Jetton T L, Chen Y, Moates J M, et al.Dual roles for glucokinase in glucose homeostasis as determined by liver and pancreatic beta cell-specific gene knock-outs using Cre recombinase[J].J Biol Chem, 1999, 274:305-315. |
| [4] | Magnuson M A, Osipovich A B.Pancreas-specific cre driver lines and considerations for their prudent use[J].Cell Metab, 2013, 18:9-20. |
| [5] | Herrera P L.Adult insulin-and glucagon-producing cells differentiate from two independent cell lineages[J].Development, 2000, 127:2317-2322. |
| [6] | Desgraz R, Herrera P L.Pancreatic neurogenin 3-expressing cells are unipotent islet precursors[J].Development, 2009, 136:3567-3574. |
| [7] | Gu G, Dubauskaite J, Melton D A.Direct evidence for the pancreatic lineage: NGN3+ cells are islet progenitors and are distinct from duct progenitors[J].Development, 2002, 129:2447-2457. |
| [8] | Gannon M, Herrera P L, Wright C V.Mosaic Cre-mediated recombination in pancreas using the pdx-1 enhancer/promoter[J].Genesis, 2000, 26:143-144. |
| [9] | Yoshida S, Takakura A, Ohbo K, Abe K, Wakabayashi J, Yamamoto M, et al.Neurogenin3 delineates the earliest stages of spermatogenesis in the mouse testis[J].Dev Biol, 2004, 269:447-458. |
| [10] | Schonhoff S E, Giel-Moloney M, Leiter A B.Neurogenin 3-expressing progenitor cells in the gastrointestinal tract differentiate into both endocrine and non-endocrine cell types[J].Dev Biol, 2004, 270:443-454. |
| [11] | Wicksteed B, Brissova M, Yan W, Opland D M, Plank J L, Reinert R B, et al.Conditional gene targeting in mouse pancreatic-cells:analysis of ectopic Cre transgene expression in the rain[J].Diabetes, 2010, 59:3090-3098. |
| [12] | Song J, Xu Y, Hu X, Choi B, Tong Q.Brain expression of Cre recombinase driven by pancreas-specific promoters[J].Genesis, 2010, 48:628-634. |
| [13] | Lee J Y, Ristow M, Lin X, White M F, Magnuson M A, Hennighausen L.RIP-Cre revisited, evidence for impairments of pancreatic beta-cell function[J].J Biol Chem, 2006, 281:2649-2653. |
| [14] | KÜhn R, Schwenk F.Advances in gene targeting methods[J].Curr Opin Immunol, 1997, 9:183-188. |
| [15] | Nagy A, Mar L.Creation and use of a Cre recombinase transgenic database[J].Methods Mol Biol, 2001, 158:95-106. |
| [16] | Zhang Y, Buchholz F, Muyrers J P, Stewart A F.A new logic for DNA engineering using recombination in Escherichia coli[J].Nat Genet, 1998, 20:123-128. |
| [17] | Zhang Y, Muyrers J P, Testa G, Stewart A F.DNA cloning by homologous recombination in Escherichia coli[J].Nat Biotechnol, 2000, 18:1314-1317. |
| [18] | Zhang Y, Muyrers J P, Rientjes J, Stewart A F.Phage annealing proteins promote oligonucleotide-directed mutagenesis in Escherichia coli and mouse ES cells[J].BMC Mol Biol, 2003, 4:1. |
 2014, Vol. 35
2014, Vol. 35


