2. 第二军医大学长海医院肛肠外科, 上海 200433
△共同第一作者
*通信作者
2. Department of Colorectal Surgery, Changhai Hospital, Second Military Medical University, Shanghai 200433, China
△Co-first authors.
*Corresponding authors
锯齿型结直肠癌(serrated colorectal adenocarcinoma,SCA)是近年来新确认的一种呈现锯齿形态的特殊结直肠癌(CRC)亚型,这种亚型可以由高度增生性息肉、广基锯齿型息肉、传统锯齿型息肉以及混合型息肉演化而来[1]。上述息肉向锯齿型肠癌演变的分子过程呈现独特的“锯齿”信号通路,不同于传统的结肠腺瘤样息肉病蛋白(adenomatous polyposis coli,APC)演变通路[2]。结直肠锯齿型息肉患者更容易进展为结直肠恶性病变,其血清中有更高水平的癌相关自身抗体[3]。锯齿型结直肠癌倾向于具有K-RAS或BRAF基因突变,不具有染色体不稳定特征,但具有高水平的微卫星不稳定以及甲基化表型特征[2, 4]。从细胞学上看,锯齿型结直肠癌缺乏坏死特征但呈现低分化特征,往往具有大量黏蛋白分泌且肿瘤细胞周围伴有明显的淋巴细胞渗透[2, 4]。尽管一项锯齿型结直肠癌全基因组表达谱研究发现锯齿型结直肠癌与其他结肠直肠癌亚型预后没有差别[5],但是,一项最近的有关结直肠癌全基因组表达谱分型的研究却发现:一种与锯齿型息肉特异性表达谱相似的结肠癌亚型具有化疗耐药和预后不良等临床特征[6]。除了研究结果不一致外,两项研究的共同缺点之一是样本量较小。
指纹基因群(gene expression signature,GES)是一组能够指示疾病表型、通路活性、分子亚型和治疗效果等重要特征的基因群,多通过高通量组学技术分析而得,已被多项研究科学整合并用于医学事件和分子机制的探索研究[7, 8, 9, 10, 11]。基于上述背景,本研究采用指纹基因群分析技术在一个大样本表达谱队列(n=600)中探索锯齿型结直肠癌的临床预后和治疗特征,以便为今后该类型结直肠癌的深入研究提供新的信息。
1 资料和方法 1.1 结直肠癌预后和治疗相关表达谱芯片的下载和处理
基因表达谱数据储存公共平台(gene expression omnibus,GEO)中存在大量肿瘤研究的表达谱数据。根据设计我们下载了3组芯片数据:(1)4个有预后信息的结直肠癌表达谱研究数据,包括GSE14333[12]、GSE17538[13]、GSE33113[14]和GSE37892[15],这些数据均产生自Affymetrix hgu 133plus 2.0芯片平台;(2)1个与锯齿型肿瘤相关的表达谱数据(GSE4045,Affymetrix hgu133a)及其相应文献中的226个锯齿型结直肠癌相关探针的信息[5];(3)2个有治疗效果信息的表达谱数据组GSE28702[16]和GSE5851[17],前者为Affymetrix hgu 133plus 2.0,后者为Affymetrix hug133a 2.0。对于上述芯片数据,我们首先用R软件Affy 包中的Robust multi-array average (RMA)法[18]获取各研究的mRNA表达谱数据,并去除GSE14333与GSE17538中重复的样本;然后仔细对应不同芯片平台的探针信息,并合并GSE14333、GSE17538、GSE33113、GSE37892和GSE4045等5个研究,用Sva软件包中的“ComBat”命令[19]进行批间误差校正,再用Limma软件包中的“normalize Quantiles”命令[20]和survJamda软件包中的Z-score功能[21]对数据进行标准化和Z转化。对于GSE28702和GSE5851,我们仅用Affy包对其进行RMA数据抽提,并进行Z-score转化。最后根据各芯片平台探针与已知锯齿型指纹基因群相关探针的对应情况抽提相应探针的表达谱数据以便进行后续分析。 1.2 指纹基因群评分的计算
指纹基因群评分模型主要参照以往研究[8]进行,其计算公式如下:
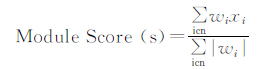
对于合并的5个芯片研究,以GSE4045[5]作为指示研究,根据该研究中锯齿型和传统型癌的病理确定结果和指纹基因群评分结果确定锯齿型、过渡型与传统型结直肠癌分组的界值,并将该界值用于大样本队列中600个CRC的亚型界定。预后分析时,对年龄、性别、TNM分期以及结直肠亚型分组等变量进行单变量和多变量Cox模型分析,对有协同作用的变量在进行分层后进行Kaplan-Meier模型分析。分析锯齿型指纹基因群评分与FOLFOX方案及西妥昔单抗疗效的关系时,采用独立样本t检验比较有反应组和无反应组的评分水平。统计分析主要用R软件和SPSS16.0来实现,检验水准(α)为0.05。 2 结 果
2.1 CRC亚型分组界值确定 GSE14333、GSE17538、GSE33113、GSE37892和GSE4045的合并数据共包含637个样本,其中指示研究GSE4045包含8个锯齿型癌和29个传统型癌。如图 1A所示,8个锯齿型癌的指纹基因群最低评分为0.701,29个传统型癌的指纹基因群最高评分0.294。根据GSE4045的研究结果,设置指纹基因群评分>0.700为锯齿型结直肠癌,指纹基因群评分≥0.300且≤0.700为过渡型结直肠癌,指纹基因群评分<0.300为传统型结直肠癌。由此,我们将600个未知分型的结直肠癌分为锯齿型(n=50)、过渡型(n=126)和传统型(n=424)3个亚型,具体如图 1B所示。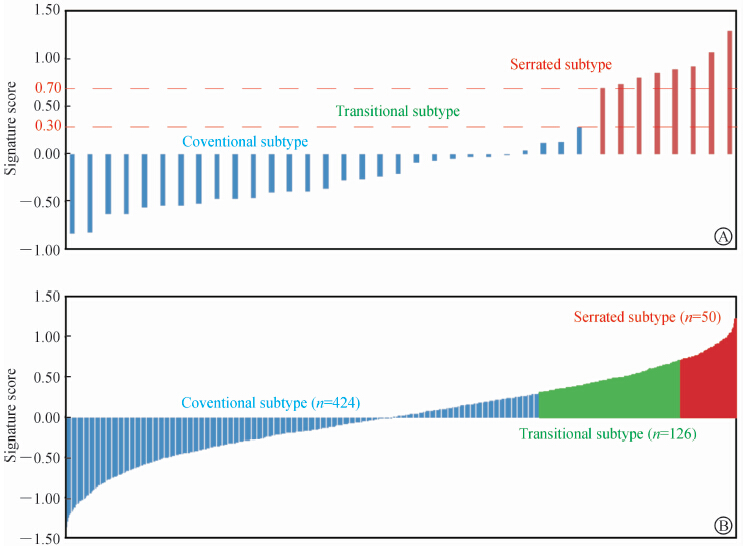 | 图1 锯齿型、过渡型和传统型结直肠癌分型的确定 Fig.1 Cut-off values of subtypes of serrated carcinoma,transitional carcinoma and conventional carcinoma of colorectal cancer A: Result of GSE4045; B: Results of GSE14333,GSE17538,GSE33113,and GSE37892 |
如表 1所示,50个锯齿型结直肠癌、126个过渡型结直肠癌和424个传统型结直肠癌在年龄和肿瘤分期构成等方面差异无统计学意义;但在性别构成上3组之间差异有统计学意义,主要表现为锯齿型结直肠癌患者中女性比例明显高于传统型(P=0.03)和过渡型结直肠癌(P=0.02)。
|
|
表 1 锯齿型、过渡型和传统型结直肠癌的人口特征和肿瘤分期特征比较 Tab 1 Clinical characteristics of serrated,transitional,and conventional subtypes of colorectal carcinoma |
600例CRC患者中,有488个具有完整复发随访信息的Ⅰ~Ⅲ期患者,本研究中预后研究主要围绕该部分样本展开。如图 2,Kaplan-Meier分析结合Log-rank检验发现锯齿型和过渡型结直肠癌在Ⅰ~Ⅲ期、Ⅱ期、Ⅲ期患者中对预后的影响非常相似,没有差别,且上述两型的预后不同于传统型结直肠癌患者的预后。基于上述研究结果,我们合并锯齿型和过渡型两组样本与传统型结直肠癌进行单因素和多因素Cox模型的分析。如表 2,单因素Cox模型发现,年龄、性别、肿瘤分期和肿瘤分型这几个变量中,仅肿瘤分期和肿瘤分型这两个变量与患者复发显著相关;进一步多因素Cox模型分析发现肿瘤分期和肿瘤分型(锯齿型+过渡型vs传统型)这两个变量是患者复发的独立危险因素,其中肿瘤分型的HR=2.118(95%CI 1.460~3.072)。比较锯齿型和传统型结直肠癌对预后的影响发现,单因素分析锯齿型相对于传统型结直肠癌其HR(95%CI)为2.081(1.179~3.674),差异有统计学意义(P=0.012);多因素分析(年龄、性别、肿瘤分期为协变量)发现其HR(95%CI)为1.792(1.011~3.177),差异有统计学意义(P=0.046)。
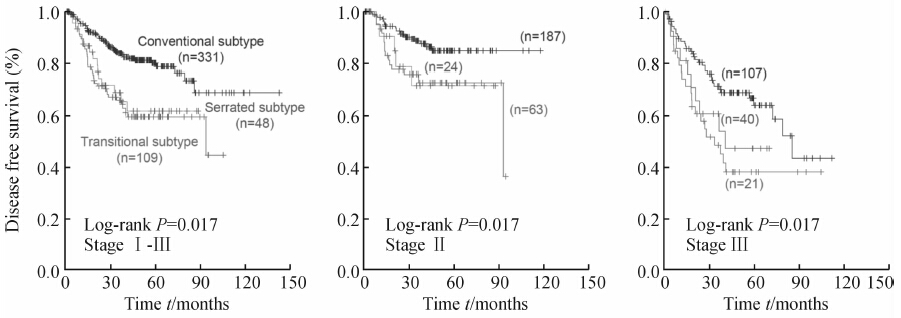 | 图2 比较锯齿型、过渡型和传统型结直肠癌在不同肿瘤分期时的预后作用 Fig.2 Prognostic values of serrated and conventional colorectal carcinoma at different tumor stages |
|
|
表 2 结直肠癌分型、分期等变量对患者预后影响的单因素和多因素Cox模型分析 Tab 3 Univaraite and multivariate Cox model analysis of effect of subtypes and stages on prognosis of patients with colorectal cancer |
2.4 不可切除锯齿型结直肠癌与姑息治疗的关系
结果(图 3)表明:GSE5851中包含25个对西妥昔单抗治疗有反应者和46个无反应者,两组的锯齿型指纹基因群评分有差异,前者低于后者(P=0.017),提示锯齿型表型对西妥昔单抗治疗有一定的抗药性;GSE28702中包含42个对FOLFOX化疗方案有反应者和41个无反应者,两组的锯齿型指纹基因群评分无差异(P=0.380),提示锯齿型表型可能与FOLFOX耐药无关。
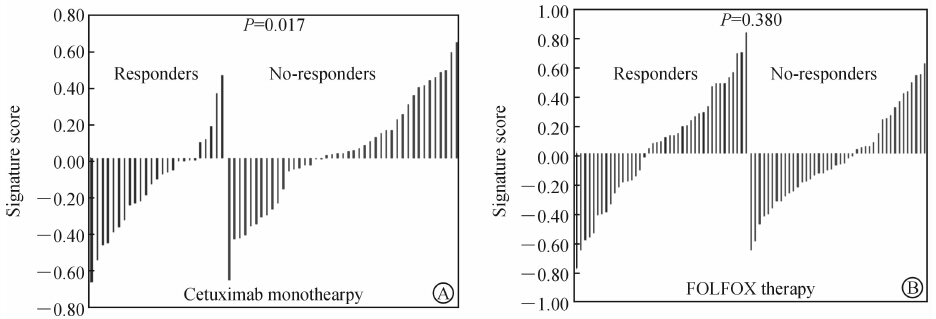 | 图3 锯齿型基因群评分与辅助治疗的关系 Fig.3 Relationship between serrated gene signature scores and adjuvant therapy effective A: GSE5851; B: GSE28702 |
具有典型锯齿形态的锯齿型结直肠癌约占结直肠癌的7%~8%[5],许多由锯齿型通路演化而来的结直肠癌并无典型的锯齿型形态。据估计锯齿型通路形成的肿瘤约占结直肠癌的30%[22]。本研究基于Laiho等[5]建立的锯齿型指纹基因群及建立该基因群所用的表达谱数据,以其中29例传统型结直肠癌的锯齿型指纹基因群评分上限和8例锯齿型结直肠癌的下限为界值,对600例未知亚型分类的结直肠癌进行了亚型预测,结果将其预测为50例锯齿型结直肠癌、126例过渡型结直肠癌和424例传统型结直肠癌。由于Laiho等[5]研究中29例传统型结直肠癌的样本量并不算太小,故这29例样本的基因群评分分布更接近于总体分布,故本研究中对传统型结直肠癌的界值划分有一定准确性;相对而言,指示研究(GSE4045)中仅8例病理上明确的锯齿型癌,由于样本量小,这8例所代表的界值划分相对容易存在误差。因此,在本研究中我们推测过渡型结直肠癌可能与锯齿型结直肠癌有相似的特征。这种利用指纹基因群对锯齿型亚型预测的方法,最大程度上识别了形态学上不典型的锯齿型结直肠癌,为本研究深入揭示锯齿型结直肠癌的重要特征提供基础。
利用600例结直肠癌患者的临床信息,我们发现锯齿型、过渡型结直肠癌相对于传统型结直肠癌,其年龄和肿瘤分期在三者间均无差异。肿瘤分期在3个结直肠癌亚型之间的无差异,提示该亚型分类可能为传统的TNM分期提供新的互补预测指标。进一步对488例具有肿瘤复发数据的Ⅰ~Ⅲ期结直肠癌进行生存分析,发现锯齿型、过渡型和传统型肿瘤分型与结直肠癌患者术后复发密切相关;由于Ⅱ期和Ⅲ期结直肠癌的预后预测更具临床价值(更能有效指导治疗),我们在分期分层后分别评级了锯齿型、过渡型和传统型结直肠癌的预后特征,结果发现锯齿型癌和过渡型亚型无论在Ⅱ期结直肠癌还是在Ⅲ期结直肠癌,均是有效的预后不良预测因子。有趣的是,锯齿型和过渡型结直肠癌对患者的复发预测几乎具有完全相同的影响。单因素和多因素Cox模型分析发现,无论是合并锯齿型和过渡型结直肠癌与传统型结直肠癌比较,还是直接比较锯齿型和传统型结直肠癌,均发现除了TNM分期以外,锯齿型和(或)过渡型结直肠癌是患者术后复发的独立危险因素。由于部分锯齿型亚型有典型的细胞形态学特征,如细胞核的嗜酸染色、细胞核呈周边型等特点[2]。因此,有效利用这些形态学改变与TNM分期信息能更好地促进临床患者预后预测。
结合一些具有药物疗效信息的表达谱数据,本研究发现西妥昔单抗治疗无效组的锯齿型指纹基因群评分显著高于治疗有效组,提示锯齿型结直肠癌可能对表皮生长因子受体(EGFR)抗体治疗不敏感。锯齿型结直肠癌往往倾向于发生K-RAS突变或BRAF突变[2, 4],进一步发现K-RAS突变和BRAF突变在小鼠模型中均能诱导锯齿型癌症表型的出现[2, 4],而携带这两种突变的患者往往对EGFR抗体治疗不敏感[24]。这些研究和我们的锯齿型指纹基因群评分研究结果相互支持。进一步研究锯齿型亚型和FOLFOX治疗的关系,结果发现两者之间无差异。由于我们的研究主要基于基因芯片的信息挖掘,而芯片等高通量数据往往容易存在明显的批间误差,故其研究结果可能存在一定的偏差。
总之,本研究提供了一种新的疾病表型和临床数据研究思路,利用系统生物学的方法建立了锯齿型结直肠癌与预后不良以及EGFR抗体药物耐药的联系,为后续深入研究提供了参考信息。
4 利益冲突
所有作者声明本文不涉及任何利益冲突。
| [1] | Noffsinger A E.Serrated polyps and colorectal cancer:new pathway to malignancy[J].Ann Rev Pathol,2009,4:343-364. |
| [2] | Bennecke M,Kriegl L,Bajbouj M,Retzlaff K,Robine S,Jung A,et al.Ink4a/Arf and oncogene-induced senescence prevent tumor progression during alternative colorectal tumorigenesis[J].Cancer Cell,2010,18:135-146. |
| [3] | Chang W,Wu L,Cao F,Liu Y,Ma L,Wang M,et al.Development of autoantibody signatures as biomarkers for early detection of colorectal carcinoma[J].Clin Cancer Res,2011,17:5715-5724. |
| [4] | Rad R,Cadiñanos J,Rad L,Varela I,Strong A,Kriegl L,et al.A genetic progression model of brafV600E-induced intestinal tumorigenesis reveals targets for therapeutic intervention[J].Cancer cell,2013,24:15-29. |
| [5] | Laiho P,Kokko A,Vanharanta S,Salovaara R, Sammalkorpi H,Järvinen H,et al.Serrated carcinomas form a subclass of colorectal cancer with distinct molecular basis[J].Oncogene,2006,26:312-320. |
| [6] | Felipe De Sousa E M,Wang X,Jansen M,Fessler E,Trinh A,de Rooij LP,et al.Poor-prognosis colon cancer is defined by a molecularly distinct subtype and develops from serrated precursor lesions[J].Nat Med,2013,19:614-618. |
| [7] | Tian S,Roepman P,Popovici V,Michaut M,Majewski I,Salazar R,et al.A robust genomic signature for the detection of colorectal cancer patients with microsatellite instability phenotype and high mutation frequency[J].J Pathol,2012,228:586-595. |
| [8] | Ignatiadis M,Singhal S K,Desmedt C,Haibe-Kains B,Criscitiello C,Andre F,et al.Gene modules and response to neoadjuvant chemotherapy in breast cancer subtypes:a pooled analysis[J].J Clin Oncol,2012,30:1996-2004. |
| [9] | Tian S,Simon I,Moreno V,Roepman P,Tabernero J,Snel M,et al.A combined oncogenic pathway signature of BRAF,KRAS and PI3KCA mutation improves colorectal cancer classification and cetuximab treatment prediction[J].Gut,2013,62:540-549. |
| [10] | Gatza M L,Lucas J E,Barry W T,Kim J W,Wang Q,Crawford M D,et al.A pathway-based classification of human breast cancer[J].Proceedings of the National Academy of Sciences,2010,107:6994-6999. |
| [11] | Watanabe T,Kobunai T,Yamamoto Y,Matsuda K,Ishihara S,Nozawa K,et al.Chromosomal instability (CIN) phenotype,CIN high or CIN low,predicts survival for colorectal cancer[J].J Clin Oncol,2012,30:2256-2264. |
| [12] | Jorissen R N,Gibbs P,Christie M,Prakash S,Lipton L,Desai J,et al.Metastasis-associated gene expression changes predict poor outcomes in patients with Dukes stage B and C colorectal cancer[J].Clin Cancer Res,2009,15:7642-7651. |
| [13] | Smith J J,Deane N G,Wu F,Merchant N B,Zhang B,Jiang A,et al.Experimentally derived metastasis gene expression profile predicts recurrence and death in patients with colon cancer[J].Gastroenterology,2010,138:958-968. |
| [14] | de Sousa E Melo F,Colak S,Buikhuisen J,Koster J,Cameron K,de Jong J H,et al.Methylation of cancer-stem-cell-associated Wnt target genes predicts poor prognosis in colorectal cancer patients[J].Cell Stem Cell,2011,9:476-485. |
| [15] | Laibe S,Lagarde A,Ferrari A,Monges G,Birnbaum D,Olschwang S.A seven-gene signature aggregates a subgroup of stage Ⅱ colon cancers with stage Ⅲ[J].OMICS,2012,16:560-565. |
| [16] | Tsuji S,Midorikawa Y,Takahashi T,Yagi K, Takayama T,Yoshida K,et al.Potential responders to FOLFOX therapy for colorectal cancer by Random Forests analysis[J].Br J Cancer,2011,106:126-132. |
| [17] | Khambata-Ford S,Garrett C R,Meropol N J,Basik M,Harbison C T,Wu S,et al.Expression of epiregulin and amphiregulin and K-ras mutation status predict disease control in metastatic colorectal cancer patients treated with cetuximab[J].J Clin Oncol,2007,25:3230-3237. |
| [18] | http://www.bioconductor.org/packages/ 2.14/bioc/html/affy.html |
| [19] | Leek J T,Johnson W E,Parker H S,Jaffe A E,Storey J D.The sva package for removing batch effects and other unwanted variation in high-throughput experiments[J].Bioinformatics,2012,28:882-883. |
| [20] | http://www.bioconductor.org/packages/2.13/bioc/ html/limma.html |
| [21] | Yasrebi H.SurvJamda:an R package to predict patients' survival and risk assessment using joint analysis of microarray gene expression data[J].Bioinformatics,2011,27:1168-1169. |
| [22] | Rex D K,Ahnen D J,Baron J A,Batts K P,Burke C A,Burt R W,et al.Serrated lesions of the colorectum:review and recommendations from an expert panel[J].Am J Gastroenterol,2012,107:1315-1329. |
| [23] | Huang C S,O'Brien M J,Yang S,Farraye F A.Hyperplastic polyps,serrated adenomas,and the serrated polyp neoplasia pathway[J].Am J Gastroenterol,2004,99:2242-2255. |
| [24] | de Roock W,Claes B,Bernasconi D,de Schutter J,Biesmans B,Fountzilas G,et al.Effects of KRAS,BRAF,NRAS,and PIK3CA mutations on the efficacy of cetuximab plus chemotherapy in chemotherapy-refractory metastatic colorectal cancer:a retrospective consortium analysis[J].Lancet Oncol,2010,11:753-762. |
 2014, Vol. 35
2014, Vol. 35


