为了提高对于乳腺癌差异基因筛选的准确率,从分子层面出发,结合拷贝数与基因表达两方面特征,分析了乳腺癌差异表达基因,研究了乳腺癌的发病机制,为乳腺癌的诊疗提供了新的研究思路.在癌症基因组图谱中下载乳腺癌的拷贝数和基因表达数据,利用R软件通过卡方检验提取乳腺癌拷贝数差异基因,结合edgeR差异基因分析算法,筛选乳腺癌差异表达基因,利用ks检验关联两方面差异基因,分析其相关性,结合string数据库构造蛋白质互作网络,筛选核心基因,通过生存分析和GO富集分析验证结果的准确性.以基因差异表达倍数大于1,p值小于0.05为标准,筛选出基因表达差异基因共有10 579个,上调基因7 543个,下调基因3 036个,经验证发现,ATAD2B等8个基因与乳腺癌的发生发展密切相关.
In order to improve the accuracy of screening differential genes for breast cancer, the differential expression genes of breast cancer was analyzed from the molecular level, combined with the characteristics of copy number and gene expression, studied the pathogenesis of breast cancer and provided new research ideas for the diagnosis and treatment of breast cancer. The cancer genome atlas database was used to download copy number and gene expression data of breast cancer, chi square test was used to extract copy number difference genes of breast cancer. Through R software, edgeR differential gene analysis algorithm was used to screen differentially expressed genes in breast cancer, ks test was used to correlate two differentially expressed genes to analyze the relationship between CNV variation and gene expression, string database was used to construct protein interaction network to screen core genes, the accuracy of the results was verified by survival analysis and go enrichment analysis. According to the standard of FDR greater than 1, p value less than 0.05, 10 579 genes were screened, 7 543 genes were up-regulated and 3 036 genes were down-regulated. It was found that eight genes such as ATAD2B were closely related to the occurrence and development of breast cancer.
目前,乳腺癌是威胁女性健康最常见的恶性肿瘤,其发病率逐年升高[1],并有年轻化的趋势,尤其是在欧美等发达国家和地区,其上升趋势较为明显.在我国,乳腺癌占女性恶性肿瘤疾病的比例是7%~10%,发病率为0.043%.
癌症的异质性会影响患者的治疗和预后反应,乳腺癌是一种异质性疾病[2],其肿瘤内分子表达的差异可能会扰乱靶向治疗,导致治疗反应的混合,这种分子异质性在原发性和转移性疾病中都得到了证实.对于乳腺癌的治疗,虽然可以从手术标本中确定肿瘤的异质性,但关键的治疗决策往往需要从核心活检标本中做出,且仅限于肿瘤的一个区域,不易重复.所以,从分子层面入手研究乳腺癌基因异质性具有很大的优势,可以从根本上提出更好的乳腺癌诊疗方案,帮助患者获得更好的医疗援助,使病情得到有效的改善.
在癌症的研究中拷贝数变异是很重要的,因为它们可能位于携带致癌基因或抑癌基因的区域,这种基因组改变通过其对表达的影响来介导表型改变.拷贝数改变,包括种系变异和体细胞拷贝数异常,这些都与疾病有关,体细胞畸变对肿瘤的发生尤为重要. CCL3L1的高拷贝数与HIV感染的低易感性相关,FCGR3B[3]的低拷贝数可增加系统性红斑狼疮和类似炎症性自身免疫性疾病的易感性.姜鑫钊等[4]应用聚合酶链式反应—限制性内切酶多态性技术,对乳腺癌样本与正常样本进行了对照检测,发现APOBEC3拷贝数的变异与乳腺癌的发病风险存在较大关联,等位基因del是乳腺癌发病的危险因素;Freeman等[5]经研究发现PTEN基因拷贝数的缺失会导致基因抑癌性的失活.
DNA转录成RNA是基因表达的关键遗传过程,转录的干扰直接影响mRNA的表达,进而影响蛋白质的产生,导致病理状态. Chen等[6]对影响浸润性乳腺癌的基因做了研究,发现Her-2/neu基因在表达方面有明显的过表达现象;李莹莹等[7]通过免疫组织化法检测乳腺癌组织中的基因表达情况,讨论了其与乳腺癌预后的关系,发现了PD-1等可以作为提示肿瘤预后的靶标;董赟等[8]应用荧光原位杂交检测法,检测了原发性乳腺癌中HER-2基因的表达情况,探讨了原发性乳腺癌中HER-2基因表达的差异性;史源等[9]采用实时荧光定量聚合酶链反应检测技术,检测了乳腺组织中BRMS1的基因表达水平,发现BRMS1基因的低表达与乳腺癌的发展与转移密切相关.
基于以往的研究发现,对于乳腺癌基因价值的挖掘大多基于单方面特征,然而,现有研究表明,基于单方面基因变异引发乳腺癌的情况较为局限,乳腺癌的发生发展过程由很多因素共同作用产生.通过对乳腺癌拷贝数与基因表达的联合分析得到乳腺癌的差异基因,并且对于筛选出的核心差异基因进行了生存分析和GO富集分析,结合基因突变概率和生存率,验证核心差异基因的准确性.相较于以往根据单特性的方式对于基因的筛选具有更高的准确性.
1 材料和方法 1.1 数据来源乳腺癌样本的拷贝数、基因表达数据来源于癌症基因组图谱(TCGA,the cancer genome atlas)数据库,从TCGA网站可以获得TCGA level3公开的数据.通过TCGA官网下载乳腺癌拷贝数数据的metadata文件和manifest文件,由于乳腺癌样本的拷贝数数据过大,需要通过GDC下载工具进行cart文件下载,笔者研究的主要是体细胞发生变化的基因,所以在下载过程中只选择Masked Copy Number Segment类型的数据.基因表达数据下载方式与拷贝数下载方式一致,其中对于乳腺癌拷贝数数据下载的样本数为2 229例,对于基因表达数据下载的样本数为1 222例.
1.2 拷贝数变异筛选拷贝数变异可能存在于癌症的致癌基因或抑癌基因区域,这种基因组的改变会对基因表达造成一定的影响,所以在癌症研究中具有很大的意义.主要利用R统计软件对原始数据进行处理.
已下载的拷贝数数据主要包括TCGA数据id、所处染色体区段的起始位置和终止位置,区段中的探针数以及相应的mRNA表达值.首先对原始拷贝数数据进行基因注释,利用基因的位置信息和拷贝数的位置信息筛选出在相应染色体区段存在的基因,形成以基因名为行名,样品id为列名,相互对应值为拷贝数值的表格.读取每个基因的拷贝数值,分别统计基因在癌症样品和正常样品中的拷贝数变化值,以得到每个基因在正常样品中拷贝数发生变化的样品数(normalCNV),正常样品中拷贝数没有发生变化的样品数(normalWild),癌症样品中拷贝数发生变化的样品数(tumorCNV),癌症样品中拷贝数没有发生变化的样品数(tumorWild),分别计算正常样本和癌症样本中,拷贝数发生变异和没有发生变异的概率,作为卡方检验的输入值,用于计算卡方检验统计值和p值,其中卡方检验统计值越大,拷贝数变异基因致癌率越高.利用Bonferroni校正方法对p值进行校正,以矫正后的p值小于0.05作为标准进行分析,p值越小,基因发生拷贝数变异的概率越高,从而获取拷贝数差异基因.
1.3 基因表达变异筛选将原始数据整理成矩阵文件,由于TCGA下载后的基因表达原始数据是独立的count文件,利用metadata文件得到数据下载文件名和样品名的对应关系,进一步区分该样本是癌症样本还是正常样本,最终得到一个M×N的矩阵,行代表的是基因id,列代表样品,其中元素xmn(m=1,2,…,M;n=1,2,…,N)代表第n个样品中第m个基因的表达值.然后,利用R软件edgeR包对基因表达矩阵进行差异表达分析,获得差异表达基因.
1.4 拷贝数与基因表达关联分析为了分析拷贝数变异对基因表达的影响,选取乳腺癌中拷贝数变异和差异表达方向一致的基因进行分析.将拷贝数差异基因矩阵文件和表达差异基因矩阵文件合并,由于样品名前4个字段代表样品信息,后4个字段代表实验信息,拷贝数和基因表达的实验信息具有一定的差异性,所以按照样品名前4个字段将其进行合并,选取基因和样品的交集,得到新的矩阵文件.新的矩阵文件中,其中的样品名的第4个字段0开头代表该样品是癌症样品数据,1、2开头代表正常样本数据,主要研究拷贝数变异对基因表达差异的影响,所以优先过滤正常样本数据,对癌症样品数据进行ks检验(Kolmogorov-Smirnov test),得到的p值为1代表拷贝数与表达之间不具有相关性,以p值为0.05作为标准,p值越小代表它们之间的相关性越强.
1.5 构建蛋白质互作网络分析核心差异基因选中乳腺癌差异表达基因,利用string在线数据库,将可信度设为0.7,并去掉其中的游离节点,得到蛋白质互作网络(PPI,protein-protein interaction network),并下载其中的基因关系对文件.使用R语言,利用PPI网络基因关系对得到其中的核心基因.
2 结果 2.1 乳腺癌中显著的拷贝数变异区域利用TCGA乳腺癌拷贝数数据,筛选拷贝数变异基因,其中在TCGA官网上下载的拷贝数数据共有2 229个,normal样本数为1 116个,tumor样本数为1 113个,通过卡方检验,以矫正后的p值小于0.05作为标准进行分析. 图 1所示为拷贝数变异圈图中1号染色体到4号染色体中变异基因的部分截取.其中外圈代表染色体编号,内圈最底部代表拷贝数丢失区域,内圈中部、上部代表拷贝数扩增区域.
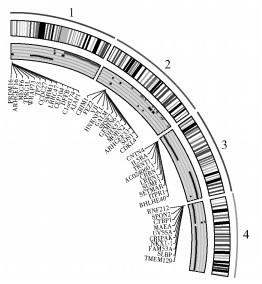
|
图 1 1~4号染色体拷贝数变异部分圈图 |
为了分析基因在癌症样本与正常样本中不同的表达情况,下载TCGA mRNA基因表达数据1 222个,其中R软件分析得到的正常样本数目为113个,癌症样品数为1 109个,以差异倍数大于1,即差异倍数为2倍以上.矫正后的p值小于0.05为过滤标准,利用R软件edgeR包进行分析,得到具有显著差异表达基因共有10 579个,其中上调基因7 543个,下调基因3 036个.如图 2所示,其中横坐标为矫正后的p值,横坐标越大,基因表达差异越明显,纵坐标为差异倍数,越往两边发散表示基因表达的差异越大,其中中间虚线上半部分表示基因表达上调,下半部分表示基因表达下调.
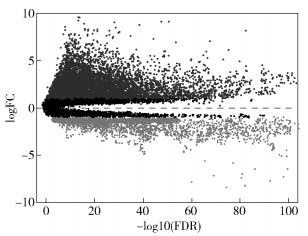
|
图 2 基因表达火山图 |
为了探索拷贝数发生变异的同时,表达也发生变化的基因,将乳腺癌拷贝数变异的11 216个基因结合表达发生变化的10 579个基因,利用ks检验出有76个基因具有显著相关性(p < 0.05).
2.4 蛋白质互作网络构建筛选核心差异基因通过string数据库导入差异表达基因后,构造出蛋白质互作网络,并利用cytoscape软件对网络进行编辑,核心差异基因之间的连接关系如图 3所示.利用R软件统计核心基因的排序情况如图 4所示,其中纵坐标代表核心基因名称,横轴数值代表核心基因的连接情况,数值越大代表连接节点数越多,基因越核心.
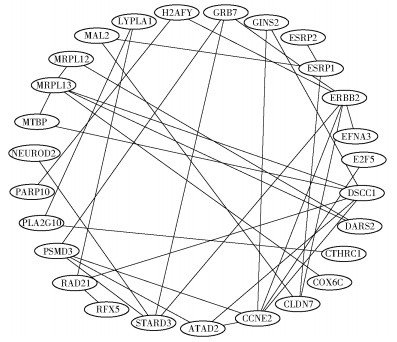
|
图 3 乳腺癌差异表达基因蛋白质互作网络 |
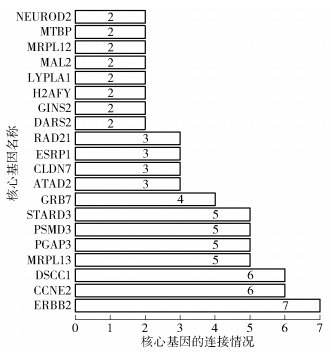
|
图 4 蛋白质互作网络核心基因统计 |
通过对乳腺癌基因拷贝数和基因表达数据分析,得到其中具有显著差异的核心基因包括:ERBB2、CCNE2、DSCC1、MRPL13、PGAP3、MAL2、MRPL12、MTBP、NEUROD2.拷贝数与基因表达具有一定的相关性,当拷贝数扩增时,基因表达上调,例如:ATAD2B、MRPL13、PGAP3、PSMD3、GRB7、LYPLA1、MAL2、NEUROD2、STARD3等,其拷贝数与基因表达之间的关系如图 5所示.
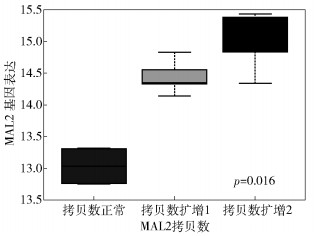
|
图 5 差异表达核心基因拷贝数与基因表达关系 |
将核心差异基因结合患者的生存状态进行生存分析,利用TCGA数据库下载乳腺癌临床信息文件,提取其中患者的随访时间或死亡时间以及生存状态,结合基因表达数据,利用R软件survival包进行生存分析,得到图 6所示的生存曲线.研究结果显示,这些与乳腺癌患者生存率相关的基因,在患者中的过度表达与其生存率低相关,表明这些基因很可能与乳腺癌预后不良有关.同时利用cosmic数据库,提取单一差异基因在乳腺癌中的突变概率,与其发生突变概率的最高基因位点信息,通过基因突变概率最高的位点,可为以后的研究提供更为准确的方向,同时结合患者观察2周后的生存率,绘制出如下表格.从表 1可以直观地看出,基因突变概率高的患者生存率较低.
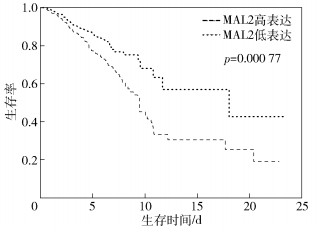
|
图 6 差异表达核心基因生存分析 |
|
|
表 1 核心差异基因 |
为了进一步研究核心差异基因的基因功能信息以便后续对于乳腺癌的治疗更具针对性,对其进行GO富集分析,利用R软件读取差异表达基因,将基因名转化成向量,通过与org. Hs. eg. db R包的比对,将差异表达基因名与基因id进行匹配,并过滤少部分没有匹配基因id的基因,进而利用ClusterProfiler R包,将基因id与数据库中基因的功能信息进行比对,进行GO富集分析.分析可知,差异表达基因主要富集在如图 7所示的区域.其中,横坐标代表富集在每部分的基因占输入总基因数目的比例,圆圈大小代表GO的数目.其中差异表达基因主要富集在分子功能处,其p值较小,富集程度较高,包括蛋白质的结合、维生素受体结合、钙离子结合、RNA聚合酶Ⅱ核心启动子序列与DNA的结合等.
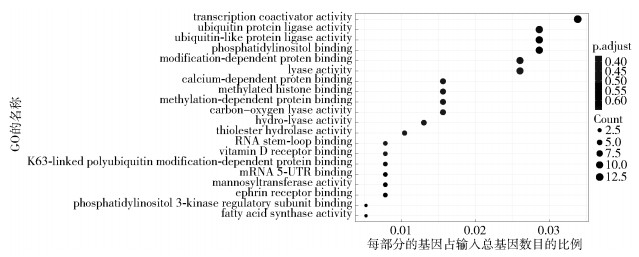
|
图 7 差异表达基因富集分析 |
结合以往研究,对核心差异基因进行分析,GRB7基因编码一个多域信号转导分子,是HER-2扩增子核心的一部分,GRB7通常与HER-2共扩增和过度表达,在体外和体内对曲妥珠单抗和拉帕替尼的HER-2阳性乳腺癌细胞的生长中起着重要作用[10]. H2AFY基因参与了RNA聚合酶Ⅰ启动子的转录调控,同时它也是Plcy1的下游靶点,参与了乳腺癌的肺转移形成[11].目前,LYPLA1在乳腺癌中的生物学机制尚不清楚,但经证实,shRNA通过抑制LYPLA1在体外的表达,可产生抑制NSCLC细胞增殖、迁移和侵袭的效果[12]. MAL2是MAL蛋白脂质家族的一员,在细胞转化中起着重要作用,实验研究表明,MAL2可以调节EMT,抑制MAL2的表达,可降低乳腺癌细胞株增殖、迁移和侵袭[13].
NEUROD2拷贝数增加与高肿瘤分级、高核分裂计数和5年生存率下降有关,并且NEUROD2拷贝数增加是独立的预后因素[14]. ATAD2B属于高度进化保守的蛋白质家族,微阵列分析和免疫组化分析均显示胶质母细胞瘤和少突胶质瘤中ATAD2B有显著的表达,乳腺癌免疫染色强烈,因此,ATAD2B可能在肿瘤发展中起作用[15]. PGAP3作为糖基磷脂酰肌醇(GPI)特异性磷脂酶的一员,调节糖基磷脂酰肌醇锚区的脂质重塑[16],在乳腺癌样本中检测出PGAP3和ERBB2位点特异性扩增,鉴于ERBB2是一种大家所熟知的参与乳腺癌的基因,所以PGAP3的拷贝数变异很可能参与乳腺癌的形成[17],STARD3与ERBB2位于同一基因座[18],它的过度表达会有助于增加乳腺癌细胞的增殖、迁移和侵袭[19].还有一些基因,在目前的研究中并未显示与乳腺癌有直接联系,但是已经确定与其他癌症具有一定联系,可以进一步研究它们与乳腺癌的预后价值.例如:MRPL13是一种线粒体核糖体蛋白,MRPL13的丢失会导致线粒体DNA的丢失[20],并最终导致线粒体编码蛋白的能力丧失,在一项研究中,降低肝癌中MRPL13的表达是调节线粒体核糖体和氧化磷酸化缺乏的关键因素,氧磷缺乏可调节肝癌细胞的侵袭性活性[21]. PSMD3是RNA聚合酶的一个亚单位[22],可以诱导cox2、s100a4/fsp-1和波形蛋白基因在肾脏中的表达,这有助于糖尿病介导的染色质转换,并促进糖尿病肾脏的转录变化[23].
4 结束语通过对GRB7、H2AFY等筛选出的差异表达基因进行生存分析和功能富集分析,并结合以往的研究成果可以看出,通过对乳腺癌拷贝数与基因表达差异分析所得到的核心差异基因具有较高的研究价值.通过结合乳腺癌拷贝数和基因表达两方面的特征,以蛋白质互作网络为基础,进而筛选乳腺癌核心差异基因,相对于单一特征的筛选,结果更为准确.针对筛选出的核心基因,进行GO富集分析和生存分析,在验证其准确性的同时阐述其研究价值.本方法在分子层面进行乳腺癌基因的预测,增加了治疗的可靠性,提高了患者的治疗效率,对于乳腺癌在日后发生、发展过程中的预测、治疗和预后提供了更好的保障, 并为更多的研究人员对于分子层面进行癌症相关分析工作提供了新的诊疗思路.
| [1] |
邹杰民.基于乳腺癌基因组数据的分析与可视化平台实现[D].长沙: 湖南大学, 2017.
|
| [2] |
Mahrooghy M, Ahmed A, Daye D, et al. Pharmacokinetic tumor heterogeneity as a prognostic biomarker for classifying breast cancer recurrence risk[J]. IEEE Transactions on Biomedical Engineering, 2015, 62(6): 1585-1587. DOI:10.1109/TBME.2015.2395812 |
| [3] |
Xu Yanxun, Zhang Jie, Yuan Yuan, et al. A bayesian graphical model for integrative analysis of TCGA data[J]. International Workshop on Genomic Signal Processing and Statistics, 2012(12): 2-4. |
| [4] |
姜鑫钊, 张明龙, 杜媛媛, 等. APOBEC3基因拷贝数变异与乳腺癌易感性的关联[J]. 基因组学与应用生物学, 2019(3): 1315-1321. Jiang Xinzhao, Zhang Minglong, Du Yuanyuan, et al. Correlation between copy number variation of APOBEC3 gene and breast cancer susceptibility[J]. Genomics and Applied Biology, 2019(3): 1315-1321. |
| [5] |
Freeman S S, Allen S W, Ganti R, et al. Copy number gains in EGFR and copy number losses in PTEN are common events in osteosarcoma tumors[J]. Cancer, 2008, 113(6): 1453-1461. DOI:10.1002/cncr.23782 |
| [6] |
Sheng Chen, Feifei Gu, Kang Li, et al. A hybrid of B and T lymphoblastic cell line could potentially substitute dendritic cells to efficiently expand out Her-2/neu-specific cytotoxic T lymphocytes from advanced breast cancer patients in vitro[J]. Journal of Hematology & Oncology, 2017, 10(1): 63. |
| [7] |
李莹莹, 董丽儒, 李宇阳, 等. PD-1/PD-L1在非特殊型浸润性乳腺癌中的表达及意义[J]. 广东医学, 2018(11): 1690-1693. Li Yingying, Dong Liru, Li Yuyang, et al. The expression and significance of PD-1/PD-L1 in nonspecific invasive breast cancer[J]. Guangdong Medical Journal, 2018(11): 1690-1693. |
| [8] |
董赟.单双侧原发性乳腺癌HER-2基因扩增差异及与临床预后的关系分析[D].南昌: 南昌大学, 2016.
|
| [9] |
史源, 胡建民. 乳腺癌患者血清BRMS1mRNA的表达及意义分析[J]. 临床研究, 2019, 27(11): 108-109. Shi Yuan, Hu Jianmin. Expression and significance of BRMS1mRNA in breast cancer patients[J]. Clinical Research, 2019, 27(11): 108-109. |
| [10] |
Luoh S W, Wagoner W, Wang Xiaoyan, et al. GRB7 dependent proliferation of basal-like, HER-2 positive human breast cancer cell lines is mediated in part by HER-1 signaling[J]. Mol Carcinog, 2019, 58(5): 1-7. |
| [11] |
Dong Ying, Zhang Ting, Li Xining, et al. Comprehensive analysis of coexpressed long noncoding RNAs and genes in breast cancer[J]. J Obstet Gynaecol Res, 2018, 45(2): 428-437. |
| [12] |
Mohammed A, Zhang Caixin, Zhang Shuwen, et al. Inhibition of cell proliferation and migration in non-small cell lung cancer cells through the suppression of LYPLA1[J]. Oncology Reports, 2019, 41(2): 973-975. |
| [13] |
Bhandari A, Shen Yanyan, Sindan N, et al. MAL2 promotes proliferation, migration, and invasion through regulating epithelial-mesenchymal transition in breast cancer cell lines[J]. Biochem Biophys Res Commun, 2018, 504(2): 434-439. DOI:10.1016/j.bbrc.2018.08.187 |
| [14] |
Lacle M M, Moelans C B, Kornegoor R, et al. Chromosome 17 copy number changes in male breast cancer[J]. Cell Oncol, 2015, 38(3): 1-9. |
| [15] |
Leachman Na T, Brellier F, Ferralli J, et al. ATAD2B is a phylogenetically conserved nuclear protein expressed during neuronal differentiation and tumorigenesis[J]. Develop Growth Differ, 2010, 52(9): 747-755. DOI:10.1111/j.1440-169X.2010.01211.x |
| [16] |
Sio Y Y, Anantharaman R, Lee S Q E, et al. The asthma-associated PER1-like domain-containing protein 1(PERLD1) haplotype influences soluble glycosylphosphatidylinositol anchor protein (sGPI-AP) levels in serum and immune cell proliferation[J]. Scientific Reports, 2020, 10(1): 1-11. DOI:10.1038/s41598-019-56847-4 |
| [17] |
Pan Xiaoyong, Hu Xiaohua, Zhang Yuhang, et al. Identification of the copy number variant biomarkers for breast cancer subtypes[J]. Molecular Genetics and Genomics, 2019, 294: 95-110. DOI:10.1007/s00438-018-1488-4 |
| [18] |
Felicio1 P S, Bidinotto L T, Melendez M E, et al. Genetic alterations detected by comparative genomic hybridization in BRCAX breast and ovarian cancers of Brazilian population[J]. Oncotarget, 2018, 9: 27525-27534. DOI:10.18632/oncotarget.25537 |
| [19] |
Qi Feng, Qin Wenxing, Zang Yuansheng, et al. Molecular mechanism of triple-negative breast cancer-associated brca1 and the identification of signaling pathways[J]. Oncology Letters, 2019, 17(3): 2905-2914. |
| [20] |
Cámara Y, Asin-Cayuela J, Park C B, et al. MTERF4 regulates translation by targeting the methyltransferase NSUN4 to the mammalian mitochondrial ribosome[J]. Cell Metabolism, 2011, 13(5): 527-539. DOI:10.1016/j.cmet.2011.04.002 |
| [21] |
Wang Ke, Li Ling, Fu Liang, et al. Integrated bioinformatics analysis the function of RNA binding proteins (RBPs) and their prognostic value in breast cancer[J]. Original Research Article, 2019, 10: 1-11. |
| [22] |
Gao H, Duan Y, Fu X, et al. Comparison of efficacy of SHENQI compound and rosiglitazone in the treatment of diabetic vasculopathy analyzing multi-factor mediated disease-causing modules[J]. PLoS ONE, 2018, 13(12): 1-18. |
| [23] |
Gao Hong, Duan Yuhong, Fu Xiaoxu, et al. Comparison of efficacy of SHENQI compound and rosiglitazone in the treatment of diabetic vasculopathy analyzing multi-factor mediated disease-causing modules[J]. PLoS One, 2018, 13(12): 1-18. |


