为了提升超声图像中的神经分割效果,提出了一种新的网络结构残差U型网络.相比于现有的U-net网络,残差U型网络加深了网络结构,提高了网络的表达能力;通过对每层参数进行规范化处理,减少了训练时间,提高了神经分割效果.实验结果表明,残差U型网络在分割效果比U-net网络提升了约13%,比SegNet网络提升了约7%.
To improve the efficiency of neural segmentation in ultrasound images, we propose a new neural structure the U-shape residual network. Compared with U-net network, this structure deepens the original structure to improve the expression ability. By standardizing the parameters of each layer, the structure reduces the training time and improve the segmentation effect. According to the results, the U-shape residual network segmentation effect increased by 13% compared with U-net network and improved about 7% compared with SegNet network.
超声图像作为一种重要的现代医疗手段,已经成为医生判断病人身体状况的重要标准.同时超声图像对于临床手术的实施也具有着重要的意义[1].传统的图像分割方法通过人工的手段对于图像中的特征信息进行提取,这种方式不仅需要大量的时间和精力,同时还需要相关领域的专业知识.相比于传统方法,采用卷积神经网络对医疗图像进行处理不需要人工提取特征信息.
卷积神经网络作为近年来最为高效的图像处理方法之一,具有准确性高、预处理较少等优势,目前广泛应用于语音识别和医学图像处理领域.将卷积神经网络应用到超声图像分割中[2-8],有助于提高分割神经区域的准确性.由于超声图像呈像的特殊性,图像中含有噪声信息,会对图像的分割造成一定的影响.通过加深卷积神经网络的深度有助于提取图像中更高维度的特征,在一定程度上可减小噪声的影响.但是实验结果表明,随着网络深度的增加,容易出现网络性能退化的问题[9],笔者提出了一种新的神经分割网络结构——残差U型网络,该网络在原有U-net网络基础上引入了残差结构,增加了网络深度,提升网络的表达能力.
实验结果证明,使用残差U型网络对超声图像中神经区域的分割效果高于U-net网络和SegNet网络结构,训练时间略高于U-net网络,远小于SegNet网络,约为SegNet网络的1/5.
1 相关工作传统的卷积神经网络,通常是对整个图像进行预测,但对于医疗图像还远远不够.医疗图像作为最难处理的图像之一,需要对于图像中的语义信息和信息所处的位置同时进行判断,这就需要对图像中的每个像素进行分类判断.
Ciresan等[10]于2012年提出用滑动窗口的方式对每个像素点进行预测,这种方法取得了很大的成功.但这种方式不仅需要大量的计算,训练缓慢,而且如果窗口设置过大,准确率会下降,窗口设置过小,获取的内容不充分.
2015年Ronneberger等[11]提出通过U-net网络对生物医疗图像进行分割,该网络共23层,分为收缩网络和扩张网络两部分.收缩网络中每一层由2个3×3的卷积层和一个2×2的池化层组成,在进行每次下采样的过程中对通道数进行加倍操作.扩张网络与收缩网络相对应,形成了一个U型结构,扩张网络的每一层由2个3×3的卷积层和一个2×2的卷积层对图像进行放大. U-net网络的主要贡献在于通过构建收缩网络和扩张网络,对于图像进行像素级的分析,同时由于收缩网络和扩张网络的对称性,可以在每一次扩张的过程中对于缺失的信息进行一定的补充.
2016年Badrinarayanan等[12]提出了SegNet网络,同样采用对称的结构对图像进行处理. SegNet网络分为编码层和解码层2部分.编码层的结构与VGG16网络相类似,但在每次池化操作的过程中,采用编码器对图像中丢失的信息进行保存.解码层则是相当于图像还原的过程,在还原过程中将编码层存储的信息释放出来,对于边缘信息进行补充.
U-net网络和SegNet网络均采用了编码-解码的形式对图像进行像素级处理,在图像处理方面均取得了不错的效果.
2 残差U型网络 2.1 网络结构改进后的网络结构如图 1所示,分为收缩网络和扩张网络2部分.收缩网络的每一层主要由2个3×3的卷积层、一个1×1的卷积层和一个步数为2的2×2的池化层组成,在进行完每个卷积操作之后,通过Relu函数进行激活,最后进行池化操作.每次下采样采集的特征数量为上一次的1倍.扩张网络的每一层与收缩网络层次相对应,每一层扩张网络由2个3×3的卷积层、一个1×1的卷积层和一个步数为2的2×2的池化层组成,在进行卷积之前,需要将获得的输入矩阵与其所对应收缩层的输出矩阵合并,进行卷积操作,通过Relu函数进行激活,在每个扩张层的最后增加一个2×2的上采样操作对图像进行放大,每次上采样采集的特征为上一次的一半.最后,加入1×1的卷积网络确定该特征图所对应的结果,通过Sigmod分类器进行分类.改进后的网络一共32层.
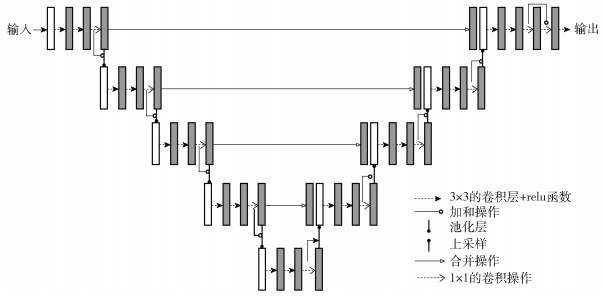
|
图 1 残差U型网络结构 |
相比于原网络,新型网络的主要贡献在于在网络结构中融入了残差结构,增加了网络深度,提升了网络的表达能力,使其能够更加准确地表达出每个像素点的信息.
2.2 残差结构残差网络结构如图 2所示.残差网络提出了一种拟合残差映射的方法,即不直接将卷积结果作为输出,而是采用残差映射的方式进行计算.残差网络通过将多个卷积层级联的输出与输入相加的方式对图片进行特征提取,减少了训练参数.
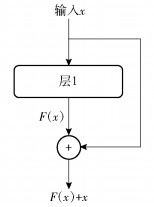
|
图 2 残差网络结构 |
网络结构可表示为
| $ H\left( x \right) = F\left( x \right) + bx $ | (1) |
这里H(x)和x表示该网络的输出及输入,F(x)为经过MLP卷积层之后的输出结果,b表示对于原有输入结果的保留程度,本次试验中设置为0.1.
MLP卷积层的引入不但简化了深度学习模型,而且增强了网络的表达能力,加深网络的深度.
残差结构的引入使得U-net的网络结构更加深入,通过MLP卷积层可以加大对于每个像素点特征卷积的局部感受野,采用残差的结构也避免了由于网络层数加深引起的网络性能退化问题.
3 实验 3.1 实验数据本次实验采用部分臂神经丛数据来进行实验,训练样本有5 000组超声图像,测试样本有5 000组.臂神经丛由第5节颈椎神经(C5)到第1节胸椎神经(T1)的前支构成.是头、颈、上肢内连接锁骨、上臂、前臂、手的神经丛的名称.主要支配上肢和肩背、胸部的感觉和运动[13]. 图 3所示为部分臂神经丛的超声图像,图中标记出来的部分为需要识别出的神经区域.
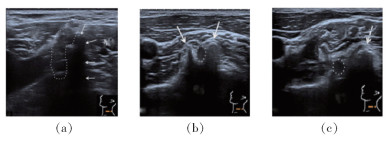
|
图 3 超声图像 |
本次试验中将输入图片大小处理为64×80,对于输入进来的图片,通过计算输入矩阵的均值和方差,对数据进行归一化操作,提升训练速度.
3.3 训练模型本次训练中采用Adam作为优化函数,学习速率为10-5,采用高斯分布的方式设置神经网络的初始权重.在NVIDIA Tesla K40 GPU下进行训练.
训练中使用的损失函数为
| $ L\left( {X, Y} \right) = \frac{1}{N}\sum\limits_{i = 1}^N {S\left( {X, Y} \right)} $ | (2) |
其中:X表示预测值,Y表示参考值,S(X, Y)表示2个模型之间的相似程度,
| $ S\left( {X, Y} \right) = 1 - \frac{{2\left| {X \cap Y} \right| + k}}{{\left| X \right| + \left| Y \right| + k}} $ | (3) |
由于部分超声图像中没有神经区域,所以会出现空图的现象,再次加入平滑值k对函数进行修正.损失函数介于0~1之间,损失函数越小,模型的鲁棒性越好.
4 实验结果及分析 4.1 评价标准本次实验中采用Dice系数(dice coefficient)和ROC曲线来对模型的进行评价.
Dice系数是一种集合相似度函数,用来评判两个样本之间的相似程度,2个样本相似度越好,Dice系数越大. Dice系数如式(4)所示.
| $ S = \frac{{2\left| {X \cap Y} \right|}}{{\left| X \right| + \left| Y \right|}} $ | (4) |
其中:|X∩Y|表示两个样本之间的相交部分或重叠部分,|X|+|Y|表示预测值与参考值的总量.本次实验中当预测值与参考值完全相同时,Dice系数为1;当预测值与参考值不相关时,Dice系数为0.所以Dice系数越大,表明2个图像相似度越高,模型越准确.
ROC曲线也称受试者工作特征曲线,是反映敏感性和特异性连续变量的综合指标,该图像以敏感性为纵坐标、(1-特异性)为横坐标绘制成曲线,曲线下面积越大,准确性越高.
在分类问题中,将实例分为正例和负例两部分,将正例预测成为正例,称之为真正类(TP, true positive),将负例预测成为正例,称为假正类(FP, false positive);反之,将负例预测成负例,称之为真负类(TN, true negative),将负例预测成为负例,称为假负类(FN, false negative)
在ROC曲线中,以假正类率(FPR, false positive rate)作为横轴,真正类率(TPR, true positive rate)作为纵轴,对图像进行绘制.如式(5)(6)所示.其中PFTR表示假正类率, PTPR表示真正类率,SFP表示假正类,STP表示真正类,STN表示真负类,SFN表示假负类.
| $ {P_{{\rm{FTP}}}} = \frac{{{S_{{\rm{FP}}}}}}{{{S_{{\rm{FP}}}} + {S_{{\rm{TN}}}}}} $ | (5) |
| $ {P_{{\rm{TPR}}}} = \frac{{{S_{{\rm{TP}}}}}}{{{S_{{\rm{TP}}}} + {S_{{\rm{FN}}}}}} $ | (6) |
表 1~表 3所示分别为U-net网络、SegNet网络和残差U型网络的训练结果.
|
|
表 1 U-net网络结构测试结果 |
|
|
表 2 SegNet网络结构测试结果 |
|
|
表 3 残差U型网络结构测试结果 |
从图中可以看出残差U型网络网络分割效果明显高于SegNet与U-net网络.与U-net网络相比,残差U型网络的网络分割效果提升了13%,与SegNet相比,效果提升了7%.
4.2.2 训练时间评估表 4所示为3种网络训练参数以及训练时间的比较,可以看出,残差U型网络的网络训练参数高于U-net网络,远小于SegNet网络;网络训练时间略高于U-net网络,但远小于SegNet网络,训练时间约为SegNet网络的1/5.
|
|
表 4 3种不同网络的训练参数 |
图 4所示为训练过程中为损失函数与迭代次数的关系.
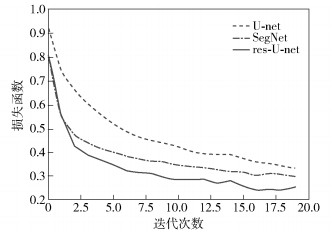
|
图 4 网络迭代次数与损失函数的关系 |
通过对比3种模型的训练过程,残差U型网络在训练过程中下降得更多,准确率高于U-net与SegNet网络.可以得出结论,残差U型网络的鲁棒性优于U-net网络与SegNet网络,实验结果也证明残差U型网络的网络分割效果高于U-net网络和SegNet网络.
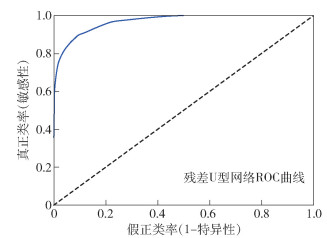
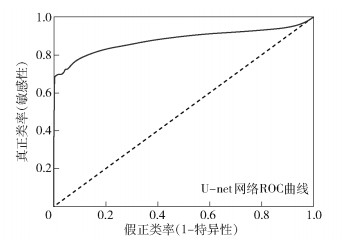
4.2.4 ROC曲线图 5~图 7分别为3种网络的ROC曲线图.可以看出,残差U型网络的面积大于SegNet网络和U-net网络,具有更高的准确性.
|
图 5 残差U型网络的网络ROC曲线 |

|
图 6 SegNet网络ROC曲线 |
|
图 7 U-net网络ROC曲线 |
图 8所示为采用3种网络结构对于同一超声图像的分割结果.可以看出,残差U型网络的分割效果好于U-net网络和SegNet网络.

|
图 8 3种不同的网络对超声图像神经网络的预测 |
实验结果表明,残差U型网络的分割效果优于U-net网络和SegNet网络,与U-net网络相比,残差U型网络的网络分割效果提升了13%,训练时间有所增加;与SegNet网络相比,残差U型网络的网络分割效果提升了7%,训练时间约为SegNet的1/5.
5 结束语笔者提出采用深度学习的方法对超声图像中的神经区域进行分割.目前已有的深度学习网络主要有U-net网络和SegNet网络,但是由于超声图像的呈像特点,这2种网络的效果并不是十分理想.为了更好地分割超声图像中的神经区域,提出了残差U型网络结构.该结构在U-net网络基础上通过引入残差结构,加深了网络结构深度,提高了神经分割效果.实验结果证明,残差U型网络的网络分割效果较U-net网络和SegNet网络有了显著提升.
| [1] | Hafiane A, Vieyres P, Delbos A. Phase-based probabilistic active contour for nerve detection in ultrasound images for regional anesthesia[J]. Computers in Biology & Medicine, 2014, 52(3): 88–95. |
| [2] | Xu Z, Huang J. Detecting 10, 000 cells in one second[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer International Publishing, 2016: 676-684. |
| [3] | Xu Z, Huang J. Efficient lung cancer cell detection with deep convolution neural network[C]//International Workshop on, Patch-Based Techniques in Medical Imaging. Cham: Springer International Publishing, 2015: 79-86. |
| [4] | Pan H, Xu Z, Huang J. An effective approach for robust lung cancer cell detection[C]//International Workshop on, Patch-based Techniques in Medical Imaging. Cham: Springer International Publishing, 2015: 87-94. |
| [5] | Zhu X, Yao J, Zhu F, et al. WSISA: making survival prediction from whole slide histopathological images[C]//IEEE Conference on Computer Vision and Pattern Recognition(CVPR). Honolulu: IEEE Computer Society, 2017: 6855-6863. |
| [6] | Yao J, Zhu X, Zhu F, et al. Deep correlational learning for survival prediction from multi-modality data[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer International Publishing, 2017: 406-414. |
| [7] | Wang S, Yao J, Xu Z, et al. Subtype cell detection with an accelerated deep convolution neural network[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer International Publishing, 2016: 640-648. |
| [8] | Yao J, Wang S, Zhu X, et al. Imaging biomarker discovery for lung cancer survival prediction[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer International Publishing, 2016: 649-657. |
| [9] | He K, Zhang X, Ren S, et al. Deep residual learning for image recognition[C]//IEEE Conference on Computer Vision and Pattern Recognition(CVPR). Las Vegas: IEEE Computer Society, 2016: 770-778. |
| [10] | Dan C C, Giusti A, Gambardella L M, et al. Deep neural networks segment neuronal membranes in electron microscopy images[J]. Advances in Neural Information Processing Systems, 2012, 25(1): 2843–2851. |
| [11] | Ronneberger O, Fischer P, Brox T. U-Net: convolutional networks for biomedical image segmentation[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer International Publishing, 2015: 234-241. |
| [12] | Badrinarayanan V, Kendall A, Cipolla R. SegNet:a deep convolutional encoder-decoder architecture for scene segmentation[J]. IEEE Transactions on Pattern Analysis & Machine Intelligence, 2017, 39(12): 2481–2495. |
| [13] | Lapegue F, Faruchbilfeld M, Demondion X, et al. Ultrasonography of the brachial plexus, normal appearance and practical applications[J]. Diagn Interv Imaging, 2014, 95(3): 259–275. doi: 10.1016/j.diii.2014.01.020 |


