2. 分子动态与稳态结构国家重点实验室, 北京分子科学国家研究中心, 中国科学院化学研究所, 北京 100190;
3. 中国科学院大学, 北京 100049
2. State Key Laboratory for Structural Chemistry of Unstable and Stable Species, Beijing National Laboratory for Molecular Sciences, Institute of Chemistry, Chinese Academy of Sciences, Beijing 100190, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China
核磁共振(nuclear magnetic resonance,NMR)波谱仪是现代化学及材料科学研究人员获得分子精确结构、展开相关科学研究的必备大型仪器,是推测未知或确证已知化合物结构、探究分子相互作用的最为有力的工具之一,在化学、材料及药学等领域具有非常重要的应用.近年来,随着磁体场强的不断升高、梯度场技术的应用以及超低温探头等技术的发展,NMR谱图的质量得到显著提升.但是,相关领域的研究人员在获得了高质量NMR谱图之后,需要对谱图进行精确指认及解析,才能够最终达到精确探究分子结构的目的.而NMR谱图的精确解析一直是相关科研人员面临的一大难题.NMR谱图通常能够提供化学位移、耦合常数和信号相对积分强度这三类重要信息.其中,化学位移由于能够提供分子内局部化学和磁性环境(即原子核周围的化学环境)等信息,对于精确解析分子结构具有非常重要作用.对化学位移的正确预测及归属是NMR谱图精确解析最核心的工作之一.
目前,NMR化学位移的计算及预测主要有以下几种策略[1]:(1)基于取代基团及化学键距离的增量加和技术(increment-based additivity techniques)[2, 3];(2)基于已知化合物谱图数据库的化学位移检索技术[4];(3)基于第一性原理的化学位移精确计算技术[5].这几种策略各有其优势及局限性.NMRdb[6]、MestReNova[7]、ChemOffice[8]和ACD[9]等软件均是利用基于取代基团及化学键距离的增量加和技术进行化学位移预测,其突出优势是计算速度非常快,几乎在数秒钟内即可得到化学位移预测结果.但由于其本质是在基准值基础上的取代基模型拟合,因此有以下几方面的局限性. 首先,其预测的原子种类必须是其初始拟合模型基准种类之内的原子类型,如果待预测分子超出基准原子类型,则由于没有基准值存在,就不能够依据基准值和取代基的连接方式通过增量加和得到化学位移.其次,增量加和技术没有或很少考虑分子空间三维结构对化学位移的影响,因此对于化学位移受空间影响较为显著或存在非常强的环电流效应的体系,预测精度不理想.同时,增量加和技术的取代基参数基于对一系列训练集分子的拟合,因此对于训练集内的分子或者与训练集分子类型非常相似的分子具有一定的预测能力,而对于新化合物或与训练集内分子结构类型不同的结构,预测效果则不理想,特别是对于一些结构较为复杂的分子,以及用以区分结构类似的分子时,有很大的局限性.而基于已知化合物谱图数据库的化学位移检索技术在预测数据库中已有的分子时,给出的化学位移结果最为准确,因为其相当于直接从数据库获取NMR实验数据.而对于数据库中没有收录的分子,该方法通常提供化学结构类似的分子的化学位移值供用户参考,很难给出准确化学位移.由于有机分子结构多变、化学空间巨大,因此不可能构建覆盖全部化学空间的化合物谱图数据库,所以该方法预测新分子化学位移时就存在一定局限.基于第一性原理的化学位移精确计算技术从基本物理常数出发,开展对分子内秉属性的计算,最终计算得到化学位移,能够充分考虑分子的三维空间结构对化学位移的影响,因此对任意结构分子均能够开展较为准确的化学位移计算.但是第一性原理计算方法较为复杂,高性能计算软硬件的计算环境搭建较为繁琐,耗时相对较长(数十分钟至数小时或数天不等).
近年来,随着计算化学理论的重要进展,以及计算硬件设备性能的大幅提高,利用第一性原理针对有机分子开展 1H 和 13C NMR化学位移的精确计算技术也取得了很大进展,部分方法已经能够用于较大分子及较复杂分子体系的准确预测[5, 10, 11].利用第一性原理计算NMR化学位移的方法在文献[1, 5, 10-13]中有较为详细的描述.在化学位移计算中,泛函方法与基组的恰当选择对于取得适当的计算精度和有效控制计算量具有至关重要的作用,直接影响方法的实用性及使用体验.Tantillo等[13]对一系列已报道的泛函方法进行梳理总结之后,发现先利用M06-2X泛函对目标分子结构进行优化,再利用mPW1PW91泛函对优化之后的分子结构开展屏蔽张量计算,可以在计算时间与计算精度之间取得比较好的平衡,其测试的80个分子的1H与13C NMR化学位移的均方根差分别为0.136 0 ppm及2.529 8 ppm.除了有机分子外,生物大分子的化学位移计算也随着QM/MM方法的发展取得了长足进展[14-18].
虽然随着计算化学理论的发展以及计算硬件设备性能的大幅提高,密度泛函方法目前已能够对比较复杂的有机分子体系开展可信度和精确度较高的化学位移计算,计算时间也在可接受尺度内,但是利用密度泛函方法精确计算有机分子化学位移的技术仍未被广泛使用,主要原因在于该方法步骤较为复杂繁琐,需要使用者具备一定的计算机编程及计算化学基础,不能像NMRdb、ChemOffice、MestReNova及ACD这些程序或软件一样即点即用,让NMR用户耗费大量精力和时间去涉猎计算化学及编程等方向,而仅仅用于进行化学位移计算,这一点较难实现;而且复杂体系的化学位移计算仍然较为不便.
为了解决上述问题,本实验室建设了一套基于密度泛函理论的高精度化学位移计算系统平台(本单位内部IP浏览器访问)——“http://10.8.1.160/iccas”(图 1).本系统平台实现了对一系列复杂计算流程和功能的有效集成,自动完成全计算流程中牵涉到的各软件的输入、调用、输出、链接等.而且用户仅利用浏览器即可操作本系统平台,无需安装任何软件,界面友好,也无需用户具备量子化学及计算机编程基础,全部计算过程用户仅需利用鼠标点击操作即可,页面提交均是实时响应,最大程度简化了用户的操作,真正实现浏览器端的即点即用,可有效提高研究人员利用NMR开展相关研究的效率,为分子结构的高效、精确解析提供了一个辅助工具.
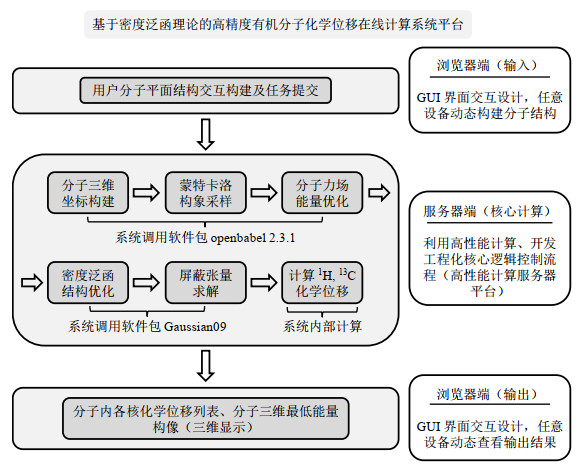
|
图 1 基于密度泛函理论的高精度有机分子NMR化学位移在线计算系统平台架构的示意图 Fig. 1 The architecture of the density functional theory-based on-line NMR chemical shift prediction platform |
本系统平台硬件采用双路Intel Xeon CPU E5-2640 v3 @ 2.60GHz(2*16核心32线程),64GB ECC校验内存.操作系统采用CentOS 7.3,网络架构基于Apache 2.2.3 server平台,采用PHP 5.2构建在线系统.分子构建及分子可视化模块采用Ketcher、JSmol和MolView模块构建.二维分子结构的三维构象采样及基于默克分子力场(Merck molecular force field,MMFF)的分子结构优化采用软件包openbabel 2.3.1,对于小分子的分子动力学模拟,MMFF是目前计算精度较高且应用较为广泛的方法之一[19, 20],本系统设定的优化条件参考文献值[19, 20].经过MMFF优化的分子结构即进入下一步基于密度泛函理论的相关计算.基于密度泛函理论的分子结构优化以及屏蔽张量计算调用软件包Gaussian 09.本系统采用Tantillo等[13]报道验证的参数集,首先在M06-2X/6-31G(d)水平优化,进而在mPW1PW91/6-31G(d)(PCM氯仿溶剂化模型)水平计算屏蔽张量,最终利用标度法,根据(1)式计算各原子化学位移数值[13],其中1H的截距和斜率分别为32.258 7和-1.040 1,13C的截距和斜率分别为193.217 9和-0.953 7[13].
| $ 化学位移 = \frac{{截距 - 屏蔽张量}}{{ - 斜率}} $ | (1) |
对于由动力学效应导致的等价原子(如甲基CH3中的三个质子)的化学位移的计算,使用者可自行按照等价原子化学位移值的平均值来估算.目前,本系统平台支持在线开展分子量小于800的浏览器端分子二维结构构建及任务投递.对于结构比较复杂、难以实现在线二维结构构建,以及分子量处于800~1 500区间内的分子,本系统支持浏览器端.mol文件(分子结构文件)上传功能,用户仅需点击鼠标将准备好的.mol文件内容上传到服务器即可实现任务投递.目前,本系统能够支持原子组成为前四周期(H~Br)主族元素的有机化合物的1H和13C和NMR化学位移计算.
2 结果与讨论 2.1 典型案例1:在线计算不饱和桥环分子的 1H 和 13C NMR化学位移首先,打开浏览器,地址栏键入“http://10.8.1.160/iccas”,打开系统平台主界面.在左侧界面构建目标分子结构,完成之后点击左侧界面上方“C-H”按钮进行自动键级分析及所有氢原子显示.进一步,点击左侧界面上方的“2D to 3D”按钮,系统会将左侧构建的分子结构转化为三维结构并在右侧显示.用户可以利用鼠标左键拖动分子转换分子的观察角度,以检查分子结构构建的准确性.如果分子结构构建确认无误,则可点击界面右上角“Start Computing!”按钮,提交计算任务(图 2).
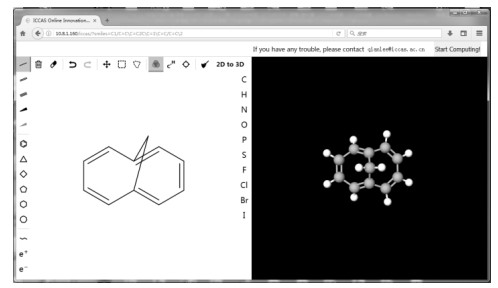
|
图 2 系统在线构建目标分子的用户图形交互界面 Fig. 2 The on-line graphical user interface of the platform for molecular building |
任务提交之后,系统自动跳转到结果显示界面.由于计算过程需要一段时间,因此如果计算尚未完成,则该页面显示“The computing work has not finished yet, please check it later.”(图 3).
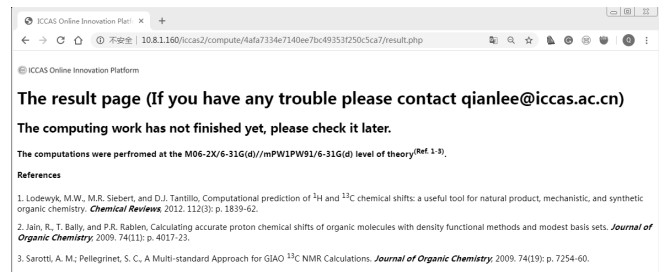
|
图 3 计算结果展示页面(计算未完成状态) Fig. 3 The result representation page (computation has not finished yet) |
此时,用户可以等待保留此页面,或者将网址拷贝下来之后,关闭界面.待计算完成之后直接键入拷贝的网址即可打开结果显示页面(本案例分子的计算大约需要3 min).计算完成之后,结果显示页面如图 4所示.
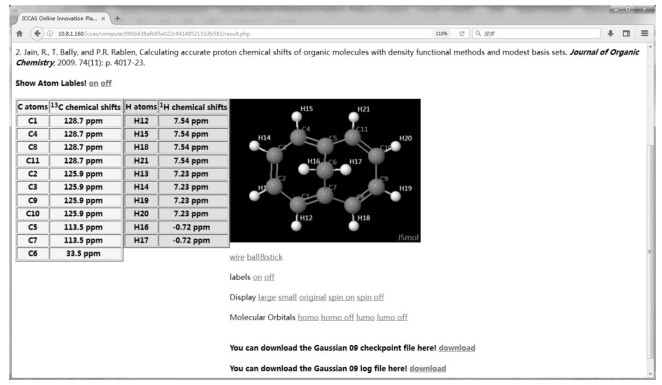
|
图 4 计算结果展示页面(计算已完成状态) Fig. 4 The result representation page (computation has finished) |
该页面左侧为分子内各原子的化学位移数值,右侧为三维显示的分子结构及原子编号.用户可以选择采用线状(wire)模型或者球棍(ball & stick)模型.
进一步,我们将本系统计算得到的两个不饱和桥环分子的 1H NMR化学位移的计算值与文献报道实验值,以及NMRdb[6]、NMRshiftDB[4]的预测值进行比较(图 5、表 1).结果显示:本系统能够较好的预测受环电流效应影响较为显著的 1H NMR化学位移,而且与NMRdb、NMRshiftDB相比,能够较为准确的处理结构较为相似的不饱和桥环分子的1H NMR化学位移.

|
图 5 利用本系统平台计算得到的两个不饱和桥环分子的 1H 和 13C NMR化学位移.(a) bicyclo[4.4.1]undeca-2, 4, 7, 9-tetraene;(b) (3Z, 5Z, 7E, 9Z, 11Z)-bicyclo[5.5.1]trideca-1, 3, 5, 7, 9, 11-hexaene,圆圈所示区域的1H NMR化学位移在结构非常类似的化合物a及b中,计算结果具有显著差异 Fig. 5 Results of the 1H and 13C NMR chemical shifts of two unsaturated bridging ring molecules predicted by the proposed system. (a) bicyclo[4.4.1]undeca-2, 4, 7, 9-tetraene; (b) (3Z, 5Z, 7E, 9Z, 11Z)-bicyclo[5.5.1]trideca-1, 3, 5, 7, 9, 11-hexaene. Hydrogens in the circles of the two similar structures show significant difference in their chemical shifts |
| 表 1 本系统计算得到的化学位移值与实验值,以及NMRdb和NMRshiftDB系统预测值的对比 Table 1 Comparison of chemical shifts predicted by NMRdb, NMRshiftDB, and the proposed system, with the experimental values |
天然产物结构一般较为复杂,且其中可能含有较多的手性原子,化学位移与空间结构密切相关,因此对这一类化合物的化学位移预测,基于加和的算法具有较大局限性.于是我们尝试利用本系统平台对于图 6(a)中的天然产物分子hexacyclinol[21, 22]进行1H和13C NMR化学位移计算,初步评估本系统所设定的默认计算方法的可靠性.我们分别对该分子的1H与13C NMR化学位移实验值与计算值进行了分析. 由于hexacyclinol [图 6(a)]的1H NMR实验谱图受到1H-1H耦合等因素的影响,化学位移重叠现象较为严重,很难得出准确的1H NMR化学位移.因此,我们着重对hexacyclinol的13C NMR化学位移实验值及预测值开展线性回归分析(表 2;图S1,可扫描文章首页OSID码或者在文章网络版查看).结果显示,利用本平台的方法组合及参数开展的hexacyclinol的13C NMR化学位移计算值与实验值呈线性相关,相关系数RC2为0.99.13C NMR化学位移计算值与实验值的平均绝对偏差为3.1 ppm、最大偏差为7.00 ppm,与文献[22]计算值较为接近.本案例从提交分子结构到获取计算结果耗时约120 min.

|
图 6 (a) 来源于植物Panus rudis的天然产物分子hexacyclinol和(b)化合物4, 4'-(4, 4-diphenyl-1, 3-butadienylidene)bis(N, N- diethylaniline)的分子结构 Fig. 6 The structure of (a) hexacyclinol, a natural product from Panus rudis, and (b) 4, 4'-(4, 4-diphenyl-1, 3-butadienylidene)bis (N, N-diethylaniline) |
| 表 2 本系统对天然产物以及分子量较大的有机分子化学位移的计算值与实验值的对比 Table 2 Comparison of chemical shifts predicted by the proposed system and those experimental value of natural product hexacyclinol and organic molecule 4, 4-(4, 4-diphenyl-1, 3-butadienylidene)bis(N, N-diethylaniline) |
由于目前基于密度泛函理论的化学位移预测大多针对较为简单的分子体系(通常分子量小于300),对于分子量较大的分子应用较少.因此,我们尝试利用我们的系统对分子量为500.72的分子4, 4'-(4, 4-diphenyl-1, 3- butadienylidene)bis(N, N-diethylaniline) [图 6(b)]进行1H和13C NMR化学位移计算.该分子由碳、氢、氮三种元素构成,其中有芳环、不饱和及饱和碳原子.利用本平台工具的方法组合及参数针对该分子进行1H和13C NMR化学位移计算,结果显示:计算值与实验值呈显著线性相关(表 2;图S2,可扫描文章首页OSID码或者在文章网络版查看).R2分别为0.99(RH2)和0.99(RC2).1H NMR化学位移计算值与实验值的平均绝对偏差为0.06 ppm,最大偏差为0.23 ppm;13C NMR化学位移计算值与实验值的平均绝对偏差为0.88 ppm,最大偏差为3.07 ppm.本化合物从提交分子结构到获取计算结果耗时约48 min.本系统平台计的算耗时与分子结构密切相关,通常情况下计算耗时随分子体系的增大而延长;柔性分子比刚性分子需要更多的计算时间.而本化合物计算耗时小于hexacyclinol,主要是由于分子刚性较大、结构收敛较快.
2.3 本系统平台特点近年来,随着计算化学理论取得重要进展,以及计算硬件设备性能的大幅提高,利用第一性原理针对有机分子开展 1H 和 13C NMR化学位移的精确计算技术也取得了很大进展,部分方法已经逐步能够用于较大分子及较复杂的分子体系的准确预测[5, 10, 11].但是利用密度泛函方法精确计算有机分子化学位移的技术仍未被相关NMR应用研究者广泛应用.究其原因主要在于密度泛函方法精确计算化学位移技术步骤较为复杂繁琐,对于研究有机化学、天然产物化学以及材料化学的研究人员而言,复杂体系的计算研究仍然较为不便.
例如,要开展相关计算工作,研究人员首先要配置一套高性能服务器或者超算环境,安装配置相应的计算软件.在此基础上,研究人员首先要构建二维分子结构,再将此二维结构存储为下一步软件可识别的输入文件格式,再利用具有三维构象采样以及分子力学优化功能的软件包(如openbabel 2.3.1)开展分子三维构象采样及优化,并输出分子坐标文件.进一步,需要将分子坐标文件制作为密度泛函计算软件所识别的坐标格式,并综合评估计算精度以及计算时间选取合适的泛函方法和基组,制作密度泛函计算所用的输入文件,并投递任务,利用相关软件(如Gaussian 09)开展基于密度泛函的分子几何结构优化以及屏蔽张量的计算.计算完成后,需要用户从结果文件中提取出分子内各原子的屏蔽张量值,然后选取合适的标度参数,根据标度参数和屏蔽张量值进行化学位移值的计算.至此计算得到了包含各个原子的化学位移的输出文件.但由于输出文件中的原子通常采用坐标和编号表示,比较抽象,用户从输出文本文件本身无法直观获知分子三维结构.因此还需将输出结果进行可视化、用户需要从输出文件中提取分子中各个原子的坐标并转化成为可视化软件可识别的格式,并用可视化软件打开,并操作软件以显示出分子三维结构以及各个原子的编号.至此用户才能够将计算得到的化学位移与分子中的原子一一对应.上述一系列过程较为复杂,对于非计算化学专业的人员非常不友好.小分子NMR实验的用户大多是实验科学家,专门购买一台高性能服务器用来计算化学位移也不太可行.同时,让NMR用户耗费大量精力和时间去涉猎高性能服务器软硬件配置、计算化学、编程等方向的内容,而仅仅用于开展化学位移计算也较难实现.用户可能更需要一种快速、简便、有效的途径来得到较高精度的化学位移计算值.
本文构建的在线计算系统平台工具实现了对一系列复杂计算流程功能的有效集成,用户界面友好,无需用户安装任何软件,仅仅利用电脑甚至手机、平板等的浏览器配合鼠标操作即可构建分子投递任务,平台系统自动在服务器端完成全计算流程中牵涉到的各软件的输入、调用、输出、链接等.服务器端计算完成后,系统在用户浏览器端实现化学位移与三维分子结构的可视化.上述全部过程无需用户具备量子化学及计算机编程基础,用户仅需利用鼠标点击操作即可,系统页面均是实时响应.密度泛函计算的参数选择方面,本系统平台采用了Tantillo等[13]报道验证过的参数集,整体计算精度相对可靠.利用此参数集开展计算分别应用于不饱和桥环、复杂天然产物以及合成分子体系(本文2.1节和2.2节案例)等,均得到相对可靠的计算结果.系统计算耗时与分子结构密切相关,通常情况下计算耗时随分子体系(特别是分子中电子数目)的增大以及随分子体系柔性的增大而延长.除此之外,在本系统平台提供的高级设置界面(图S3,可扫描文章首页OSID码,或者在文章网络版查看),用户可以选择不同的泛函、基函数和溶剂化模型等,可避免对于特殊体系进行过于自动化处理后的结果偏差.该系统可有效提高研究人员利用NMR技术开展相关研究的效率,为分子结构的高效、精确解析提供了一个辅助工具.
3 结论本实验室构建了一套基于“Browser-Server架构”和密度泛函理论的高精度NMR化学位移在线计算系统平台(本单位内部IP访问地址为“http://10.8.1.160/iccas”).目前,本系统平台能够支持原子组成为前四周期(H~Br)主族元素、分子量小于800的有机化合物的 1H 和 13C NMR化学位移计算.本系统平台默认的计算参数采用了Tantillo等[13]报道验证过的参数集,计算精度相对可靠.除此之外,本系统平台提供的高级设置界面可供用户自行选择不同的泛函,基函数和溶剂化模型等,从而避免不同的体系因具体情况不同而采用了过于自动化处理后的结果偏差.本系统平台界面友好,用户利用浏览器即可操作,无需安装任何软件,也无需用户具备量子化学及计算机编程基础,全部计算过程利用鼠标点击操作即可完成,且页面交互均是实时响应,最大程度简化了用户的操作,真正实现浏览器端的即点即用,可有效提高研究人员利用NMR技术开展相关研究的效率,为分子结构的高效、精确解析提供了一个辅助工具.目前本系统平台已部署于本单位核磁共振中心,面向本单位百余个课题组提供有机分子化学位移相关科学计算服务.由于本单位相关计算需求旺盛,且密度泛函理论计算的计算量较大,而本系统全部计算均在我们的服务器端完成,因此目前本系统平台仅为本单位内部IP用户提供访问.感兴趣的读者可以与我们联系开展相关合作.下一步,我们计划尝试利用更多的分子谱图数据信息作为训练集,进一步优化计算使用的参数系统,提高计算精度;另一方面,我们计划将杂核(如11B、15N、19F、29Si、31P)的化学位移计算加入到系统中来.随着硬件水平的发展与提升,本系统平台将来有望实现用于更大规模有机分子的化学位移精确计算.
致谢 感谢国家自然科学基金资助项目(21778058)、国家自然科学基金国际(地区)合作与交流基金资助项目(21911530180)以及中国科学院青年创新促进会基金的资助.
利益冲突 无
附件材料附录
图 S1 (a)来源于植物 Panus rudis 的天然产物分子 hexacyclinol 的分子结构;(b) 13C NMR 化学位移计算值与实验值的线性回归分析;(c) 13C NMR 化学位移计算值与实验值的偏差.
图 S2 化合物 4,4'-(4,4-diphenyl-1,3-butadienylidene)bis(N,N-diethylaniline)的分子结构、本系统计算的 NMR 化学位移、1H 及 13C NMR 化学位移计算值与实验值的线性回归分析及偏差.
图 S3 本系统平台提供的高级设置界面.
短视频:本系统平台操作演示.
| [1] | ABRAHAM R J, MOBLI M. Modelling 1H NMR spectra of organic compounds: theory, applications and NMR prediction software[M]. Hoboken, NJ: Wiley, 2008. |
| [2] | BEEBY J, STERNHEL S, HOFFMANN T, et al. Estimation of chemical-shifts of aromatic protons using additive increments[J]. Anal Chem, 1973, 45(8): 1571-1573. DOI: 10.1021/ac60330a048. |
| [3] | MATTER U E, PASCUAL C, PRETSCH E, et al. Estimation of chemical shifts of olefinic protons using additive increments.3. Examples of utility in nmr studies and identification of some structural features responsible for deviations from additivity[J]. Tetrahedron, 1969, 25(9): 2023-2024. DOI: 10.1016/S0040-4020(01)82823-4. |
| [4] | KUHN S, SCHLORER N E. Facilitating quality control for spectra assignments of small organic molecules: nmrshiftdb2-a free in-house NMR database with integrated LIMS for academic service laboratories[J]. Magn Reson Chem, 2015, 53(8): 582-589. DOI: 10.1002/mrc.4263. |
| [5] | CASABIANCA L B, DE DIOS A C. Ab initio calculations of NMR chemical shifts[J]. J Chem Phys, 2008, 128(5): 052201. DOI: 10.1063/1.2816784. |
| [6] | BANFI D, PATINY L. www.nmrdb.org: Resurrecting and processing NMR spectra on-line[J]. Chimia, 2008, 62(4): 280-281. |
| [7] | MESTRENOVA, MESTRELAB RESEARCH S L[CP]. Santiago de Compostela, Spain. |
| [8] | ChemOffice[CP]. CambridgeSoft Corp, Cambridge, MA, USA 02140-9802. |
| [9] | ACD[CP]. Advanced Chemistry Development, Toronto, Ontario, Candada. |
| [10] | BALLY T, RABLEN P R. Quantum-chemical simulation of 1H NMR spectra. 2. Comparison of DFT-based procedures for computing proton-proton coupling constants in organic molecules[J]. J Org Chem, 2011, 76(12): 4818-4830. DOI: 10.1021/jo200513q. |
| [11] | WILLOUGHBY P H, JANSMA M J, HOYE T R. A guide to small-molecule structure assignment through computation of (H-1 and C-13) NMR chemical shifts[J]. Nat Protoc, 2014, 9(3): 643-660. DOI: 10.1038/nprot.2014.042. |
| [12] | SEFZIK T H, TURCO D, IULIUCCI R J, et al. Modeling NMR chemical shift: A survey of density functional theory approaches for calculating tensor properties[J]. J Phys Chem A, 2005, 109(6): 1180-1187. DOI: 10.1021/jp0455780. |
| [13] | LODEWYK M W, SIEBERT M R, TANTILLO D J. Computational prediction of H-1 and C-13 chemical shifts: A useful tool for natural product, mechanistic, and synthetic organic chemistry[J]. Chem Rev, 2012, 112(3): 1839-1862. DOI: 10.1021/cr200106v. |
| [14] | HE X, WANG B, MERZ K M. Protein NMR chemical shift calculations based on the automated fragmentation QM/MM approach[J]. J Phys Chem B, 2009, 113(30): 10380-10388. DOI: 10.1021/jp901992p. |
| [15] | SWAILS J, ZHU T, HE X, et al. AFNMR: automated fragmentation quantum mechanical calculation of NMR chemical shifts for biomolecules[J]. J Biomol Nmr, 2015, 63(2): 125-139. DOI: 10.1007/s10858-015-9970-3. |
| [16] | HE X, ZHU T, WANG X W, et al. Fragment quantum mechanical calculaton of proteins and its applications[J]. Accounts Chem Res, 2014, 47(9): 2748-2757. DOI: 10.1021/ar500077t. |
| [17] | ZHU T, ZHANG J Z H, HE X. Automated fragmentation QM/MM calculation of amide proton chemical shifts in proteins with explicit solvent model[J]. J Chem Theory Comput, 2013, 9(4): 2104-2114. DOI: 10.1021/ct300999w. |
| [18] | ZHU T, HE X, ZHANG J Z H. Fragment density functional theory calculation of NMR chemical shifts for proteins with implicit solvation[J]. Phys Chem Chem Phys, 2012, 14(21): 7837-7845. DOI: 10.1039/C2CP23746F. |
| [19] | HALGREN T A, MMFF VII. Characterization of MMFF94, MMFF94s, and other widely available force fields for conformational energies and for intermolecular-interaction energies and geometries[J]. J Comput Chem, 1999, 20(7): 730-748. DOI: 10.1002/(SICI)1096-987X(199905)20:7<730::AID-JCC8>3.0.CO;2-T. |
| [20] | HALGREN T A, MMFF VI. MMFF94s option for energy minimization studies[J]. J Comput Chem, 1999, 20(7): 720-729. DOI: 10.1002/(SICI)1096-987X(199905)20:7<720::AID-JCC7>3.0.CO;2-X. |
| [21] | PORCO J A, SU S, LEI X G, et al. Total synthesis and structure assignment of (+)-hexacyclinol[J]. Angew Chem Int Edit, 2006, 45(35): 5790-5792. DOI: 10.1002/anie.200602854. |
| [22] | RYCHNOVSKY S D. Predicting NMR spectra by computational methods: Structure revision of hexacyclinol[J]. Org Lett, 2006, 8(13): 2895-2898. DOI: 10.1021/ol0611346. |
| [23] | Database for Organic Compounds[OL]. SDBS, https://sdbs.db.aist.go.jp. |
 2021, Vol. 38
2021, Vol. 38 
