阿尔兹海默疾病具有发病机制复杂、发病时间长、治愈难度大的特点,是一种严重影响患者正常生活、不可逆的神经退行性疾病[1, 2],且随着时间的推移会越来越严重.我们可以将其分为轻度认知障碍(MCI)和阿尔兹海默症(AD)两类[3],MCI是AD的前驱阶段,具有极大的不稳定性,如果对MCI进行正向干预,可以有效的延缓或减轻MCI[4].近期,国家药监管理局有条件批准了我国自主研发的创新药——甘露特钠胶囊(九期一)的上市注册,但它也只能起到改善轻度和中度阿尔兹海默疾病患者认知功能的作用.据统计,截至2019年,全球共有阿尔兹海默疾病患者超过5 200万,大约每3 s世界上就会产生1个阿尔兹海默疾病患者[5];中国有1 000多万阿尔兹海默疾病患者,这个数字是2010年(569万)的2倍,是1990年(193万)的7倍,而且我国已成为世界上阿尔兹海默疾病患者最多的国家[5].据预测,到2050年,全球阿尔兹海默疾病患者将会达到1.52亿,而我国阿尔兹海默疾病患者会达到2 800万.因此,阿尔兹海默疾病的早期诊断意义重大[6].
基于磁共振图像和计算机辅助的阿尔兹海默疾病的诊断方法可以分为两类:一类方法首先从不同的磁共振图像中通过一系列复杂的步骤提取量化特征(如局部二进制特征[7]、直方图特征[8]等),然后通过传统机器学习算法(如逻辑回归、支持向量机等)实现分类,它采用了人工定义特征,具有主观性,不能全面地提取更加抽象的特征.另一类方法通过人工智能端到端的深度学习方法,从大量训练数据中自动学习到更全面、更丰富的特征来处理复杂的医学诊断问题,从而避免人工选择特征的弊端[9].比如Korolev等[10]采用残差和普通卷积神经网络(residual and plain CNN)、Khvostikov等[11]采用不同模态的3D卷积神经网络(CNN)用于AD患者、MCI患者和正常老化(CN)人群的分类.
基于磁共振图像和计算机辅助的AD、MCI和CN[12]的精准诊断不仅需要受疾病影响较大的海马区域的相对局部信息[13],也需要结合灰质或白质等相对全局信息[14].有研究[15]表明白质与灰质之间存在功能连接,而且也存在与AD相关的病变区域.通过融合大脑磁共振图像多个感兴趣区域(ROIs)的特征,可以拓宽输入数据的信息范围,实现多类型多层次特征的整合.3D结构磁共振图像与2D结构磁共振图像相比,具有更加丰富的空间结构信息[10].此外,随着卷积层的加深,提取的特征虽然更抽象更具体,但它也会丢弃部分但重要的特征信息[16].通过增加中间层之间的输出和连接,可以减少特征信息的丢失,从而更好更充分的学习图像特征.
本文通过综合考虑大脑3D结构磁共振图像中海马、灰质(无海马)和白质三个不同ROIs之间的相互联系与影响,实现了相对局部区域的信息和相对全局信息的相结合.并提出了多输出3D CNN方法,可以学习到更加丰富的空间结构特征信息,减少了由神经网络的加深导致的部分原始图像重要特征信息丢失的问题,从而增强图像特征信息的传播、提高阿尔兹海默疾病诊断的准确率和鲁棒性.
1 实验部分 1.1 实验数据本文使用的磁共振图像是颅骨剥离的3D全脑磁共振图像以及相对应图像的双侧海马体的金标准mask图像,共有132例,包括25例AD、61例MCI以及46例CN:AD患者中包含12名女性和13名男性,年龄为(75.24±6.35)岁;61例MCI患者中包含13名女性和48名男性,年龄为(75.24±7.13)岁;46例CN人群中包含20名女性和26名男性,年龄为(76.52±5.79)岁.将这132例3D磁共振图像分为训练集、验证集和测试集:随机抽取4例AD患者、16例MCI患者和9例CN作为测试集;其余的21例AD患者、45例MCI患者和37例CN用作训练集和验证集.磁共振图像数据来源于阿尔兹海默病神经成像计划数据库(ADNI,ADNI主要目标是测试是否可以将正电子发射断层扫描图像和磁共振图像,与其他生物标记物评估和临床神经生理学结合起来,以监测MCI和早期阿尔兹海默疾病的进展[11]);双侧海马体的金标准mask图像数据来源于第九届中国大学生服务外包创新创业大赛的华为命题.
1.2 数据预处理首先使用SPM12工具箱[17]对3D全脑磁共振图像进行分割,得到大脑的白质与灰质[18];然后将海马组织的金标准与全脑磁共振图像相应体素相乘得到3D双侧海马组织;将海马组织金标准的体素值翻转,再与SPM12分割出的灰质对应体素相乘,得到3D无海马组织的灰质.由于存在内存资源有限、计算量和ROI尺寸不同等问题,直接输入整张3D磁共振图像是不可取的(尤其是与灰质和白质相比,海马体的体积太小).因此,我们首先找到并去除3D磁共振图像边缘,保留ROIs.然后调整ROIs至合适的尺寸:灰质和白质的体积调整为112×112×112,海马的体积调整为56×56×56.ROIs的2D切片如图 1所示.
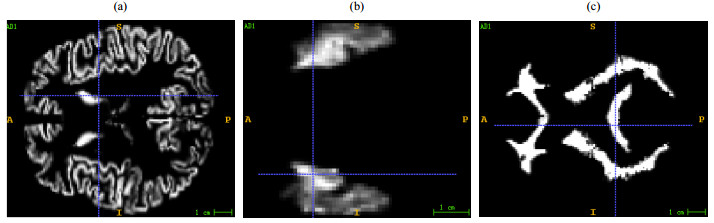
|
图 1 ROIs的2D切片.(a)灰质;(b)海马;(c)白质.A(前),P(后),S(上),I(下) Fig. 1 2D slices of ROIs.(a) Grey matter; (b) Hippocampus; (c) White matter. A (anterior), P (posterior), S (superior), I (interior) |
然而深度学习训练神经网络需要大量数据,当数据量较少时,神经网络会出现过拟合、模型泛化能力差等问题[15].此外我们使用的数据集跟其他很多医用数据集一样,有数据不平衡的问题,MCI患者数量大约是AD患者的2.5倍,而数据集的不平衡会影响模型的鲁棒性和泛化能力[19].因此我们对数据进行增强以实现数据扩充和数据平衡.对于数据扩充,我们通过对训练集与测试集数据进行补丁、旋转、翻转等操作以实现数据集的扩增;对于数据平衡,假设数据量最多的组别个数为m,将其扩增为原来的n倍,则其他类别(假设有mi个数据)需通过数据扩充的方式增加(m×n-mi)个新数据.经过数据扩充与平衡,本文得到训练集和验证集数据共1 648个,测试集数据共348个.
1.3 实验方法本文一共使用了3种方法实现阿尔兹海默疾病诊断与分类.方法一(基础3D CNN)[20]是单输入单输出的3D CNN:通过输入单个ROI,实现阿尔兹海默疾病的诊断与分类,但它诊断效果最差.方法二(三输入3D CNN)是多输入单输出的3D CNN:通过输入海马、灰质(无海马)和白质多个感兴趣区域,拓宽了输入数据的信息范围、考虑了不同ROIs之间的相互影响,诊断效果相比基础3D CNN有了较大的提升.方法三(多输出3D CNN)是多输入多输出的3D CNN:通过输入海马、灰质(无海马)和白质多个ROIs、增加中间层的输出与连接,实现多类型多层次特征的相互整合,增强了特征的传播,减少了特征信息的丢失,从而提高分类的精度,获得很好的诊断效果.
在基础3D CNN方法中,我们首先用最基本的3D CNN[20]实现AD、MCI和CN的分类(输入分别为灰质、海马、白质),它是由4个自动提取特征的卷积块(图 2)和2个全连接块组成.每个卷积块包含2个3D卷积层和1个最大池化层.卷积层的基础滤波器是16个,随着卷积块的增加,滤波器的个数依次增加1倍;每一卷积层都使用批的标准化[21],激活函数为LeakyReLu,每个卷积层的卷积核大小为3×3×3,填充方式为same,最大池化层的大小为2×2×2.4个卷积块之后的第一个全连接块由1个全连接层和1个Dropout层(丢弃的占全部神经元的比率为0.3)组成,最后一个全连接块由3个神经元和用于最终分类预测的Softmax激活函数组成(图 3).这种方法基于单一ROI,没有考虑不同ROI之间的联系与相互影响.
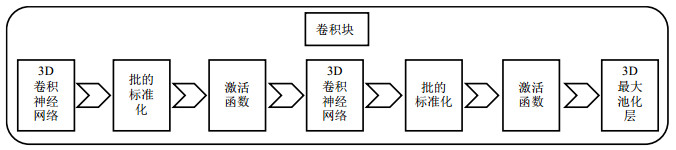
|
图 2 网络框架的卷积块结构 Fig. 2 Convolution block structure of network framework |

|
图 3 基础3D卷积神经网络结构 Fig. 3 Basic 3D CNN structure |
针对上述问题,我们使用了三输入3D CNN方法.它有3个不同的输入(输入为灰质、海马、白质)和1个输出,但在灰质和白质输入卷积块之前有个大小为2×2×2的最大池化层,以确保每个分支输入卷积层的尺寸大小一致,最后各路的特征图在卷积块之后连接到全连接层,其他结构与基础3D CNN框架一致(图 4).但三输入3D CNN方法会进入局部最优;且随着神经网络的加深,会丢失原始图像部分但重要的特征信息,从而降低诊断的准确率[22].

|
图 4 三输入3D卷积神经网络结构 Fig. 4 Three-input 3D CNN structure |
在上述三输入3D CNN方法的基础上,我们提出了多输出3D CNN方法实现AD、MCI和CN的三分类,即网络在每个卷积块之后都会有一个输出.通过融合大脑3D磁共振图像多个ROIs(灰质、海马、白质)的特征,可以拓宽输入数据的信息范围,实现多类型多层次特征的整合,对疾病进行分类预测;通过增加中间层之间的输出和连接,可以缩短输入和输出之间的距离,实现早期反馈、减少原始图像特征信息的丢失,从而优化模型,实现全局最优(图 5).

|
图 5 多输出3D卷积神经网络结构 Fig. 5 Multi-output 3D CNN structure |
实验是在GTX 1080、Ubuntu 14.04和CUDA 8.0的工作站环境中,以TensorFlow为后端的Keras实现.我们设置最大迭代次数(Epoch)为280,并使用了早期停止;使用SGD(stochastic gradient descent)作为优化函数,损失函数为交叉熵损失函数,初始学习率为1×10-5,批的大小(batch size)为4.
2.1 准确率比较FP、FN、TP、TN分别代表使用模型对AD、MCI或CN进行分类预测得到的假阳性、假阴性、真阳性和真阴性数量的个数,准确率的计算如(1)式所示:
| $ 准确率 = \left( {TP + TN} \right)/\left( {TP + TN + FP + FN} \right) $ | (1) |
利用基础3D CNN方法在测试集上基于灰质、白质和海马体组织得到的准确率分别为81.6%、79.9%、84.5%;利用三输入3D CNN方法在测试集上得到的准确率为85.1%;利用多输出3D CNN方法在测试集上得到的准确率为90.5%.由于基础3D CNN方法是基础实验,且准确率较低,所以我们不给予过多的分析.使用三输入3D CNN和多输出3D CNN方法得到的混淆矩阵如图 6所示.由混淆矩阵我们可以看出不确定性最高的是MCI,它可能被分类为CN,也可能被分类为AD,这是因为MCI介于CN和AD之间,且MCI与AD之间没有明确的界限,而且MCI具有极大的不稳定性;而CN和AD之间的界限相对明确,所以分类的准确度较高.三输入3D CNN通过拓宽输入数据的信息范围,实现多层次、多类型的信息特征相融合;局部和全局ROI信息特征的相结合;还综合考虑不同ROI之间存在的相互联系与影响.但是随着卷积层的加深,提取的特征虽然更抽象,原始图像部分重要的信息特征也会丢失.通过利用多输出3D CNN,增加中间层的连接和输出,缩短输入到输出的距离,可以有效的解决三输入单输出3D CNN的弊端,提高阿尔兹海默疾病的诊断精度.

|
图 6 使用(a)三输入3D CNN方法和(b)多输出3D CNN方法得到的混淆矩阵 Fig. 6 Confusion matrixes using (a) three-input 3D CNN method and (b) multi-output 3D CNN method |
图 7是多输出3D CNN模型的准确率和损失曲线.图 7(a)是多输出3D CNN训练集和验证集的准确率曲线图,通过曲线图我们可知训练集训练的很好,达到很高的准确率,而验证集的准确率大约只有90%,低于训练集.图 7(b)是多输出3D CNN模型训练集和验证集的损失曲线图,通过曲线图可得验证集的损失大约为0.2,而训练集的损失很小,这和图 7(a)训练集准确率很高,而验证集准确率没有训练集高相对应.

|
图 7 基于多输出3D CNN模型的(a)准确率和(b)损失曲线 Fig. 7 (a) Accuracy rate and (b) loss curves of multi-output 3D CNN model |
我们进一步使用精确率、灵敏度、特异性和F1分数(F1-score)估计神经网络模型的分类效果(表 1).其中,F1-score是机器学习中多分类问题最终测评的方法.它是精确率和召回率的调和平均数,最大为1,最小为0.精确率、灵敏度、特异性和F1-score的计算公式如(2)~(5)式所示:
| $ 精确率 = TP/\left( {TP + FP} \right) $ | (2) |
| $ 灵敏度 = 召回率 = TP/\left( {TP + FN} \right) $ | (3) |
| $ 特异性 = TN/\left( {TN + FP} \right) $ | (4) |
| $ {\rm{F}}1 - {\rm{score = }}\left( {2 \times 精确率 \times 召回率} \right)/\left( {精确率 + 召回率} \right) $ | (5) |
| 表 1 使用不同方法进行阿尔兹海默症(AD)、轻度认知障碍(MCI)和正常老化(CN)分类诊断的性能比较 Table 1 Performance comparison of classification and diagnosis for AD, MCI and CN with different methods |
Korolev等[10]的方法是基于海马体组织对本研究整个测试集分类准确率最高的模型,由表 1可知Korolev等的方法对于MCI诊断效果最差,对于CN诊断效果最好,但均低于Khvostikov等[11]的方法、三输入3D CNN方法和多输出3D CNN方法.Khvostikov等的方法对于CN、MCI和AD的诊断虽然在F1-score指标及少数几个指标上(MCI的精确率和特异性、CN的灵敏度)优于三输入3D CNN方法.但三输入3D CNN方法在其它精确率、灵敏度和特异性方面优于Khvostikov等的方法.而相比三输入3D CNN方法,多输出3D CNN方法在各方面均有一定提升.对于整个测试集三分类的平均结果,多输出3D CNN方法的精确率达到91.0%、灵敏度达到90.4%、特异性达到95.2%、F1-score达到90.5%.由此可见,使用本文提出的多输出3D CNN诊断阿尔兹海默疾病时,不仅可以综合利用多个ROIs,实现多层次多类型特征的整合,还可以减少原始图像信息的丢失,实现特征利用率的最大化.
2.3 讨论上述实验结果证明本文提出的多输出3D CNN方法可以有效的对AD、MCI和CN进行分类诊断,准确性较高、鲁棒性较好.基于深度学习的阿尔兹海默疾病诊断大多数集中在海马、灰质和白质等单一ROI,很少将海马、灰质(无海马)以及白质联合起来,而单一的信息会忽略ROIs之间可能存在的联系与相互间的影响.因此,我们通过融合大脑磁共振图像的海马、灰质和白质多个ROIs的信息特征,实现AD的精准诊断.另外,我们使用了3D磁共振图像,而不是2D磁共振图像.因为和2D磁共振图像相比,3D磁共振图像可以提供更丰富的空间结构信息,3D CNN可以从3D磁共振图像中提取出表达性更强和更有临床意义的特征,从而有利于实现AD疾病的精准诊断.
通过人工智能端到端的深度学习方法,虽然可以从大量训练数据中自动学习到很好的特征,避免传统机器学习方法人工选择特征的弊端.但CNN从原始图像提取的重要特征信息会部分丢失,且随着卷积层的加深,重要特征信息丢失也会越严重.这也是我们提出本方法的目的所在,我们通过增加中间层之间的输出和连接,缩短输入和输出之间的距离,从而减少特征信息的丢失、更好更充分的学习图像特征.但我们实验过程中也存在图像原样本量较少和不平衡的问题.原样本量较少,是由于海马体组织金标准图像难以获得;原样本量越多,模型学习到的特征就越丰富,模型的泛化能力越好.原样本量不平衡在一定程度上会影响模型的训练效果.
3 结论本文使用多输出3D CNN方法实现了AD、MCI和CN的三分类.通过综合利用大脑3D磁共振图像中海马体、灰质(无海马体)和白质3个ROIs的信息可以拓宽输入数据的信息范围,实现多层次多类型信息的互补.通过增加中间层的连接和输出,可以缩短输入到输出的距离,增强特征传播,改善信息交流,减少原始图像随卷积层的加深而造成重要特征信息丢失的问题,从而提高AD疾病的诊断效果.之后,我们将考虑不同模态与其他ROIs之间的融合.此外,该模型理论上也可以应用到其他疾病的诊断中.
利益冲突 无
| [1] |
LUO T, DUAN C. The status and research significance of Alzheimer's disease at home and abroad[J].
Clinical Medical Practice, 2013, 22(11): 839-840.
罗涛, 段晨. 阿尔茨海默病的国内外现状及研究意义[J]. 临床医药实践, 2013, 22(11): 839-840. |
| [2] | MCKHANN G, DRACHMAN D, FOLSTEIN M, et al. Clinical diagnosis of Alzheimer's disease: Report of the NINCDS-ADRDA Work Group*under the auspices of department of health and human services task force on Alzheimer's disease[J]. Neurology, 1984, 34(7): 939-939. DOI: 10.1212/WNL.34.7.939. |
| [3] | CUI R X, LIU M H. Hippocampus analysis by combination of 3D DenseNet and shapes for Alzheimer's disease diagnosis[J]. IEEE J Biomed Health Inform, 2018, 23(5): 2099-2107. |
| [4] | JAIN R, JAIN N, AGGARWAL A, et al. Convolutional neural network based Alzheimer's disease classification from magnetic resonance brain images[J]. Cogn Syst Res, 2019, 57: 147-159. DOI: 10.1016/j.cogsys.2018.12.015. |
| [5] | ALZHEIMER'S ASSOCIATION. 2018 Alzheimer's disease facts and figures[J]. Alzheimers Dement, 2018, 14(3): 367-429. DOI: 10.1016/j.jalz.2018.02.001. |
| [6] | BRON E E, SMITS M, VAN DER FLIER W M, et al. Standardized evaluation of algorithms for computer-aided diagnosis of dementia based on structural MRI: the CADDementia challenge[J]. Neuroimage, 2015, 111: 562-579. DOI: 10.1016/j.neuroimage.2015.01.048. |
| [7] | SORENSEN L, SHAKER S B, DE BRUIJNE M. Quantitative analysis of pulmonary emphysema using local binary patterns[J]. IEEE Trans Med Imaging, 2010, 29(2): 559-569. DOI: 10.1109/TMI.2009.2038575. |
| [8] | UCHIYAMA Y, KATSURAGAWA S, ABE H, et al. Quantitative computerized analysis of diffuse lung disease in high-resolution computed tomography[J]. Med Phys, 2003, 30(9): 2440-2454. DOI: 10.1118/1.1597431. |
| [9] | PLIS S M, HJELM D R, SALAKHUTDINOV R, et al. Deep learning for neuroimaging: a validation study[J]. Front Neurosci-Switz, 2014, 8: 229. |
| [10] | KOROLEV S, SAFIULLIN A, BELYAEV M, et al. Residual and plain convolutional neural networks for 3d brain mri classification[C]//2017 IEEE 14 th International Symposium on Biomedical Imaging (ISBI 2017). IEEE, 2017: 835-838. |
| [11] | KHVOSTIKOV A, ADERGHAL K, BENOS-PINEAU J, et al. 3D CNN-based classification using sMRI and MD-DTI images for Alzheimer disease studies[J]. 2018. arXiv: 1801.05968. |
| [12] |
SUN J W, YAN S J, HAN Y S, et al. Classifying the course of Alzheimer's disease with brain MR images and a method based on three-dimensional local pattern transformation[J].
Chinese J Magn Reson, 2019, 36(3): 268-277.
孙京文, 闫士举, 韩勇森, 等. 基于脑部磁共振图像三维局部模式变换特征提取进行阿尔茨海默病病程预测分类[J]. 波谱学杂志, 2019, 36(3): 268-277. |
| [13] | PLANCHE V, RUET A, COUPÉ P, et al. Hippocampal microstructural damage correlates with memory impairment in clinically isolated syndrome suggestive of multiple sclerosis[J]. Mult Scler J, 2017, 23(9): 1214-1224. DOI: 10.1177/1352458516675750. |
| [14] | ZHAO J, DING X, DU Y, et al. Functional connectivity between white matter and gray matter based on fMRI for Alzheimer's disease classification[J]. Brain Behav, 2019, 9(10): e01407. |
| [15] | FEIS R A, BOUTS M J R J, DOPPER E G P, et al. Multimodal MRI of grey matter, white matter, and functional connectivity in cognitively healthy mutation carriers at risk for frontotemporal dementia and Alzheimer's disease[J]. BMC Neurol, 2019, 19(1): 1-11. DOI: 10.1186/s12883-018-1232-z. |
| [16] | BÄCKSTRÖM K, NAZARI M, GU I Y H, et al. An efficient 3D deep convolutional network for Alzheimer's disease diagnosis using MR images[C]//2018 IEEE 15th International Symposium on Biomedical Imaging (ISBI 2018). IEEE, 2018: 149-153. |
| [17] |
ZHANG H M, CHEN S Z. A new brain function imaging analysis method-statistical parameter graph (SPM)[J].
Chinese Medical Imaging Technology, 2002, 18(7): 711-713.
张海敏, 陈盛祖. 一种新的脑功能显像分析法-统计参数图(SPM)[J]. 中国医学影像技术, 2002, 18(7): 711-713. |
| [18] |
ZHAO S Y, WANG Y J. Classification of Alzheimer's disease patient and healthy group based on magnetic resonance images and improved UNet++model[J].
Chinese J Magn Reson, 2020, 37(3): 321-331.
赵尚义, 王远军. 基于磁共振图像和改进的UNet++模型区分阿尔茨海默症患者和健康人群[J]. 波谱学杂志, 2020, 37(3): 321-331. |
| [19] | ORTIZ-SUÁREZ J M, RAMOS-POLLÁN R, ROMERO E. Exploring Alzheimer's anatomical patterns through convolutional networks[C]//12th International Symposium on Medical Information Processing and Analysis. International Society for Optics and Photonics, 2017, 10160: 101600Z. |
| [20] | TRAN D, BOURDEV L, FERGUS R, et al. Learning spatiotemporal features with 3d convolutional networks[C]//Proceedings of the IEEE international conference on computer vision. 2015: 4489-4497. |
| [21] | NORMALIZATION B. Accelerating deep network training by reducing internal covariate shift[J]. 2015. Arxiv: 1502.03167. |
| [22] | RIEKE J, EITEL F, WEYGANDT M, et al. Visualizing convolutional networks for MRI-based diagnosis of Alzheimer's disease[M]//Understanding and Interpreting Machine Learning in Medical Image Computing Applications. Springer, Cham, 2018: 24-31. |
 2021, Vol. 38
2021, Vol. 38 
