2. 昆明医科大学 药学院, 云南省天然药物药理重点实验室, 云南 昆明 620500
2. School of Pharmaceutical Science & Yunnan Key Laboratory of Pharmacology for Natural Products, Kunming Medical University, Kunming 620500, China
细胞松弛素类化合物(cytochalasin)是一类种类繁多、结构多样、活性广泛的真菌代谢产物.此类化合物最早由Carter等人[1]于1967年从真菌Helminthosporium dematioidium中分得,并因它们能够结合肌动蛋白改变其聚合反应产生肌松效果而得名.文献报道细胞松弛素主要来自毛壳属(Chaetomium)、曲霉属(Aspergillus)、拟茎点霉属(Phomopsis)真菌,常根据来源命名为aspochalasins、chaetoglobosins、phomopsichalasins等[2].细胞松弛素具有广泛的生理活性,除抑制细胞运动和分裂外,还有抗肿瘤、抗菌、抗病毒、影响心血管和神经系统功能等作用[3],是微生物次生代谢产物研究的热点领域之一[4].
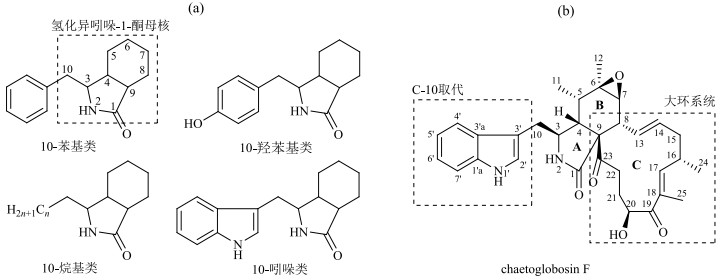
天然的细胞松弛素均以高度氢化的异吲哚-1-酮为母核,并骈连有碳环或内酯环等大环,其结构变化主要在:①异吲哚酮母核的氧化水平和取代基个数及种类不同;②耦合大环的种类及氧化水平、取代基个数和种类不同;③母核的C-10取代基不同.根据C-10取代基的不同,细胞松弛素分为四大类:10-苯基类(cytochalasins)、10-吲哚类(chaetoglobosins)、10-羟苯基类(pyrichalasins)和10-烷基类(aspochalasins、alachalasins)(图 1)[5].目前细胞松弛素主要通过波谱学方法鉴定,尤其是核磁共振(NMR)波谱技术占据了最主要的地位.但细胞松弛素结构复杂多样、鉴定困难,目前缺乏其NMR特征的总结.Chaetoglobosin F(化合物1)是分离自五加科药用植物——三七(Panax notoginseng)种子内生真菌Chaetomium globosum中的10-吲哚类细胞松弛素,结构具有代表性[6].本文以该化合物为例,综合运用1D和2D NMR技术(包括1H NMR、13C NMR、DEPT135、1H-1H COSY、HSQC、HMBC和NOESY)对其进行结构解析,并总结归纳10-吲哚类细胞松弛素的NMR特征,为该类化合物的结构鉴定和深入研究提供参考.
|
图 1 (a) 细胞松弛素的结构分类及(b) chaetoglobosin F(化合物1)的化学结构 Fig. 1 Types of cytochalasins (a) and chemical structure of chaetoglobosin F (compound 1) (b) |
NMR实验均在Bruker AM-400型超导NMR谱仪(Bruker)上完成,使用PABBO-5 mm多核宽带探头和直径为5 mm的NMR样品管(ST-500-7,NORELL);溶剂CDCl3(99.8%氘代,青岛腾龙微波科技有限公司)含0.03%四甲基硅烷(TMS).质谱于Agilent G3250AA TOF质谱仪(Agilent,Santa Clara)上测定.柱层析硅胶和薄层层析(TLC)硅胶板购于青岛海洋化工厂;乙酸乙酯、氯仿、甲醇购于天津化学试剂有限公司.TLC显色方式为紫外灯(上海宝山顾村电光ZF-20C暗箱式紫外分析仪)或茴香醛硫酸乙醇显色剂.
1.2 样品分离内生真菌PH30461分离自云南省文山壮族苗族自治州产三七种子,经内转录间隔区(ITS)测序鉴定为球毛壳菌Chaetomium globosum,菌株保存于云南大学.菌株采用PDB培养基于28 ℃摇瓶发酵7天.以乙酸乙酯萃取发酵液得总提取物46 g,采用硅胶柱色谱分离,氯仿/甲醇(v/v,1/0→0/1)溶液梯度洗脱,TLC检测合并得4个部分(Fr.A~Fr.D).Fr.B经Sephadex LH-20凝胶柱色谱(甲醇)分离,TLC检测合并得3个部分(Fr.B1~Fr.B3).Fr.B2放置析出固体,过滤后由硅胶柱色谱分离,经乙酸乙酯洗脱得化合物1(40 mg).
1.3 实验条件化合物1溶于CDCl3,NMR测试温度为(293.0±0.1)K.1H和13C NMR工作频率分别为400.13 MHz和100.61 MHz,谱宽分别为8 012.8 Hz和24 038.5 Hz.DEPT135采用仪器自带标准脉冲序列deptsp135,谱宽为16 129.0 Hz.二维谱(包括1H-1H COSY、NOESY、HSQC和HMBC谱)均采用标准脉冲程序采集.1H-1H COSY和NOESY的F2维(1H)和F1维(1H)的谱宽均为8 012.8 Hz,采样数据点阵t2 × t1 = 1 024 × 128,NOESY混合时间为0.5 s;HSQC和HMBC的F2(1H)和F1(13C)维谱宽分别为8 012.8 Hz和24 038.5 Hz,采样数据点阵t2 × t1 = 1 024 × 128.
2 结果与讨论 2.1 Chaetoglobosin F(化合物1)的NMR结构解析化合物1为白色粉末,254 nm下有紫外吸收,高分辨质谱显示分子式为C32H38N2O5(HR-ESI-MS: m/z 531.285 5 [M+H]+, 553.268 4 [M+Na]+),计算不饱和度为15.
化合物1的1H NMR谱低场区显示有1组典型的C-3′单取代吲哚环信号:δH 8.62(brs, 1H)、δH 6.99(d, J = 2.0 Hz, 1H)、δH 7.38(d, J = 8.0 Hz, 1H)、δH 7.15(t, J = 8.0 Hz, 1H)、δH 7.21(t, J = 8.0 Hz, 1H)、δH 7.49(d, J = 8.0 Hz, 1H)[7, 8].此外低场区还有2组双键质子信号,包括1个三取代双键δH 6.98(d, J = 8.4 Hz, 1H);1个二取代反式双键δH 6.35(ddd, J = 15.0/10.0/1.4 Hz, 1H)和δH 5.24(ddd, J = 15.0/10.0/2.8 Hz, 1H).1H NMR谱高场区显示4个甲基信号,其中2个与次甲基相连:δH 1.13(d, J = 7.2 Hz, 3H)、δH 1.02(d, J = 6.7 Hz, 3H);2个与季碳相连:δH 1.22(s, 3H)、δH 1.82(s, 3H).此外由HSQC可知δH 8.62(brs, 1H)和δH 6.30(brs, 1H)为活泼氢信号.
化合物1的13C NMR和DEPT135谱显示32个13C NMR信号.除吲哚环的8个碳原子外,剩余24个碳原子中包含6个季碳:其中2个酮羰基δC 203.7 (s)、δC 208.4 (s),1个酰胺羰基δC 175.0 (s)和1个双键季碳δC 134.4 (s);4个亚甲基;10个次甲基:其中2个双键碳δC 128.9 (d)、δC 133.6 (d),2个含氧取代次甲基δC 61.9 (d)、δC 72.0 (d);4个甲基:δC 12.3 (q)、δC 20.0 (q)、δC 13.0 (q)、δC 19.7 (q).
根据以上数据,结合来源菌株及文献[9]报道相关化合物NMR数据,可初步推测化合物1为10-吲哚类细胞松弛素.
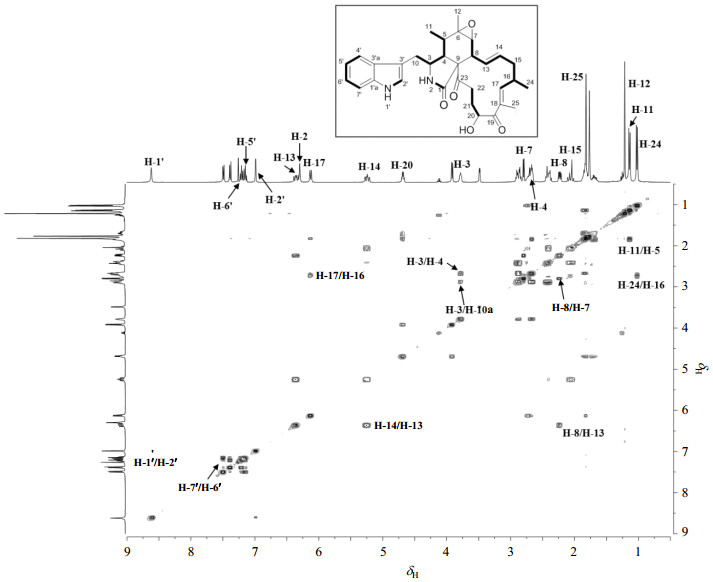
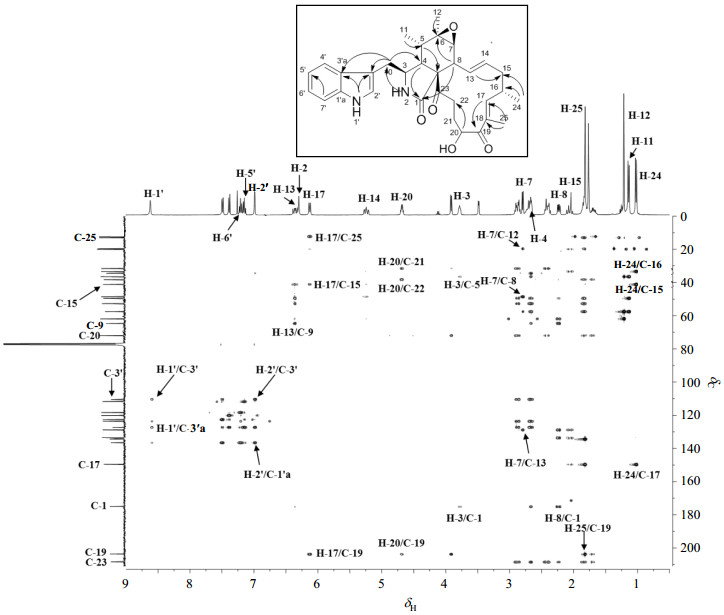
3-取代吲哚环(indol ring)除通过NMR信号与文献[7]比对外,还可根据1H-1H COSY谱(图 2)显示的H-4′/H-5′/H-6′/H-7′和H-1′/ H-2′自旋耦合系统,以及HMBC谱(图 3)显示的H-7′[δH 7.49 (d, J = 8.0 Hz, 1H)]和C-5′[δC 120.1 (d)],H-6′[δH 7.21 (t, J = 8.0 Hz, 1H)]和C-4′[δC 111.8 (d)],H-1′[δH 8.62 (brs, 1H)]和C-3′[δC 110.4 (s)]、C-3′a[δC 127.3 (s)]等相关验证.此外,HMBC谱中显示的H-10[δH 2.85 (m, 1H)和δH 2.66 (m, 1H)]与C-3′、C-3′a相关证实吲哚环通过亚甲基C-10与细胞松弛素的母核连接.
|
图 2 化合物1的1H-1H COSY 谱和主要的1H-1H COSY相关(  |
|
图 3 化合物1的HMBC谱和主要的HMBC相关(↷) Fig. 3 The HMBC spectrum and key HMBC correlations(↷) of compound 1 |
细胞松弛素母核的2-吡咯烷酮(2-pyrolidone ring,A环)变化小,其中酰胺[δH 6.30 (brs, 1H)]和δC 175.0 (s)]和羰基邻位季碳C-9[δC 64.7 (s)]可作为细胞松弛素鉴别的特征NMR信号.母核结构变化主要在于六元环(B环)氧化水平差异.1H-1H COSY谱中显示的H-10/H-3/H-4/H-5/H-11自旋耦合系统,结合HMBC谱中H-11[δH 1.13 (d, 7.2 Hz, 3H)]和C-5[δC 36.5 (d)]的相关,可确定CH3-11与C-5相连.化合物1有5个氧原子,存在于3个羰基和3个含氧取代碳[δC 57.5 (s)、δC 61.9 (d)、δC 72.0 (d)],推测有1个缩醛和1个羟基取代.根据C-6与C-7的化学位移,H-12[δH 1.22 (s, 3H)]与C-5、C-6、C-7的HMBC相关,结合1H-1H COSY相关链中断这一信息,可推测存在C(6)-O-C(7)缩醛基团[10],甲基CH3-12连接在含氧季碳C-6上.
细胞松弛素中与母核骈连大环(macro ring,C环)是构成其化学结构多样性最主要因素,大环碳数、氧化水平、取代基等差异大.首先根据不饱和度推测大环环系.化合物1不饱和度为15,已鉴定的吲哚环不饱和度为6,A环不饱和度为3,大环的5个不饱和度除去2个双键和2个羰基外仅剩余1个不饱和度,故仅有1个大环.1H-1H COSY谱显示的H-7/H-8/H-13/H-14/H-15/H-16/H-17/H-24和H-20/H-21/H-22自旋耦合系统,以及HMBC谱中H-17和C-19、H-20与C-18、C-22与C-9等相关可确定化合物1中的大环为本类细胞松弛素最常见的十三元碳环,且通过C-8和C-9与母核骈连.HMBC谱中H-7和C-13、H-16和C-14、H-15和C-17等相关进一步确定△13, 14和△17, 18位置.CH3-24的位置可由H-24与H-16的1H-1H COSY相关确定.CH3-25由HMBC谱中显示的H-25与C-17、C-19的相关可确定.羟基取代于C-20是根据其化学位移确定的,此外,HMBC谱中的H-20和C-18、H-20和C-22等相关进一步验证其位置.
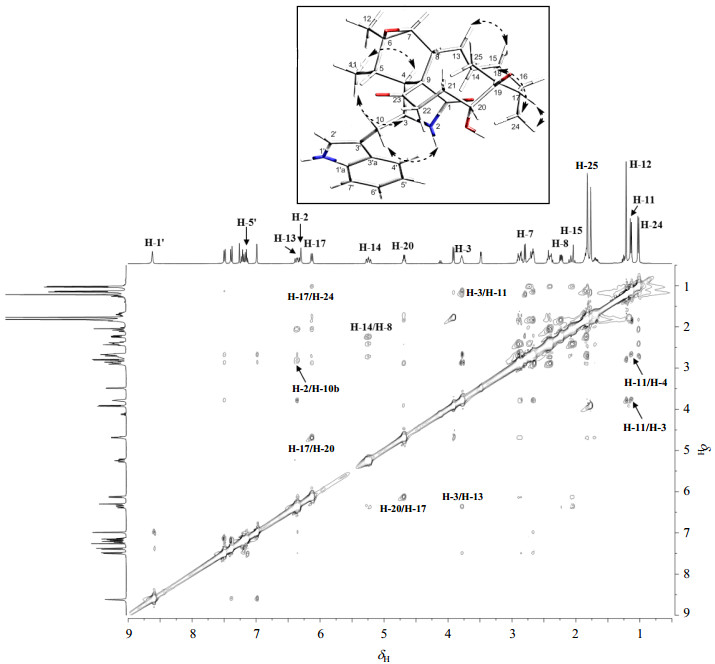
至此,化合物1平面结构得到确定,化合物1相对构型是通过耦合常数和NOESY谱(图 4)判断.表 1列出了化合物1的非邻位NOESY相关.根据H-3α与H-11的NOESY相关可判断C-10为β构型且CH3-11为α构型[9].H-8为β构型是根据H-7与H-8之间较大的耦合常数(J7, 8 = 7.2 Hz)[11]. H-13和H-14较大的耦合常数(J13, 14 = 15.0 Hz)说明△13, 14为E构型[12].NOESY谱中显示的H-17和H-20相关表明△17, 18也是E构型.CH3-24为α构型是由NOESY谱中H-24与H-25的相关确定的.至此,确定了化合物1的相对构型,其绝对构型的确定则需通过X-射线单晶衍射、电子圆二色谱(ECD)计算等方法.
|
图 4 化合物1的NOESY谱和主要NOESY相关(  |
| 表 1 化合物1的1H和13C NMR信号归属(CDCl3) Table 1 1H and 13C NMR assignments for compound 1 in CDCl3 |
10-吲哚类是天然细胞松弛素中数量较多的一类,自1973年首次报道chaetoglobosins A和B[13]起,至今发现的该类化合物已将近100个[14].该类化合物结构特征为3-取代吲哚环连接于氢化异吲哚-1-酮母核C-10上,并骈连有十三元碳环为主的大环系统,其NMR结构特征总结如下.
(1)3-取代吲哚环:该环结构变化小,NMR化学位移固定(表 2),在谱图中辨识度高.目前仅armochaetoglobin T取代有C(2′)-O-C(3′)缩醛基团,其C-2′[δC 82 (d)]、C-3′[δC 92 (d)]因sp2→sp3转化而高场位移[9].
| 表 2 10-吲哚类细胞松弛素常见结构单元的特征NMR信号 Table 2 Characteristic NMR signals of common structural units in 10-indole cytochalasins |
(2)2-吡咯烷酮:此类化合物B环几乎无变化,其酰胺羰基与季碳C-9化学位移较为固定,可视为本类化合物鉴定的特征信号.仅有少数化合物C-10连有甲基[δC 34~37 (d),δC 13~15 (q)][15].
(3)六元环系统:六元环由环己烷和CH3-11、CH3-12共同组成,氧化水平较高,不同位置取代有双键、羟基、缩醛等基团.双键常见△5, 6、△6, 7,部分为△6, 12端烯 [16].缩醛多为C(6)-O-C(7),极少数形成C(3)-O-C(6)(表 2).羟基有OH-12、OH-7β、OH-6和OH-5等,OH-7β最为常见,还可见少许OAc-7[δH 2.0 (q);δC 172 (s),21 (q)][9]、酮羰基-7[δC 208 (s)][17].少数化合物有取代基构型变化,如CH3-12一般为α型,但少数因存在C(3)-O-C(6)、OH-6α而为β型.
(4)大环系统:与10-苯基类细胞松弛素相比,本类化合物大环的环系变化较小,这是两类细胞松弛素的区别之一.环系数目可依据高分辨质谱所得分子式计算不饱和度,根据已鉴定不饱和基团、片段中环系进行推算.多数为1个十三元碳环(5/6/13环系),少数存在环系变化,如十一元环(5/6/11环系)、C(19)-C(20)氧化断裂开环[18]、C(17)-C(21)联结(5/6/10/6环系)等情况[9].在本类化合物的结构鉴定中,大环的环系主要根据1H-1H COSY相关链及HMBC相关确定.
大环系统本质上属于聚酮结构,氧化水平差异导致其结构丰富的多样性,富含双键、甲基、羟基、羰基等基团[19].不同位置的基团受到邻近结构影响化学位移差异较大,如双键△17, 18、△21, 22易与邻近羰基、羟基形成共轭结构,导致整个共轭体系的化学位移发生变化.反式二取代双键△13, 14的邻近结构变化小,可作为细胞松弛素鉴定的特征NMR信号.
综上所述,10-吲哚类细胞松弛素结构解析时可分为10-吲哚环、氢化异吲哚-1-酮母核(A、B环)和大环系统(C环)三个部分.各部均有一些化学位移相对固定的特征NMR信号,从这些特征NMR信号入手,结合2D NMR谱的相关,可对该类化合物进行准确的鉴定和归属.
3 总结本文通过综合运用1D和2D NMR技术,分析和归属了复杂真菌天然产物chaetoglobosin F(化合物1)所有1H和13C NMR信号,运用NOESY技术和偶合常数对其空间结构进行了确定.此外,本文还初步总结归纳了10-吲哚细胞松弛素的NMR特征,为10-吲哚类细胞松弛素的结构解析和深入研究提供了参考.
| [1] | ALDRIDGE D C, ARMSTRONG J J, SPEAKE R N, et al. The cytochalasins, a new class of biologically active mould metabolites[J]. Chem Commun, 1967, 1: 26-27. |
| [2] | YAN B C, WANG W G, HU D B, et al. Phomopchalasins A and B, two cytochalasans with polycyclic-fused skeletons from the endophytic fungus Phomopsis sp.shj2[J]. Org Lett, 2016, 18(5): 1108-1111. DOI: 10.1021/acs.orglett.6b00214. |
| [3] | SCHERLACH K, BOETTGER D, REMME N, et al. The chemistry and biology of cytochalasans[J]. Nat Prod Rep, 2010, 27(6): 869-886. DOI: 10.1039/b903913a. |
| [4] | ZHU H C, CHEN C M, TONG Q Y, et al. Asperflavipine A:a cytochalasan heterotetramer uniquely defined by a highly complex tetradecacyclic ring system from Aspergillus flavipes QCS12[J]. Angew Chem Int Edit, 2017, 56(19): 5242-5246. DOI: 10.1002/anie.201701125. |
| [5] | ISHIUCHI K, NAKAZAWA T, YAGISHITA F, et al. Combinatorial generation of complexity by redox enzymes in the chaetoglobosin A biosynthesis[J]. J Am Chem Soc, 2013, 135(19): 7371-7377. DOI: 10.1021/ja402828w. |
| [6] | LI W, YANG X Q, YANG Y B, et al. Anti-phytopathogen, multi-target acetylcholinesterase inhibitory and antioxidant activities of metabolites from endophytic Chaetomium globosum[J]. Nat Prod Res, 2016, 30(22): 2616-2619. DOI: 10.1080/14786419.2015.1129328. |
| [7] |
YANG E B, MA L B, WANG J H, et al. NMR signal assignment and solvent effects of N-substituted indoles[J].
Chinese J Magn Reson, 2013, 30(2): 256-263.
杨二冰, 马利波, 王金辉, 等. 吲哚衍生物NMR数据归属及其溶剂效应研究[J]. 波谱学杂志, 2013, 30(2): 256-263. DOI: 10.3969/j.issn.1000-4556.2013.02.010. |
| [8] |
ZHAO M, PENG S Q. Proton NMR and stereochemistry of synthetic indole alkaloids[J].
Chinese J Magn Reson, 1995, 12(1): 71-78.
赵明, 彭师奇. 合成吲哚生物碱的1H NMR及立体化学[J]. 波谱学杂志, 1995, 12(1): 71-78. |
| [9] | CHEN C M, TONG Q Y, ZHU H C, et al. Nine new cytochalasan alkaloids from Chaetomium globosum TW1-1(Ascomycota, Sordariales)[J]. Sci Rep, 2016, 6: 18711. DOI: 10.1038/srep18711. |
| [10] | IWAMOTO C, YAMADA T, ITO Y, et al. Cytotoxic cytochalasans from a Penicillium species separated from a marine alga[J]. Tetrahedron, 2001, 57(15): 2997-3004. DOI: 10.1016/S0040-4020(01)00153-3. |
| [11] | THOHINUNG S, KANOKMEDHAKUL S, KANOKMEDHAKUL K, et al. Cytotoxic 10-(indol-3-yl)-[13]cytochalasans from the fungus Chaetomium elatum ChE01[J]. Arch Pharm Res, 2010, 33(8): 1135-1141. DOI: 10.1007/s12272-010-0801-5. |
| [12] |
YIN T P, CHEN Y, LUO P, et al. Structural elucidation and NMR spectral assignments of two C 19-diterpenoid alkaloids[J].
Chinese J Magn Reson, 2018, 35(1): 90-97.
尹田鹏, 陈阳, 罗萍, 等. 两个C19-二萜生物碱的结构鉴定和NMR信号归属[J]. 波谱学杂志, 2018, 35(1): 90-97. |
| [13] | SEKITA S, YOSHIHIRA K, NATORI S, et al. Structures of chaetoglobosin A and B, cytotoxic metabolites of Chaetomium globosum[J]. Tetrahedron Lett, 1973, 14(23): 2109-2112. DOI: 10.1016/S0040-4039(01)86820-9. |
| [14] | ZHU X W, ZHOU D L, LIANG F Y, et al. Penochalasin K, a new unusual chaetoglobosin from the mangrove endophytic fungus Penicillium chrysogenum V11 and its effective semi-synthesis[J]. Fitoterapia, 2017, 123: 23-28. DOI: 10.1016/j.fitote.2017.09.016. |
| [15] | CHRISTIAN O E, COMPTON J, CHRISTIAN K R, et al. Using jasplakinolide to turn on pathways that enable the isolation of new chaetoglobosins from Phomospis asparagi[J]. J Nat Prod, 2005, 68(11): 1592-1597. DOI: 10.1021/np050293f. |
| [16] | CUI C M, LI X M, LI C S, et al. Cytoglobosins A-G, cytochalasans from a marine-derived endophytic fungus, Chaetomium globosum QEN-14[J]. J Nat Prod, 2010, 73(4): 729-733. DOI: 10.1021/np900569t. |
| [17] | XUE M, ZHANG Q, GAO J M, et al. Chaetoglobosin Vb from endophytic Chaetomium globosum:absolute configuration of chaetoglobosins[J]. Chirality, 2012, 24(8): 668-674. DOI: 10.1002/chir.22068. |
| [18] | CHEN C M, WANG J P, LIU J J, et al. Armochaetoglobins A-J:cytochalasan alkaloids from Chaetomium globosum TW1-1, a fungus derived from the terrestrial arthropod Armadillidium vulgare[J]. J Nat Prod, 2015, 78(6): 1193-1201. DOI: 10.1021/np500626x. |
| [19] | YIN T P, XING Y, CAI L, et al. A new polyketide glycoside from the rhizospheric Clonostachys rogersoniana associated with Panax notoginseng[J]. J Asian Nat Prod Res, 2017, 19(12): 1258-1263. DOI: 10.1080/10286020.2017.1314271. |
 2019, Vol. 36
2019, Vol. 36 
