2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
泛素(ubiquitin, Ub)是一种广泛存在于真核细胞中的信号分子.它是由76个氨基酸组成的分子量较小的蛋白质,几乎参与生命体中所有的生理过程[1].在泛素参与的生理过程中,泛素在特异性连接酶的作用下通过共价键与底物连接,对底物进行泛素化.而泛素作为一种信号分子,能够被上千种受体识别[2].因此,泛素能够携带底物与功能各异的蛋白质相互作用,行使复杂多样的生物学功能,在这些功能中就包括重要的蛋白质降解生理功能.
细胞中错误折叠的、异常的蛋白质能够被泛素化,随后被运送至泛素蛋白酶体系统降解,作为细胞内的“垃圾”被清除.蛋白质降解功能的异常可能诱发神经退行性疾病,例如帕金森病和老年性痴呆[3-5].在对异常蛋白质降解时,通常被泛素化的异常蛋白与蛋白质酶体是通过一个“转运因子”进行连接,将底物运送至活性中心进行水解.转运因子通常是一个由多个不同功能结构域组成的蛋白质,在这些结构域中往往包含一个泛素结合结构域,用于连接泛素化的蛋白底物.这些泛素结合结构域之间具有较低的氨基酸序列相似性,但是均能够与泛素结合形成复合体,其相互作用的亲和力从3 μmol/L到2 mmol/L不等[6].在种类繁多的泛素结合结构域中,有一些具有高度相似的折叠方式,它们由三个α-螺旋组成,通过疏水相互作用形成螺旋束,具有该结构特征的泛素结合结构域被叫作泛素结合域(ubiquitin-associated domain,UBA,图 1),它们能够与泛素表面由L8、I44和V70组成的疏水口袋相互作用形成蛋白质复合体.
|
图 1 泛素结合域(UBA)的结构(PDB编号为1DV0) Fig. 1 UBA structure shown in cartoon (PDB code: 1DV0) |
Ubiquilin-1(Ubql-1)是一种参与蛋白质降解的重要的转运因子[7].Ubql-1通过其碳端的UBA与细胞内泛素化的蛋白质聚集体相互作用,将其“转运”至蛋白酶体进行水解反应[6].与Ubql-1相似,Rad23A作为转运因子参与蛋白酶体催化的多聚泛素化底物的降解过程.在Rad23A中有两个UBA结构域,一个位于Rad23A的中部(UBA1),另外一个则位于其碳端(UBA2)[8].Rad23A的UBA2结构域和泛素非共价结合,通过“加帽”泛素来抑制多聚泛素蛋白链的延长,从而限制了蛋白酶体催化蛋白水解[9].因此,泛素与UBA的相互作用提供了一类重要的泛素识别机制.
泛素的复杂性不仅仅体现在能够被种类繁多的受体识别,还体现在泛素本身还能够被磷酸化[10].组学研究显示几乎泛素表面所有丝氨酸都能够被磷酸化[11-14],但是目前唯一被发现的能够磷酸化泛素的激酶是PINK1,它能够特异性磷酸化泛素的S65位.PINK1泛素磷酸化激酶功能的发现实现了在体外对磷酸化泛素(phosphorylated ubiquitin,pUb)的研究,同时也将泛素化和磷酸化这两种重要的翻译后修饰方式紧密的联系在一起,为传统的泛素相关研究开辟了全新的探索方向.
磷酸化直接影响泛素的结构,使泛素在溶液中呈现两种互相转换的构象,即舒展态(pUb relaxed state, pUbrelax)和收缩态(pUb retracted state, pUbretract)[15].舒展态与非磷酸化泛素的蛋白质空间结构非常类似;与舒展态相比,收缩态碳端β-折叠向其氮端收缩了两个氨基酸,造成与碳端β-折叠相关的氢键网络的变化[16].磷酸化在改变泛素溶液结构的同时,还增强了泛素对溶液pH环境的敏感性.收缩态和舒展态具有不同的pKa,因此溶液中pH值的变化能够直接改变两种构象之间的平衡,变换两种构象之间的比例.由此可见,泛素S65位的磷酸化赋予了泛素全新的特性.
磷酸化改变了泛素的溶液结构,而这种结构的变化往往伴随着蛋白质功能的变化.已有文献[17]报道指出pUb能够从酶活底物转变成激活因子.因此,磷酸化能够通过改变泛素的结构进而调控泛素的功能.泛素与UBA之间的相互作用作为一种重要的受体对泛素的识别机制,已经被广泛研究.但是,由于PINK1对泛素磷酸化的功能近期才被发现,磷酸化对泛素与UBA相互作用的影响与调控的研究尚未全面开展.目前,文献[18]报道中指出通过蛋白质体外结合实验发现磷酸化很有可能会提高泛素与Rad23A之间的亲和力.本文使用核磁共振(nuclear magnetic resonance,NMR)方法[19]对磷酸化泛素与Rad23A-UBA2之间的相互作用以及磷酸化泛素与Ubql-1-UBA之间的相互作用进行了检测.已有报道[20]指出UBA与泛素之间的解离常数为μmol/L级别,而NMR恰好是检测此范围内蛋白质之间相互作用的有力手段.我们发现Rad23A-UBA2与Ubql-1-UBA都特异性的与磷酸化泛素的舒展态相互作用.与Rad23A-UBA2相比,Ubql-1-UBA与磷酸化泛素之间有更高的亲和力.而且,在与Ubql-1-UBA相互作用时,随着溶液中Ubql-1-UBA浓度升高,磷酸化泛素的收缩态会向舒展态转换,而这种转换可能是较高分子间亲和力造成的.
1 实验部分 1.1 样品制备 1.1.1 蛋白表达与纯化将人源泛素、Rad23A-UBA2、Ubql-1-UBA、PINK1蛋白的基因克隆到大肠杆菌(BL21 star)表达载体中,获得大量的重组表达.
用LB培养基来表达不需要同位素标记的泛素靶蛋白及各种酶,培养温度为37 ℃,待吸光光度值OD600=0.6~0.9时,加入异丙基硫代半乳糖苷(isopropyl-β-d-thiogalactoside,IPTG)开始诱导.泛素蛋白采用0.5 mmol/L IPTG 37 ℃诱导表达4 h;Rad23-UBA2、Ubql-1-UBA采用0.5 mmol/L IPTG 23 ℃过夜诱导20 h;而泛素磷酸化激酶PINK1则采用0.1 mmol/L IPTG 18 ℃低温诱导表达24 h.收取菌液后离心(7 000 rpm)10 min,收集沉淀.然后高压破碎重悬的菌液,高速离心(20 000 rpm)20 min后,取上清进行蛋白质纯化.
蛋白质纯化采用标准层析色谱柱方法,根据融合表达的蛋白质所带标签和电荷的不同,联合应用离子交换柱和分子筛等高分辨柱.泛素蛋白初步纯化采用SP SepharoseTM Fast Flow(FFSP)阳离子交换树脂(GE Healthcare),根据目标蛋白与杂蛋白带电荷的不同将部分杂蛋白除去;然后采用分子筛Superdex-100(S100,GE Healthcare),根据蛋白的大小对目标蛋白进一步纯化.Ubql-1-UBA蛋白带负电荷,初步纯化采用DEAE阴离子交换树脂(GE Healthcare),再使用S100对目标蛋白进一步纯化.Rad23A-UBA2与Trix促融标签融合表达,且带有His标签,故采用Ni柱对其进行初步纯化,再过S100进行精纯.PINK1的质粒带有谷胱甘肽S-转移酶(glutathione S-transferase,GST)亲和标签,采用GST亲和柱(GE Healthcare)进行层析浓缩后,直接用HiPrep 26/10脱盐柱(GE Healthcare)置换缓冲液,-80 ℃冻存.整个过程都在低温条件下进行,以保证酶的活性.
纯化后的蛋白样品通过十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate polyacrylamide gel electrophoresis,SDS-PAGE)鉴定纯度,用电喷雾-四极杆-飞行时间质谱仪(ESI-Q-TOF-MS,Agilent6530)检测样品的精确分子量,以判断是否为目标蛋白.
用于NMR检测的蛋白使用U-15NH4Cl和D-glucose作为细菌生长的唯一氮源和碳源,将大肠杆菌在M9基础培养基中培养,来获得稳定同位素标记的泛素蛋白.培养温度为37 ℃,采用0.5 mmol/L IPTG诱导表达4 h.纯化、鉴定方式与非标记蛋白方法一致.
1.1.2 磷酸化泛素蛋白样品制备我们使用PINK1对泛素单体进行磷酸化,磷酸化位点为S65:将泛素蛋白、PINK1、ATP、MgCl2以1:0.01:50:50(物质的量之比)的比例混合后,25 ℃水浴12 h或者37 ℃恒温水浴反应5 h,反应体系含20 mmol/L Tris、1 mmol/L DTT,pH 8.0,泛素蛋白的终浓度约为0.2 mmol/L.然后通过阴离子交换层析柱分离磷酸化和未磷酸化的泛素蛋白样品.最后通过高分辨质谱鉴定磷酸化泛素蛋白.
1.1.3 NMR滴定实验样品制备缓冲体系含20 mmol/L HEPES和150 mmol/L NaCl,pH 7.4.测量UBA(滴定相)浓度,保证其物质的量浓度 > 2 mmol/L(若浓度未达到要求,则用透析缓冲液润洗Amicon管后继续浓缩,直到滴定相的浓度为被滴定相的10倍以上,以防止在滴定过程中体系体积变化太大),被滴定相为泛素(或磷酸化泛素)蛋白样品统一配制,蛋白浓度为100 μmol/L,锁场溶剂为(10% D2O+90% H2O),体积为500 μL,缓冲体系为20 mmol/L HEPES、150 mmol/L NaCl,pH 7.4.
1.2 NMR实验按滴定相与被滴定相物质的量之比为0:1、0.1:1、0.2:1、0.3:1、0.5:1、0.75:1、1:1、1.25:1、1.5:1、2:1依次采集1H- 15N HSQC谱图.NMR实验在配备低温探头的Bruker 600型NMR谱仪上进行,实验温度为298 K.采样数据点阵t2×t1 = 1 024×256,累加次数为16,1H、15N维的谱宽分别为9 597 Hz、1 702 Hz.
2 结果与讨论 2.1 磷酸化对泛素与Rad23A-UBA2之间相互作用的调控我们首先采集了未加入UBA的15N同位素标记的磷酸化泛素的1H-15N HSQC谱图.通过对谱图上主链氨基酸信号进行分析[21],我们发现信号个数超过120个,多于非磷酸化泛素氨基酸个数(76个),说明磷酸化泛素在溶液中呈现出两种构象,即pUbrelax和pUbretract.此外,通过对NMR信号强度的分析,我们发现来自上述两种构象的信号强度比为1:1,说明在20 mmol/L HEPES、150 mmol/L NaCl、pH 7.4、298 K条件下,pUbrelax与pUbretract在溶液中各占50%.而上述观察到的现象也与已发表文献[16]报道的结论一致.
我们将Rad23A-UBA2与泛素的物质的量之比为1.25:1时相对未加入靶蛋白时的化学位移扰动(chemical shift perturbation,CSP)[(1)式]描绘到泛素的表面.(1)式中,Xi、Yi分别为滴定到第i张谱时的各信号1H、15N维化学位移,X0、Y0位未加入UBA时各信号对应的1H、15N维化学位移[图 2(a)].可以观测到,Ub与Rad23A-UBA2之间的相互作用界面主要集中在β-折叠区域,包含L8-I44-V70 loop区疏水口袋,远离S65磷酸化位点.泛素磷酸化后,作用界面并未发生变化,依然集中在β1-、β3-、β4-折叠与相连接的loop区[I13、R42、L50、L67-V71等氨基酸所在区域,图 2(b)中已标记为橙色].

|
图 2 (a) Rad23A-UBA2在pH 7.4、298 K条件下滴定pUb的1H-15N HSQC谱,UBA的浓度变化用不同颜色加以区分;(b) Rad23A-UBA与泛素的复合体结构(PDB编号为5XBO),CSP较大的氨基酸残基显示为橙色,L8-I44-V70输水口袋已用蓝色标记,S65磷酸化位点用球棍模型标出;(c)滴定相(Rad23A-UBA2)与被滴定相(pUb或Ub)物质的量之比达到1.25:1时的Ub、pUbrelax与pUbretract的CSP;(d) Rad23A-UBA与Ub、pUbrelax相互作用KD的拟合结果 Fig. 2 (a) The superimposed 1H-15N HSQC spectra monitoring the titration of Rad23A-UBA2 into pUb at 298 K and pH 7.4, UBA concentration changes are labeled by different colors. (b) The structure of the Rad23A-UBA2/Ub complex shown in cartoon (PDB code: 5XBO), the regions with large CSPs are mapped on to the surface of ubiquitin in orange. The hydrophobic pocket formed by L8-I44-V70 is highlighted as blue, and S65 phosphorylation site is shown as sticks and spheres. (c) The CSP profiles of Ub, pUbrelax, and pUbretract shown in column when the molar ratio between Rad23A-UBA2 and Ub or pUb is 1.25:1. (d) The fitting of KD of Rad23A-UBA2/Ub and Rad23A-UBA2/ pUbrelax |
| $ CSP = \sqrt {0.5 \times {{({X_i} - {X_0})}^2} + 0.1 \times {{({Y_i} - {Y_0})}^2}} $ | (1) |
我们挑选处于作用界面的氨基酸残基,以体系中UBA的浓度为x轴、氨基酸残基的CSP为y轴作散点图.采用(2)式对这些点进行全局拟合.p为被滴定相的浓度(100 μmol/L);化学计量比n=1;a、b分别为化学位移扰动变化的最小值和最大值,计算UBA与Ub之间的平衡解离常数KD(即公式中的k).
| $ y = \left[ {\frac{{x + p}}{{n + k}} - \sqrt {{{\left( {\frac{{x + p}}{{n + k}}} \right)}^2} - \frac{{4xp}}{n}} } \right] \times \frac{{(b - a) \times n}}{{2p}} + a $ | (2) |
蛋白质之间的相互作用会造成NMR信号的展宽,降低谱图的质量,进而影响后续数据分析.我们选择滴定终点为滴定相与被滴定相物质的量之比为2:1,在此浓度下我们能够获得足够的蛋白质复合体比例用于分析,并且谱图质量较高.从磷酸化泛素的CSP [图 2(c)]可以看出,Rad23A-UBA2的引入只造成了pUbrelax的化学位移扰动,而其pUbretract的NMR信号则几乎不受到Rad23A-UBA2的影响.这说明Rad23A-UBA2特异性与pUbrelax相互作用,而与pUbretract之间没有明显的相互作用.与非磷酸化泛素相比较,pUbretract的碳端β-折叠向其氮端收缩两个氨基酸,而pUbretract的V70正好处在结构发生变化的区域.该氨基酸的变化可能造成了泛素表面由L8-I44-V70组成的疏水口袋的结构改变,进而破坏了pUbretract和Rad23A-UBA2的相互作用.而pUbrelax和非磷酸化泛素结构高度相似,可以与Rad23A-UBA2相互作用.
但是从CSP与UBA的浓度拟合获得KD来分析,磷酸化几乎没有改变泛素与Rad23A-UBA2之间的亲和力,拟合获得的Rad23A-UBA2/Ub、Rad23A-UBA2/pUbrelax的KD分别为(145.96±5.73)μmol/L、(151.77±5.62)μmol/L.由于pUbrelax的三级结构与非磷酸化泛素的结构类似,且磷酸化位点S65距离蛋白质之间的作用界面较远,所以磷酸化对pUbrelax与Rad23A-UBA2之间的相互作用影响较小,蛋白质之间的亲和力仍然处在μmol/L级别.
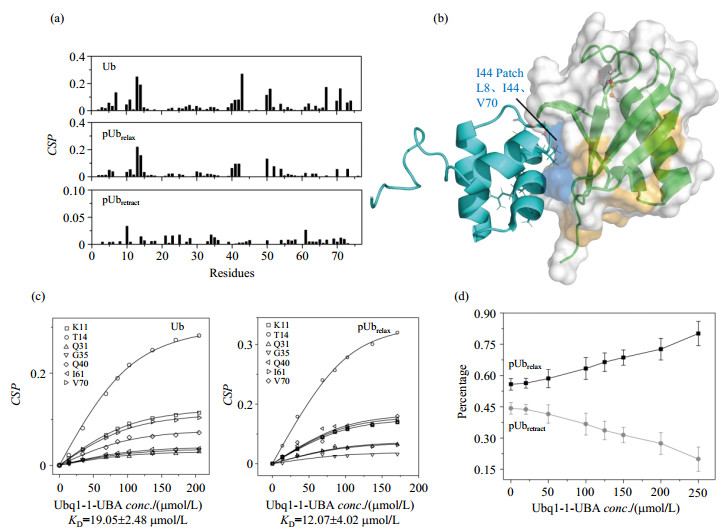
2.2 磷酸化对泛素与Ubql-1-UBA之间相互作用的调控我们用未标记的Ubql-1-UBA滴定15N稳定同位素标记的泛素或磷酸化泛素(图 3),采用相同的处理方式,我们将Ubql-1-UBA与Ub的物质的量之比为1.25:1时的CSP描绘到泛素的表面(图 4),结果显示在加入Ubql-1-UBA后,泛素的G10氨基酸附近、Q40-L50氨基酸以及碳端区域具有明显的连续的化学位移变化,并且Ubql-1-UBA对pUbrelax造成了与非磷酸化泛素相似的CSP变化.这说明非磷酸化泛素和pUbrelax通过相同的互作界面与Ubql-1 UBA结合,而此相互作用界面是保守的泛素与其配体的结合区域.

|
图 3 Ubql-1-UBA在pH 7.4、298 K条件下滴定pUb的1H-15N HSQC谱,UBA的浓度变化用不同颜色加以区分 Fig. 3 The superimposed 1H-15N HSQC spectra monitoring the titration of Ubql-1-UBA into pUb at 298 K and pH 7.4, UBA concentration changes are labeled by different colors |
|
图 4 (a) Ubql-1-UBA与泛素滴定比例为1.25:1时Ub、pUbrelax与pUbretract的化学位移扰动;(b) Ubql-1-UBA与泛素的复合体结构(PDB编号为2JY6),CSP较大的氨基酸残基显示橙色,结合界面L8-I44-V70区域显示蓝色,S65磷酸化位点用球棍模型标出;(c) Ub和pUbrelax分别与Ubql-1-UBA结合的KD拟合结果;(d) pUbrelax与pUbretract的峰强随UBA滴定浓度增加的变化趋势图 Fig. 4 (a) The CSP profiles of pUbrelax, pUbretract and Ub induced by addition of Ubql-1-UBA with a molar ratio of 1.25:1; (b) The cartoon structure of the complex of Ubql-1-UBA and Ub (PDB code: 2JY6), and the regions with large CSP values are mapped onto Ub surface in orange. The hydrophobic pocket formed by L8-I44-V70 is highlighted as blue, and S65 phosphorylation site is shown as sticks and spheres; (c) The KD of Ubql-1-UBA/Ub and Ubql-1-UBA/pUbrelax fitted based on the CSPs induced by addition of Ubql-1-UBA; (d) The signal intensity changes of pUbrelax and pUbretract induced by the addition of Ubql-1-UBA |
对不同滴定相浓度下CSP的拟合显示Ubql-1-UBA与非磷酸化泛素和pUbrelax的KD分别为(19.05± 2.48)μmol/L和(12.07±4.02)μmol/L.由此可以推断磷酸化对泛素与Ubql-1-UBA之间的相互作用造成了微弱的影响.而蛋白质之间亲和力的细小变化与保守的蛋白质互作界面相关,磷酸化未改变泛素与Ubql-1-UBA的相互作用区域,进而保留了泛素与Ubql-1-UBA之间结合的方式与强度.
与Rad23A-UBA2的实验结果相同,在加入Ubql-1-UBA之后,pUbretract的化学位移并没有发生明显的变化,这说明Ubql-1-UBA与pUbretract之间也没有相互作用,而特异性选择与其pUbrelax结合.这种构象的选择性相互作用可能与磷酸化泛素两种构象之间结构差异有关.
值得注意的是,随着滴定实验中Ubql-1-UBA的浓度增加,pUbretract化学位移尽管不变,但其信号强度逐渐下降,这说明虽然Ubql-1-UBA没有和pUbretract相互作用,但是Ubql-1 UBA的加入导致后者在溶液中的比例发生了变化.通过进一步比较在不同浓度Ubql-1 UBA时pUbrelax和pUbretract的比例发现,溶液中Ubql-1-UBA的浓度与pUbrelax在溶液中的比例呈正比,而与pUbretract呈反比.因此我们推断在溶液中磷酸化泛素与Ubql-1-UBA的相互作用能够诱导pUbretract向pUbrelax转换,而此现象未曾在Rad23A-UBA2体系中观察到.我们推测pUbrelax与Rad23A-UBA2结合形成复合体,该复合体中pUbrelax的结构能级与自由状态下的pUbrelax的结构能级类似,因此未发生态之间的转换.但是和Ubql-1-UBA结合后,这种复合体中pUbrelax的结构能级可能比自由状态下pUbrelax和pUbretract低,并且可能伴随着结构的变化,因此结构向能级较低的构象转换.
3 结论本文通过NMR方法对非磷酸化泛素和磷酸化泛素与Rad23A-UBA2和Ubql-1-UBA之间的相互作用进行了表征,为磷酸化后的泛素的靶点的筛选、参与信号调控的作用机制的研究提供了实验基础.实验结果表明Rad23A-UBA2与Ubql-1-UBA都特异性与pUbrelax相互作用,而磷酸化未对上述UBA与pUbrelax之间的相互作用的方式与强度造成较大影响.虽然在与Rad23A-UBA2和Ubql-1-UBA均未与pUbretract显示明显的相互作用,但是与Ubql-1-UBA的加入能够驱使pUbretract向pUbrelax的转换.我们推测,pUbrelax与Rad23A-UBA2能结合形成复合体,该复合体中pUbrelax的结构能级与自由状态下的pUbrelax的结构能级类似,因此未发生态之间的转换.而与Rad23A-UBA2相比较,Ubql-1-UBA与pUbrelax形成的复合体中,pUbrelax的结构能级可能比自由状态下pUbrelax和pUbretract的都低,并且可能伴随着结构的变化,因此结构向能级较低的构象转换.
| [1] | KOMANDER D, RAPE M. The ubiquitin code[J]. Annu Rev Biochem, 2012, 81: 203-229. DOI: 10.1146/annurev-biochem-060310-170328. |
| [2] | DIKIC I, WAKATSUKI S, WALTERS K J. Ubiquitin-binding domains-from structures to functions[J]. Nat Rev Mol Cell Biol, 2009, 10(10): 659-671. DOI: 10.1038/nrm2767. |
| [3] | FOROUD T, UNIACKE S K, LIU L, et al. Heterozygosity for a mutation in the parkin gene leads to later onset Parkinson disease[J]. Neurology, 2003, 60(5): 796-801. |
| [4] | DENG H X, CHEN W, HONG S T, et al. Mutations in UBQLN2 cause dominant X-linked juvenile and adult-onset ALS and ALS/dementia[J]. Nature, 2011, 477(7363): 211-215. DOI: 10.1038/nature10353. |
| [5] | EL AYADI A, STIEREN E S, BARRAL J M, et al. Ubiquilin-1 regulates amyloid precursor protein maturation and degradation by stimulating K63-linked polyubiquitination of lysine 688[J]. Proc Natl Acad Sci U S A, 2012, 109(33): 13416-13421. DOI: 10.1073/pnas.1206786109. |
| [6] | ZHANG D N, RAASI S, FUSHMAN D. Affinity makes the difference:nonselective interaction of the UBA domain of Ubiquilin-1 with monomeric ubiquitin and polyubiquitin chains[J]. J Mol Biol, 2008, 377(1): 162-180. |
| [7] | HANAOKA E, OZAKI T, OHIRA M, et al. Molecular cloning and expression analysis of the human DA41 gene and its mapping to chromosome 9q21.2-q21.3[J]. J Hum Genet, 2000, 45(3): 188-191. DOI: 10.1007/s100380050209. |
| [8] | WITHERS-WARD E S, MUELLER T D, CHEN I S, et al. Biochemical and structural analysis of the interaction between the UBA(2) domain of the DNA repair protein HHR23A and HIV-1 Vpr[J]. Biochemistry, 2000, 39(46): 14103-14112. DOI: 10.1021/bi0017071. |
| [9] | HEESSEN S, MASUCCI M G, DANTUMA N P. The UBA2 domain functions as an intrinsic stabilization signal that protects Rad23 from proteasomal degradation[J]. Mol Cell, 2005, 18(2): 225-235. DOI: 10.1016/j.molcel.2005.03.015. |
| [10] | KOYANO F, OKATSU K, KOSAKO H, et al. Ubiquitin is phosphorylated by PINK1 to activate parkin[J]. Nature, 2014, 510(7503): 162-166. DOI: 10.1038/nature13392. |
| [11] | LEE H J, NA K, KWON M S, et al. Quantitative analysis of phosphopeptides in search of the disease biomarker from the hepatocellular carcinoma specimen[J]. Proteomics, 2009, 9(12): 3395-3408. DOI: 10.1002/pmic.v9:12. |
| [12] | ZHOU H J, DI PALMA S, PREISINGER C, et al. Toward a comprehensive characterization of a human cancer cell phosphoproteome[J]. J Proteome Res, 2013, 12(1): 260-271. DOI: 10.1021/pr300630k. |
| [13] | SCHWEPPE D K, RIGAS J R, GERBER S A. Quantitative phosphoproteomic profiling of human non-small cell lung cancer tumors[J]. J Proteomics, 2013, 91: 286-296. DOI: 10.1016/j.jprot.2013.07.023. |
| [14] | TSAI C F, WANG Y T, YEN H Y, et al. Large-scale determination of absolute phosphorylation stoichiometries in human cells by motif-targeting quantitative proteomics[J]. Nat Commun, 2015, 6: 6622. DOI: 10.1038/ncomms7622. |
| [15] | WAUER T, SWATEK K N, WAGSTAFF J L, et al. Ubiquitin Ser65 phosphorylation affects ubiquitin structure, chain assembly and hydrolysis[J]. EMBO J, 2015, 34(3): 307-325. |
| [16] | DONG X, GONG Z, LU Y B, et al. Ubiquitin S65 phosphorylation engenders a pH-sensitive conformational switch[J]. Proc Natl Acad Sci U S A, 2017, 114(26): 6770-6775. |
| [17] | SWATEK K N, KOMANDER D. Ubiquitin modifications[J]. Cell Res, 2016, 26(4): 399-422. |
| [18] | SWANEY D L, RODRÍGUEZ-MIAS R A, VILLÉN J. Phosphorylation of ubiquitin at Ser65 affects its polymerization, targets, and proteome-wide turnover[J]. EMBO Rep, 2015, 16(9): 1131-1144. DOI: 10.15252/embr.201540298. |
| [19] |
ZHANG N X, WU J, ZHANG H Q, et al. Protein-protein interactions studied by NMR-A review using the ubiquitin-proteasome[J].
Chinese J Magn Reson, 2012, 29(2): 182-189.
张乃霞, 吴娟, 张华群, 等. NMR技术在蛋白质-蛋白质相互作用研究中的应用-泛素-蛋白水解酶体通路研究实例介绍[J]. 波谱学杂志, 2012, 29(2): 182-189. DOI: 10.3969/j.issn.1000-4556.2012.02.003. |
| [20] | ZHANG D, RAASI S, FUSHMAN D. Affinity makes the difference:nonselective interaction of the UBA domain of Ubiquilin-1 with monomeric ubiquitin and polyubiquitin chains[J]. J Mol Biol, 2008, 377(1): 162-180. |
| [21] |
LI S L, ZHU Q J, LIU M L, et al. Characteristics of protein NMR resonances and chemical shift assignments[J].
Chinese J Magn Reson, 2017, 34(2): 137-147.
李双利, 朱勤俊, 刘买利, 等. 蛋白质分子核磁共振谱峰的特性及其化学位移归属[J]. 波谱学杂志, 2017, 34(2): 137-147. |
 2019, Vol. 36
2019, Vol. 36 
