2. 厦门大学 化学化工学院, 厦门大学高场核磁研究中心, 福建 厦门 361005;
3. 运动科学研究与传播中心, 厦门理工学院, 福建 厦门 361024
2. College of Chemistry and Chemical Engineering, High-Field Nuclear Magnetic Resonance Research Center, Xiamen University, Xiamen 361005, China;
3. Research and Communication Center of Exercise and Health, Xiamen University of Technology, Xiamen 361024, China
肿瘤是一种代谢性疾病,癌基因表达对肿瘤细胞代谢的影响是目前肿瘤研究的热点之一[1].Sirtuin7(SIRT7)属于组蛋白去乙酰基酶家族,是一种烟酰胺腺嘌呤二核苷酸(NAD+)依赖性的高度保守的去乙酰基酶[2].SIRT7基因位于第17号染色体长臂25区3带(17q25.3),其编码蛋白主要定位于细胞核[3].研究表明,SIRT7蛋白可以通过将启动子上的组蛋白H3K18去乙酰化,参与细胞的多种生理过程,包括rRNA转录与修饰、细胞衰老和凋亡,以及DNA损伤修复等[4].此外,SIRT7蛋白也可以与其它蛋白,如p53(K382diAC)[5]等相互作用,对其进行转录后的去乙酰化修饰.还有研究发现SIRT7蛋白除了具有去乙酰化转移酶活性以外,还具有去长链脂肪酰化[6]和去琥珀酰化转移酶活性[7]的功能,从而在细胞应激与代谢等生理过程中发挥重要作用.随着研究的深入,目前可以证实SIRT7基因的异常表达与心血管疾病、糖尿病以及多种肿瘤的发生发展密切相关[5, 8].尽管如此,SIRT7基因调控疾病发生和发展的分子机制并未完全阐明.在肿瘤研究中,越来越多的研究发现SIRT7基因具有癌基因属性.在胃癌、乳腺癌等肿瘤组织中,SIRT7基因均被发现存在异常表达,且这种异常表达与肿瘤细胞的增殖侵袭密切相关[9, 10].在胶质瘤研究[11]中,研究者发现与正常脑组织相比,SIRT7基因在瘤组织中的表达明显上调;同时,抑制SIRT7基因表达可以显著抑制胶质瘤细胞的增殖和侵袭能力.由此可以推测SIRT7基因很可能是胶质瘤恶性进展过程中的重要癌基因之一.胶质瘤恶性进展伴随着肿瘤细胞代谢特征的改变,但其分子机制仍未完全阐明.而且目前,SIRT7基因对胶质瘤细胞代谢特征的影响及作用机制尚未见相关报道.
本文以SIRT7基因稳定低表达的胶质瘤细胞株为研究对象,利用核磁共振氢谱(1H NMR)对该细胞株的水溶性萃取物进行代谢组学分析,寻找胶质瘤细胞中与SIRT7基因表达相关的特征性代谢物和代谢通路,重点探讨SIRT7基因表达变化对胶质瘤细胞恶性特性及代谢特征的影响.研究结果将为阐明恶性进展中胶质瘤细胞代谢特征改变的分子机制提供有利的理论依据.
1 实验部分 1.1 细胞培养本课题组利用shRNA干扰技术转染恶性胶质瘤细胞系U251,获得了稳定低表达SIRT7基因的转染shRNA-SIRT7质粒的细胞株Sh7和正常表达SIRT7基因的转染对照序列的细胞株ShC,两种细胞株均液氮保存;体外复苏后,使用含10%血清的DMEM完全培养基,在37 ℃以及5% CO2培养箱内进行培养;细胞培养1~2天进行换液;当细胞汇合度达到80%时,进行细胞传代.
1.2 蛋白质免疫印迹(Western blot)检测细胞中SIRT7蛋白表达Western blot检测使用的抗体购于CST公司.细胞株Sh7和ShC接种在6孔板,培养48 h,提取两组细胞总蛋白并用双辛酸(BCA)法测定蛋白浓度;两种样本分别取20 μg总蛋白进行十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(SDS-PAGE),并用增强化学发光法(ECL)显影检测SIRT7蛋白表达情况,使用β-actin蛋白作为内参.
1.3 逆转录-聚合酶链反应(RT-PCR)检测细胞中SIRT7mRNA表达TRIzol试剂盒及cDNA逆转录试剂盒购于宝生生物公司(Takara).Sh7和ShC接种在6孔板,培养48 h,采用TRIzol试剂盒提取各组细胞总RNA并逆转录为cDNA.SIRT7基因的上游引物序列为:5’-CAG GGA GTA CGT GCG GGT GT-3’,下游引物序列为5’-TCGGTCGCCGCTTCCCAGTT-3’;内参基因GAPDH的上游引物序列为:5’-AGCCACATCGCTCAGACAC-3’;下游引物序列为5’-GCCCAATACGACCAAATCC-3’.实验采用SYBR Green法进行荧光定量PCR扩增.
1.4 3-(4, 5-二甲基噻唑-2)-5-(3-羧甲基苯基)-2-(4-磺苯基)-2氢-四唑内盐(MTS)法测定细胞增殖MTS试剂购于Promega公司.Sh7和ShC接种在96孔板(每孔接种4 000个细胞).将细胞接种的时间点设定为0 h,分别在接种后24 h、48 h、72 h三个时间点检测细胞的增殖能力.向接种有细胞的96孔板中加入20 μL MTS试剂,在37 ℃培养箱继续培养4 h后,在490 nm波长下测量每个样品孔的吸光度,作为判断细胞增殖的依据。
1.5 水溶性细胞萃取液制备Sh7和ShC分别接种于10 cm培养皿,每组细胞样本设置6个平行样.细胞培养至汇合度约90%[12]时,利用直接刮取法制备细胞萃取液,主要操作步骤如下:倒掉细胞培养液,在培养皿中加入2 mL冷甲醇(﹣20 ℃)对细胞进行灭活,利用细胞刷将细胞刮离培养皿,并将细胞甲醇溶液转移到15 mL离心管,并加入2 mL氯仿和1.95 mL的超纯水.漩涡振荡5 min,静置15 min;在转速14 000 rpm、4℃条件下,离心30 min,取上层液体,并用氮吹仪吹干,重新加入500 μL D2O和50 μL D2O配制的含0.1% 2, 2, 3, 3-d(4)-3-(三甲基硅基)丙酸钠(TSP)的磷酸盐缓冲液(1.5 mol/L K2HPO4/NaH2PO4, pH=7.4),混合均匀后在转速14 000 rpm、4 ℃条件下离心15 min.随后取上清500 μL加入到5 mm的NMR样品管中,用于后续分析.
1.6 NMR实验1H NMR谱图在Bruker AvanceIII 600 MHz NMR谱仪上采集,实验温度是298 K,采用的脉冲序列为[(RD)-90˚-t1-90˚-τm-90˚-ACQ](来自Bruker Biospin pulse program library):t1=6.6 μs,τm=120 ms,水峰压制在等待时间(RD=3 s)中进行.采集点数(TD)为32 k,谱宽(SW)为10 kHz,累加次数(NS)为256次,空采次数(DS)为16次.使用Topspin 2.1(Bruker Biospin)处理谱图,自由感应衰减(FID)信号在傅里叶变化之前进行指数窗函数处理,线宽因子(lb)为0.3 Hz.
1.7 数据分析细胞增殖、蛋白定量等检测值用平均值±标准差(mean±SD)表示,并使用GraphPad Prism5软件中的t检验或非参数检验进行组间统计学分析,p < 0.05表示有统计学差异.
使用MestReNova 6.5(Mestrelab Research S.L,Spain)对NMR谱图进行手动相位和基线校正,并以TSP定标(δH 0.00)后,进行分段积分.积分区域为δH 0.2~9.5,水峰区域(δH 4.5~5.2)被剔除以消除水峰抑制效应对谱线积分的影响,积分间隔为1.8 Hz.采用总面积归一化方式得到X矩阵.利用SIMCA-P12+软件(Umetrics Inc.,Umea,Sweden)对X矩阵进行多变量分析.在进行多变量分析之前,采用帕莱托换算(Pareto Scaling)对数据实行缩放换算.特征性代谢物寻找主要采用如下方式:首先进行无监督的主成分分析(PCA),初步判断两组之间的代谢模式是否存在明显差异;然后通过有监督的偏最小二乘判别分析(PLS-DA)进一步验证PCA结果的可靠性,并通过7次循环交互验证(7-fold cross validation)和置换排列验证(RPT)的方法验证PLS-DA模型的可靠性;之后使用正交偏最小二乘分析(OPLS-DA)进行特征性变量(bin)的筛选.筛选标准主要依据第一预测主成分(Predictive component)的变量权重重要排序(VIP)值和相关系数(r).因为实验样品数量为12,所以在显著性p=0.05和p=0.01时的相关系数阀值的绝对值为0.532和0.661.对VIP≥1和|r| > 0.532或0.661变量进行归纳,从而找出对Sh7和ShC两组差异有贡献的特征性代谢物.本文利用ChenomxNmrSuite软件(Chenomx Inc.,Edmonton,Canada),结合我们课题组已发表的文献数据[12]及HMDB数据库(http://www.hmdb.ca/)对特征性代谢物进行信号归属和指认.代谢通路分析利用网站服务器MetaboAnalyst(https://www.metaboanalyst.ca/MetaboAnalyst/faces/home.xhtml)进行.
2 结果与讨论 2.1 胶质瘤细胞株SIRT7基因的表达水平与增殖能力检测我们首先检测了Sh7和ShC两种细胞株中SIRT7基因在蛋白和mRNA水平的表达情况,并在传代培养24 h、48 h、72 h时检测了两种细胞的增殖能力.如图 1所示,shRNA-SIRT7质粒转染可以显著降低胶质瘤细胞中SIRT7基因在蛋白[图 1(a)]和mRNA水平[图 1(b)]的表达量.MTS增殖实验[图 1(c)]结果证实,SIRT7基因低表达的胶质瘤细胞株的增殖能力弱于对照组;至传代培养48 h时,两组细胞的增殖能力已存在显著的统计学差异(p < 0.05).因此,根据细胞生长特点,后续代谢组学实验选取传代培养48 h的细胞样品进行分析.
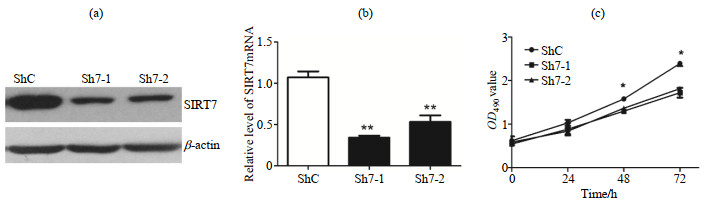
|
图 1 Sh7和ShC两种细胞株中SIRT7基因在蛋白和mRNA水平的表达情况,以及增殖能力的比较. (a) Western blot检测SIRT7蛋白表达水平;(b) RT-PCR检测SIRT7mRNA表达水平;(c) MTS实验检测细胞增殖能力 Fig. 1 Comparison of SIRT7 protein expression level, SIRT7mRNA expression level and cell proliferation between Sh7 and ShC cell lines. (a) Western blot analysis of SIRT7 protein expression level; (b) RT-PCR analysis of SIRT7mRNA expression level; (c) MTS analysis of cell proliferation |
为了明确SIRT7基因低表达是否引起胶质瘤细胞代谢改变,我们对传代培养48 h的低表达SIRT7基因组(Sh7)和对照组(ShC)的细胞水溶性萃取液进行1H NMR谱图采集与分析.获得谱图(论文网络版图S1)后,我们首先进行无监督的PCA分析.由PCA得分图(图 2)可知,Sh7与ShC细胞株的代谢模式存在明显差异.在PCA分析之后,又通过有监督的PLS-DA进一步验证PCA结果的可靠性.由PLS-DA得分图[图 3(a)]可知,在以第一主成分和第二主成分构成的空间(tp1/tp2)里,Sh7和ShC可以较好区分,而且RPT结果[图 3(b)]表明二分类模型没有出现过拟合现象,具有很好的数据预测能力.以上结果说明,与ShC对照组相比,Sh7细胞株的代谢模式发生了明显变化.
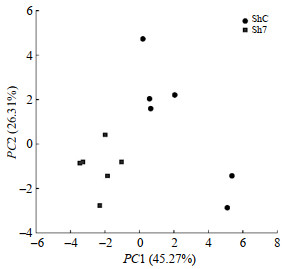
|
图 2 Sh7和ShC细胞水溶性萃取液主成分分析(PCA)的得分图 Fig. 2 PCA scores plot of aqueous metabolites extraction from Sh7 and ShC cell lines |
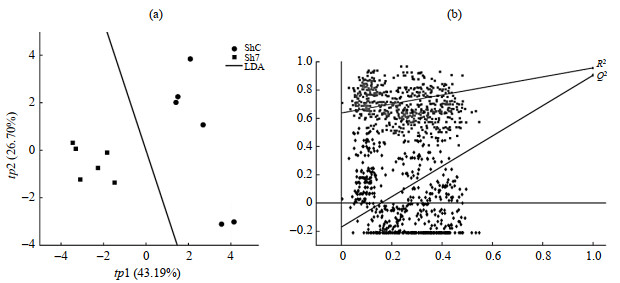
|
图 3 Sh7和ShC细胞水溶性萃取液的PLS-DA分析. (a) PLS-DA得分图,LDA表示线性判别分析;(b)置换排列验证实验 Fig. 3 The PLS-DA results of aqueous extracts from Sh7 and ShC cell lines. (a) The PLS-DA scores plot, LDA represents linear discriminant analysis; (b) Response permutation test |
我们进一步通过OPLS-DA得分图与载荷图(图 4),对区分两组细胞代谢模式有贡献的特征性代谢物进行了筛选,共发现22个特征性代谢物(表 1).进一步以这些特征性代谢物在各组样品中的归一化后的积分值作为其相对浓度进行t检验分析,结果与载荷图分析一致.与对照组相比,SIRT7基因低表达胶质瘤细胞株中含量增加的代谢物有12种:丁酸盐(butyrate)、乳酸(lactate)、丙氨酸(alanine)、谷氨酸(glutamate)、谷氨酰胺(glutamine)、谷胱甘肽(glutathione)、肌酸(creatine)、磷酸肌酸(creatine phosphate)、牛磺酸(taurine)、甘氨酸(glycine)、腺嘌呤核糖核苷酸(AMP)、延胡索酸盐(fumarate);含量降低的代谢物有10种:缬氨酸(valine)、亮氨酸(leucine)、异亮氨酸(isoleucine)、赖氨酸(lysine, )、乙酰胺(acetamide)、天冬氨酸(aspartate)、磷酸胆碱(PC)、酪氨酸(tyrosine)、苯丙氨酸(phenylalanine)和甲酸盐(formate).

|
图 4 Sh7和ShC细胞水溶性萃取液的OPLS-DA得分图(左)与载荷图(右).左图中LDA表示线性判别分析.右图中红色表示差异最为显著(|r| > 0.661且VIP > 1);橘色表示差异显著(0.532 < |r| < 0.661且VIP > 1);蓝色表示差异不显著(NS) Fig. 4 The OPLS-DA scores plot (left) and the coefficients-coded loadings plot (right) of aqueous metabolites extraction from Sh7 and ShC cell lines. LDA (left) represents linear discriminant analysis. The red color (right) indicates that the variables are very significant (|r| > 0.661 and VIP > 1); orange color indicates that the variables are significant (0.532 < |r| < 0.661 and VIP > 1); blue color indicates that the variables are insignificant (NS) |
| 表 1 特征性代谢物的相关信息 Table 1 Characteristic metabolites information |
这些与SIRT7基因低表达相关的特征性代谢物中有很多是已报道的胶质瘤恶性表型相关的标志性代谢物.有文献[13, 14]报道,高恶性程度胶质瘤细胞中乳酸含量降低与细胞维持高侵袭能力和抗凋亡能力相关.SIRT7基因低表达胶质瘤细胞的增殖和侵袭能力减弱[11],反映在代谢变化上可能会发生乳酸含量增加.我们的组学分析结果证实了这一推测.此外,高恶性程度胶质瘤细胞中牛磺酸含量降低很可能与其抗凋亡和抗氧化功能增强有关[15].SIRT7基因低表达胶质瘤细胞中的牛磺酸含量增加.推测SIRT7基因低表达可能会影响细胞氧化应激代谢途径进而影响胶质瘤细胞的增殖和侵袭能力.肌酸是细胞能量存储和利用的重要化合物.其浓度在能量代谢减退时增加,在能量代谢增加时降低.高恶性程度胶质瘤细胞中肌酸含量呈现降低趋势,而SIRT7基因低表达胶质瘤细胞中肌酸含量增加,这很可能与其能量代谢改变有关.综上,癌基因SIRT7基因低表达抑制胶质瘤细胞增殖和侵袭等恶性特性的同时,也使得肿瘤细胞内一些恶性程度相关的代谢物含量发生改变.
2.4 代谢通路富集分析单一分析每种代谢物浓度的改变,很难全面反映细胞整体代谢特征的变化.为了更好地研究SIRT7基因低表达胶质瘤细胞株代谢特征的全貌,我们利用网站服务器上系统生物学分析软件MetaboAnalyst对所获得的细胞水溶性萃取物中的代谢物信息进行整合和通路富集分析[16, 17],结果如图 5所示.
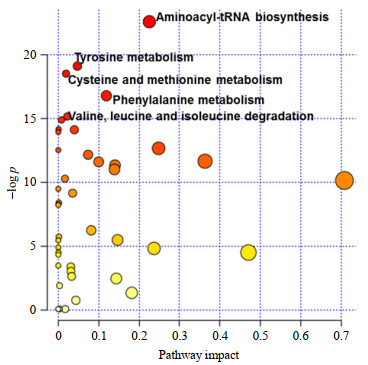
|
图 5 代谢通路富集分析 Fig. 5 Pathway enrichment analysis |
通过对特征性代谢物的富集分析,我们发现SIRT7基因低表达对胶质瘤细胞代谢的影响集中在氨酰-tRNA生物合成、酪氨酸代谢、半胱氨酸和蛋氨酸代谢、苯丙氨酸代谢、支链氨基酸降解代谢等代谢通路上.其中SIRT7基因低表达与氨酰-tRNA生物合成代谢通路的相关性(论文网络版附件表S1)最高.有文献[4]报道,SIRT7基因定位于核仁,SIRT7基因表达水平与核糖体合成密切相关.而众所周知核糖体的合成促进蛋白质的翻译.蛋白质翻译过程中氨基酸的活化与搬运离不开氨酰-tRNA的作用.此外,细胞核不仅是核糖体作用的主要场所,也是监测细胞应激信号的重要场所.另外,通路富集分析显示SIRT7基因低表达对多种氨基酸代谢通路都有显著影响,这种影响很可能与SIRT7蛋白的去乙酰化酶活性相关.有研究[18]表明,SIRT7基因表达可以调节细胞内质网应激及线粒体能量代谢.研究者发现SIRT7蛋白可以去乙酰化线粒体中心调节子GABPβ1,影响其转录活性,调控细胞能量代谢.通过对SIRT7基因低表达胶质瘤细胞株代谢特征的分析,我们发现代谢组学分析结果与SIRT7基因功能研究结果具有潜在的一致性.为了进一步阐明SIRT7表达对胶质瘤细胞代谢的调控机制,后续我们还将开展关键性代谢酶的检测以及代谢通路验证等相关工作.
3 结论我们利用基于NMR的代谢组学方法分析了SIRT7基因低表达胶质瘤细胞株的代谢特征,指认了22种与癌基因SIRT7表达有相关性的特征性代谢物,其中包括乳酸、牛磺酸等胶质瘤恶性表型相关的标志性代谢物.代谢组学分析结果显示胶质瘤细胞中癌基因SIRT7表达很可能是通过调控氨酰-tRNA生物合成、酪氨酸代谢、半胱氨酸和蛋氨酸代谢、苯丙氨酸代谢、支链氨基酸降解代谢等代谢通路从而影响胶质瘤细胞的增殖、侵袭等恶性表型.上述研究结果为进一步阐明癌基因SIRT7调控胶质瘤细胞代谢的作用机制提供了理论依据.
| [1] | COLLER H A. Is cancer a metabolic disease?[J]. Am J Pathol, 2014, 184(1): 4-17. |
| [2] | CHALKIADAKI A, GUARENTE L. The multifaceted functions of sirtuins in cancer[J]. Nat Rev Cancer, 2015, 15(10): 608-624. DOI: 10.1038/nrc3985. |
| [3] | VOELTER-MAHLKNECHT S, LETZEL S, MAHLKNECHT U. Fluorescence in situ hybridization and chromosomal organization of the human Sirtuin 7 gene[J]. Int J Oncol, 2006, 28(4): 899-908. |
| [4] | MICHISHITA E, PARK J Y, BURNESKIS J M, et al. Evolutionarily conserved and nonconserved cellular localizations and functions of human SIRT proteins[J]. Mol Biol Cell, 2005, 16(10): 4623-4635. DOI: 10.1091/mbc.e05-01-0033. |
| [5] | VAKHRUSHEVA O, SMOLKA C, GAJAWADA P, et al. Sirt7 increases stress resistance of cardiomyocytes and prevents apoptosis and inflammatory cardiomyopathy in mice[J]. Circ Res, 2008, 102(6): 703-710. DOI: 10.1161/CIRCRESAHA.107.164558. |
| [6] | TONG Z, WANG Y, ZHANG X Y, et al. SIRT7 is activated by DNA and deacetylates histone H3 in the chromatin context[J]. ACS Chem Biol, 2016, 11(3): 742-747. DOI: 10.1021/acschembio.5b01084. |
| [7] | LI L, SHI L, YANG S D, et al. SIRT7 is a histone desuccinylase that functionally links to chromatin compaction and genome stability[J]. Nat Commun, 2016, 7: 12235. DOI: 10.1038/ncomms12235. |
| [8] | LEE N, KIM D K, KIM E S, et al. Comparative interactomes of SIRT6 and SIRT7:Implication of functional links to aging[J]. Proteomics, 2014, 14(13, 14): 1610-1622. |
| [9] | ZHANG S, CHEN P, HUANG Z A, et al. Sirt7 promotes gastric cancer growth and inhibits apoptosis by epigenetically inhibiting miR-34a[J]. Sci Rep, 2015, 5: 9787. DOI: 10.1038/srep09787. |
| [10] | LI D, LI L F. MicroRNA3666 inhibits breast cancer cell proliferation by targeting sirtuin 7[J]. Mol Med Rep, 2017, 16(6): 8493-8500. DOI: 10.3892/mmr.2017.7603. |
| [11] | MU P F, LIU K, LIN Q Y, et al. Sirtuin 7 promotes glioma proliferation and invasion through activation of the ERK/STAT3 signaling pathway[J]. Oncol Lett, 2019, 17(2): 1445-1452. |
| [12] |
SHAO W, GU J P, HUANG C H, et al. Metabolic profiles of CHG5 and U87 glioma cell lines derived by 1H NMR[J].
Chinese J Magn Reson, 2014, 31(1): 40-48.
邵巍, 顾金苹, 黄彩华, 等. NMR分析胶质瘤细胞系CHG5和U87的代谢轮廓[J]. 波谱学杂志, 2014, 31(1): 40-48. DOI: 10.3969/j.issn.1000-4556.2014.01.005. |
| [13] | COLEN C B, SHEN Y M, GHODDOUSSI F, et al. Metabolic targeting of lactate efflux by malignant glioma inhibits invasiveness and induces necrosis:an in vivo study[J]. Neoplasia, 2011, 13(7): 620-632. DOI: 10.1593/neo.11134. |
| [14] | SHAO W, GU J P, HUANG C H, et al. Malignancy-associated metabolic profiling of human glioma cell lines using 1H NMR spectroscopy[J]. Mol Cancer, 2014, 13: 197. DOI: 10.1186/1476-4598-13-197. |
| [15] | LI T, LIU L B, WU H L, et al. Anti-herpes simplex virus type 1 activity of Houttuynoid A, a flavonoid from Houttuynia cordata Thunb[J]. Antiviral Res, 2017, 144: 273-280. DOI: 10.1016/j.antiviral.2017.06.010. |
| [16] | GOEMAN J J, VAN DE GEER S A, DE KORT F, et al. A global test for groups of genes:testing association with a clinical outcome[J]. Bioinformatics, 2004, 20: 93-99. DOI: 10.1093/bioinformatics/btg382. |
| [17] | XIA J, WISHART D S. MetPA:A web-based metabolomics tool for pathway analysis and visualization[J]. Bioinformatics, 2010, 26(18): 2342-2344. DOI: 10.1093/bioinformatics/btq418. |
| [18] | SUN L H, FAN G L, SHAN P P, et al. Regulation of energy homeostasis by the ubiquitin-independent REGgamma proteasome[J]. Nat Commun, 2016, 7: 12497. DOI: 10.1038/ncomms12497. |
 2019, Vol. 36
2019, Vol. 36 
