2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
细胞色素c是一个位于线粒体内外膜之间的血红蛋白,不仅在呼吸链中起电子传递作用[1-3],而且在细胞凋亡中扮演着重要角色,其发挥相应功能的分子机制研究广受关注[3-5].在氧化态与还原态的转变过程中,细胞色素c将电子从细胞色素c还原酶传递到细胞色素c氧化酶[2, 6].在受到凋亡刺激后,细胞色素c从线粒体被释放至细胞质,参与形成凋亡小体,凋亡小体激活半胱天冬酶,半胱天冬酶激活效应胱门蛋白酶,最后导致细胞凋亡.
细胞色素c是一个典型的多构象蛋白,而且是广为使用的蛋白质折叠和结构/功能关系研究的模型蛋白[2].它在发挥功能的过程中通常伴随着多种构象转换,并且其构象易受环境、与配体的相互作用、温度等多种因素的影响[7-9].在自然态,细胞色素c的Heme基团通过两个硫醚键与蛋白的两个半胱氨酸(Cys)残基C14、C17的支链相连[10].在发生氧化还原构象转变时,Heme基团中心的铁离子在二价与三价之间转换[11],同时,与周围基团支链的键合发生变化,配位的变化使得氧化态的细胞色素c的结构相比还原态具有更高的柔性[12].细胞色素c与心磷脂结合时,其构象由紧凑变得更为舒展[13-16],导致细胞色素c从线粒体内膜释放至细胞质,再与细胞凋亡因子Apaf-1结合,促使细胞凋亡[17, 18].
总而言之,细胞色素c的构象变化与其功能密切相关,研究其构象变化对于我们了解其功能机制至关重要.表征细胞色素c蛋白构象的方法包括X射线、光谱技术、冷冻电镜等[19].而核磁共振(NMR)是在原子水平反映蛋白多时间尺度动力学变化的理想工具[20, 21].NMR表征蛋白构象经常进行同位素标记,如13C或者15N标记,并采用2D NMR谱(如1H-15N HSQC谱图)使信号分散,以观测蛋白的构象变化[22].但同位素标记的NMR技术的应用受到很多的限制:首先,很多蛋白过量表达困难,标记成本高;其次,很多翻译后修饰蛋白在过量表达后,往往失去了其应有的翻译后修饰,因而可能会对其构象或者相互作用造成影响,如野生型酿酒酵母iso-1细胞色素c蛋白[23],其甲基化修饰在常用的原核体系E. Coli表达时无法实现.
基于甲基的NMR检测技术是研究大分子量蛋白质结构及动力学的理想工具.由于13C核的旋磁比高于15N核,且甲基具有较高的质子浓度,通常处在分子表面,而且较强的柔性使其通常具有较长的横向弛豫时间(T2),因此通常在天然丰度下就可获得1H-13C相关信号.目前,天然同位素丰度下的基于甲基1H-13C相关的NMR波谱已被用于一些蛋白质的结构检测,如单克隆抗体的评估等[24].
在本研究中,我们尝试采用1H-13C HSQC NMR技术观测支链甲基的变化,表征天然同位素丰度下野生型酵母细胞色素c(yeast iso-1 cytochrome c,y1 Cytc)的构象变化.为了准确归属野生型y1 Cytc的支链甲基信号,我们首先利用13C/15N标记的突变体细胞色素c[y1 Cytc (K72A/C102T)],对氧化和还原态细胞色素c的支链甲基进行了归属.与野生型相比,该突变体更加稳定,K72位的突变使其受pH的影响更小[25],C102位的突变有助于避免二聚[26],因而便于信号采集和归属.然后,我们通过信号比对,对天然丰度野生型细胞色素c的氧化态与还原态的支链甲基进行了归属.最后,通过监测甲基信号在氧化还原过程中的变化过程,我们对野生型细胞色素c的氧化还原构象变化进行了表征,其结果与突变体蛋白的氧化还原构象变化一致,这表明该方法可应用于天然丰度蛋白质的结构变化表征.由于野生型细胞色素c存在翻译后修饰,因此表征野生型细胞色素c的构象变化更具有实际意义.
1 实验部分 1.1 实验试剂无水磷酸二氢钠(NaH2PO4)、无水磷酸氢二钠(Na2HPO4)、铁氰化钾K3[Fe(CN)6]、EDTA-2Na购于国药集团化学试剂有限公司,Tirs购于Baitg公司,溶菌酶(Lysozyme)购于Biosharp公司,核酸内切酶(DNase)购于Thermo Scientific科技有限公司,重水(D2O)购于Sigma-Aldrich公司,TECP购于Aladdin生化科技股份有限公司.
1.2 突变体y1 Cytc (K72A/C102T)的表达纯化天然丰度的突变体y1 Cytc (K72A/C102T)表达操作流程如下[26]:将质粒转入到大肠杆菌BL21(DE3)感受态细胞,涂至氨苄平板中,37 ℃条件培养8 h,挑单接种至10 mL含100 μg/mL氨苄的TB培养基中,37 ℃、220 rpm条件下培养约12 h;再将菌液全部转入1 L含100 μg/mL氨苄的TB培养基中,37 ℃、220 rpm条件下培养36 h;然后在4 ℃、6 000 rpm条件下离心15 min(Beckman JA-10转子)收集沉淀后的菌体.纯化过程如下[27]:菌体沉淀重悬于含5 mmol/L EDTA的50 mmol/L Tris(pH 7.5)缓冲液中,加入溶菌酶(1.5 mg/g湿菌体)与微量DNase消化1 h,使用高压破菌仪破碎,细菌裂解液在20 000 rpm、4 ℃下(Beckman JA-25.5转子)离心30 min收集上清.于上清中慢慢加入150 g/L的(NH4)2SO4晶体,盐析3~6 h,20 000 rpm、4 ℃下(Beckman JA-25.5转子)离心30 min收集上清.上清于20 mmol/L PB缓冲液(pH 7.0)中透析过夜.透析后的样品通过0.22 μm超滤膜过滤后,上样至用缓冲液A(20 mmol/L PB,pH 7.0)平衡后的SP阳离子柱,用20%~80%线性梯度的缓冲液B(缓冲液A+1 mol/L NaCl)洗脱.将得到的样品用3 k分子量蛋白浓缩管浓缩至5 mL以下,经0.22 μm超滤膜过滤后上样至用缓冲液C(缓冲液A+150 mmol/L NaCl)平衡后的pg75柱,使用缓冲液C洗脱,收集80 mL出峰位置的样品峰.最终纯化好的样品用去离子水(ddH2O)于3 k分子量蛋白浓缩管换洗3次,用冷冻干燥机冻干,置于-0 ℃冰箱待用.
13C/15N双标的突变体y1 Cytc (K72A/C102T)的表达如下[23, 26]:将质粒转入到大肠杆菌BL21(DE3)感受态细胞,涂至氨苄平板中,37 ℃生长8 h,挑单菌落接种至5 mL含100 μg/mL氨苄的TB培养基中,37 ℃、220 rpm的摇床中培养约8 h;6 000 rpm,4 ℃(Beckman JA-10转子)离心10 min收集沉淀后的菌体,菌体用含100 μg/mL氨苄M9培养基(其中1 L M9含1 g 15N标记的氯化铵和5 g 13C标记的葡萄糖)重悬,转入50 mL含100 μg/mL氨苄的M9培养基中,37 ℃、220 rpm条件下培养8 h;再转入500 mL含100 μg/mL氨苄的M9培养基中,37 ℃、220 rpm条件下培养24 h;最后于4 ℃、6 000 rpm条件下离心15 min(Beckman JA-10转子)收集菌体.M9培养基组成与参考文献[23]所用一致.后续纯化过程同天然丰度的y1 Cytc (K72A/C102T).
1.3 野生型y1 Cytc的表达纯化野生型y1 Cytc购于Sigma-Aldrich公司,在实验之前经SP阳离子柱与pg75柱纯化,纯化条件同上所述.
1.4 NMR实验为了使蛋白质处于稳定的氧化或者还原态,额外的氧化剂和还原剂分别被加入至NMR缓冲溶液中,使蛋白维持不同的状态,便于信号归属.氧化态野生型y1 Cytc和突变体y1 Cytc (K72A/C102T)的缓冲液为:50 mmol/L PB,10 mmol/L K3[Fe(CN)6],10% D2O,pH 7.0.还原态的野生型y1 Cytc和突变体y1 Cytc (K72A/C102T)的缓冲液为:50 mmol/L PB,10 mmol/L TECP,10% D2O,pH 7.0.
氧化态与还原态13C/15N双标的突变体y1 Cytc (K72A/C102T)的NMR谱图均在配备了5 mm HCN超低温探头的Bruker Avance Ⅲ 600 MHz NMR谱仪上采集,实验温度为298 K.主链归属实验包括2D 1H-15N HSQC、3D HNCA、HNCOCA、HNCACB、CBCA(CO)NH和HBHA(CO)NH.侧链归属实验包括2D 1H-13C HSQC(aliphatic and aromatic)、3D(H)CCH-TOCSY.具体采样参数见表 1.
| 表 1 13C/15N双标的突变体y1Cytc (K72A/C102T) NMR实验采样条件 Table 1 NMR experimental parameters for the assignments of 13C/15N double-labeled y1 Cytc (K72A/C102T) |
为了获得更高的灵敏度,天然同位素丰度的野生型y1 Cytc和突变体y1 Cytc (K72A/C102T)的1H-13C HSQC谱图在Bruker Avance Ⅲ 850 MHz NMR谱仪上采集,实验温度为298 K,1H与13C维的谱宽分别为20 ppm和50 ppm,采样点数为2 048×128,累加次数为32.
1.5 NMR数据处理NMR数据使用NMRpipe软件处理[28],采用Sparky软件进行分析.主链及侧链信息通过PINE服务器(http://pine.nmrfam.wisc.edu)自动归属,加以人工校正完成.
2 结果与讨论 2.1 突变体y1 Cytc (K72A/C102T)侧链甲基归属对于突变体y1 Cytc (K72A/C102T)氧化态和还原态蛋白,我们分别获得了50个支链甲基中的38个和41个甲基的归属,详见表 2.与高等真核生物Cytc相比,y1 Cytc的N端含有5个额外残基,这里编号为-5至-1[29].可能是因为顺磁效应的影响,我们得到的氧化态蛋白支链的归属信息少于还原态.Fe2+、Fe3+的外层电子排布分别是3d6、3d5,未成对电子数分别是4、5,其中有4个电子于Heme基团中参与配位,故Heme基团中未成对电子数分别为0、1,自旋分别为0、1/2,故氧化态的Cytc具有强的顺磁性.
| 表 2 13C/15N双标的突变体y1 Cytc (K72A/C102T)的侧链甲基归属 Table 2 The assignments of the side-chain methyl groups of the mutant y1 Cytc (K72A/C102T) double-labeled by 13C/15N |
从表 2中可以看出,多数y1 Cytc (K72A/C102T)侧链的甲基在氧化还原过程中无明显的化学位移变化,表明该蛋白在氧化态和还原态之间变化时,其主要结构保持不变.但仍有一些基团发生了较大的化学位移变化,如L9、L15、L58、L85、L94、和L98的δ甲基,A51、A72的β甲基,V28、V57的γ甲基,I35、I53、I75的δ和γ甲基,和T78、T96、T102的γ甲基.这些基团的13C维化学位移变化均超过0.3 ppm,1H维化学位移变化均超过0.2 ppm,表明上述基团所处位置的结构在氧化还原过程中可能发生了构象转变.
2.2 野生型的y1 Cytc的侧链甲基归属天然同位素丰度野生型y1 Cytc侧链甲基归属主要是通过与天然丰度的突变体y1 Cytc (K72A/C102T)蛋白的1H-13C HSQC谱图化学位移比对完成(图 1).其中天然丰度的突变体的1H-13C HSQC谱图归属是通过与表 2的归属信息比对完成.从图 1看出:野生型y1 Cytc与其突变体y1 Cytc (K72A/C102T)的多数谱峰化学位移基本相同,表明其主要结构可能没有大的变化.但是野生型蛋白的部分信号,特别是氧化态蛋白在δC 40处的亚甲基信号和突变体信号相比变化明显:野生型蛋白质的信号分散度和强度较为均一,信号数量明显少于突变体;而突变体蛋白质的信号不仅数量多,而且强弱差异明显,说明可能有部分低丰度构象存在.野生型y1 Cytc与突变体y1 Cytc (K72A/C102T)的最大差别有两点:首先,野生型蛋白的72位赖氨酸是三甲基化修饰的;其次,野生型蛋白的102位是半胱氨酸.这些特殊修饰或者基团的存在可能会引发相应的结构或者构象发生变化.这一结果表明突变可能会造成部分结构的改变,因此,采用天然提纯的蛋白质直接作为检测对象可能更为准确的反应蛋白质的构象变化.
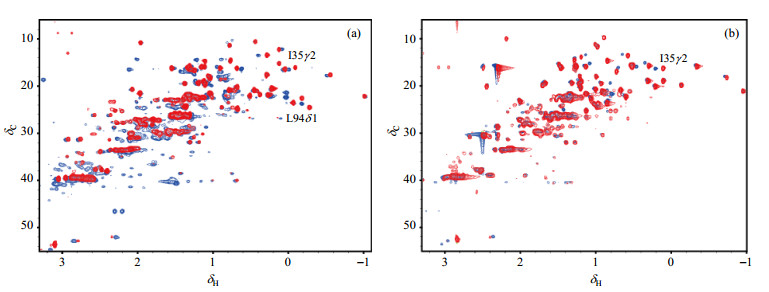
|
图 1 (a) 氧化态和(b)还原态的天然丰度的野生型y1 Cytc(红色)与突变体y1 Cytc (K72A/C102T)(蓝色)的1H-13C HSQC谱图 Fig. 1 Overlay of 2D 1H-13C HSQC spectra of wild type y1 Cytc (red) and mutant y1 Cytc (K72A/C102T) (blue) under natural abundance in (a) oxidized state and (b) reduced state |
图 2为野生型y1 Cytc的侧链甲基谱图及部分主要残基甲基信号的归属.我们的实验结果发现,与亚甲基、次甲基相比,甲基的谱线分散度好,且谱线窄、信号强.在氧化和还原态转换过程中,甲基分布在分子表面,其化学位移对环境具有较高的敏感性,很多甲基的化学位移变化明显.与之相对,亚甲基区域信号重叠严重,信号识别和指认困难.此外,我们所采集的是天然丰度蛋白质的谱图,所用蛋白浓度为2 mmol/L,二维1H-13C HSQC谱的采集时间小于1 h,表明基于甲基的NMR检测具有足够灵敏度,是表征天然同位素丰度蛋白质的构象和与配体相互作用的理想探针.当然,需要指出的是该实验的灵敏度和同位素标记的蛋白质相比仍然较低,因此在后续应用中还需要进一步优化,比如结合其他快速采样技术,如SOFAST和非均匀采样等,以便在更短的时间内实现相应谱线的采集,进而实现天然同位素丰度蛋白更短时间尺度构象转变过程的检测,获得更为广泛的应用.
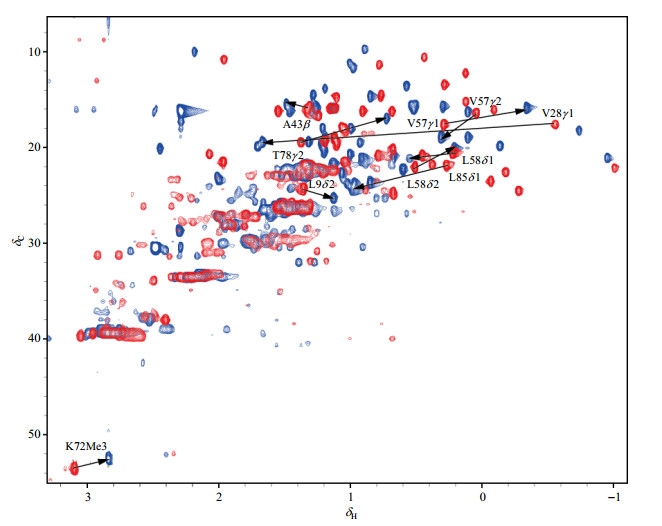
|
图 2 天然丰度的氧化态(红色)与还原态(蓝色)的野生型y1 Cytc的1H-13C HSQC谱图 Fig. 2 Overlay of 2D 1H-13C HSQC spectra of wild type y1 Cytc at natural abundance in oxidized (red) and reduced (blue) states, respectively |
另外,从图 2还可以看出:修饰甲基的信号分布特殊,位于δC 52处,该区域是支链次甲基信号分布范围,由于次甲基信号质子数少,弛豫速率较甲基快,因此相应信号的灵敏度远低于甲基.次甲基等信号的弱化使得修饰甲基和其他甲基相比,背景更少,信号识别更为容易;而且,修饰甲基在氧化和还原态之间转换时,其化学位移变化明显,表明该基团可被用于相应蛋白质构象变化的观测和分析;另外,氧化态蛋白质的修饰甲基信号不仅和其还原态有明显区别,其信号附近还有个较弱的信号,可能是新的低分布构象.由于蛋白质在突变以及同位素标记表达时无法实现翻译后修饰,因此在突变及同位素标记可能会对天然提纯的野生型蛋白质的真实构象变化产生影响,而用天然提纯蛋白质来表征相应的构象转换可更加准确的反映蛋白质本身的构象变化.
从图 2还可以看出化学位移发生较大变化的支链甲基主要为A43的β、L9的δ 2、V28的γ1、V57的γ 1、V57的γ 2、L58的δ 1、L58的δ 2、T78的γ 2、L85的δ 1甲基,以及K72的翻译后修饰甲基.以上残基在y1 Cytc结构中所处的位置如图 3所示.从图 3中可以看出,发生构象变化的残基主要集中在靠近Heme基团的一些loop区,以及N端的α螺旋,这一结果与文献[25]发现相符.有关文献[30]表明,y1 Cytc的构象变化主要与其Heme基团的配位变化有关,还原态状态下Heme基团分别与M80和H18于轴向配位,形成氧化态时,M80则由K73/79取代,从而造成相应的构象变化,因此其结构变化通常发生在含有K73和M80的70~85这段Ω-loop区域,这与我们的实验结果相匹配,即L85和T78的支链甲基化学位移均发生较大的变化.另外,我们还发现L9基团的化学位移变化明显,L9位于C端,没有和Heme配位,其化学位移变化可能是由于Heme配位改变,引发蛋白质整体结构松散程度变化造成的,这与氧化和还原态在该结构区域的变化相符合.此外,在细胞色素c从还原态到氧化态的过程中,由Fe2+到Fe3+产生的顺磁效应也会导致Heme基团附近基团的化学位移的变化.研究表明,N端α螺旋对结构非常重要,其位置变化与很多的构象变化相关,如二聚等,并且其配位的改变通常会引发结构的整体变化,这与我们的观测相符合.
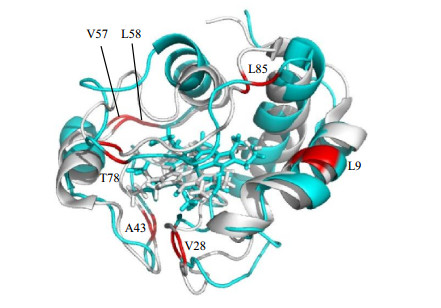
|
图 3 氧化态(灰色)与还原态(蓝色)y1 Cytc的结构(PDB号分别为2LIR、2LIT),及NMR检测到的可能参与构象变化的残基(红色) Fig. 3 The structure of oxidized (grey) and reduced (blue) y1 Cytc (PDB ID: 2LIR, 2LIT), and the residues detected those are involved in conformation transition (red) |
由于突变表达可能会对蛋白质的结构产生部分影响,如对于我们所研究的细胞色素c,如图 1所示,δH 0.30与δC 20.0附近,部分基团如I35 γ 2、L94 δ 1化学位移的变化与突变体存在明显差异,因此用天然蛋白质直接进行结构表征具有重要的应用价值,我们的研究表明,基于甲基的NMR检测能够用于天然提纯蛋白质的构象变化研究.
3 结论我们利用多维NMR实验对突变体y1 Cytc (K72A/C102T)蛋白的侧链甲基进行了归属,进而通过1H-13C HSQC谱图的对比,对天然提纯的野生型y1 Cytc侧链甲基信号进行了归属,并对氧化-还原构象变化进行了分析,发现基于甲基检测的1H-13C HSQC实验可在较短的时间内获得天然同位素丰度蛋白质的谱图,所观测到的蛋白结构变化信息也与PDB结构图中信息相符合,表明甲基NMR信号采集是观测天然丰度蛋白质的理想探针,可用于难以表达的重要功能蛋白以及翻译后修饰蛋白的构象变化研究.
| [1] | BANCI L, BERTINI I, GRAY H B, et al. Solution structure of oxidized horse heart cytochrome c[J]. Biochem, 1997, 36(32): 9867-9877. DOI: 10.1021/bi970724w. |
| [2] | FETROW J S, BAXTER S M. Assignment of N-15 chemical shifts and N-15 relaxation measurements for oxidized and reduced iso-1-cytochrome c[J]. Biochem, 1999, 38(14): 4480-4492. DOI: 10.1021/bi9827417. |
| [3] | KALPAGE H A, BAZYLIANSKA V, RECANATI M A, et al. Tissue-specific regulation of cytochrome c by post-translational modifications:respiration, the mitochondrial membrane potential, ROS, and apoptosis[J]. Faseb J, 2019, 33(2): 1540-1553. DOI: 10.1096/fj.201801417R. |
| [4] | LIU X S, KIM C N, YANG J, et al. Induction of apoptotic program in cell-free extracts:Requirement for dATP and cytochrome c[J]. Cell, 1996, 86(1): 147-157. DOI: 10.1016/S0092-8674(00)80085-9. |
| [5] | ZAIDI S, HASSAN M I, ISLAM A, et al. The role of key residues in structure, function, and stability of cytochrome-c[J]. Cell Mol Life Sci, 2014, 71(2): 229-255. DOI: 10.1007/s00018-013-1341-1. |
| [6] | ALVAREZ-PAGGI D, HANNIBAL L, CASTRO M A, et al. Multifunctional cytochrome c:Learning new tricks from an old dog[J]. Chem Rev, 2017, 117(21): 13382-13460. DOI: 10.1021/acs.chemrev.7b00257. |
| [7] | OVIEDO-ROUCO S, CASTRO M A, ALVAREZ-PAGGI D, et al. The alkaline transition of cytochrome c revisited:Effects of electrostatic interactions and tyrosine nitration on the reaction dynamics[J]. Arch Biochem Biophys, 2019, 665: 96-106. DOI: 10.1016/j.abb.2019.02.016. |
| [8] | MUNEESWARAN G, KARTHEESWARAN S, MUTHUKUMAR K, et al. Temperature-dependent conformational dynamics of cytochrome c:Implications in apoptosis[J]. J Mol Graph Model, 2018: 79140-148. |
| [9] | DONG A, BROWN C, BAI S, et al. Acid-enhanced conformation changes of yeast cytochrome c coated onto gold nanoparticles, a FT-IR spectroscopic analysis[J]. Int J Biol Macromol, 2018: 112591-597. |
| [10] | DANIELSON T A, STINE J M, DAR T A, et al. Effect of an imposed contact on secondary structure in the denatured state of yeast iso-1-cytochrome c[J]. Biochem, 2017, 56(51): 6662-6676. DOI: 10.1021/acs.biochem.7b01002. |
| [11] | BAISTROCCHI P, BANCI L, BERTINI I, et al. Three-dimensional solution structure of Saccharomyces cerevisiae reduced iso-1-cytochrome c[J]. Biochem, 1996, 35(43): 13788-13796. DOI: 10.1021/bi961110e. |
| [12] | IMAI M, SAIO T, KUMETA H, et al. Investigation of the redox-dependent modulation of structure and dynamics in human cytochrome c[J]. Biochem Biophys Res Commun, 2016, 469(4): 978-984. DOI: 10.1016/j.bbrc.2015.12.079. |
| [13] | BARAYEU U, LANGE M, MENDEZ L, et al. Cytochrome c autocatalyzed carbonylation in the presence of hydrogen peroxide and cardiolipins[J]. J Biol Chem, 2019, 294(6): 1816-1830. DOI: 10.1074/jbc.RA118.004110. |
| [14] | MOHAMMADYANI D, YANAMALA N, SAMHAN-ARIAS A K, et al. Structural characterization of cardiolipin-driven activation of cytochrome c into a peroxidase and membrane perturbation[J]. BBA-Biomembranes, 2018, 1860(5): 1057-1068. DOI: 10.1016/j.bbamem.2018.01.009. |
| [15] | AMACHER J F, ZHONG F, LISI G P, et al. A compact structure of cytochrome c trapped in a lysine-ligated state:loop refolding and functional implications of a conformational switch[J]. J Am Chem Soc, 2015, 137(26): 8435-8449. DOI: 10.1021/jacs.5b01493. |
| [16] | HANSKE J, TOFFEY J R, MORENZ A M, et al. Conformational properties of cardiolipin-bound cytochrome c[J]. Proc Natl Acad Sci USA, 2012, 109(1): 125-130. DOI: 10.1073/pnas.1112312108. |
| [17] | KOBAYASHI H, NAGAO S, HIROTA S. Characterization of the cytochrome c membrane-binding site using cardiolipin-containing bicelles with NMR[J]. Angew Chem Int Ed Engl, 2016, 55(45): 14019-14022. DOI: 10.1002/anie.201607419. |
| [18] | QI H, JIANG Y, YIN Z Y, et al. Optimal pathways for the assembly of the Apaf-1.cytochrome c complex into apoptosome[J]. Phys Chem Chem Phys, 2018, 20(3): 1964-1973. DOI: 10.1039/C7CP06726G. |
| [19] | JIN J P. Protein structure, function, and regulation in biological movement[J]. Arch Biochem Biophys, 2006, 456(2): 99-101. DOI: 10.1016/j.abb.2006.11.017. |
| [20] | KAY L E. NMR studies of protein structure and dynamics-a look backwards and forwards[J]. J Magn Reson, 2011, 213(2): 492-494. DOI: 10.1016/j.jmr.2011.08.010. |
| [21] |
LI H, KAMATARI Y O, KITAHARA R, et al. High-pressure nmr for studying protein structure and dynamics[J].
Chinese J Magn Reson, 2016, 33(1): 1-26.
李华, KAMATARIY O, KITAHARAR, 等. 高压NMR在蛋白质结构和动力学研究中的应用[J]. 波谱学杂志, 2016, 33(1): 1-26. |
| [22] |
RAN M L, QIN L Y, TANG C, et al. Regulation of inter-protein interactions between ubiquitin and ubiquitin-associated domains in Rad23A/ubiquilin-1 by phosphorylation[J].
Chinese J Magn Reson, 2019, 36(1): 15-22.
冉梦琳, 覃凌云, 唐淳, 等. 磷酸化调控泛素单体与Rad23A/Ubiquilin-1中泛素结合域互作的检测[J]. 波谱学杂志, 2019, 36(1): 15-22. |
| [23] | MORAR A S, KAKOURAS D, YOUNG G B, et al. Expression of N-15-labeled eukaryotic cytochrome c in Escherichia coli[J]. J Biol Inorg Chem, 1999, 4(2): 220-222. DOI: 10.1007/s007750050307. |
| [24] | ARBOGAST L W, DELAGLIO F, TOLMAN J R, et al. Selective suppression of excipient signals in 2D H-1-C-13 methyl spectra of biopharmaceutical products[J]. J Biomol NMR, 2018, 72(3/4): 149-161. |
| [25] | LAN W X, WANG Z H, YANG Z Z, et al. Conformational toggling of yeast iso-1-cytochrome c in the oxidized and reduced states[J]. Plos One, 2011, 6(11). |
| [26] | SZABO C M, SANDERS L K, LE H C, et al. Expression of doubly labeled Saccharomyces cerevisiae iso-1 ferricytochrome c and H-1, C-13 and N-15 chemical shift assignments by multidimensional NMR[J]. Febs Letters, 2000, 482(1/2): 25-30. |
| [27] | POLLOCK W B R, ROSELL F I, TWITCHETT M B, et al. Bacterial expression of a mitochondrial cytochrome c. Trimethylation of Lys72 in yeast iso-1-cytochrome c and the alkaline conformational transition[J]. Biochem, 1998, 37(17): 6124-6131. DOI: 10.1021/bi972188d. |
| [28] | DELAGLIO F, GRZESIEK S, VUISTER G W, et al. NMR pipe-A multidimensional spectral processing system based on unix pipes[J]. J Biomol NMR, 1995, 6(3): 277-293. |
| [29] | LETT C M, ROSU-MYLES M D, FREY H E, et al. Rational design of a more stable yeast iso-1-cytochrome c[J]. BBA-Protein Struct M, 1999, 1432(1): 40-48. DOI: 10.1016/S0167-4838(99)00071-0. |
| [30] | SUN P, WANG Q W, YUAN B, et al. Monitoring alkaline transitions of yeast iso-1 cytochrome c at natural isotopic abundance using trimethyllysine as a native NMR probe[J]. Chem Comm, 2018, 54(89): 12630-12633. DOI: 10.1039/C8CC07605G. |
 2019, Vol. 36
2019, Vol. 36 
