2. 波谱与原子分子物理国家重点实验室, 武汉磁共振中心(中国科学院 精密测量科学与技术创新研究院), 湖北 武汉 430071;
3. 深圳市第二人民医院 医学影像科, 广东 深圳 518037;
4. 深圳大学总医院 医学影像科, 广东 深圳 518055
2. State Key Laboratory of Magnetic Resonance and Atomic and Molecular Physics, National Center for Magnetic Resonance in Wuhan(Innovation Academy for Precision Measurement Science and Technology, Chinese Academy of Sciences), Wuhan 430071, China;
3. Department of Radiology, The First Affiliated Hospital of Shenzhen University, Shenzhen 518037, China;
4. Department of Radiology, Shenzhen University General Hospital, Shenzhen 518055, China
前列腺癌是目前男性群体最常见的癌症之一.2018年,美国新增前列腺癌病例164 690例,死亡29 430例[1].1998年~2008年,中国的前列腺癌发病率也呈明显上升趋势[2, 3].有研究[4]预测至2030年,全球将会有1 700 000例前列腺癌患者.因此前列腺癌的早期检测对于增加前列腺癌患者的存活率和提高前列腺癌患者的生活质量至关重要.
当前前列腺癌的临床诊断方式主要包括前列腺特异性抗原(Prostate Specific Antigen,PSA)血清检测、直肠指检(Digital Rectal Examination,DRE)、经直肠超声检查(Transrectal Ultrasound,TRUS)、经直肠超声引导下穿刺活检和磁共振成像(Magnetic Resonance Imaging,MRI).其中,经直肠超声引导穿刺活检是目前诊断前列腺癌最可靠的方式.但这种传统的诊断方式不仅让患者十分痛苦,而且可能出现采样区域不是病变区域的情况[5, 6],进而延误病情治疗,或者出现过度诊断和治疗的情况.
MRI是一种无创、准确地检测前列腺癌的方式[7, 8].然而对磁共振图像的准确解读要求放射科医师必须具备大量的专业知识和丰富的经验,而且非常耗时.针对磁共振图像的自动化诊断能有效解决上述问题.卷积神经网络(Convolutional Neural Network,CNN)自20世纪90年代就被用于各类疾病的医学图像的诊断,例如肺结节X光图像的诊断[9, 10]、乳腺癌X光图像的诊断[11]等.近年来,深度CNN更在医学图像的分割和分类方面体现出了十分优异的效果,已有研究[12, 13]表明该方法可用于辅助前列腺癌诊断.
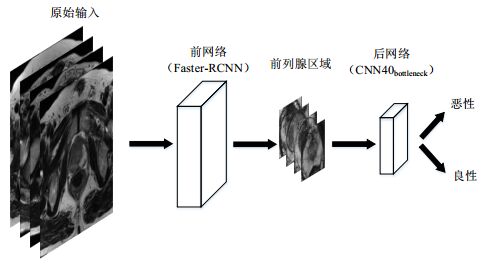
级联CNN可以实现在不同场景下的针对性训练,本文根据基于磁共振图像的前列腺癌临床诊断的步骤,采用级联CNN,以T2加权图像为数据集,实现对前列腺良性病变和恶性病变的分类.级联CNN分为前网络与后网络两个部分.前网络采用以VGG16[14]为骨架的Faster-RCNN[15],对磁共振图像进行分割.选择Faster-RCNN是因为它检测目标时具有相当高的准确率,同时其具有的区域候选网络(Region Proposal Network,RPN)结构可以勾勒出前列腺区域的锚盒,便于提取前列腺区域作为后网络输入.后网络以ResNet结构作为参考,通过瓶颈结构[16]串联组成,同时引入全局平均池化[17]和批量标准化层[18]增加传递效率,并减少参数量.
1 基于级联CNN的前列腺癌磁共振图像诊断原理用于诊断前列腺癌磁共振图像中除包含前列腺外,还有其他组织和器官,而病变区域主要集中在前列腺组织.若直接对磁共振图像进行训练,会受到其他区域信息的干扰.本文提出利用级联CNN对磁共振图像进行分析:前网络采用Faster-RCNN识别提取磁共振图像中的出前列腺区域,排除其他组织和器官对诊断的干扰;后网络使用基于ResNet-50改进的分类网络CNN40bottleneck对前列腺癌磁共振图像进行良恶性分类.基于级联CNN的前列腺癌磁共振图像诊断步骤见图 1所示.
|
图 1 基于级联CNN的前列腺癌磁共振图像诊断步骤 Fig. 1 Prostate cancer diagnosis based on cascaded CNN and magnetic resonance image |
前网络采用了Faster-RCNN网络[15](图 2)提取前列腺部分,该网络可以分为以下几个部分:(1)CNN,该部分使用VGG16网络实现特征图的提取;(2)RPN,该部分通过锚盒在特征图上滑动寻找可能包含目标的候选区域;(3)感兴趣区域池化,该部分对各种不同尺寸的候选区域做池化处理以统一尺寸;(4)softmax分类器,该部分对感兴趣区域池化部分输出的特征向量进行分类.经过实验验证,前网络对于前列腺区域的检测准确率可以达到接近100%.

|
图 2 Faster-RCNN原理图 Fig. 2 Schematic diagram of Faster-RCNN |
CNN隐藏层中参数的传递过程很多时候会造成协变量的偏移状况,这会使得不同卷积核的输出分布发生变化,导致梯度消失或者梯度爆炸,直观表现即包括过拟合等.批量标准化[18]的基本思想是通过算法将隐藏层输入拉回(0, 1)的正态分布,简称BN层,该层的优点有:(1)能够更好的拟合隐藏层间输入和设置较高的学习率,梯度传递更有效,学习时间减少;(2)不需要过多的调参,使网络可以适应不合适的初始参数;(3)BN层和激活层连接在一起,可以让输入值落在激活函数对输入比较敏感的区域,增加网络的非线性.算法的具体步骤如下:
步骤一:输入激活层的数据
步骤二:得到层输入均值
| ${\mu _B} = \frac{1}{n}\mathop \sum \limits_{i = 1}^n {x_i}$ | (1) |
步骤三:得到层输入方差:
| $\sigma _B^2 = \frac{1}{n}\mathop \sum \limits_{i = 1}^n {({x_i} - {\mu _B})^2}$ | (2) |
步骤四:将输入数据进行规范:
| ${\hat x_i} = \frac{{{x_i} - {\mu _B}}}{{\sqrt {\sigma _B^2 + \varepsilon } }}$ | (3) |
步骤五:对
| $\frac{{\partial l}}{{\partial {{\hat x}_i}}} = \frac{{\partial l}}{{\partial {y_i}}} \cdot \gamma $ | (4) |
| $\frac{{\partial l}}{{\partial \sigma _B^2}} = \mathop \sum \limits_{i = 1}^n \frac{{\partial l}}{{\partial {{\hat x}_i}}} \cdot ({x_i} - {\mu _B}) \cdot \frac{{ - 1}}{2}{(\sigma _B^2 + \varepsilon )^{ - \frac{3}{2}}}$ | (5) |
| $\frac{{\partial l}}{{\partial {\mu _B}}} = \left( {\mathop \sum \limits_{i = 1}^n \frac{{\partial l}}{{\partial {{\hat x}_i}}} \cdot \frac{{ - 1}}{{\sqrt {\sigma _B^2 + \varepsilon } }}} \right) + \frac{{\partial l}}{{\partial \sigma _B^2}} \cdot \frac{{\sum\limits_{i = 1}^n { - 2({x_i} - {\mu _B})} }}{n}$ | (6) |
| $\frac{{\partial l}}{{\partial {x_i}}} = \frac{{\partial l}}{{\partial {{\hat x}_i}}} \cdot \frac{1}{{\sqrt {\sigma _B^2 + \varepsilon } }} + \frac{{\partial l}}{{\partial \sigma _B^2}} \cdot \frac{{2({x_i} - {\mu _B})}}{n} + \frac{{\partial l}}{{\partial {\mu _B}}} \cdot \frac{1}{n}$ | (7) |
| $\frac{{\partial l}}{{\partial \gamma }} = \mathop \sum \limits_{i = 1}^n \frac{{\partial l}}{{\partial {y_i}}} \cdot {\hat x_i}$ | (8) |
| $\frac{{\partial l}}{{\partial \beta }} = \mathop \sum \limits_{i = 1}^n \frac{{\partial l}}{{\partial {y_i}}}$ | (9) |
其中l为层间传递梯度,n为数据维度.
步骤六:输出:
| ${y_i} = \gamma {\hat x_i} + \beta = B{N_{\gamma \cdot \beta }}({x_i})$ | (10) |
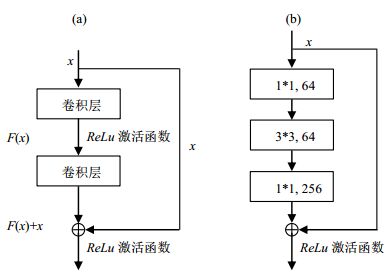
残差结构[16]如图 3(a)所示,x表示输入,
|
图 3 (a) 残差学习;(b)瓶颈结构 Fig. 3 (a) Residual learning; (b) Bottleneck |
全局平均池化[17]如图 4(a)所示,该操作是将最后一层卷积层输出的n张特征图分别求平均值,将均值作为特征参数放进分类器进行分类.全连接如图 4(b)所示,是将最后的一层卷积层输出的特征图中每一个点投射到一维向量上,输入到全连接层中.使用全局平均池化的网络参数量比使用全连接的网络少,同时不易出现过拟合.

|
图 4 (a) 全局平均池化;(b)全连接 Fig. 4 (a) Global average pooling; (b) Fully connected layer |
Inception[19]是2014年由Szegedy提出的网络结构,初次使用于GoogLeNet.Inception首先通过不同大小的卷积核分别对特征图进行特征提取,然后合并特征结构.这种结构能够避免选择卷积核大小的过程,同时能从不同大小的卷积核获取不同的特征,结合不同大小卷积层的特征能够进一步得到更好的特征,从而提高准确度.Inception-V1的结构如图 5所示,由1*1、3*3或5*5三个大小卷积核提取特征后,合并特征进行判断.Inception网络还在不停的发展[20, 21],目前最新的为Inception-V4[21].本文将对比Inception结构与瓶颈结构的在本文场景的使用效果.

|
图 5 Inception-V1结构 Fig. 5 Framwork of Inception-V1 |
本文使用的后网络参考ResNet[16]的思想构建,称作CNN40bottleneck,根据输入图像的各类特性提出了有针对性的优化,从而减小网络体积、加快训练速度,同时保证网络分类效果.前网络裁切得到的前列腺部分的尺寸大小不一,而后网络要求输入的图像尺寸统一.本文将后网络输入图像的尺寸统一成64*64,该尺寸不仅保留了图像特征,而且能够减小计算量,使得训练更快.
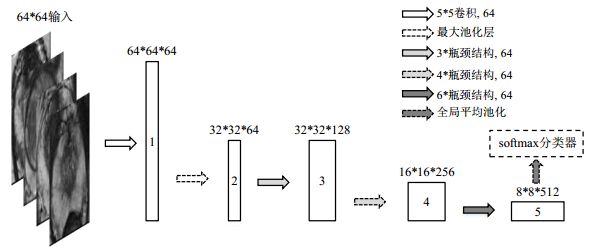
本文使用的后网络的结构如图 6所示,共有40层卷积层,实线方框为特征图.在第一层使用5*5卷积进行特征提取得到1号特征图;再通过最大池化层(Max Pooling)将特征图变为2号特征图(32*32*64);接着将2号特征图放入3、4、6组串连在一起的瓶颈结构,分别得到编号为3、4、5的三个特征图,每个瓶颈结构依次包含了一个1*1卷积层、一个3*3卷积层与一个1*1卷积层,其中数量为4、6的瓶颈结构组里第一个瓶颈结构中的3*3卷积的步长为2,将特征图尺寸分别变成16*16*256的4号特征图和8*8*512的5号特征图,最后将8*8的特征图进行全局平均池化,得到512维特征向量,将其作为输入放入softmax分类器进行分类.该网络的损失函数为交叉熵损失函数,如(11)式所示,其中y为期望输出,p为实际输出:
| ${l_{(p,y)}} = - \left[ {y\ln p + (1 - y)\ln (1 - p)} \right]$ | (11) |
|
图 6 采用了瓶颈结构的40层分类卷积神经网络 Fig. 6 40-layers CNN using bottleneck |
本文使用的后网络CNN40bottleneck与ResNet50结构的对比如表 1所示,针对本文训练图片数据量较少的特点,本文网络CNN40bottleneck减少了网络的层结构,同时在第一层卷积层缩小了卷积核大小,在瓶颈结构的卷积层上减少了通道数.
| 表 1 本文使用的后网络CNN40bottleneck与ResNet50对比 Table 1 Comparison between CNN40bottleneck used in this research and ResNet50 |
本文采用的数据来源为两个部分,一部分由深圳市第二人民医院提供,由西门子prisma3.0T MRI系统采集,分辨率为384*384,数据均为前列腺部位的横断位T2加权像,其中包含确诊的前列腺癌的病例22例(包含前列腺癌病灶特征的图像116张),良性14例(148张);另一部分为PROSTATEx竞赛[22]的公开数据集,经过筛选使用其中有前列腺癌病灶特征的图像84张,良性病例的图像280张.两部分数据汇总得恶性病例的磁共振图像200张与良性病例的磁共振图像428张.
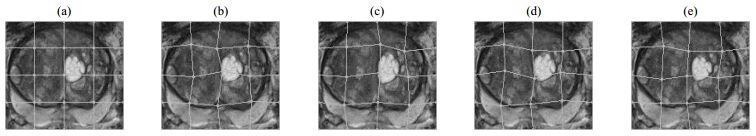
两次从上述病例图像中抽取样本图像组成2组数据集:第一组抽取良性病例和恶性病例的磁共振图像各45张作为测试集;第二组抽取良性病例和恶性病例的磁共振图像各55张作为测试集.每组中余下的磁共振图像通过基于移动最小二乘法的图像变换方式实现数据增广[23],并将其作为训练集.第一组的训练集增广后共得良性病例的磁共振图像1 400张、恶性病例的磁共振图像1 422张;第二组的训练集增广后共得良性病例的磁共振图像1 328张、恶性病例的磁共振图像1 344张.图像变换的效果如图 7所示,图 7(a)为根据前网络输出裁剪的图像,图 7(b)~7(e)为基于移动最小二乘法进行图像变换后的图像.
|
图 7 (a) 切割后的前列腺区域的磁共振图像与(b)~(e)对应的变换后图像 Fig. 7 (a) Cut prostate magnetic image and (b)~(e) its deformed images |
本文使用的硬件平台为:intel i7 8700k;Nvidia1080ti.软件平台为:Windows10;Keras/Tensorflow;Python 3.6.各项训练参数设置为:循环次数(epoch)1 000次;计算批次大小(batch size)32;学习率(learning rate)初值0.001,并以(1-epoch/maxepoch)速率衰减,maxepoch为最大循环次数.
本实验主要使用了6种网络进行测试,并对比各类参数.这6种网络模型分别是:本文网络CNN40bottleneck;没有使用批量标准化的本文网络CNN40bottleneck_nobn;使用全连接没有使用全局平均池化的本文网络CNN40bottleneck_fc;将瓶颈结构替换成如图 5所示的Inception-V1结构后的网络CNN40Inception;通用网络ResNet50;目前Inception系列中分类效果最好的Inception-V4.
2.3 评价标准基于磁共振图像的自动化诊断系统应该要满足以下两个条件:(1)低假阳性率(False Positive Rate,FPR),假阳性的出现可能会造成过度治疗;(2)高的真阳性率(True Positive Rate,TPR),真阳性率高可以帮助患者进行及时的治疗以提高生还率.
精确性、敏感性、特异性、f1-score可以反应出模型对真阳性和假阳性的判断效果.受试者工作特征(Receiver Operating Characteristic,ROC)曲线平面的横坐标是FPR,纵坐标是TPR,曲线下面积(Area Under Roc Curve,AUC)值为ROC右下方的面积,能够直观体现模型整体性能,数值越大表明分类效果越好.
本文采用精确性、敏感性、特异性、f1-score和AUC值联合评估本文使用的级联CNN网络的性能,并通过对比网络的训练时间以及参数量评估网络效率.
3 结果与讨论 3.1 训练时间与参数量6种网络的训练时间以及参数量如表 2所示.相较于ResNet50、Inception-V4两个通用模型,CNN40bottleneck的训练时间(4 654 s)与参数量(16.8 MB)显著减少;作为本文网络基础的ResNet50训练时间(23 023 s)大约是CNN40bottleneck的5倍,参数量(180 MB)大约是CNN40bottleneck的10倍,说明CNN40bottleneck较大地削减了网络整体层数与通道数.CNN40bottleneck_nobn的训练时间最短(3 098 s),由此得知批量标准化操作会一定程度增加网络训练时间;参数量(16.5 MB)与CNN40bottleneck相近.CNN40bottleneck_fc相较CNN40bottleneck训练时间更长(4 685 s)、参数量(17.3 MB)更多,说明全连接比全局平均池化使用更多的参数量,且需要更多的训练时间.CNN40Inception相较于使用瓶颈结构的网络训练时间更长(5 610 s),且占用了更多参数量(40.7 MB).上述结果表明网络架构的调整,以及应用瓶颈结构与全局平均池化,能够缩短训练时间、减小参数量.
| 表 2 不同方法的训练时间与参数量比较 Table 2 Comparison of training time and parameters among different methods |
图 8为CNN40bottleneck与通用模型ResNet50、Inception-V4分别在两组数据集上得到的ROC曲线,6种模型最终得到的各项分类性能指标如表 3所示.本文提出的优化网络CNN40bottleneck得到的AUC值为0.972 6,与ResNet50(AUC=0.934 3)、Inception-V4(AUC=0.939 5)两种公开通用模型相比,分类性能更优.本文在削减了层数量以及通道数后,分类性能反而进一步提升,由此我们猜想在像素密度较低的数据集上,参数量过多的网络有可能会有过多的特征输入,然而其中许多特征是冗余的,会对分类器分类产生干扰.使用了Inception模块的CNN40Inception(AUC=0.927 8)的各项指标都不及CNN40bottleneck,说明瓶颈结构相较于Inception对分类效果有提升作用.CNN40bottleneck_fc(AUC=0.948 5)的各项指标均不如本文网络,说明本数据集上全局平均池化是更好的特征选择方法.CNN40bottleneck_nobn(AUC=0.964 4)的分类效果在本文所比较的网络中仅次于CNN40bottleneck,说明批量标准化是能够提升网络分类效果的,但是在本文数据集像素密度小、层数少的情况下,协变量偏移较少,对网络的分类效果提升不是特别明显.综上所述,CNN40bottleneck在本文所比较的网络中分类效果最优;整体网络结构的调整以及瓶颈结构、批量标准化、全局平均池化的应用不仅能大幅度的缩减训练时间与参数量,而且可以提升分类性能.
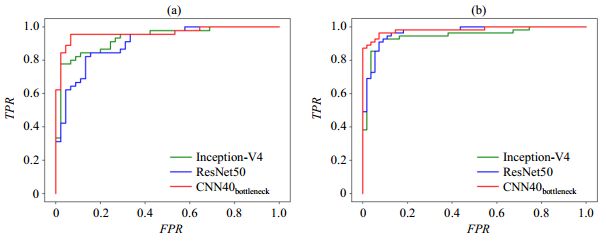
|
图 8 (a) 第一组和(b)第二组数据集的ROC曲线 Fig. 8 ROC curves from the (a) first and (b) second datasets |
| 表 3 不同方法的性能比较 Table 3 Comparison of evaluate indexes obtained by different methods |
本文提出了一种用于前列腺癌磁共振图像自动分类的级联CNN方法,其中前网络用于提取与分割前列腺区域,以排除磁共振图像中非前列腺区域的干扰,前网络的输出经增广后作为后网络的训练数据,后网络以ResNet50为参考,削减网络层数与通道数以减少模型参数并缩短训练的时间,同时使用批量标准化与全局平均池化优化网络以提升分类效果.结果显示,本文提出的分类网络CNN40bottleneck在基于磁共振图像的前列腺良型病变和恶性病变分类中,AUC值能够达到0.972 6,能够有效的对前列腺癌进行诊断,同时该网络的低参数、训练速度快等特点能作为更大数据集的训练或多模态训练[11, 13]的基础.
| [1] | SIEGEL R L, MILLER K D, DVM A J. Cancer statistics, 2018[J]. Ca:A Cancer Journal for Clinicians, 2018, 68(1): 11. |
| [2] |
HAN S J, ZHANG S W, CHEN W Q, et al. Analysis of the status and trends of prostate cancer incidence in China[J].
Chinese Clinical Oncology, 2013, 18(4): 330-334.
韩苏军, 张思维, 陈万青, 等. 中国前列腺癌发病现状和流行趋势分析[J]. 临床肿瘤学杂志, 2013, 18(4): 330-334. DOI: 10.3969/j.issn.1009-0460.2013.04.009. |
| [3] |
YANG J Y, YANG M Z, WEI W. Epidemiological study on the occurrence and development of prostate cancer[J].
J Clin Urology, 2017, 9: 74-78.
杨进益, 杨明州, 魏伟. 前列腺癌发生发展的流行病学研究进展[J]. 临床泌尿外科杂志, 2017, 9: 74-78. |
| [4] | MADDAMS J, UTLEY M, MØLLER H. Projections of cancer prevalence in the United Kingdom, 2010-2040[J]. Brit J Cancer, 2012, 107(7): 1195-1202. DOI: 10.1038/bjc.2012.366. |
| [5] | PENG Y, JIANG Y, YANG C, et al. Quantitative analysis of multiparametric prostate MR images:differentiation between prostate cancer and normal tissue and correlation with Gleason score--a computer-aided diagnosis development study[J]. Radiology, 2013, 267(3): 787-796. DOI: 10.1148/radiol.13121454. |
| [6] | ROOIJ M D, HAMOEN E H J, FÜTTERER J J, et al. Accuracy of multiparametric MRI for prostate cancer detection:A meta-analysis[J]. AJR Am J Roentgenol, 2014, 202(2): 343-351. DOI: 10.2214/AJR.13.11046. |
| [7] | FEHR D, VEERARAGHAVAN H, WIBMER A, et al. Automatic classification of prostate cancer Gleason scores from multiparametric magnetic resonance images[J]. Proc Natl Acad Sci U S A, 2015, 112(46): 6265-73. DOI: 10.1073/pnas.1505935112. |
| [8] | PENG Y, JIANG Y, YANG C, et al. Quantitative analysis of multiparametric prostate mr images:differentiation between prostate cancer and normal tissue and correlation with gleason score--a computer-aided diagnosis development study[J]. Radiology, 2013, 267(3): 787-796. DOI: 10.1148/radiol.13121454. |
| [9] | HASEGAWA A, LO S C B, FREEDMAN M T, et al. Convolution neural-network-based detection of lung structures[J]. Proceedings of SPIE-The International Society for Optical Engineering, 1994: 654-662. |
| [10] | LO S B, LOU S A, LIN J S, et al. Artificial convolution neural network techniques and applications for lung nodule detection[J]. IEEE Trans Med Imaging, 1995, 14(4): 711-718. DOI: 10.1109/42.476112. |
| [11] | SAHINER B, CHAN H P, PETRICK N, et al. Classification of mass and normal breast tissue:a convolution neural network classifier with spatial domain and texture images[J]. IEEE Trans Med Imaging, 1996, 15(5): 598-610. DOI: 10.1109/42.538937. |
| [12] | YANG X, LIU C Y, WANG Z W, et al. Co-trained convolutional neural networks for automated detection of prostate cancer in multi-parametric MRI[J]. Med Image Anal, 2017, 42: 212-227. DOI: 10.1016/j.media.2017.08.006. |
| [13] | WANG Z W, LIU C Y, CHENG D P, et al. Automated detection of clinically significant prostate cancer in mp-MRI images based on an end-to-end deep neural network[J]. IEEE Trans Med Imaging, 2018, 37(5): 1127-1139. DOI: 10.1109/TMI.2017.2789181. |
| [14] | SIMONYAN K, ZISSERMAN A. Very deep convolutional networks for large-scale image recognition[J]. Computer Science, 2014. arXiv: 1409.1556. https://arxiv.org/abs/1409.1556 |
| [15] | REN S, HE K, GIRSHICK R, et al. Faster R-CNN: towards real-time object detection with region proposal networks[C]//International Conference on Neural Information Processing Systems. 2015. https://arxiv.org/pdf/1506.01497.pdf |
| [16] | HE K M, ZHANG X Y, REN S Q, et al. Deep residual learning for image recognition[C]//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. IEEE, 2016: 770-778. https://www.computer.org/csdl/proceedings-article/cvpr/2016/8851a770/12OmNxvwoXv |
| [17] | LIN M, CHEN Q, YAN S C. Network in network[J]. Computer Science, 2013: 196-204. |
| [18] | IOFFE S, SZEGEDY C. Batch normalization: accelerating deep network training by reducing internal covariate shift[C]//International Conference on International Conference on Machine Learning. JMLR.org, 2015. http://proceedings.mlr.press/v37/ioffe15.html |
| [19] | SZEGEDY C, LIU W, JIA Y, et al. Going deeper with convolutions[C]//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. 2015: 1-9. https://www.oalib.com/references/21244713 |
| [20] | SZEGEDY C, VANHOUCKE V, IOFFE S, et al. Rethinking the inception architecture for computer vision[C]//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. 2016. 10.1109/CVPR.2016.308 |
| [21] | SZEGEDY C, IOFFE S, VANHOUCKE V, et al. Inception-v4, Inception-ResNet and the impact of residual connections on learning[C]//Thirty-First AAAI Conference on Artificial Intelligence. 2017: 711-718. https://www.researchgate.net/publication/301874967_Inception-v4_Inception-ResNet_and_the_Impact_of_Residual_Connections_on_Learning |
| [22] | GEERT L, OSCAR D, JELLE B, et al. Prostatex challenge data. The cancer imaging archive[OL].https://doi.org/10.7937/K9TCIA.2017.MURS5CL. |
| [23] | SCHAEFER S, MCPHAIL T, WARREN J. Image deformation using moving least squares[C]//ACM Siggraph. 2006: 711-718. 10.1145/1179352.1141920 |
 2020, Vol. 37
2020, Vol. 37 
