目前,磁共振成像(MRI)是膝关节的主要影像学检查方法.高场与超高场MRI具有图像信噪比高和空间分辨率高的优点,为膝关节疾病的进一步精确诊断提供了强有力的工具.然而,3 T以上B1+与电场在人体组织中分布的不均匀性增强,造成图像质量降低;而且电场的非均匀分布导致人体组织中可能出现局部射频功率沉积(SAR)超标的情况[1],对人体组织造成伤害,从而成为MRI扫描过程中一个关键的安全因素.
对于膝关节高场成像而言,国际上已经开展了一些局部SAR的研究[2-4].而国内迄今主要是全局SAR的研究[5],局部SAR仅限于胚胎、盆腔等[6].与全局SAR不同,局部SAR一般而言不能通过在体实测得到,不同个体的局部SAR精准估计需要通过电磁仿真实现[7-9].为了达到实用的目的,要求能够快速而精确的获得个体的膝关节模型.这就需要在磁共振图像的基础上进行图像分割,标定各像素所属的组织类型,从而重建出膝关节模型.
一些情况下,基于低场磁共振图像或小翻转角扫描获得的高场图像重建病人的膝关节模型,能够降低高场扫描中SAR超标的风险.但由于这些图像的信噪比与对比度较低,因此要分割出所有组织非常困难.然而,对于SAR估计而言,有研究[10-12]表明采用“肌肉-脂肪-肺”模型可以达到一定的近似程度,即考虑组织的介电性能(介电常数与电导率),在重建中与肌肉、脂肪、肺接近的组织分别归类为肌肉、脂肪与肺.
卷积神经网络(CNN)是当前发展迅速的深度学习方法,在膝关节分割方面已有一定的应用[13-15].2015年,Ronneberger等[16]以CNN为基础提出了一种改进的全卷积网络U-Net,其在传统全卷积网络的编解码结构上增加了浅层编码结果与深层解码结果的跳跃连接,边缘分割比较精细.
本研究采用U-Net对低场矢位膝关节图像进行分割与标注,考虑到膝关节中骨骼的比例与骨骼的介电性能,采用“肌肉-脂肪-骨骼”模型对组织进行简化归类.重建出的膝关节模型置于3 T的正交鸟笼线圈中进行电磁仿真,所得到的SAR10g(局部SAR的一种指标,即10 g组织中SAR的平均值)与手动标注全部组织所重建的模型进行比较判断.实验表明,采用U-Net能够得到较好的分割图像.同时,仿真结果验证了组织简化用于SAR估计的可行性,并且本方法构建的模型的SAR10g的最大值接近于手动分割的结果,热点区域基本不变.
1 材料与方法 1.1 数据集本研究采用的膝关节矢位磁共振图像在嘉恒医疗装备有限公司的0.35 T MRI系统JC35P上扫描得到,成像时间在2016~2017年.训练集15人、测试集14人,受试者平均年龄约51岁.训练与测试集中各有约1/2的受试者存在半月板、韧带损伤或者骨质增生等问题.扫描均获得所有受试者知情与同意,并经过温州医科大学附属第一医院伦理委员会的许可.
磁共振图像扫描采用矢位T1加权的自旋回波序列,使用90°翻转脉冲.图像层厚为4.5 mm、层间距为0.5 mm、层数处于19~24之间、脉冲间隔时间(TR)为600 ms(19层)~750 ms(24层)、回波时间(TE)为16 ms、成像视野(FOV)为230×230 mm2,图像重建分辨率为384×384.既往研究表明,对于3 T系统的SAR估计而言,5 mm的模型分辨率可以满足要求[10, 11, 17].所有图像均经过预增强,灰度分布调整到0~4 095之间.
组织的手动标注由两名有多年经验的影像科医生在Photoshop软件上完成,基本上按照完整的膝关节组织类型来标注,包括肌肉、脂肪、松质骨、皮质骨、半月板、软骨、积液等.韧带与肌腱由于占有像素较少,且不明显,因此作为肌肉的一部分.
1.2 组织的简化归类膝关节是人体的一个复杂的部位,包含若干种类型的组织,对其进行精确的分割是极其困难与耗时的.研究表明在SAR电磁仿真中,可以采用“肌肉-脂肪-肺”模型进行组织简化:人体器官大部分组织的介电性能与肌肉接近,脂肪与肌肉相差非常明显,肺处于两者之间[18].这样通过电磁仿真可以在一定近似度上实现对局部SAR的估计.
在膝关节中,骨骼占有一定的比例,而骨骼的介电性能处于脂肪与肌肉之间.因此,这里将上述模型修改为“肌肉-脂肪-骨骼”模型.图像分为背景、肌肉、脂肪以及骨骼四种区域,除脂肪与骨骼外的其他组织均归类为肌肉,以此为基础进行训练与测试,以及局部SAR的估计.
1.3 U-Net网络结构图 1给出了一个典型的U-Net网络结构(参考文献[16]的Fig.1),它是一种具有全卷积形式的改进型CNN.传统的CNN由于下采样,特征图不断收缩,所以相对而言更适合图像识别而非分割.而U-Net在收缩路径之后添加了与之对称的扩张路径,在每一个下采样操作对称的位置进行上采样操作(一般进行转置卷积计算,也称为反卷积),这样在扩张路径上特征图尺寸会不断恢复,因此就有较大的可能得到原始图像的分割结果.此外,这种网络结构能够在相对较小的数据集上进行训练.

|
图 1 U-Net网络结构的示例,这里图像大小为384×384,卷积层数为4(依据文献[16]重制) Fig. 1 An illustration of the U-Net network with input image size of 384×384, and convolution layers of 4 (Reproduced from Ref. [16]) |
在一台桌面电脑上完成U-Net网络的全部训练与测试,其配置为:四核Intel i7-4770处理器,16 GB内存,一块GPU显卡,型号为GeForce GTX TITAN.电脑上运行64位Windows操作系统,网络运行的软件平台为TensorFlow,编程语言为Python.
U-Net网络训练中,经反复调整得到优化的结构与参数.卷积层数为4,下采样操作的数量为3,卷积核的大小为7×7,初始的卷积核数量为16.网络训练采用梯度下降算法[19, 20],使用的误差函数为交叉熵函数E,定义为:
| $ E = - \sum\nolimits_{x \in X} {t(x)} \lg [p(x)] $ | (1) |
x是来自一批次输入X中的每一个输入,t(x)是预测对象的真实值,p(x)是网络输出结果通过softmax函数预测的值.softmax函数的作用是将输出结果作映射,映射后所有类别的输出范围为[0, 1],且和为1,其定义为:
| $ {\rm{soft}}\max {(p)_i} = \frac{{\exp ({p_i})}}{{\sum\nolimits_{k = 1}^K {\exp ({p_k})} }} $ | (2) |
pi表示第i类输出,k表示求和时来自所有类别K的第k类.
在带有卷积层的神经网络中,一般建议将初始权重设为均值为0的高斯分布[16].因此在这里我们通过标准差为
为了评估网络的分割效果,采用骰子系数(DCC)进行定量分析,以手动标注为参考.用A表示网络分割结果,M表示手动标注的结果,则DCC定义如下:
| $ DCC = \frac{{2 \times |M \cap A|}}{{|M| + |A|}} $ | (3) |
其中运算符| |表示统计区域中像素的数量.
1.6 SAR仿真与计算SAR的电磁场仿真采用Remcom XFDTD软件(Remcom Inc.,State College,PA)进行,硬件平台为前面所述的桌面电脑.线圈模型来自于一个3 T的正交鸟笼膝线圈[21],该线圈有8根杆,长度为180 mm、直径为175 mm,采用集总元件,重建的膝关节模型置于线圈中心.SAR定义为单位质量组织中所吸收的射频功率,由(4)式给定:
| $ SAR = \frac{\sigma }{{2\rho }}|\vec E{|^2} $ | (4) |
σ为电导率;ρ为组织的质量密度;电场强度
对于1个特定体素p,其局部SAR(这里为SAR10g)定义为:
| $ SA{R_{10{\rm{g}}}}(p) = \frac{{\sum\nolimits_{q \in V} {{m_q} \cdot SAR(q)} }}{{\sum\nolimits_{q \in V} {{m_q}} }} $ | (5) |
这里V是以p为中心的质量等于10 g的区域,q是V中的任一体素.
2 实验结果为简便起见,这里将包含完整组织类型的手动标注称为手动标注(完整),依据“肌肉-脂肪-骨骼”进行组织简化的手动标注称为手动标注(简化).
2.1 图像分割结果为验证U-Net网络的分割性能,将测试集图像输入训练好的网络,其输出逐层地与手工标注(简化)结果进行比较.采用DCC作为定量评价标准,结果如表 1所示.表 1显示,基于U-Net的分割结果在三种组织上的DCC平均值都大于0.92.
| 表 1 测试集基于U-Net分割结果与手动标注(简化)的骰子系数(DCC) Table 1 DCC calculation based on U-Net segmentation and manual delineation (muscle-fat-bone) using the test set |
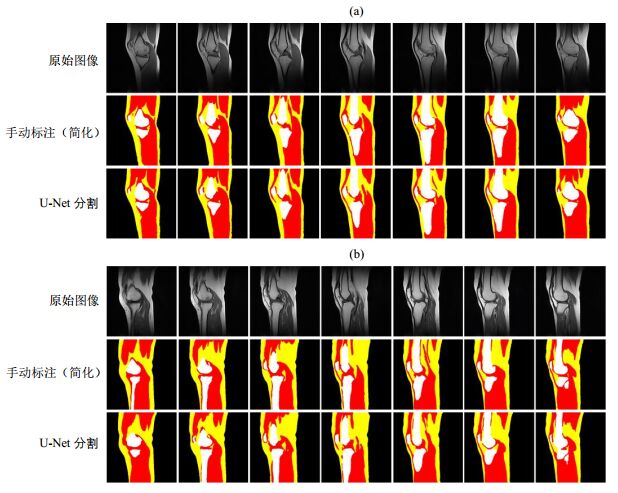
图 2给出了两个例子,分别显示了两名受试者的网络分割与手动标注(简化)图像.图 2(a)为一名正常的受试者(女性,年龄21岁),图 2(b)为一名半月板损伤并有积液的受试者(女性,年龄58岁),为便于显示,这里给出了中间连续7层的图片.
|
图 2 两名女性受试者的U-Net分割与手动标注(简化)图像.(a)和(b)分别为一名21岁和58岁的女性受试者.红色、黄色、白色区域分别对应肌肉、脂肪与骨骼 Fig. 2 The images of U-Net segmentation and manual delineation (muscle-fat-bone) for two female subjects. (a)21-year old; (b)58-year old. The muscle, fat, and bone are labeled in red, yellow, and white, respectively |
对于测试集,根据其U-Net分割与手动标注(完整)结果分别重建了膝关节模型,将该模型分别置于鸟笼线圈中进行了电磁场仿真,再根据仿真得到的电场强度计算其SAR10g.找出SAR10g的最大值Max(SAR10g),并计算U-Net分割与手动标注(完整)得到的Max(SAR10g)的相对误差(无量纲),然后在整个测试集范围计算相对误差的平均值与标准差,结果如表 2所示.为了验证组织简化的合理性,手动标注(完整)与手动标注(简化)所构建的模型也进行了仿真与结果分析.
| 表 2 基于U-Net分割与手动标注得到的测试集的Max(SAR10g)的相对误差 Table 2 The relative errors of Max(SAR10g) values from U-Net segmentation and manual delineation using the test set |
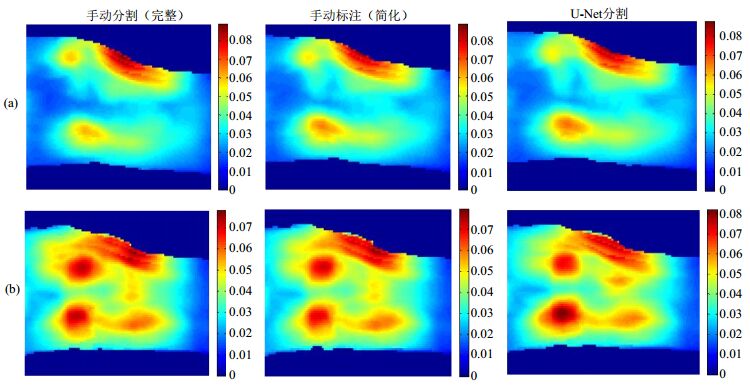
图 2所示的两名受试者的网络分割与手动标注所重建膝关节的SAR10g如图 3所示,由于层数较多,这里显示的是SAR10g的最大密度投影(MIP)图.
|
图 3 两名女性受试者(与图 2相同)膝关节SAR10g分布的MIP. (a)一名21岁女性受试者,手动分割(完整)、手动标注(简化)以及U-Net分割得到的Max(SAR10g)分别为0.088 7 W/kg、0.088 5 W/kg以及0.087 5 W/kg;(b)一名58岁的女性受试者,手动分割(完整)、手动标注(简化)以及U-Net分割得到的Max(SAR10g)分别为0.078 1 W/kg、0.079 6 W/kg以及0.081 8 W/kg.每个图最右边的色条表示图中不同的颜色对应的SAR10g(单位为W/kg) Fig. 3 The SAR10g distribution (MIP) of the knee models of the two female subjects mentioned in Fig. 2. (a)21-year old, the Max(SAR10g) values constructed from manual delineation (full tissues), manual delineation (muscle-fat-bone) and U-Net segmentation are 0.088 7 W/kg, 0.088 5 W/kg, and 0.087 5 W/kg separately. (b)58-year old, the Max(SAR10g) values constructed from manual delineation (full tissues), manual delineation (muscle-fat-bone) and U-Net segmentation are 0.078 1 W/kg, 0.079 6 W/kg and 0.081 8 W/kg separately. The right color bar of each sub-figure represents the SAR10g value corresponding to different colors (unit: W/kg) |
从图 3可以看出,U-Net分割与手动标注(简化)的Max(SAR10g)均接近手动标注(完整)的结果,并且SAR10g的热点区域位置基本相同.
3 讨论 3.1 U-Net分割、组织简化与SAR估计U-Net是一种全卷积的神经网络,相比传统CNN,由于增加了上采样层以及下、上采样层之间的连接通道,其更适合于完成精细的图像分割工作.相比半自动分割方法,它能够实现全自动的分割,提高实时性,减轻医生或研究人员的工作量.因此,在临床与科研方面具有很大的应用潜力.
对于膝关节而言,由于包含的组织类型较多,采用U-Net进行完整组织的图像分割具有较大的难度,花销数十小时的训练时间也难以收敛,分割效果较差,即使增加网络层数其改善也不明显.考虑到SAR估计的应用背景,电磁场仿真主要取决于介质的介电常数与电导率,肌肉、脂肪以及骨骼的这两种参数相互之间差别较大,而肌肉与膝关节其他大部分组织比较接近,这样构成“肌肉-脂肪-骨骼”模型用于组织的简化分类.U-Net依据该模型对矢位T1加权自旋回波图像进行训练与测试,不到10 h就能达到收敛,并且具有较好的分割效果,单幅384×384图像的平均分割时间小于0.3 s.与依据“肌肉-脂肪-骨骼”手动标注的图像相比,三种组织的DCC平均值均大于0.92,展现了一定程度的良好的分割性能.
仿真与计算初步表明,依据“肌肉-脂肪-骨骼”手动标注并重建的膝关节模型的局部SAR的最大值,接近按照完整组织手动标注并重建的结果,相对误差平均值小于3%,热点区域基本不变,这验证了组织简化在SAR估计方面的有效性.基于组织简化的U-Net图像分割所重建出的膝关节模型,其Max(SAR10g)的最大值相对误差平均值小于4%(见表 2),热点区域也基本不变,表明U-net分割所重建出的膝关节模型具有较高的相似度,能够较好地应用于SAR估计.
3.2 不足与展望CNN训练集的大小对网络性能有较大影响,在本研究中,训练集人数为15,尽管超过10(一般公认的人数底线),但仍希望能够明显地扩大人员数量,增大在年龄、体型、病变情况等方面的覆盖面.同时,测试集的样本数量也需要扩大,以充分验证所提方法的可信度.在今后的工作中增加数据集规模的同时,希望针对性的增加带有病变组织的膝关节图像,以验证本方法对于膝关节疾病组织的分割敏感性与特异性.鉴于此,本研究尚属膝关节SAR估计的一个初步性的工作.
由于低场MRI在信噪比与分辨率等方面的不足,研究所用的图像难以清晰地反映关节以及相邻腿部组织的细节,同时模型也不能用于7 T MRI系统的SAR仿真之中.今后希望能够获得高场高分辨的数据集,层厚为2 mm,甚至1 mm,这样不仅可以对U-Net网络性能进行高可信度的评估,而且所建模型能够用于7 T甚至9.4 T MRI系统的SAR仿真,显著地扩大所提方法的适用面.
实验结果表明,单个U-Net分割效果总体较好,但也有部分图像的DCC有待提高.为了进一步改善网络的分割精度,除了加大训练集的人员数量外,可以考虑调整网络的结构,采用多U-Net网络并行结构,这样预期可以取得更好的效果.
4 结论本文针对高场膝关节MRI中局部SAR估计的现实需求,提出了一种基于低场磁共振图像的分割方法以重建膝关节模型.依据“肌肉-脂肪-骨骼”模型进行组织简化,采用U-Net进行图像的自动分割,实验结果表明所重建的模型能够获得与手动标注模型接近的SAR10g分布,从而为高场膝关节扫描的SAR实时而准确的估计提供了有潜在价值的手段.
| [1] | PADORMO F, BEQIRI A, HAJNAL J V, et al. Parallel transmission for ultrahigh-field imaging[J]. NMR Biomed, 2016, 29(9): 1145-1161. DOI: 10.1002/nbm.3313. |
| [2] | BOSS A, GRAF H, BERGER A, et al. Tissue warming and regulatory responses induced by radio frequency energy deposition on a whole-body 3-Tesla magnetic resonance imager[J]. J Magn Reson Imaging, 2007, 26(5): 1334-1339. DOI: 10.1002/jmri.21156. |
| [3] | JIN J, WEBER E, DESTRUEL A, et al. An open 8-channel parallel transmission coil for static and dynamic 7T MRI of the knee and ankle joints at multiple postures[J]. Magn Reson Med, 2018, 79(3): 1804-1816. DOI: 10.1002/mrm.26804. |
| [4] | GAGLIARDI V, RETICO A, BIAGI L, et al. Subject-specific knee SAR prediction using a degenerate birdcage at 7 T[C]. 2018 IEEE International Symposium on Medical Measurements and Applications (MeMeA), 2018: 1-5. [DOI:10.1109/MeMeA.2018.8438709]. |
| [5] | 燕新强, 薛蓉, 丰宝桐, 等.一种多通道磁共振成像设备的SAR实时监测系统及方法: 中国, CN201410503941.7[P]. 2014-12-24. |
| [6] |
HUANG Q H, GAO Y, XIN X G. Study on the law of B1 field homogeneity and SAR inside human body varying with field strength at high and ultra-high field MR[J].
Chin J Biomed Eng, 2013, 32(1): 21-27.
黄绮华, 高勇, 辛学刚. 高场和超高场MR下人体内B1场均匀性及SAR随场强变化规律的研究[J]. 中国生物医学工程学报, 2013, 32(1): 21-27. DOI: 10.3969/j.issn.0258-8021.2013.01.004. |
| [7] | GRAESSLIN I, HOMANN H, BIEDERER S, et al. A specific absorption rate prediction concept for parallel transmission MR[J]. Magn Reson Med, 2012, 68(5): 1664-1674. DOI: 10.1002/mrm.24138. |
| [8] | GRAESSLIN I, VERNICKEL P, BÖRNERT P, et al. Comprehensive RF safety concept for parallel transmission MR[J]. Magn Reson Med, 2015, 74(2): 589-598. DOI: 10.1002/mrm.25425. |
| [9] | VAN DEN BERGEN B, VAN DEN BERG CA, BARTELS LW, et al. 7 Tesla body MRI:B1 shimming with simultaneous SAR reduction[J]. Phys Med Biol, 2007, 52: 5429-5441. DOI: 10.1088/0031-9155/52/17/022. |
| [10] | HOMANN H, BÖRNERT P, EGGERS H, et al. Toward individualized SAR models and in vivo validation[J]. Magn Reson Med, 2011, 66(6): 1767-1776. DOI: 10.1002/mrm.22948. |
| [11] | WOLF S, DIEHL D, GEBHARDT M, et al. SAR simulations for high-field MRI:how much detail, effort, and accuracy is needed?[J]. Magn Reson Med, 2013, 69(4): 1157-1168. DOI: 10.1002/mrm.24329. |
| [12] | WU T N, SHAO Q, YANG L. Simplified segmented human models for whole body and localised SAR evaluation of 20 MHz to 6 GHz electromagnetic field exposures[J]. Radiat Prot Dosimetry, 2013, 153(3): 266-272. DOI: 10.1093/rpd/ncs105. |
| [13] | PRASOON A, PETERSEN K, IGEL C, et al. Deep feature learning for knee cartilage segmentation using a triplanar convolutional neural network[C]. International conference on MICCAI 2013, 2013: 246-253. https://rd.springer.com/chapter/10.1007/978-3-642-40763-5_31 |
| [14] | LIU F, ZHOU Z Y, JANG H, et al. Deep convolutional neural network and 3D deformable approach for tissue segmentation in musculoskeletal magnetic resonance imaging[J]. Magn Reson Med, 2018, 79(4): 2379-2391. DOI: 10.1002/mrm.26841. |
| [15] | ZHOU Z Y, ZHAO G Y, KIJOWSKI R, et al. Deep convolutional neural network for segmentation of knee joint anatomy[J]. Magn Reson Med, 2018, 80(6): 2759-2770. DOI: 10.1002/mrm.27229. |
| [16] | RONNEBERGER O, FISCHER P, BROX T. U-net: Convolutional networks for biomedical image segmentation[C]. International Conference on MICCAI 2015, 2015: 234-241. https://link.springer.com/chapter/10.1007/978-3-319-24574-4_28 |
| [17] | COLLINS C M, SMITH M B. Spatial resolution of numerical models of manand calculated specific absorption rate using the FDTD method:a study at 64 MHz in a magnetic resonance imaging coil[J]. J Magn Reson Imaging, 2003, 18(3): 383-388. DOI: 10.1002/jmri.10359. |
| [18] | HASGALL P A, DI GENNARO F, BAUMGARTNER C, et al. IT'IS Database for thermal and electromagnetic parameters of biological tissues[R]. Version 4.0, 2018-5-15. [DOI:10.13099/VIP21000-04-0.itis swiss/database]. |
| [19] | HINTON G, SRIVASTAVA N, SWERSKY K. Neural networks for machine learning lecture 6a: Overview of mini-batch gradient descent[R]. Toronto University, 2012: 14. |
| [20] | YUAN K, YING B, VLASKI S, et al. Stochastic gradient descent with finite samples sizes[C]. 2016 IEEE 26th International Workshop on Machine Learning for Signal Processing (MLSP), 2016: 1-6. https://ieeexplore.ieee.org/abstract/document/7738878 |
| [21] | PETERSON D M, CARRUTHERS C E, WOLVERTON B L, et al. Application of a birdcage coil at 3 Tesla to imaging of the human knee using MRI[J]. Magn Reson Med, 1999, 42(2): 215-221. DOI: 10.1002/(SICI)1522-2594(199908)42:2<215::AID-MRM1>3.0.CO;2-8. |
 2020, Vol. 37
2020, Vol. 37 
