正常大脑由白质、灰质和脑脊液三种软组织构成,而肿瘤会导致脑部软组织产生异常.脑胶质瘤是成人脑肿瘤中最常见的原发性恶性肿瘤,发病率高、治愈率低[1].磁共振成像(Magnetic Resonance Imaging,MRI)是临床诊断脑胶质瘤的技术之一.胶质瘤的磁共振图像呈现为一片异质的脑瘤区域,通常包含4个部分:坏死区域、增强区域、水肿区域、非胶质瘤病灶区域.由于胶质瘤结构复杂,因此多模态磁共振图像能更清晰地显示不同模态的结构信息,但胶质瘤不同模态的磁共振图像差别较大、强调的信息不同.目前利用小波分解进行图像融合的方法可以把不同模态的图像进行融合,这样可以使图像包含尽可能少的冗余信息、最大程度丰富有效信息,使图像尽可能清晰[2].
近年来,有研究者提出了区域生长法、水平集法、模糊聚类法和机器学习等脑肿瘤分割方法[3].但是在分割脑肿瘤过程中,大多数方法需要医生手动分割.区域生长法需要在图像中提前设置一个种子点,Shih等[4]提出自动选取种子点的方法,但是如果将种子点设置在正常区域会产生错误的分割结果,且图像的噪声和灰度分布不均匀会导致过分割或欠分割.Yamasaki等[5]将GrowCut算法用于脑肿瘤分割,但该方法仍需设置种子区域.水平集法是基于主动轮廓模型的分割方法,需要选择最优初始轮廓.Rana等[6]采用快速包围盒算法选择肿瘤区域的初始轮廓,利用水平集提取肿瘤边界,但是错误选择初始轮廓会产生较大的误差.模糊聚类法一般和K均值或C均值(K均值是依据误差平方和最小化的原则进行聚类,而模糊的C均值是K均值聚类算法的推广,其基本依据是加权误差平方和最小化.两者都是通过迭代的方法划分类别.)联合使用,但该方法需要明确数据分布的先验知识,假设的数据分布和真实数据分布接近时才可分割出鲁棒性较好的结果[7, 8].传统的脑瘤分割方法不仅过多地依赖医学领域的先验知识,而且在分割过程中耗费大量的人力和时间.而最近比较热门的深度学习方法具有自我学习能力、通用性和高效性等特点,因此基于深度学习的医学图像自动分割具有重要的研究价值.2006年,Hinton等[9]提出神经网络训练算法并改进后,得到大数据的支持,深度学习开始广泛应用于图像、语音识别等领域.研究表明,基于深度学习的计算机辅助诊断可给医生提供参考诊断结果,还可避免医生在诊断中的主观错误[10-14].目前常用基于深度学习的胶质瘤分割方法主要是基于卷积神经网络(Convolutional Neural Network,CNN)的全自动分割,需要大量的样本和标记信息训练网络模型以提高分割精度[15].
脑胶质瘤分割通常是根据图像的灰度、纹理、亮度和对比度等区分出正常组织和病变区域,目的是提取并研究胶质瘤解剖结构,定位胶质瘤的感兴趣区域,测出肿瘤的大小和体积以进行针对性治疗.Havaei等[16]提出基于CNN的脑肿瘤分割方法,利用图像的局部和全局特征,且在最后一层采用卷积层,使运行速度提高40倍.Pereira等[17]提出基于CNN的胶质瘤分割方法,与Havaei等的方法不同,他们采用3×3的小型卷积核,网络结构更深、降低了过拟合几率、减少了网络参数.上述方法虽然解决了MRI数据标签不平衡的问题,但存储开销很大、消耗计算机的算力,而且获得的概率信息是一维(1D)的,不能标识每个像素点的类别.Ronneberger等[18]提出UNet网络,使用编码器和解码器结构(其中编码器用来特征提取,解码器用于上采样,因为网络结构像U型,故称UNet网络),补足图片信息、提高图像分辨率,使分割结果更加细致准确.上述方法均使用2D CNN来处理2D图像块,或采用3D正交的2D图像块提取上下文信息,未利用3D体素信息.2016年,Kamnitsas等[19]提出3D CNN分割方法,利用3D的条件随机场进行处理获得了更加精确的分割结果.Casamitjana等[20]结合网络的局部和全局特征训练3D CNN,使用多分辨率网络结构分割脑肿瘤,提高了网络分割精度.Chen等[21]基于3D CNN模型使用两种不同大小的卷积核,提取图像的多尺度信息,该方法在时间、空间和鲁棒性上均优于目前已有的3D CNN架构.Mehta等[22]提出端到端的回归分割3D CNN用于脑肿瘤MRI体素的合成,其峰值信噪比和结构相似性都高于之前的方法.
本文针对当前基于深度学习的脑胶质瘤分割方法中需要大量的训练样本、计算机算力不足的问题,通过多模态数据融合减少训练样本、降低内存占用、增加网络训练速度.同时在UNet网络的基础上构建3D-UNet模型,实现多模态磁共振图像脑胶质瘤及瘤内不同区域的分割,以提高分割精度.相比2D-UNet模型,我们采用3D图像块对3D体素进行卷积和反卷积,结合3D体素的局部细节特征和全局轮廓特征生成像素级别的分割结果.并采用批量归一化(Batch Normalization,BN),提高训练速度、减弱正则化[23, 24].我们选取了Brats2018(Brain Tumor Segmentation 2018)数据集分别进行融合前和融合后的全自动分割对比实验,并对实验结果进行了定性和定量评价.
1 方法 1.1 基于小波分解的多模态图像融合图像融合分为像素级融合、特征级融合和决策级融合三个层次.像素级融合是将各源图像或其变换图像中对应像素点的灰度值进行融合,获取的融合结果信息最为丰富,是当前融合的主要研究方向;特征级融合将一些关注的特征集从原始数据中抽取出来进行融合,易丢失图像中的重要信息,融合后图像失真较严重;决策级融合是从每一份独立的原始数据中获取决策,再将它们合并形成一个全局性的决策,工作过程复杂且不易实现.而基于像素级的融合方法可以主要分为三大类:基于小波变换的融合算法,基于塔分解算法的融合方法和基于梯度场的融合方法.由于本文处理的是大批量数据,融合过程要求简单、快速、有效,所以采用了基于离散小波变换(Discrete Wavelet Transformation,DWT)的融合算法[25].我们对T1、Tlce、T2、Flair四种模态的3D MRI数据直接进行小波融合,图 1为融合流程图.
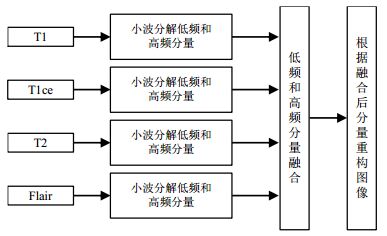
|
图 1 基于小波分解的图像融合流程 Fig. 1 Image fusion process based on wavelet decomposition |
DWT通过特定的低通和高通滤波器将图像分解为多个子带,这些子带又可以分为包含近似信息的低频分量和包含边缘细节的高频分量,DWT分解的一层结构如图 2所示.对四幅图像中的低频分量(LLL)采用加权融合规则进行融合.设图像A、B、C、D经过j层小波变换分解后,低频分量加权融合准则如(1)式所示,Fj(x, y, z)是融合低频分量,Lj(x, y, z)是各图像分解得到低频分量:
| $ {F_j}(x,y,z) = {\alpha _1}{L_{Aj}}(x,y,z) + {\alpha _2}{L_{Bj}}(x,y,z) + {\alpha _3}{L_{Cj}}(x,y,z) + {\alpha _4}{L_{Dj}}(x,y,z) $ | (1) |

|
图 2 三阶离散小波分解结构.H:高频分量;L:低频分量 Fig. 2 Wavelet decomposition structure of third-order discrete. H: high-frequency component; L: low-frequency component |
其中α1、α2、α3和α4分别是四个低频分量的融合系数,α1+α2+α3+α4=1,本文实验中设置α1=0.10、α2=0.10、α3=0.40、α4=0.40、j=1.
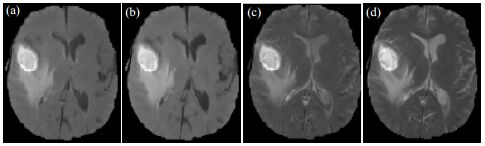
由于Flair和T1图像的病灶边界模糊不清,而T1ce和T2图像的病灶更清晰,因此在后续实验中应取Flair和T1的权重低于T1ce和T2.对LLL采用加权平均法做了四组实验,表 1为融合过程中选取的具体系数,E为融合图的信息熵,E越大表示融合图像的信息量越大[26].图 3为不同系数的融合结果对比图.当α1=0.10、α2=0.10、α3=0.40、α4=0.40时,E值最高,为5.746 1,最高.如图 3(d)所示,融合图的肿瘤区域更明亮,水肿区域轮廓结构也更加清晰.
| 表 1 低频分量不同的融合系数对比 Table 1 Comparison of fusion coefficients with different low-frequency components |
|
图 3 不同系数的融合结果.(a) Group 1; (b) Group 2; (c) Group 3; (d) Group 4(Groups 1~4与表 1相同) Fig. 3 Fusion results of different coefficients. (a) Group 1; (b) Group 2; (c) Group 3; (d)Group 4 (Groups 1~4 were the same with Table 1) |
高频分量(LLH、LHL、LHH等)融合准则如(2)式和(3)式所示,
| $ H_{mj}^h = {e^{\left| {H_{mj}^h(x,y,z) - \mu _{mj}^h} \right| - {{(\sigma _{mj}^h)}^2}}} $ | (2) |
| $ {H_j}(x,y,z) = \frac{{H_{mj}^h \cdot H_{mj}^h(x,y,z)}}{{\sum\limits_{m = 1}^4 {H_{mj}^h} }} $ | (3) |
最后,对融合系数进行小波逆变换操作,重构图像,得到融合图像.对融合图像采用信息熵E评价,如(4)式所示.
| $ E = - \sum\limits_{i = 0}^{L - 1} {{p_i}{{\log }_2}{p_i}} $ | (4) |
其中,L表示图像灰度级别,pi表示灰度值i像素占总像素比例.
图 4是Brats2018数据集中Brats18_2013_10_1患者四种模态的磁共振图像及其融合图,Flair、T1、T1ce、T2图像和融合图的信息熵分别为5.745 9、5.550 0、5.339 7、5.738 8和5.746 1,融合图的信息熵最大,所含信息量最大.从图 4(e)可看出,融合图结合了四种模态磁共振图像的有效信息,补充胶质瘤病灶部分,使边缘轮廓更加清晰,有利于进一步的分割.
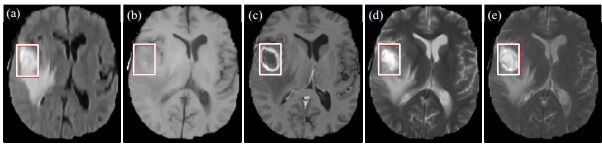
|
图 4 融合效果图.(a) Flair; (b) T1; (c) T1ce; (d) T2; (e)融合图像 Fig. 4 Fusion effect images. (a) Flair; (b) T1; (c) T1ce; (d) T2; (e) Fusion image |
本文基于2D-UNet构建3D-UNet网络,架构流程如图 5所示.首先融合四种模态图像,然后从融合后的图像中提取400个大小为64×64×64的图像块作为网络的输入.训练时,为计算损失函数和反向传播,需将金标准(Ground Truth,GT)添加到图像块中.训练网络包括左侧的特征提取和右侧的上采样两部分.特征提取部分每层包括两个3×3×3的卷积核,每次卷积操作后采用BN和Relu激活函数;与传统2D-UNet网络不同的是,本文在每个维度添加感受野为3×3×3,步长为2的最大池化;上采样部分包括两个3×3×3的卷积和2×2×2的反卷积;最后一层采用1×1×1的卷积核将输出的特征通道降至4.将特征提取部分的高分辨率细节特征,与上采样后的等分辨率轮廓特征进行通道维度拼接以增加特征信息.

|
图 5 3D-UNet架构流程 Fig. 5 Architecture progress of 3D-UNet |
利用融合图像和金标准来训练网络,分割脑胶质瘤及瘤内结构,从截断的正态分布中输出标准偏差为0.1的随机值来初始化权重参数.提取图像块时,对金标准中标记为病变部分的所有像素进行采样,非病变像素在其周围提取图像块,以解决医学图像数据类别不平衡的问题.由于最大池化仅提取了相邻区域的最大特征,但分割需检测图像的细微特征,故在最大池化的感受野之间存在重叠以保持图像细节信息.由于池化层会丢失图像信息,降低图像分辨率且不可逆,对图像分割任务有影响,而上采样可以补足一些图片的信息,但是信息补充不够完全,所以需要与分辨率较高的图片拼接.随着卷积次数增多,提取的特征也更加有效、更加抽象.为降低过拟合,在上采样之前加入dropout正则化,丢弃率为0.5[27].由于图像特征分为坏死、增强、水肿和非胶质瘤病灶区域,故将网络输出层设置为4通道.后处理阶段使用基于连通区域的轮廓提取方法以降低假阳性,本文提出的算法流程见图 6.

|
图 6 本文提出算法的流程图 Fig. 6 Flowchart of algorithm proposed in this research |
由于医学图像的类别不平衡问题,当训练集的样本数据和目标样本集分布不一致时,训练得到的模型无法很好地泛化.神经网络中每层输入经过一系列操作后会与原来对应的输入数据分布不同,故需矫正训练样本.BN可规范化某些层或所有层的输入,固定输入的均值和方差,所以我们在激活函数之前添加了BN.每个批次训练期间,前向传播时,对输入权值进行标准化并保存其均值和方差,输出不变.反向传播时,根据BN层中的均值和方差对卷积层和Relu层进行链式求导,获取梯度和当前学习速率并更新权重.测试时,每个BN层对训练集中的所有数据求取总体均值和方差,然后根据训练集中整体的无偏估计计算BN层的输出.实验结果表明,在训练和测试过程中使用BN可提高训练速度并减少正则化.(5)~(8)式是BN传导过程,其中,
| ${\mu _B} = \frac{1}{m}\sum\limits_{i = 1}^m {{x_i}} $ | (5) |
| $\sigma _B^{\rm{2}} = \frac{1}{m}\sum\limits_{i = 1}^m {{{({x_i} - {\mu _B})}^2}} $ | (6) |
| ${\hat x_i} = \frac{{{x_i} - {\mu _B}}}{{\sqrt {\sigma _B^{\rm{2}} + \varepsilon } }}$ | (7) |
| ${y_i} = \gamma {\hat x_i} + \beta \equiv B{N_{\gamma ,\beta }}({x_i})$ | (8) |
在最后一层中结合交叉熵代价函数和损失函数Softmax来更新权重参数并产生预测的概率分布,Softmax的定义如下:
| ${p_k}(x) = \frac{{\exp [{a_k}(x)]}}{{\sum\limits_{k = 1}^K {\exp [{a_k}(x)]} }}$ | (9) |
其中,
本文使用Tensorflow中的Adam算法作为梯度下降法,该算法是一种自适应学习率算法,对每个参数都计算自适应的学习率,具有较好的鲁棒性[28].Adam算法存储梯度指数衰减均值
| ${m_t} = {\beta _1}{m_{t - 1}} + (1 - {\beta _1}){g_t}$ | (10) |
| ${v_t} = {\beta _2}{v_{t - 1}} + (1 - {\beta _2})g_t^2$ | (11) |
其中,
对
| ${\hat m_t} = \frac{{{m_t}}}{{1 - \beta _1^t}}$ | (12) |
| ${\hat v_t} = \frac{{{v_t}}}{{1 - \beta _2^t}}$ | (13) |
其中,β的上标t表示t时刻的指数衰减率,β与t值无关,但不同t时刻可修改β;
| ${\theta _t} = {\theta _{t - 1}} - \frac{\alpha }{{\sqrt {{{\hat v}_t}} + \varepsilon }}{\hat m_t}$ | (14) |
其中
本文实验环境为CUDA 9.0并行计算架构、CuDNN9.0 GPU加速库.GPU型号为内存24 GB的Quadro P6000.我们采用1.6.0版本的开源Tensorflow框架,该框架创建了基本的CNN架构,用户可在此基础上开发自己的网络架构.现利用Brats2018数据库中所有训练集,共285例数据,其中包括210例高级别脑胶质瘤(High Grade Glioma,HGG)和75例低级别脑胶质瘤(Low Grade Glioma,LGG),每例数据包含T1、T1ce、T2、Flair四种模态和金标准.Brats2018数据库中图像均去颅骨,严格与T1图像对齐并插值
实验过程中,训练集用来拟合模型,通过设置分类器的参数训练分类模型.验证集是当通过训练集训练出多个模型后,为了找出效果最佳的模型,使用各个模型对验证集数据进行预测,并记录准确率,选出效果最佳的模型所对应的参数,即调整模型参数.测试集是通过训练集和验证集得出最优模型后,使用测试集进行模型预测,用于衡量最优模型的性能和分类能力.训练集、验证集、测试集常见的划分方法有:留出法、交叉验证法和自助法.我们采用留出法来划分,留出法常见的做法是将2/3~4/5的样本用于训练.因此我们将训练集、验证集、测试集按3:1:1的比例随机分配.
2.2 预处理我们使用N4ITK对融合后的MRI数据做偏置场校正、标准化图像灰度[29].为解决数据不平衡的问题,采用数据抽取的方式抽取预处理后的图像,为保证图像病灶和非病灶分布一致,抽取每个图像块大小为

|
图 7 预处理效果对比图.(a)HGG1预处理前;(b)HGG1预处理后;(c)HGG2预处理前;(d)HGG2预处理后 Fig. 7 Pre-processing effect images. (a) HGG1, before pre-processing; (b) HGG1, after pre-processing; (c) HGG2, before pre-processing; (d) HGG2, after pre-processing |
数据库中HGG分割结果包含水肿、增强和坏死区域,LGG的增强和坏死区域较难区分,临床中并未严格区分增强和坏死的金标准,为便于算法性能统一比较,将增强和坏死区域合并统计为脑胶质瘤核心区域的分割精度.融合后图像均为155层,最终的定量评价结果是所有层分割结果的平均值.采用Dice相似系数(Dice Similarity Coefficient,DSC)、阳性预测值(Positive Predictive Value,PPV)和敏感性(Sensitivity,Sen)作为定量评价指标对胶质瘤及瘤内结构的平均分割效果进行客观评价,计算方式如(15)~(17)式所示:
| $DSC\left( {{P_*},{T_*}} \right) = \frac{{|{P_*} \cap {T_*}|}}{{\left( {|{P_*}| + |{T_*}|} \right)/2}}$ | (15) |
| $PPV\left( {{P_*},{T_*}} \right) = \frac{{|{P_*} \cap {T_*}|}}{{|{P_*}|}}$ | (16) |
| $Sen\left( {{P_*},{T_*}} \right) = \frac{{|{P_*} \cap {T_*}|}}{{|{T_*}|}}$ | (17) |
其中,
本文利用构建的3D-UNet网络实现脑部磁共振图像胶质瘤及瘤内结构的分割,融合前后的分割结果对比如图 8所示.图 8(a)和8(b)分别是两例HGG[(Brats18_CBICA_AAB_1,HGG1)和(Brats18_2013_10_1,HGG2)]和两例LGG[(Brats18_TCIA10_625_1,LGG1)和(Brats18_2013_9_1,LGG2)]病例未融合和融合后的分割对比图.
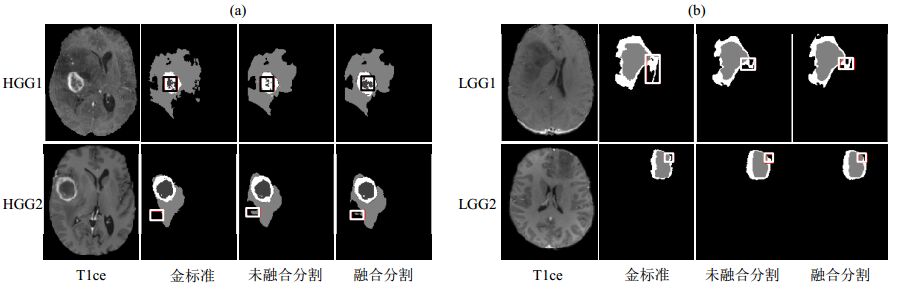
|
图 8 分割结果与金标准对比.(a) HGG;(b)LGG Fig. 8 The Tlce MR images, and segmentations of ground truth, non-fusion and fusion.(a) HGG; (b) LGG |
图 8(a)第一列为T1ce图像;第二列为各病例的金标准,即GT;第三列为未融合的HGG分割结果;第四列为融合后的HGG分割结果.其中,灰色代表水肿区域,白色代表增强区域,黑色代表坏死区域.由于Flair和T1的肿瘤区域模糊不清,而T2过于明亮,不利于肿瘤区域显示,故利用T1ce作对比.图 8(a)第一行未融合的分割结果与金标准相比,坏死区域和水肿区域边界影像特征不明显,增强区域与金标准接近,而融合后的分割结果坏死部分与金标准高度相似,提高了分割精度;第二行未融合的分割结果与金标准相比,增强区域和坏死区域与金标准相近,但水肿区域的边界假阳性较高,而融合后的水肿区域边界更接近金标准,减少了假阳性.
图 8(b)的第一列为T1ce图;第二列为各病例的金标准;第三列为未融合的LGG分割结果;第四列为融合后的LGG分割结果.其中,白色代表水肿区域,灰色代表核心区域.图 8(b)第一行未融合的分割结果与金标准相比,核心区域较接近,水肿区域边界出现欠分割,而融合后水肿区域边界更接近金标准,提高了分割准确率;第二行未融合的分割结果与金标准相比,水肿区域边界和核心区域出现过分割,而融合后水肿区域边界更接近金标准,提高了分割精度.可以看出本文算法分割结果与金标准相似度较高,对水肿区域基本能够分割准确,对核心区域分割略有偏差,整体分割效果较理想.
3.2 分割算法性能评价为进一步证明本文算法的有效性,分别选取其中10例HGG和10例LGG的水肿、核心区域和整体分割结果进行了定量评价,从表 2和表 3可看出,本文算法取得了较好的分割结果.未融合HGG和LGG水肿部分的平均DSC分别为85.52%和86.74%,而融合后分别为86.44%和89.30%;未融合HGG和LGG核心区域的平均DSC分别为83.76%和64.71%,而融合后该部分平均DSC分别为86.52%和74.96%;未融合HGG和LGG整体的平均DSC分别为86.16%和88.28%,而融合后分别为89.96%和91.31%.由于HGG的增强和坏死部分组织差异较明显,而LGG该影像特征不明显,各部分组织都比较模糊,故HGG相比于LGG的核心区域分割结果相对较好.
| 表 2 融合后和未融合HGG分割结果的DSC指标分析 Table 2 DSC analysis for HGG segmentation of fusion and non-fusion images |
| 表 3 融合后和未融合LGG分割结果的DSC指标分析 Table 3 DSC analysis for LGG segmentation of fusion and non-fusion images |
我们采用留出法随机分配Brats2018数据库中的数据,并将1/5的数据作为测试集,表 4定量对比了57例测试集融合后和未融合的平均分割结果,可看出融合后的分割结果明显优于未融合.本文算法的DSC的平均值达到85.92%,Sen为86.02%,PPV为83.97%.由于肿瘤的核心区域边界较清晰,而且融合后胶质瘤的核心区域得到增强,因此该部分准确率优于水肿区域.整体的分割结果还包括分割全脑与背景区域的精度,而全脑和背景区域的灰度差别较明显,故整体的平均DSC高于水肿区域和核心区域.融合后的图像在增加有效信息的同时,小部分噪声得到了增强,造成胶质瘤水肿区域出现边界模糊的情况,因此Sen和PPV的整体分割精度差距不明显.融合后,每例数据分割程序的平均运行时间为348 s,未融合每例数据分割程序的平均运行时间为399 s,平均运行时间约减少50 s,57例测试集分割的平均运行时间约减少2 850 s,整体运行速度约提高20%.
| 表 4 融合后与未融合结果性能对比 Table 4 Performance of comparison between fusion and non-fusion |
表 5定量对比了后处理阶段各个病变区域的平均分割结果.在实验的后处理阶段,首先生成二值图像,通过寻找中心像素的最大连通区域,得到每个病变的边界框,将不同的病变分离出来.后处理后的病变轮廓更清晰,平均DSC、Sen和PPV分别达到了85.92%、86.02%和83.97%,故采用基于连通区域的轮廓提取的结果优于处理之前.
| 表 5 后处理前后结果性能对比 Table 5 Performance comparison between before and after post-processing |
由于LGG坏死区域边界模糊不清,这里故将增强和坏死区域统一为核心区域,并计算了水肿区域的DSC.文献[30]应用MNI152中的脑分割图谱和人脑连接组计划中的纤维束追踪数据对Brats2018中的数据进行分割和生存预测,且结合了3D-UNet和难分负样本挖掘对像素分类,分割结果中核心和整体区域的DSC分别为74.90%和87.50%.该方法采用
本文提出了一种结合小波融合的3D-UNet网络实现脑胶质瘤及瘤内结构的分割算法.首先,将脑胶质瘤MRI的T1、T1ce、T2、Flair四种模态磁共振图像融合,可补充显示脑胶质瘤病灶区域且抑制病灶周围的冗余信息.其次,提取待分类的图像块,增加了输入网络的数据量,解决数据类别不平衡的问题.然后采用
| [1] | MENZE B H, JAKABA, BAUER S, et al. The multimodal brain tumor image segmentation benchmark (BRATS)[J]. IEEE Trans Med Imaging, 2015, 34(10): 1993-2024. DOI: 10.1109/TMI.2014.2377694. |
| [2] | DENG A, WU J, YANG S. An image fusion algorithm based on discrete wavelet transform and canny operator[M]. Berlin Heidelberg: Springer, 2011. |
| [3] | LEVINE M D, SHAHEEN S I. A modular computer vision system for picture segmentation and interpretation[J]. IEEE Trans Pattern Anal Mach Intell, 1981, 3(5): 540-556. DOI: 10.1109/tpami.1981.4767147. |
| [4] | SHIH F Y, CHENG S. Automatic seeded region growing for color image segmentation[M]. London: Butterworth-Heinemann, 2005. |
| [5] | YAMASAKI T. GrowCut-based fast tumor segmentation for 3D magnetic resonance images[C]//Medical Imaging: Image Processing. San Diego, California, USA: International Society for Optics and Photonics, 2012. https://www.spiedigitallibrary.org/conference-proceedings-of-spie/8314/1/GrowCut-based-fast-tumor-segmentation-for-3D-magnetic-resonance-images/10.1117/12.911649.short?SSO=1 |
| [6] | RANA R, BHADAURIA H S, SINGH A. Brain tumour extraction from MRI images using bounding-box with level set method[C]//Sixth International Conference on Contemporary Computing. Noida, India: IEEE, 2013. 10.1109/IC3.2013.6612212 |
| [7] | SELVAKUMAR J, LAKSHMI A, Arivoli T. Brain tumor segmentation and its area calculation in brain MR images using K-mean clustering and Fuzzy C-mean algorithm[C]//International Conference on Advances in Engineering. Singapore: IEEE, 2012. 10.1002/uog.14209 |
| [8] | CLARKE L P, VELTHUIZEN R P, CAMACHO M A, et al. MRI segmentation:methods and applications[J]. Magn Reson Imaging, 1995, 13(3): 343-368. DOI: 10.1016/0730-725X(94)00124-L. |
| [9] | LECUN Y, BENGIO Y, HINTON G. Deep learning[J]. Nature, 2015, 521(7553): 436-444. DOI: 10.1038/nature14539. |
| [10] | ZHANG W L, LI R J, DENG H T, et al. Deep convolutional neural networks for multi-modality isointense infant brain image segmentation[J]. Proc IEEE Int Symp Biomed Imaging, 2015, 108: 1342-1345. |
| [11] | LIAO S, GAO Y Z, OTO A, et al. Representation learning:A unified deep learning framework for automatic prostate MR segmentation[J]. Med Image Comput Comput Assist Interv, 2013, 16(2): 254-261. DOI: 10.1007/978-3-642-40763-5_32. |
| [12] | GUO Y R, GAO Y Z, SHEN D G. Deformable MR prostate segmentation via deep feature learning and sparse patch matching[J]. IEEE Trans Med Imaging, 2016, 35(4): 1077-1089. DOI: 10.1109/TMI.2015.2508280. |
| [13] | WU G R, KIM M, WANG Q, et al. Unsupervised deep feature learning for deformable registration of MR brain images[J]. Med Image Comput Comput Assist Interv, 2013, 16(2): 649-656. DOI: 10.1007/978-3-642-40763-5_80. |
| [14] | XIAO Z, HUANG R H, DING Y, et al. A deep learning-based segmentation method for brain tumor in MR images[C]//2016 IEEE 6th International Conference on Computational Advances in Bio and Medical Sciences (ICCABS). Atlanta, Georgia, USA: IEEE, 2016. 10.1109/ICCABS.2016.7802771 |
| [15] |
WANG H Z, ZHAO D, YANG L Q, et al. An approach for training data enrichment and batch labeling in AI+MRI aided diagnosis[J].
Chinese J Magn Reson, 2018(4): 447-456.
汪红志, 赵地, 杨丽琴, 等. 基于AI+MRI的影像诊断的样本增广与批量标注方法[J]. 波谱学杂志, 2018(4): 447-456. |
| [16] | HAVAEI M, DAVY A, WARDE-FARLEY D, et al. Brain tumor segmentation with deep neural networks[J]. Med Image Anal, 2015, 35: 18-31. DOI: 10.1016/j.media.2016.05.004. |
| [17] | PEREIRA S, PINTO A, ALVES V, et al. Brain tumor segmentation using convolutional neural networks in MRI images[J]. IEEE Trans Med Imaging, 2016, 35(5): 1240-1251. DOI: 10.1109/TMI.2016.2538465. |
| [18] | RONNEBERGER O, FISCHER P, BROX T. U-Net: Convolutional networks for biomedical image segmentation[C]//International Conference on Medical Image Computing and Computer-assisted Intervention. Munich, Germany: Springer, 2015.9351: 234-241. 10.1007/978-3-319-24574-4_28 |
| [19] | KAMNITSAS K, LEDIG C, NEWCOMBE V F J, et al. Efficient multi-scale 3D CNN with fully connected CRF for accurate brain lesion segmentation[J]. Med Image Anal, 2017, 36: 61-78. DOI: 10.1016/j.media.2016.10.004. |
| [20] | CASAMITJANA A, PUCH S, ADURIZ A, et al. 3D convolutional neural networks for brain tumor segmentation: a comparison of multi-resolution architectures[C]//International Workshop on Brainlesion: Glioma. Quebec, Canada: Springer, 2017. https://www.researchgate.net/publication/317087910_3D_Convolutional_Neural_Networks_for_Brain_Tumor_Segmentation_A_Comparison_of_Multi-resolution_Architectures |
| [21] | CHEN L L, WU Y, DSOUZA A M, et al. MRI tumor segmentation with densely connected 3D CNN[C]//Medical Imaging 2018: Image Processing. Houston, Texas, United States: International Society for Optics and Photonics, 2018, 10574: 105741F. 10.1117/12.2293394 |
| [22] | MEHTA R, ARBEL T. RS-Net: Regression-segmentation 3D CNN for synthesis of full resolution missing brain MRI in the presence of tumours[C]//International Workshop on Simulation and Synthesis in Medical Imaging. Granada, Spain: Springer, 2018. https://link.springer.com/chapter/10.1007/978-3-030-00536-8_13 |
| [23] | SZEGEDY C, VANHOUCKE V, IOFFE S, et al. Rethinking the inception architecture for computer vision[C]//2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Las Vegas, NV, USA: IEEE, 2016: 2818-2826. |
| [24] | IOFFE S, SZEGEDY C. Batch normalization:Accelerating deep network training by reducing internal covariate shift[J]. ICML, 2015. |
| [25] | CIAMPI M. Medical image fusion for color visualization via 3D RDWT[C]//IEEE International Conference on Information Technology & Applications in Biomedicine. Corfu, Greece: IEEE, 2010. |
| [26] |
CHAI Q H, SU G Q, NIE S D. Compressive sensing low-field MRI reconstruction with dual-tree wavelet transform and wavelet tree sparsity[J].
Chinese J Magn Reson, 2018, 35(4): 486-497.
柴青焕, 苏冠群, 聂生东. 双树小波变换与小波树稀疏联合的低场CS-MRI算法[J]. 波谱学杂志, 2018, 35(4): 486-497. |
| [27] | SRIVASTAVA N, HINTON G, KRIZHEVSKY A, et al. Dropout:A simple way to prevent neural networks from overfitting[J]. J Mach Learn Res, 2014, 15(1): 1929-1958. |
| [28] | BOCK S, GOPPOLD J, WEI M. An improvement of the convergence proof of the ADAM-optimizer[J]. Computer Science, 2018. arXiv: 1804.10587 https://arxiv.org/abs/1804.10587 |
| [29] | TUSTION N J, AVANTS B B, COOK P A, et al. N4ITK:improved N3 bias correction[J]. IEEE Trans Med Imaging, 2010, 29(6): 1310-1320. DOI: 10.1109/TMI.2010.2046908. |
| [30] | KAO P Y, NGO T, ZHANG A, et al. Brain tumor segmentation and tractographic feature extraction from structural MR images for overall survival prediction[C]//International MICCAI Brainlesion Workshop. Granada, Spain: Springer, 2018. https://link.springer.com/chapter/10.1007/978-3-030-11726-9_12 |
| [31] | WENINGER L, RIPPEL O, KOPPERS S, et al. Segmentation of brain tumors and patient survival prediction: methods for the BraTS 2018 challenge[C]//International MICCAI Brainlesion Workshop. Granada, Spain: Springer, 2018. https://link.springer.com/chapter/10.1007/978-3-030-11726-9_1 |
 2020, Vol. 37
2020, Vol. 37 
