2. 中国科学院计算技术研究所, 北京 100190;
3. 复旦大学附属华山医院, 上海 200040;
4. 西门子医疗系统有限公司, 上海 201318;
5. 上海理工大学 光电信息与计算机工程学院, 上海 200093
2. Institute of Computing Technology, Chinese Academy of Sciences, Beijing 100190, China;
3. Huashan Hospital, Fudan University, Shanghai 200040, China;
4. Siemens Healthineers, Shanghai 201318, China;
5. School of Optical-Electrical and Computer Engineering, University of Shanghai for Science and Technology, Shanghai 200093, China
众所周知,对于任何领域的人工智能(AI)研究或者产品开发而言,训练样本的获取都是核心问题之一.没有训练样本,再好的AI算法也无法实现其价值;AI产品的质量是由其所拥有的训练样本数量所决定的[1].
目前关于AI的技术开发有个误区,即认为AI主要做神经网络构建和编程工作.其实不然,AI从业者2/3以上的工作量是在获取或标注训练样本.Google等公司不断向世界分享它们在算法和神经网络结构上的最新成果,但对其训练数据集却很少公开,即使公开也只是其中很少一部分[2].由此可见,神经网络构建和算法对于AI固然重要,但是如何获取更多的训练样本可能更重要.目前AI领域对于算法和网络构建倾注了全力,但关注训练样本的人却很少.对于公知领域的数据样本勾画和标注,只需要普通劳务人员即可实现,因此出现了一批专门为AI服务的数据公司,甚至是产业园.但是对于需要专业知识的样本获取和标注,依靠通用数据公司是无法完成的.
磁共振图像的特点决定了其标准图像种类众多,同一种疾病的图像种类更多.现阶段对于基于人工智能+磁共振成像(AI+MRI)的影像诊断,解决训练数据短缺的问题是当务之急,需要更多的志愿者或阳性病例来更快地训练提升AI影像的性能. AI+MRI迫切需要快速高效的标准图像和异常标注样本图像的输入[3].输入学习的样本种类越多,AI对某张具体图像的分析判断才越准确.
但现有的AI+MRI影像诊断研究与产品化过程中,存在着训练样本数量和样本类型无法满足需求的瓶颈问题.主要原因如下:
(1)依靠医院端获取的大量病例存在着医疗数据安全的政策法规等具体问题[4].(2)医院和医生对于需要花费大量时间和精力提供大量标注样本进行产品开发缺乏动力.(3)现有医疗数据标准化没有形成,质量参差不齐.(4)大样本量的采集时间和高成本也是AI+MRI诊断产品化的障碍.相对于数字放射摄影(DR)、计算机断层扫描(CT)的单参数图像,磁共振图像是多参数图像,除了常规的多种权重图像外,还有灌注、弥散、血流等图像,以及各种定量图像,如弛豫时间定量、化学参数定量、生理参数定量、化学交换饱和转移(CEST)、磁化率敏感等.即使是常规图像,不同厂家的序列参数不同,图像也是有差异的.也就是说,正常组织同一个部位的标准图像已有多种,非正常组织的图像数量就进一步增加.对AI而言,扫描和标注更多种类的磁共振图像无疑意味着经费和时间的巨大消耗.但是,若AI学习的图像样本不够多,其适应性和准确性也就难以提高,这也是AI+MRI难以产品化的原因之一.
此外,与公知领域的AI应用(比如基于语音识别的同译、基于人脸识别的安防、基于场景图像识别的无人驾驶)相比,AI医学影像是一个需要专业标注的领域.国内外研究小组对这个问题进行了一些解决方法的探索.比较常用的是借鉴公知领域处理一些不常见物体(如飞机)的方法,比如对一张飞机图片进行拉伸、旋转、色彩变幻、叠加云彩或噪声等方法生成其他样本,进行样本增广.上海交通大学人工智能实验室[5]通过旋转平移等几何变换针对有限数量的正样本作数据多样性增广,类似于医生通过不同的视角、不同的上下文去分析结节区域,并利用生成对抗网络(GAN),从随机噪声中产生新的结节正样本,学习生成新形态的结节样本,深度增广数据的多样性,提升模型泛化能力.Jenni等人[6]在AI识别无脊椎动物的训练样本时,采用了类似多角度拍摄样本照片的方法实现样本增广.在人脸识别方面,对一张人脸通过GAN生成不同表情人脸图像,进行增广样本,但其是否能提升性能还是存疑的.但这两种方法不适用于MRI样本增广.也有一些研究小组另辟蹊径,通过其他办法获取大样本.比如被誉为人工智能创始人之一的斯坦福大学吴恩达(Andrew Ng)小组[7]开源了含有4万张人体上肢端(包括肩膀、肱部、手肘、前臂、手腕、手、指端)的X光片的数据集(命名为MURA),并用这个数据集训练卷积神经网络,用以寻找并定位X光片的异常部位.最后训练结果是,该模型在手指和手腕X光片的诊断表现比放射科医生好,但其他部位则比放射科医生差.值得注意的是,这4万张图来自近15 000篇论文,其中9 067篇为正常上肢骨骼肌X光片的研究,5 915篇为异常上肢骨骼肌的研究.即该团队不是直接从医院获取医疗影像,而是从公开渠道获取样本.可见不同AI应用领域,获取样本和增广样本的方法都是不同的,需要根据具体领域知识采用针对性的增广方法.
本文利用临床MRI设备对志愿者或阳性病例进行正常或重点病灶区的定量扫描,将获取的高分辨率的各向同性的纵向弛豫时间(T1)、横向弛豫时间(T2)、质子密度(Pd)和表观扩散系数(ADC)等物理信息的多维数据矩阵作为原始数据样本.同时,我们开发了虚拟MRI技术平台,对原始样本进行虚拟扫描,实现不同序列和不同参数下的多种类磁共振图像输出.利用该平台,只需选取边界区分度好的一种类型图像,建立MASK矩阵,就可低成本、高效率的产生多类型标注图像样本.
1 基本理论与方法本工作的基本思路是:通过定量MRI技术获取正常志愿者和少量阳性病例重点病灶区的物理信息,然后利用虚拟MRI技术对正常或病灶区信息进行虚拟数据采集和成像,基于不同的成像序列和参数,输出大量磁共振图像.这些图像在分辨率、信噪比、权重、b值等方面有所不同,可极大增广训练样本的种类和数量.由于信息来源于同一个病例,因此只需要一次标注即可覆盖本次生成的所有样本标注.主要流程如图 1所示.
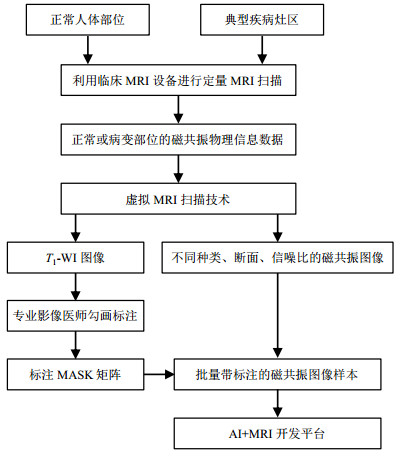
|
图 1 AI+MRI训练样本自动增广和批量标注平台开发流程 Figure 1 Platform development diagram for AI+MRI training data enrichment and batch labeling |
正常人体组织(后续可采集重点病灶)区域的定量MRI物理、化学及生物信息可通过高场临床MRI扫描仪来获取.采用不同的定量扫描技术,可以实现不同信息的定量测量.比如通过T1 Mapping技术,获得每个体素的T1信息;通过T2 Mapping技术,获得每个体素的T2信息;通过多b值的扩散加权图像可以得到每个体素的ADC信息.本项目中,定量扫描的数据要作为后续虚拟MRI扫描的样品模型,需要包括整个病灶区,并且要求是无间隔扫描,同时空间分辨率最好达到1 mm*1 mm* 1 mm.这对扫描技术提出了很高的要求.本项目采用西门子Prisma3T MRI扫描仪作为采集设备,具体扫描技术和参数如下:
(1)T1 Mapping使用MP2RAGE序列,脉冲间隔重复时间(TR)=5 000 ms,回波时间(TE)=2.98 ms,反转时间(TI)=707/2 500 ms,翻转角(flip angle)=4°/5°,成像视野(FOV)=240×256 mm2,192 sagittal slices, voxel size=1×1×1 mm3;
(2)T2 Mapping使用multiple spin echo序列,TR=9 650 ms,TE= 13.8/27.6/41.4/55.2/69.0 ms,flip angle=180°,FOV=256×256 mm2,128 sagittal slices, voxel size=1×1×2 mm3;
(3)ADC Mapping使用RESOLVE序列,TR= 20 040 ms,TE= 67 ms,flip angle=180°,b values =0/1 000 s/mm2,FOV=256×256 mm2,128 sagittal slices,voxel size=1×1×2 mm3,读出方向分段采集.
1.2 虚拟MRI扫描技术模型建立虚拟MRI扫描技术可以按照二维快速傅里叶变换(2D-FFT)成像理论模型进行模型物理信息原始信号的虚拟采集,进行K空间填充后再经FFT变换得到磁共振图像.基于Hunchison的2D-FFT成像理论,进行虚拟数据采集后的原始K空间数据S是一个四重积分:
| $ S[n, m, k, l] = k{M_{\rm{0}}}\sum\limits_{n = 0}^{N - 1} {\sum\limits_{m = 0}^{M - 1} {\sum\limits_{k = 0}^{N - 1} {\sum\limits_{l = 0}^{M - 1} \rho (k, l)} } } {{\rm{e}}^{{\rm{j}}(2{\rm{ \mathsf{ π} }}/N)kn}}{{\rm{e}}^{{\rm{j}}(2{\rm{ \mathsf{ π} }}/M)ml}} $ | (1) |
M、N分别表示相位编码步数和频率编码的采样点数;k、l表示二维的空间积分项,分别表示选层后的样品的横向(与频率编码方向一致)和纵向(与相位编码方向一致)空间位置;n、m表示二维时间积分项,分别表示频率编码时间和相位编码时间;KM0表示体素的磁化矢量大小.图像重建则对(1)式中的S进行FFT即可.由于实际AI应用时会遇到不同信噪比的图像,因此可在(1)式的信号上叠加不同程度的噪声来获取有信噪比差异的图像样本.
对于不同序列的虚拟数据采集,需要将(1)式中的KM0项进行相应修改来实现,如:
(1)自旋回波(SE)序列,KM0为:
| $ k{M_0} = A\rho ({\rm{H}})[1 - {\rm{exp}}( - TR/{T_1})]{\rm{exp}}( - TE/{T_2}) $ | (2) |
其中,ρ(H)为质子密度,对应原始数据的Pd信息;A为放大倍数.
(2)梯度回波(GRE)序列,kM0分为去除剩余磁化(FLASH)和利用剩余磁化(bFFSP)两类,分别为:
| $ k{M_0} = A\rho ({\rm{H}})\frac{{{\rm{exp}}( - TE/T_2^*)[1 - {\rm{exp}}( - TR/{T_1})]\sin \alpha }}{{1 - {\rm{exp}}( - TR/{T_1})\cos \alpha }} $ | (3a) |
| $ k{M_0} = A\rho ({\rm{H}})\frac{{{\rm{exp}}( - TE/T_2^*)[1 - {\rm{exp}}( - TR/{T_1})\sin \alpha ]}}{{1 - {\rm{exp}}( - TR/{T_1}){\rm{exp}}( - TR/{T_2}) - \left[ {{\rm{exp}}( - TR/{T_1}) - {\rm{exp}}( - TR/{T_2})\cos \alpha } \right]}} $ | (3b) |
(3)反转恢复(IR)序列,kM0为:
| $ k{M_0} = A\rho ({\rm{H}})\left[ {1 - 2{\rm{exp}}( - TI/{T_1})} \right]\{ 1 - {\rm{exp}}[ - (TR - TI)/{T_1}]\} {\rm{exp}}( - TE/{T_2}) $ | (4) |
(4)扩散加权成像(DWI)序列,可在(2)式基础上,增加exp(-bD)因子体现不同b值的衰减影响即可,D即为原始数据中的ADC.
其他序列的虚拟采集均可以通过完善数据填充模型和对应的信号幅度规律来实现.在以下研究中,我们将正常人脑进一步分隔为灰质、白质、脑脊液三种组织类型,并对三种组织各自统一赋典型值后作为原始信息进行研究,这些结果可以普适性地拓展到其他细分组织及三维像素水平模板,以及全身其他部位的扫描数据输入.
1.3 MRI虚拟扫描平台开发完成物理信息和虚拟数据采集模型建模之后,可以通过图形用户界面(GUI)编程实现类似真机的数据采集和图像重建界面.由于matlab具备强大的数据矩阵运算能力,因此本项目采用matlab2016a版本来实现GUI编程,实现后的主功能界面如图 2所示:
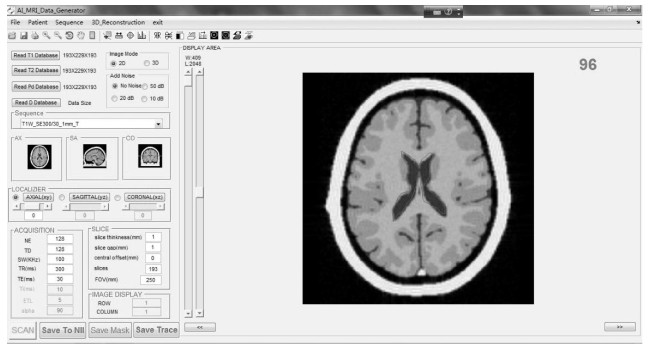
|
图 2 虚拟MRI扫描和训练样本标注平台的主功能界面 Figure 2 The operation interface of virtual MRI scanning and training data labeling platform |
软件功能包括读取原始样本的各种三维物理信息数据(T1、T2、Pd、ADC等).可以按照样本需求,选择2D或3D模式,也可以选择不同噪声程度选项,以及不同的虚拟扫描序列和参数组合.如T1WI_SE300/30_1 mmT,表示采用SE序列、TR=300 ms、TE=30 ms、1 mm层厚的横断面T1权重像.对于2D样本模式,则可选择不同断面(横断面、矢状面、冠状面)以及相应的倾斜角度.此后可对相应序列参数和结构参数进行设置,最后单击扫描(SCAN)按钮,即可显示相应的图像样本.可以对获得的图像进行基本处理,包括测距、放大缩小、移动、直方图显示、图像旋转、负片、电影浏览、直方图均衡、窗宽窗位调节和勾画标注等.基本处理后,如果是2D模式,可通过菜单栏下的save as DICOM菜单将图像保存为标准的医学图像格式.如果是3D模式,可单击Save to NII按钮,将三维图像存储为nii格式.经过医师逐层勾画后,可单击Save Mask将勾画结果保存为标注模版数据库,与样本一起作为训练样本输送给AI系统.
2 结果与讨论我们开发的自动MRI训练样本图像生成和批注平台,在读入原始样本物理信息后,选择不同序列和序列参数,可虚拟扫描得到不同类型、任意权重、任意断面的磁共振图像,如T1WI、T2WI、T2*WI、PDWI、STIR、T1-Flair、T2-Flair、black water、water image和DWI等等.
图 3(a)~3(e)分别为正常脑部组织同一断面物理信息经虚拟扫描得到的T1WI、T2WI、T1-Flair、T2-Flair和STIR图像.
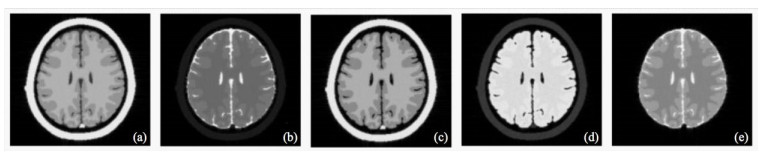
|
图 3 正常脑部组织同一层面采用不同序列进行虚拟MRI扫描获得的图像. (a) T1WI; (b) T2WI; (c) T1-Flair; (d) T2-Flair; (e) STIR Figure 3 The images of a normal brain model for the same slice acquired by virtual MRI scanning using different sequences (a) T1WI; (b) T2WI; (c) T1-Flair; (d) T2-Flair; (e) STIR |
通过本平台在正常颅脑模型基础上构建一个脑膜瘤模型,经过不同序列虚拟MRI扫描可得到同一种病变的灰度对比差异如图 4(a)~4(d)所示.
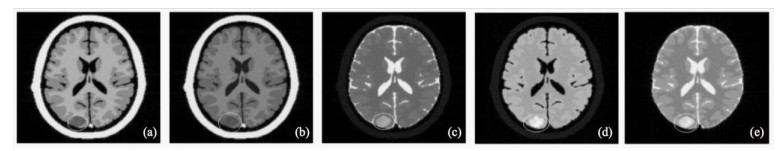
|
图 4 脑膜瘤组织模型同一层面采用不同序列进行虚拟MRI扫描获得的图像. (a) T1WI; (b) T2WI; (c) T1-Flair; (d) T2-Flair; (e) STIR Figure 4 The images of a meningioma model for the same slice acquired by virtual MRI scanning using different sequences. (a) T1WI; (b) T2WI; (c) T1-Flair; (d) T2-Flair; (e) STIR |
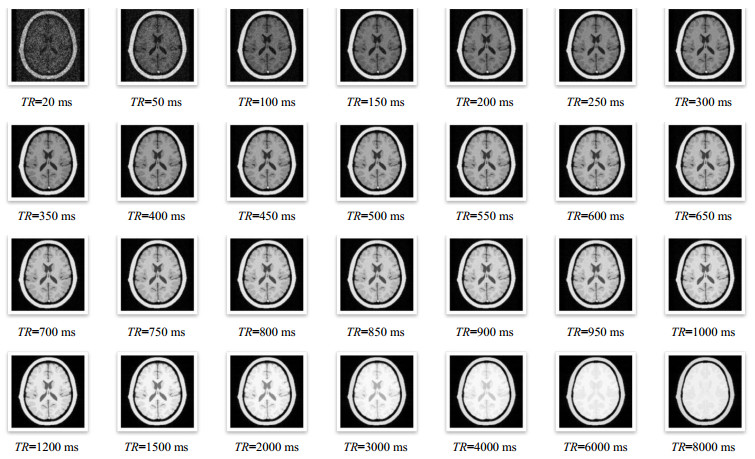
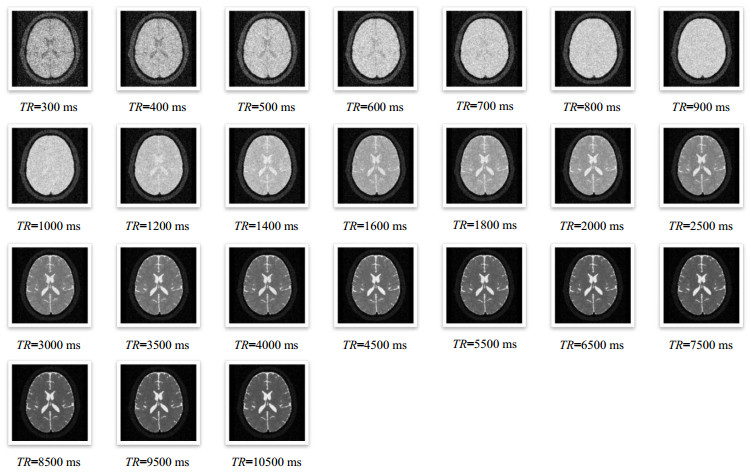
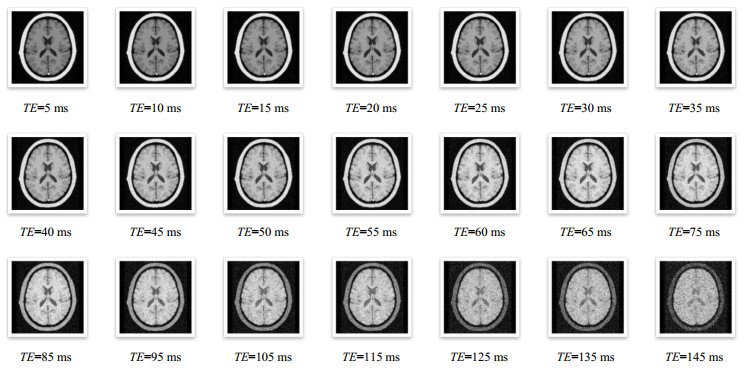
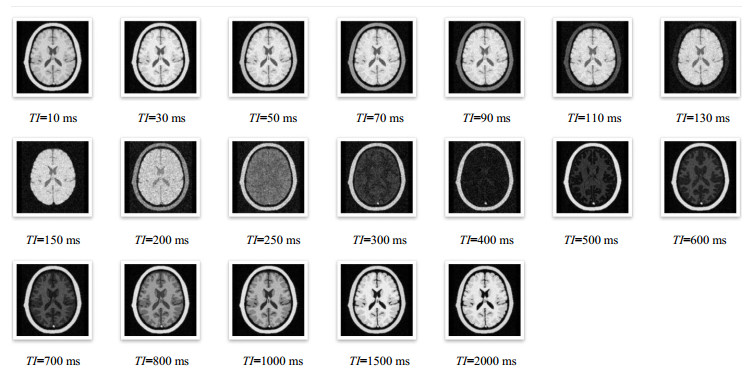
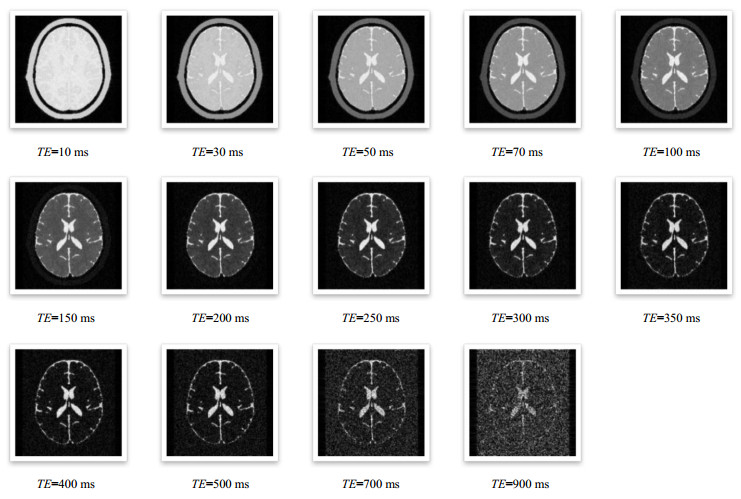
在不同序列参数下,同种模型利用同种序列也可以得到对比度和信噪比逐渐变化的图像.这种效果差异可以弥补设备差异、技师偏好等带来的实际图像差异.图 5所示为正常人脑SE序列图像随参数变化(TE=10 ms,TR=20~8 000 ms)从T1WI到PDWI变化效果图;图 6为正常人脑SE序列图像随参数变化(TE=145 ms,TR=300~10 500 ms)的T2WI变化效果图;图 7为正常人脑SE序列图像随参数变化(TR=100 ms和200 ms,TE=5~145 ms)的图像效果变化图;图 8为正常人脑IR序列图像随参数变化(TR=500 ms,TE=30 ms,TI=10~2 000 ms)的变化效果图;图 9为正常人脑SE序列图像随参数变化(TR=10 500 ms,TE=10~900 ms)的水成像效果图.
|
图 5 正常人脑SE序列图像随参数(TE=10 ms, TR=20~8 000 ms)从T1权重像到质子权重像的变化 Figure 5 Gradual chang from T1-weighted images to proton weighted images of normal human brain acquired by virtual MRI scanning using different SE sequence parameters (TE=10 ms, TR=20~8 000 ms) |
|
图 6 正常人脑SE序列T2权重像随参数变化(TE=145 ms, TR=300~10 500 ms) Figure 6 The T2-weighted images of normal human brain acquired by virtual MRI scanning using different SE sequence parameters (TE=145 ms, TR=300~10 500 ms) |
|
图 7 正常人脑SE序列图像随参数变化(TR=100 ms, TE=5~65 ms; TR=200 ms, TE=75~145 ms) Figure 7 The images of normal human brain acquired by virtual MRI scanning using different SE sequence parameters (TR=100 ms, TE=5~65 ms; TR=200 ms, TE=75~145 ms) |
|
图 8 正常人脑IR序列随参数变化(TR=500 ms,TE=30 ms,TI=10~2 000 ms)的图像 Figure 8 The images of normal human brain acquired by virtual MRI scanning using different IR sequence parameters (TR=500 ms, TE=30 ms, TI=10~2 000 ms) |
|
图 9 正常人脑SE序列水成像效果图随参数变化(TR=10 500 ms,TE=10~900 ms) Figure 9 The water imaging of normal human brain acquired by virtual MRI scanning using different SE sequence parameters (TR=10 500 ms, TE=10~900 ms) |
本研究有助于解决基于AI+MRI的影像诊断研究中样本需求量大与临床实际难以提供大量标注样本的客观矛盾,时解决了临床医生提供大量样本的劳动力过大、动力不足,以及AI数据需求方数据资源花费巨大的问题.对于单一病例,进行一次标注后,后续增广的图像无需再次手工标注,可以大量节省人力、时间和财力.
除了一次标注覆盖同一病例全部样本图像之外,本方法进行虚拟样本增广的效率也远远高于临床设备.一般使用临床设备获取3~5种图像序列(每个序列15张图像),耗时为8~20 min.经测试,本方法增广一种序列的193张图像,采集时间约为5 s.一个正常人体全脑的定量扫描时间约0.5 h.如果仅针对重点灶区,定量扫描时间为8~10 min.理论上,任意参数组合可虚拟采集得到无限多幅图像,但为了避免AI训练时出现过拟合,经过定量扫描后虚拟扫描的图像种类选择50种.
人类疾病有2 000多种,目前的AI+影像辅助诊断模式中,都是针对某一种疾病进行特定的样本训练.因此针对每种疾病的AI诊断,都需要获得相应的数据集.本研究成果作为一种通用型磁共振图像样本增广工具,后续只需要采集一定数量的不同程度该疾病阳性病例的物理信息数据,再借助专业影像医生的一次勾画标注,即可获得批量的带标注的训练样本图像.
相比人体MRI技术而言,目前动物(如宠物狗、赛马、警太等动物)相关的MRI技术、疾病图谱以及诊断的基础更加薄弱,技术人员和医生更加稀少,且检查价格更高,样本量也更稀少.因此本研究除了可以对于人体疾病开展AI+影像诊断样本增广和标注外,也可以满足宠物的AI诊断应用.应用本方法和平台产生磁共振脑图像样本集已经用于超分辨图像的AI应用中,其效果优于采用自然图像效果.本数据集仅用于AI研究使用,如采用请注明出处.
| [1] | ELIZEBETH G. AI firms lure academics[J]. Nature, 2016, 532(4): 422-423. |
| [2] | TING D S W, LIU Y, BURLINA P, et al. AI for medical imaging goes deep[J]. Nat Med, 2018, 24(5): 539-540. DOI: 10.1038/s41591-018-0029-3. |
| [3] | MAXMEN A. AI researchers embrace Bitcoin technology to share medical data[J]. Nature, 2018, 555(7696): 293-294. DOI: 10.1038/d41586-018-02641-7. |
| [4] | JIANG K L, GIUSEPPE C, RAYMOND T. Training data enrichment for infrequent dis-course relations//Proceedings of COLING 2016[C]. Osaka: Proc Intl Soc, 2016. |
| [5] | JENNI R, EKATERINA R, KRISTIAN M, et al. Data enrichment in fine-grained classification of aquatic macroinvertebrates//2016 ICPR 2nd Workshop on Computer Vision for Analysis of Underwater Imagery[C]. Cancun: Proc Intl Soc, 2016 |
| [6] | ILVEL D, HUANG A, MADDISON C, et al. Mastering the game of Go with deep neural networks and tree search[J]. Nature, 2016, 529(7587): 484-489. DOI: 10.1038/nature16961. |
| [7] | RAJPURKAR P, IRVIN J, BAGUL A, et al. MURA Dataset: Towards radiologist-level abnormality detection in musculo-skeletal//1st Conference on Medical imaging with Deep Learning[C]. Amsterdam, 2018 |
 2018, Vol. 35
2018, Vol. 35 
