2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
肠道菌群是寄居在宿主肠道所有微生物聚集体的总称,其种类繁多,数目惊人[1].它作为“超级生物体”(superorganism),与宿主形成了紧密的共生关系.大量研究表明,肠道菌群分布和功能的改变与多种疾病,如内分泌代谢性疾病、肾脏疾病、神经性疾病、心血管疾病以及癌症[2-4]的发生发展密切相关,原因在于肠道菌群不仅有助于宿主从食物中获取营养物质和能量,还可产生许多重要信号因子和能量底物,如胆汁酸(BA)、胆碱、短链脂肪酸等.这些小分子代谢物又影响着宿主的免疫、消化、能量代谢和营养等许多生理活动,其中胆汁酸作为体内代谢通路“肝肠循环”的重要参与者而得到了更多关注.
胆汁酸是宿主胆固醇代谢过程中所产生的一系列固醇类物质.按其结构可分为结合胆汁酸和游离胆汁酸,按其来源可分为初级胆汁酸和次级胆汁酸.其中约90%的胆汁酸通过肝肠循环与牛磺酸和甘氨酸共轭形成胆酸盐.大约5%~10%的胆汁酸的生物转化依赖于肠道细菌的降解,还有一些以粪便形式排出.它们不仅能乳化脂肪、促进脂溶性物质的消化与吸收,还可与细菌细胞膜上的磷脂结合破坏菌膜,达到抗细菌粘附和中和内毒素的效果,从而抑制小肠细菌的过度生长[5].在肠道中,各种形式的胆汁酸充分发挥各自的生理功能.其中,肠道上段胆汁酸主要与脂类的消化吸收有关,肠道下段胆汁酸在肠道菌群作用下发生转化,并在肠粘膜中按主动运输或被动运输机理被重新吸收,一小部分随食物残渣排出体外.同时,胆汁酸可以作为重要的信号分子,通过激活核法尼醇X受体(FXR)和G蛋白偶联膜受体-5(TGR-5)等依赖的信号转导通路[6, 7],调节体内的能量代谢平衡和炎症反应.此外,胆汁酸还可以通过激活肠道免疫应答基因直接或间接调节肠道微生物的组成[8].随着人们对宿主肠道菌群的关注日益增加,胆汁酸与宿主肠道菌群之间相互依赖、相互竞争、相互抑制的复杂关系在多个工作中被报道[9].研究发现,胆汁酸代谢物的异常表达与体内肠道系统疾病[10]、非酒精性脂肪肝[11]、代谢性疾病[12]、癌症[13]、神经退行性疾病[14]等密切相关.而且,调节宿主胆汁酸的水平可以缓解或治疗上述疾病的发生[15, 16].前人的研究表明,胆汁酸的分布及表达与多种因素如宿主的年龄、饮食和身体质量指数相关[17],且胆汁酸代谢组呈现明显的性别差异性应答[18].此外,因肠道组织结构及肠道菌群分布的差异,不同肠段对营养物质的吸收以及外源性药物的蓄积也各不相同[19].因此,生理条件下不同性别宿主肠道各区段及粪样提取物胆汁酸分布、表达及其生物学功能的探寻对研究肠道微生态相关的疾病显得非常必要.
随着人们对胆汁酸检测技术的不断提高以及对胆汁酸认识的不断深入,近年来发展了不少检测胆汁酸的方法[20, 21].本文选用了实验室常用的模型动物——C57BL/6小鼠为动物模型,采取较为成熟的基于超高效液相色谱-三重四极杆-质谱联用(UPLC-QqQ-MS)靶向代谢组分析与基于核磁共振(NMR)非靶向代谢组分析相结合的检测方法,通过比较同年龄不同性别C57BL/6小鼠肠道各区段不同胆汁酸的含量及粪样提取液的代谢组,研究生理状态下雌、雄小鼠肠道菌群相关代谢物的分布规律及差异.本研究将为不同性别模型小鼠胆汁酸的代谢表型及其与肠道菌群的相互作用提供基础研究数据.
1 实验部分 1.1 药品与化学试剂本实验所用的29种胆汁酸标准品及其氘代内标均购于美国Steraloids Inc.主要包括胆酸(CA)、鹅脱氧胆酸(CDCA)、脱氧胆酸(DCA)、石胆酸(LCA)、熊脱氧胆酸(UDCA)、α-鼠胆酸(α-MCA)、β-鼠胆酸(β-MCA)、ω-鼠胆酸(ω-MCA)、猪脱氧胆酸(HDCA)、别胆酸(ACA)、猪胆酸(HCA)、3-羟基-7-氧代-5β-胆酸(NCA)、甘氨脱氧胆酸(GDCA)、甘氨鹅脱氧胆酸(GCDCA)、甘氨石胆酸(GLCA)、甘氨熊脱氧胆酸(GUDCA)、甘氨胆酸(GCA)、甘氨猪脱氧胆酸(GHDCA)、牛磺胆酸(TCA)、牛磺脱氧胆酸(TDCA)、牛磺熊脱氧胆酸(TUDCA)、牛磺石胆酸(TLCA)、牛磺α-鼠胆酸(T-α-MCA)、牛磺β-鼠胆酸(T-β-MCA)、牛磺ω-鼠胆酸(T-ω-MCA)、牛磺猪胆酸(THCA)、12-酮脱氧胆酸(12-keto DCA)、异脱氧胆酸(iso-DCA)和异石胆酸(iso-LCA).[2, 2, 4, 4-D4]-DCA(DCA-d4)、[2, 2, 4, 4-D4]-CA(CA-d4)、[2, 2, 4, 4-D4]-CDCA(CDCA-d4)、[2, 2, 4, 4-D4]-LCA(LCA-d4)、[2, 2, 4, 4-D4]-GCDCA(GCDCA-d4)和[2, 2, 4, 4-D4]-GCA(GCA-d4)作为内标用于后期的定量分析.质谱分析用的HCOOH、高效液相色谱(HPLC)纯级的CH3CN和CH3OH采购自德国Sigma-Aldrich Co.超纯水(电阻率大于18.2 MΩ·cm)取自德国Merck Millipore Elix Advantage超纯水仪.
重水(D2O,99%氘代)购于德国Sigma-Aldrich Co;2, 2, 3, 3-氘代三甲基硅烷丙酸钠(TSP)购于美国Cambridge Isotope Laboratories, Inc.;分析纯级NaN3购自福晨(天津)化学试剂有限公司;分析纯级K2HPO4·3H2O和NaH2PO4·2H2O购自上海国药集团化学试剂有限公司.
1.2 动物实验及样品收集无特定病原体(SPF)级别C57BL/6小鼠由南京大学模式动物研究所提供,共计26只(其中雌性小鼠12只,雄性小鼠14只),饲养于武汉大学实验动物中心,实验动物设施使用证明编号为SYXK(鄂)398-2006.动物随机分笼饲养,自由饮食饮水,饲养环境温度为20~25 ℃,湿度为50%~70%,每12 h光照-黑暗交替一次.待实验动物饲养至9月龄时将其处死,并按如下分段标准收集小鼠肠壁组织:胃幽门部往后约12 cm作为十二指肠;剩下肠段分3份,前2/3作为空肠,后1/3作为回肠.盲端组织视为盲肠;盲肠部往下约3 cm作为结肠.收取每部分肠道中段的1~2 cm组织样品用于检测.将收集到的肠段组织的内容物挤出,然后用小剪刀将肠壁组织纵向剪开,在生理盐水中漂洗两遍后用滤纸蘸干水分,液氮速冻后置于-80 ℃冰箱保存.小鼠粪样于动物处死前一天进行收集,液氮速冻后置于-80 ℃冰箱保存.
1.3 样品制备 1.3.1 肠壁组织胆汁酸样品制备内标溶液配置:溶剂由甲醇和水组成(V甲醇:V水=9:1),含浓度为0.5 μmol/L的DCA-d4、CA-d4、CDCA-d4、LCA-d4、GCDCA-d4和GCA-d4共6种氘代内标.
称取5 mg左右肠壁组织,加入1 mL提取液(V甲醇:V水=2:1,含0.005% HCOOH,-20 ℃预冷24 h)、50 μL内标溶液,涡旋混匀,使用组织破碎仪(QIAGEN,Germany)进行匀浆(20 Hz,90 s);对匀浆后的样品使用液氮快速冻融三次,再次匀浆(20 Hz,90 s)后离心(10 000 g,4 ℃)15 min;将所得上清进行过滤(尼龙66针式过滤头,直径为13 mm,孔径为0.22 μm);最后将滤液转移至色谱样品瓶中待UPLC-QqQ-MS检测.
1.3.2 粪样提取物样品制备参照优化的实验方法[22]进行粪样代谢物提取.称取约50 mg小鼠粪便,加入600 μL 0.1 mol/L磷酸盐缓冲液[pH = 7.4;V水:V重水 = 7:3;含0.1 mol/L K2HPO4·3H2O和NaH2PO4·2H2O,以及0.03 mol/L NaN3和0.002%(w/v)的TSP].涡旋30 s混匀后液氮冻融三次;然后使用组织破碎仪进行匀浆(20 Hz,90 s);离心(15 930 g,4 ℃)10 min取上清;重复一次上述提取过程,并将两次上清液混合后再次进行离心;最后取600 μL上清至NMR样品管待NMR检测.
1.4 样品检测 1.4.1 肠壁组织中胆汁酸UPLC-QqQ-MS检测肠壁组织中胆汁酸UPLC-QqQ-MS采样参数参照文献[23],并进行了优化.使用1290 UPLC-6460 QqQ联用仪(Agilent Technologies, USA),配备Agilent Jet Stream电喷雾离子源.色谱柱采用Kinetex® Core-Shell 2.6 µm C18 column(100 mm×2.1 mm, 2.6 µm, Phenomenex, USA).进样量为1 µL,柱温设置为45 ℃.流动相A为含有0.005% HCOOH的超纯水,流动相B为含有0.005% HCOOH的CH3CN,流速为0.6 mL/min.洗脱梯度为:0~2 min,77%~67% A;2~6 min,67%~66% A;6~11 min, 66%~30% A.因胆汁酸离子化时更容易产生准分子离子峰[M-H]-,在负离子检测模式下有更强的响应,因而我们选择各胆汁酸的[M-H]-作为其多反应监测(MRM)模式下的子离子,以提高其检测灵敏度.干燥气温度是350 ℃,干燥气流量为10 L/min,雾化气压为20 psi,鞘气温度为350 ℃,鞘气流量为10 L/min,毛细管电压为3 500 V.
1.4.2 粪样提取物NMR检测粪样提取物的NMR信号采集在配备超低温探头的Bruker AVIII 600 MHz NMR谱仪上完成.实验温度为298 K,采用noesygppr1d(RD-90˚-t1-90˚-tm-90˚-ACQ)脉冲序列.等待时间(RD)设为2 s,混合时间(tm)设为100 ms,t1为4 μs,90˚脉宽(p1)约为10 μs,采样时间(AQ)是1.36 s,采样点数(TD)为32 k,累加扫描128次.
1.5 数据分析 1.5.1 胆汁酸组成的数据分析将所采集色谱质谱图导入Mass Hunter软件(Agilent Technologies, USA),对积分面积进行手动微调,并用内标法得到胆汁酸含量.以各肠段组织湿重对胆汁酸含量进行归一化,通过SPSS软件(V20.0,IBM,USA)进行独立样本双尾t检验或非参数检验(95%置信度),得到不同性别C57BL/6小鼠各肠道组织胆汁酸浓度的差异,p < 0.05提示组间有统计学差异.
1.5.2 粪样提取物数据分析粪样提取物1H NMR谱图经傅里叶变换、基线和相位校正后,以TSP进行化学位移定标(δ 0.00).利用Amix软件对谱图进行积分,积分区间为δ 0.5~9.5(去除δ 4.67~5.03区间的水峰残余信号),积分间隔为1.2 Hz.对所得积分数据采取重量归一化处理后,导入SIMCA-P软件进行多变量分析.通过主成分分析(PCA)观察样本整体分布情况,通过偏最小二乘法-判别分析(PLS-DA)进行模型分类和交叉检验,通过正交偏最小二乘法-判别分析[24](O-PLS-DA)寻找在组间有差异的代谢物.此外,O-PLS-DA模型数据还通过CV-ANOVA进行模型验证,使用matlab软件绘制Pearson相关系数负载图(coefficient plots),以便直观展示在性别组间发生显著变化的代谢物[25].
2 结果与讨论 2.1 雌性和雄性C57BL/6小鼠各区段肠壁组织胆汁酸含量的分布及变化规律不同性别C57BL/6小鼠肠壁组织各区段胆汁酸的分布如表 1所示.在所有肠壁组织中,总共检测出19种胆汁酸,按其结构可分为7种结合型胆汁酸(其中有5种牛磺酸结合型胆汁酸和2种甘氨酸结合型胆汁酸)和12种游离胆汁酸.按其来源可分为10种初级胆汁酸和9种次级胆汁酸.值得注意的是,初级胆汁酸和次级胆汁酸的分类与物种密切相关[8].如鼠胆酸(MCA)仅存在于啮齿动物中,初级胆汁酸CDCA经羟基化作用转化成α-MCA,UDCA经羟基化作用转化成β-MCA[26],后者在肠道菌群作用下形成ω-MCA.Sayin等人[26]的研究工作表明在小鼠体内,由于其细胞色素C表型CYP2C70具有高氧化型表达特性,无须通过肠道菌群的参与也可在肝脏产生UDCA,因此UDCA在小鼠组织中归为初级胆汁酸[7].但由于人体组织的细胞色素P450酶(CYP2C9)缺乏氧化作用,无法将CDCA转化为UDCA,因此UDCA在人体组织中归为次级胆汁酸.
| 表 1 不同性别C57BL/6小鼠各区段肠道组织的胆汁酸含量(μg/mg, n雌性=12, n雄性=14) Table 1 Bile acid concentration (μg/mg, nfemale=12, nmale=14) of the different intestinal segments of female and male C57BL/6 mice |
通过比较空肠、回肠、盲肠及结肠四个肠段的胆汁酸含量可见(表 1),无论是雄性小鼠还是雌性小鼠,胆汁酸在不同肠段的分布差异较大.其中在肠道上段(空肠)以牛磺酸结合型胆汁酸和初级胆汁酸为主,并以TCA含量最高(雄性小鼠:1158.0±418.4 μg/mg,雌性小鼠:859.2±140.3 μg/mg).肠道下段(盲肠)以游离胆汁酸和次级胆汁酸为主,并以DCA含量最高(雄性小鼠:96.2±11.6 μg/mg,雌性小鼠:96.0±14.6 μg/mg).大量的文献表明,结合型胆汁酸的结合方式与物种特异性密切相关.例如,在人体中游离胆汁酸优先与甘氨酸进行结合[27],而在啮齿类动物模型中呈现主要与牛磺酸结合的趋势.这种种属特异性结合的方式主要是因为将胆汁酸催化为相应的N-酰基胆汁酸络合物的主要酶类(胆汁酸辅酶A,氨基酸N-酰基转移酶)在不同物种间的表达有差异[28, 29].另外,次级胆汁酸DCA含量的高低与宿主盲肠内梭菌目细菌的含量呈正相关[30].
分别比较雌性小鼠和雄性小鼠不同肠段中胆汁酸含量的差别,发现胆汁酸在不同肠段中呈现显著的性别差异性应答(表 1),并以肠道下段的差异更为显著.其中,肠道上段仅表现为雄性小鼠回肠iso-DCA的水平高于雌性小鼠,其余胆汁酸均无显著性别差异.在盲肠组织中,可见雌性小鼠初级胆汁酸(p < 0.05)及其占总胆汁酸含量(p < 0.05)较雄性小鼠呈显著性增加,并以α-MCA(p < 0.01)和β-MCA(p < 0.01)的变化最为显著.尽管雌性小鼠次级胆汁酸ω-MCA(p < 0.01)、iso-DCA(p < 0.05)以及LCA(p < 0.05)含量相对于雄性小鼠呈增加趋势,但是由于初级胆汁酸的增加更为显著,使得次级胆汁酸占总胆汁酸的含量相对减少.在结肠组织中,主要表现为雌性小鼠G-BAs(p < 0.05)和β-MCA(p < 0.05)的含量较雄性小鼠呈显著性增加,以及iso-DCA含量的轻度降低(p < 0.05).
此外,选取总胆汁酸、结合胆汁酸、游离胆汁酸等其它不同分类的胆汁酸含量及其占总胆汁酸的百分比,并以空肠胆汁酸含量及占比为基准[31],观察了在不同性别C57BL/6小鼠中,自上而下不同肠道区段各指标相对于空肠胆汁酸含量的变化曲线(图 1).通过变化率曲线可见雌性小鼠与雄性小鼠中不同类型胆汁酸含量及其在总胆汁酸中的占比曲线趋势基本相同,除了甘氨酸结合型胆汁酸的差异较大[32].这可能与甘氨酸在不同性别宿主的差异性表达有关[32].此外,由变化率曲线可见空肠肠壁组织的总胆汁酸含量高于其它几个肠段,并随着肠道走向自上而下逐渐降低,主要表现为结合型胆汁酸,特别是以牛磺酸结合型胆汁酸的降低最为显著.其中,从空肠到回肠组织中其总胆汁酸含量降低得最为显著(约为70%~80%,雄性小鼠),从回肠到盲肠组织总胆汁酸含量降低约10%(雄性小鼠),盲肠到结肠的变化几乎无明显差异,这与雄性SD大鼠肠道内容物的NMR代谢组研究结果一致[33].而游离胆汁酸占总胆汁酸的含量呈现逐渐上升的趋势.另外,通过初级胆汁酸、次级胆汁酸及其在总胆汁酸中的含量变化,可见肠段从小肠至大肠延续过程中,初级胆汁酸逐渐降低,次级胆汁酸逐渐升高,这也从生理学角度证实了肠道下段肠道微生物对胆汁酸的脱羟基化和去偶联作用[34].
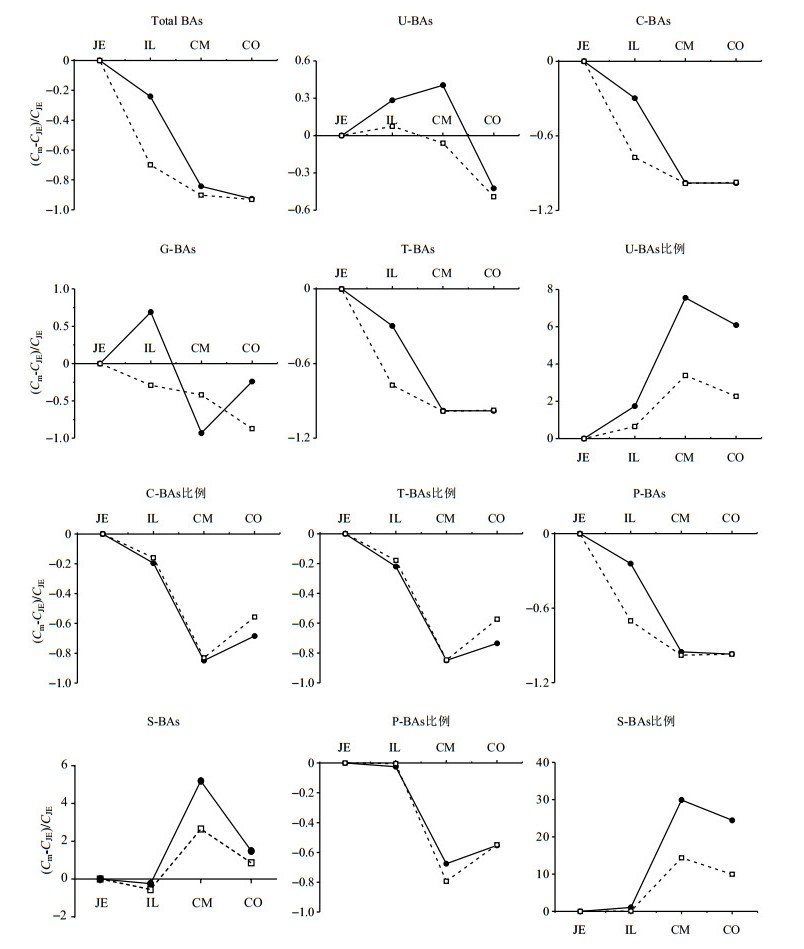
|
图 1 不同性别C57BL/6小鼠肠壁各区段各类胆汁酸及其占总胆汁酸比例相对于空肠的变化率. JE:空肠; IL:回肠; CM:盲肠; CO:结肠.纵坐标中Cm表示空肠、回肠、盲肠或结肠中各类胆汁酸或其占总胆酸的比例. CJE表示空肠中各类胆汁酸或其占总胆酸的比例. ●雌性 □雄性 Figure 1 The rate of change of bile acids and their percentages in total bile acids in different intestinal segments with respective to that in jejunal tissue of female and male C57BL/6 mice. JE: Jejunum; IL: Ileum; CM: Cecum; CO: Colon. Cm: the content of different types of bile acids or their percentages in total bile acids in JE, IL, CM and CO; CJE: the content of different types of bile acids or their percentages in total bile acids in JE. ●Female □Male |
众所周知,胆汁酸通过肝肠循环的高效作用形式可对约95%的胆汁酸进行重吸收,剩下约5%的胆汁酸会随粪便排出体外.所以粪样提取物的代谢组中除了宿主的食物信息以外,必然包含了胆汁酸信息,同时也包含了宿主和肠道菌群共代谢信息,这些信息均可通过无靶向的NMR方法检测到.同时,由于NMR检测平台样品前处理简单、结果重现性好、能提供丰富的结构信息等优点,因此本文观察了肠道组织色谱检测平台和粪样提取物NMR检测平台的代谢组信息,希望借此能比较不同的代谢组平台对胆汁酸信息的提取.
已有文献报道在粪样提取物NMR代谢组中,可鉴定出近40种代谢物[35],主要包括胆汁酸、短链脂肪酸、单糖、氨基酸以及三羧酸循环的中间产物.其中短链脂肪酸与肠道微生物对食物的酵解有关[36],氨基酸及糖类可能与菌群的内源性代谢产物有关[37],胆汁酸为宿主-微生物经过肠道菌群重吸收后通过粪样排出体外.因此,粪样提取代谢物不仅能反映肠道的健康状况同时也与肠道菌群生态平衡有密切关系.
不同性别小鼠粪样提取物的代谢组分析结果如图 2所示.由OPLS-DA模型散点图及模型指标(R2X=0.731,Q2=0.792,pCV-ANOVA=0.001 4)可见,雌、雄小鼠粪样代谢组存在显著差异.其中胆汁酸在雌雄小鼠粪样中的含量有显著性差异.与雌性小鼠相比,雄性小鼠粪样中胆汁酸含量较低,这可能与雌、雄小鼠的菌群微生态差异有关.除胆汁酸代谢物外,酪氨酸及丙酸盐、尿嘧啶、戊酸盐也存在差异,这可能与肠道微生物对食物的酵解以及菌群的内源性代谢产物有关.值得注意的是,由于检测灵敏度的差异,粪样代谢组NMR分析与LC-MS分析所检测到的胆汁酸类别不完全相同.分峰拟合分析推测粪样提取物NMR代谢组中可能含有TCA、CA、DCA和T-β-MCA等胆汁酸[38],但其具体的代谢物分类还有待于标准品的进一步确认.这也进一步提示了在胆汁酸分析中两种代谢组分析平台互为补充的特点.
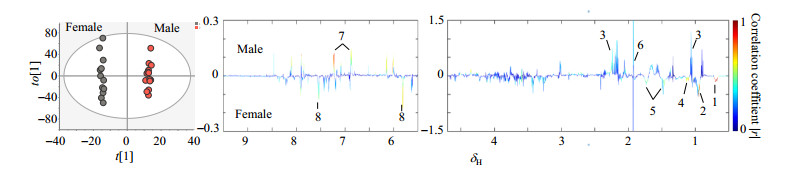
|
图 2 不同性别C57BL/6小鼠粪样提取物(NMR检测)的O-PLS-DA得分图(左)和相关系数负载图(右).编号对应代谢物:1.胆汁酸;2. α-酮-β-甲基-戊酸盐;3.丙酸盐;4. α-酮-异戊酸;5.戊酸盐;6.乙酸;7.酪氨酸;8.尿嘧啶 Figure 2 O-PLS-DA analysis of fecal extracts metabolome (detected by NMR) from female and male C57BL/6 mice. Scores plots (left) and loading plots (right). 1. Bile acids; 2. α-keto-β-methyl-valerate; 3. propionate; 4. α-keto-isovalerate; 5. valerate; 6. acetate; 7. tyrosine; 8. uracil |
本文通过基于HPLC-QqQ-MS的靶向代谢组结合基于NMR的非靶向代谢组学的方法研究了不同性别C57BL/6小鼠肠道组织胆汁酸相关代谢组的差异性应答.研究表明在正常的生理情况下,小鼠肠道组织共检测出19种胆酸代谢物,粪样提取物代谢组共检出30余种代谢物.研究结果发现,不同性别小鼠肠道胆汁酸的整体水平未发现显著差异,但是肠组织各区段的分布差异明显,且以不同性别小鼠盲肠和结肠的胆汁酸水平和粪样提取物的差异最为显著.本研究的后续工作可以考虑通过变性梯度凝胶电泳、16S rRNA基因测序等分子生物学手段[39]检测肠道菌群的分布特征,以综合研究不同性别宿主生理状况下胆汁酸与菌群分布的直接联系.本文通过对C57BL/6小鼠肠道组织及粪样提取物的代谢组分析,提供了C57BL/6小鼠的肠道胆盐基础代谢组数据,可为后续的以C57BL/6小鼠为实验动物模型的其它代谢组学实验提供了基础信息,并可为更全面地认识不同性别宿主-肠道菌群共代谢的代谢表型变化提供了基础数据.
| [1] | XU J, GORDON J I. Honor thy symbionts[J]. Proc Natl Acad Sci U S A, 2003, 100(18): 10452-10459. DOI: 10.1073/pnas.1734063100. |
| [2] | MARTIN F P, DUMAS M E, WANG Y L, et al. A top-down systems biology view of microbiome-mammalian metabolic interactions in a mouse model[J]. Mol Syst Biol, 2007, 3: 112. |
| [3] | CLAUS S P, TSANG T M, WANG Y L, et al. Systemic multicompartmental effects of the gut microbiome on mouse metabolic phenotypes[J]. Mol Syst Biol, 2008, 4: 219. |
| [4] | WIKOFF W R, ANFORA A T, LIU J, et al. Metabolomics analysis reveals large effects of gut microflora on mammalian blood metabolites[J]. Proc Natl Acad Sci U S A, 2009, 106(10): 3698-3703. DOI: 10.1073/pnas.0812874106. |
| [5] | PARLESAK A, SCHAECKELER S, MOSER L, et al. Conjugated primary bile salts reduce permeability of endotoxin through intestinal epithelial cells and synergize with phosphatidylcholine in suppression of inflammatory cytokine production[J]. Crit Care Med, 2007, 35(10): 2367-2374. DOI: 10.1097/01.CCM.0000284586.84952.FB. |
| [6] | RAMIREZ-PEREZ O, CRUZ-RAMON V, CHINCHILLA-LOPEZ P, et al. The role of the gut microbiota in bile acid metabolism[J]. Ann Hepatol, 2017, 16(Suppl. 1:s3-105.): a15-s20. |
| [7] | SAYIN S I, WAHLSTROM A, FELIN J, et al. Gut microbiota regulates bile acid metabolism by reducing the levels of tauro-beta-muricholic acid, a naturally occurring FXR antagonist[J]. Cell Metab, 2013, 17(2): 225-235. DOI: 10.1016/j.cmet.2013.01.003. |
| [8] | WAHLSTROM A, SAYIN S I, MARSCHALL H U, et al. Intestinal crosstalk between bile acids and microbiota and its impact on host metabolism[J]. Cell Metab, 2016, 24(1): 41-50. DOI: 10.1016/j.cmet.2016.05.005. |
| [9] | STALEY C, WEINGARDEN A R, KHORUTS A, et al. Interaction of gut microbiota with bile acid metabolism and its influence on disease states[J]. Appl Microbiol Biotechnol, 2017, 101(1): 47-64. DOI: 10.1007/s00253-016-8006-6. |
| [10] | HOFMANN A F. The continuing importance of bile acids in liver and intestinal disease[J]. Arch Intern Med, 1999, 159(22): 2647-2658. DOI: 10.1001/archinte.159.22.2647. |
| [11] | RAO A, KOSTERS A, MELLS J E, et al. Inhibition of ileal bile acid uptake protects against nonalcoholic fatty liver disease in high-fat diet-fed mice[J]. Sci Transl Med, 2016, 8(357): 357ra122. DOI: 10.1126/scitranslmed.aaf4823. |
| [12] | THOMAS C, PELLICCIARI R, PRUZANSKI M, et al. Targeting bile-acid signalling for metabolic diseases[J]. Nat Rev Drug Discov, 2008, 7(8): 678-693. DOI: 10.1038/nrd2619. |
| [13] | NAGENGAST F, GRUBBEN M, VAN MUNSTER I. Role of bile acids in colorectal carcinogenesis[J]. Eur J Cancer, 1995, 31(7, 8): 1067-1070. |
| [14] | RAMALHO R M, VIANA R J, LOW W C, et al. Bile acids and apoptosis modulation:an emerging role in experimental Alzheimer's disease[J]. Trends Mol Med, 2008, 14(2): 54-62. DOI: 10.1016/j.molmed.2007.12.001. |
| [15] | WATANABE M, HOUTEN S M, WANG L, et al. Bile acids lower triglyceride levels via a pathway involving FXR, SHP, and SREBP-1c[J]. J Clin Invest, 2004, 113(10): 1408-1418. DOI: 10.1172/JCI21025. |
| [16] | JIANG C T, XIE C, LV Y, et al. Intestine-selective farnesoid X receptor inhibition improves obesity-related metabolic dysfunction[J]. Nat Commun, 2015, 6: 10166. DOI: 10.1038/ncomms10166. |
| [17] | XIE G X, WANG Y X, WANG X N, et al. Profiling of serum bile acids in a healthy Chinese population using UPLC-MS/MS[J]. J Proteome Res, 2015, 14(2): 850-859. DOI: 10.1021/pr500920q. |
| [18] | BENNION L J, DROBNY E, KNOWLER W C, et al. Sex differences in the size of bile acid pools[J]. Metabolism, 1978, 27(8): 961-969. DOI: 10.1016/0026-0495(78)90140-3. |
| [19] | NICKEN P, HAMSCHER G, BREVES G, et al. Uptake of the colon carcinogen 2-amino-1-methyl-6-phenylimidazo[4, 5-b]pyridine by different segments of the rat gastrointestinal tract:its implication in colorectal carcinogenesis[J]. Toxicol Lett, 2010, 196(1): 60-66. DOI: 10.1016/j.toxlet.2010.04.002. |
| [20] | YUAN B F, ZHU Q F, GUO N, et al. Comprehensive profiling of fecal metabolome of mice by integrated chemical isotope labeling-mass spectrometry analysis[J]. Anal Chem, 2018, 90(5): 3512-3520. DOI: 10.1021/acs.analchem.7b05355. |
| [21] | GARCIA-CANAVERAS J C, DONATO M T, CASTELL J V, et al. Targeted profiling of circulating and hepatic bile acids in human, mouse, and rat using a UPLC-MRM-MS-validated method[J]. J Lipid Res, 2012, 53(10): 2231-2241. DOI: 10.1194/jlr.D028803. |
| [22] |
CHEN L, SONG K, WANG Y L. Effects of attenuated salmonella typhimurium infection on fecal metabonome in mice[J].
Chinese J Magn Reson, 2014, 31(3): 349-363.
陈璐, 宋侃, 王玉兰. 感染减毒鼠伤寒沙门氏菌对小鼠粪样代谢组的影响-WIPM和Bruker 500 MHz核磁共振波谱仪检测结果的比较[J]. 波谱学杂志, 2014, 31(3): 349-363. DOI: 10.3969/j.issn.1000-4556.2014.03.006. |
| [23] | WANT E J, COEN M, MASSON P, et al. Ultra performance liquid chromatography-mass spectrometry profiling of bile acid metabolites in biofluids:application to experimental toxicology studies[J]. Anal Chem, 2010, 82(12): 5282-5289. DOI: 10.1021/ac1007078. |
| [24] |
HU Y L, HAO F H, WANG Y L. NMR-based metabonomic analyses on spleen tissues of 4T1 tumor-bearing mice subjected to chemotherapies with different drug delivery strategies[J].
Chinese J Magn Reson, 2018, 35(1): 8-21.
胡依黎, 豪富华, 王玉兰. 基于NMR的4T1荷瘤小鼠脾脏受不同给药方式影响的代谢组学研究[J]. 波谱学杂志, 2018, 35(1): 8-21. |
| [25] |
SONG Y P, LI N, XUE H S, et al. Metabonomics analysis of brown adipose and white adipose tissues[J].
Chinese J Magn Reson, 2016, 33(2): 208-223.
宋懿朋, 李宁, 薛海斯, 等. 棕色脂肪组织和白色脂肪组织的代谢组学研究[J]. 波谱学杂志, 2016, 33(2): 208-223. |
| [26] | TAKAHASHI S, FUKAMI T, MASUO Y, et al. Cyp2c70 is responsible for the species difference in bile acid metabolism between mice and humans[J]. J Lipid Res, 2016, 57(12): 2130-2137. DOI: 10.1194/jlr.M071183. |
| [27] | FALANY C N, JOHNSON M R, BARNES S, et al. Glycine and taurine conjugation of bile acids by a single enzyme. Molecular cloning and expression of human liver bile acid CoA:amino acid N-acyltransferase[J]. J Biol Chem, 1994, 269(30): 19375-19379. |
| [28] | FALANY C, FORTINBERRY H, LEITER E, et al. Cloning, expression, and chromosomal localization of mouse liver bile acid CoA:amino acid N-acyltransferase[J]. J Lipid Res, 1997, 38(6): 1139-1148. |
| [29] | ZHANG R, BARNES S, DIASIO R B. Differential intestinal deconjugation of taurine and glycine bile acid N-acyl amidates in rats[J]. Am J Physiol, 1992, 262(2): G351-G358. |
| [30] | MULLER V M, ZIETEK T, ROHM F, et al. Gut barrier impairment by high-fat diet in mice depends on housing conditions[J]. Mol Nutr Food Res, 2016, 60(4): 897-908. DOI: 10.1002/mnfr.v60.4. |
| [31] | WU J F, XU W X, MING Z P, et al. Metabolic changes reveal the development of schistosomiasis in mice[J]. PLoS Negl Trop Dis, 2010, 4(8): e807. DOI: 10.1371/journal.pntd.0000807. |
| [32] | WANG Y L, TANG H R, NICHOLSON J K, et al. A metabonomic strategy for the detection of the metabolic effects of chamomile (Matricaria recutita L.) ingestion[J]. J Agric Food Chem, 2005, 53(2): 191-196. DOI: 10.1021/jf0403282. |
| [33] | TIAN Y, ZHANG L M, WANG Y L, et al. Age-related topographical metabolic signatures for the rat gastrointestinal contents[J]. J Proteome Res, 2012, 11(2): 1397-1411. DOI: 10.1021/pr2011507. |
| [34] | RIDLON J M, KANG D J, HYLEMON P B. Bile salt biotransformations by human intestinal bacteria[J]. J Lipid Res, 2006, 47(2): 241-259. DOI: 10.1194/jlr.R500013-JLR200. |
| [35] |
TIAN Y, TANG H R. Identification and structural determination of saccharides in rat feces[J].
Chinese J Magn Reson, 2012, 29(3): 361-371.
田园, 唐惠儒. 大鼠粪样中几种糖类物质的结构确定[J]. 波谱学杂志, 2012, 29(3): 361-371. DOI: 10.3969/j.issn.1000-4556.2012.03.005. |
| [36] | DEN BESTEN G D, VAN EUNEN K, GROEN A K, et al. The role of short-chain fatty acids in the interplay between diet, gut microbiota, and host energy metabolism[J]. J Lipid Res, 2013, 54(9): 2325-2340. DOI: 10.1194/jlr.R036012. |
| [37] | BACKHED F, LEY R E, SONNENBURG J L, et al. Host-bacterial mutualism in the human intestine[J]. science, 2005, 307(5717): 1915-1920. DOI: 10.1126/science.1104816. |
| [38] | ZHAO Y, WU J F, LI J V, et al. Gut microbiota composition modifies fecal metabolic profiles in mice[J]. J Proteome Res, 2013, 12(6): 2987-2999. DOI: 10.1021/pr400263n. |
| [39] | FRAHER M H, O'TOOLE P W, QUIGLEY E M. Techniques used to characterize the gut microbiota:a guide for the clinician[J]. Nat Rev Gastroenterol Hepatol, 2012, 9(6): 312-322. DOI: 10.1038/nrgastro.2012.44. |
 2018, Vol. 35
2018, Vol. 35 
