2. 第三军医大学第三附属医院野战外科研究所 病理科, 重庆 400042
2. Department of Pathology, Institute of Surgery Research, Daping Hospital, Third Military Medical University, Chongqing 400042, China
胶质母细胞瘤(glioblastoma,GBM)是颅内最常见的、恶性程度最高的原发性肿瘤[1].与其他恶性肿瘤相似,肿瘤内异质性是GBM发展、治疗耐受形成和复发的直接原因[2].GBM肿瘤内异质性体现在影像学表现、组织病理、基因改变及表达形式等多方面,其异质性的遗传基础在于基因的突变和染色体的异常[3].
近几年来,Verhaak等[3]将GBM分为四种亚型,分别为:经典型(classic,CL),间充质型(mesenchymal,MSE),前神经元型(proneural,PN)和神经源型(neural,NL).其中CL型是GBM最常见的基因畸变类型,通常伴随表皮生长因子受体(epidermal growth factor receptor,EGFR)的过表达.MES型的病理特征出现类似于间质的组织结构,如:骨、滑膜、平滑肌等,通常伴随着同源性磷酸酶-张力蛋白(phosphatase and tensin homolog deleted on chromosome ten,PTEN)基因的丢失.PN型与神经发育基因突变和良好的预后相关,大多表现为异柠檬酸脱氢酶-1(isocitrate dehydrogenase 1,IDH-1)与抑癌基因TP53的突变.IDH是三羧酸循环中关键限速酶,在细胞氧化损伤反应中发挥着重要的调控作用,IDH突变的WHO Ⅱ、Ⅲ、Ⅳ级胶质瘤的预后良好,患者存活期明显延长.TP53在G1/S控制点起负调节细胞生长周期,介导DNA损伤后的细胞应激反应,维持遗传稳定性,决定细胞是否进行程序化死亡,胶质瘤细胞TP53突变,导致细胞不能阻滞于G1期以修复损伤的DNA,促进细胞恶性转化.NL型的特征是各种神经元标记物的表达,其中神经丝轻链(neurofilament light chain,NEFL)是该类型最重要的分子指标.不同亚型的GBM对治疗的敏感性及其预后均有显著差异,因此术前对GBM不同分子的表达情况进行分析将有助于制定更为有效、合理的个性化治疗方案.
磁共振成像(magnetic resonance imaging, MRI)作为一种公认的无创诊断技术,已被广泛用于胶质瘤的诊断当中.然而近几年的临床研究证实,即使是相同病理类型的GBM,MRI的影像学数据也存在很大差异.导致这种现象的因素可能是由于GBM的遗传异质性引起的[4],因此对MRI影像学数据和分子标记物的表达进行相关性分析,进而对临床疗效和预后进行评估,对临床GBM患者治疗方案的选择具有重要的指导意义.
1 实验方法 1.1 研究对象与取材本研究经过伦理委员会机构审查和批准,并且与病人签署知情同意书.回顾性分析2009年5月~2015年3月在大坪医院经手术病理证实的60例GBM的临床资料及影像学数据,其中男36例,女24例.年龄34~79岁,平均年龄为(54.2±12.4)岁,临床症状包括呕吐、头痛、神经变化、四肢无力、意识障碍和语言障碍等,正常对照组取自10例胶质瘤标本的周围正常脑组织.本研究的纳入标准为:1)位置均为幕上;2)肿瘤无扩散,患者年龄在18周岁以上;3)新诊断的GBM手术前未经放、化疗等干预;4)术前行MRI常规扫描,并获得有效的原始数据;5)病人知情并同意进入此项研究,签署同意书.排除标准为:多发肿瘤、脑膜脑炎和结核患者,孕期妇女,精神障碍患者,不能控制的糖尿病患者以及严重心肺疾病患者.
1.2 病理检测标本均采用4%中性甲醛固定、石蜡包埋后进行薄层切片,片厚为5 mm,切片要求含肿瘤组织不少于25%.所有切片对5种分子标记物(PTEN、EGFR、TP53、IDH-1、NEFL)分别进行免疫组化EnVision二步法检测.5种分子标记物单克隆抗体与二抗均购自于北京中杉金桥生物技术有限公司.阴性对照以磷酸盐缓冲液(PBS)代替一抗进行孵育.
1.3 MRI扫描与图像处理MRI扫描采用Siemens verio 3.0T(Magnetom Verio,Siemens Medical Solutions,Erlangen,Germany)超导型MRI扫描仪,所有患者均行平扫及增强扫描,对比剂采用钆喷替酸葡甲胺(Gd-DTPA,北京北陆医药化工公司),剂量为0.2 mmol/kg,注射流速为4.5 mL/s.常规MRI扫描参数如下:TSE-T2WI:TR = 4 900 ms,TE = 92 ms,SL = 5.0 mm,FOV = 23 cm×23 cm;T1WI增强扫描参数如下:TR = 250 ms,TE = 2.48 ms,SL = 5.0 mm,FOV = 23 cm×23 cm;液体衰减反转恢复序列(fluid attenuated inversion recovery sequence, FLAIR):TR = 8 500 ms,TE = 88 ms,SL = 3.0 mm,FOV = 23 cm×23 cm.扫描方位包括T1WI矢状位、T1WI和TLDWI轴位,及T1WI增强冠状位.
1.4 数据处理GBM的常规MRI形态学指标:对患者MRI数据进行形态学测量分析,总结出水肿区最长径(edema longest diameter,ELD)、肿瘤区最长径(tumor longest diameter,TLD)、强化与肿瘤长径比例(CTE/TLD)3个形态学指标(图 1).具体测量方法如下.ELD:T1-FLAIR图像上,于肿瘤实质旁选取3个较为显著水肿区域测得水肿最长径,取均值.TLD:T2WI图像上测得3个肿瘤最长径,取均值.CTE/TLD:T1WI增强扫描图像测得3个肿瘤区域最大径,取均值,与TLD之比即为CTE/TLD.TLD、ELD和CTE/TLD的测量和计算均由3位医师分别使用双盲法独立完成.3人计算后平均值作为最后的分析数值.
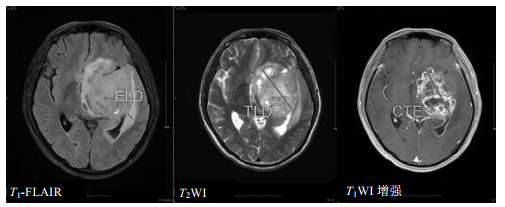
|
图 1 基于影像的形态学指标测量示图 Figure 1 The schematic diagram of imaging index |
GBM病理学特征:以染色强度结合阳性细胞数百分比综合计分是病理切片分析的常规半定量分析.所有读片均由两名资深病理学工作者采用双盲法阅片,光镜下随机观察8个高倍(400×)视野,每个视野计数100个细胞,数出阳性细胞数目,换算成百分比,取其平均值.将瘤细胞阳性比率分为:0级,无阳性细胞;1级(弱阳性),瘤细胞阳性率<10%;2级(中等阳性),瘤细胞阳性率10%~50%;3级(强阳性),瘤细胞阳性率>50%.瘤细胞染色程度分为:0级,无染色;1级,弱染色,外观轻度变黄;2级,中度染色,呈棕黄色;3级,明显染色,呈棕色.染色指数(staining index,SI),计算为染色程度×瘤细胞阳性比率[5].高表达组定义为SI>4,低表达组定义为SI≤4.
1.5 统计分析采用SPSS 19.0统计软件对影像学指标和组化指标分别进行Pearson相关分析和多元logistics回归分析.进行受试者工作(receiver operating characteristic, ROC)曲线分析,寻找影像学指标的cut-off值,以评价敏感性和特异性.所有数据都以(平均值±标准差)形式显示,p<0.05设置为显著性水平.组内相关系数(interclass correlation coefficient, ICC)分析用来比较测试者测量值的一致性.
| $ ICC = \frac{{MSR - MSE}}{{MSR + (k - 1) + \frac{k}{n}(MSC - MSE)}} $ | (1) |
(1) 式中MSR为随机区组(即被观察对象)间的均方(即方差);MSE为误差的均方;MSC表示列变量均方;k为重复次数;n为被观察的例数.ICC值介于0~1之间,等于个体的变异度除以总的变异度:0表示不可信;1表示完全可信.一般认为ICC低于0.4表示信度较差,大于0.75表示信度良好.
2 结果与讨论 2.1 GBM特征分子表达图 2显示GBM特征分子的免疫组化染色图.突变型TP53阳性细胞表现为细胞核呈棕色或棕黑色着色,阳性表达率为52%(31例),其中弱阳性表达率为32%(19例),中等阳性表达率为15%(9例),强阳性表达率为5%(3例).NEFL阳性细胞胞质中可见棕黄色染色颗粒,NEFL的阳性表达率为63%(38例),其中弱阳性表达率为35%(21例),中等阳性表达率为13%(8例),强阳性表达率为15%(9例).EGFR表达阳性的肿瘤细胞胞质中可见棕黄色颗粒,阳性表达率达77%(46例),其中强阳性表达率为22%(13例),中等阳性表达率为54%(25例),弱阳性表达率仅为13%(8例).PTEN表达阳性的肿瘤细胞镜下其细胞质中可见棕黄色颗粒,PTEN的阳性表达率为78%(47例),其中弱阳性表达率为7%(4例),中等阳性表达率为22%(13例),强阳性表达率为50%(30例).5个分子指标中,IDH-1阳性表达率最低,其表达阳性细胞胞质中可见呈棕黄色染色的颗粒,阳性表达率为33%(20例).其中,强阳性表达率5%(1例),中等阳性表达率(6例),均呈弱阳性表达率22%(13例).
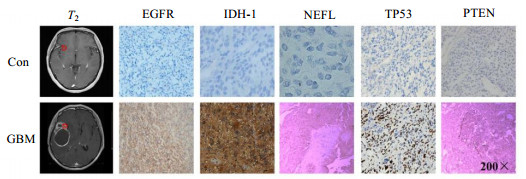
|
图 2 GBM相关分子指标分析 Figure 2 The analyze of GBM related molecular biomarkers |
表 1表示不同的GBM特征分子表达及相关组别的基于影像的肿瘤形态学指标.
| 表 1 各分子不同表达组ELD、CTE/TLD和TLD值的比较(平均值±标准差) Table 1 Comparation of ELD, CTE/TLD and TLD values in groups with different molecular expression level (mean ± standard deviation) |
由表 1可见:首先,GBM特征分子表达率高低不同,其MRI形态学指标都有显著性差异,提示GBM特征分子变化与肿瘤形态学变化较为一致;其次,GBM特征分子表达增高伴随特定肿瘤形态学指标增加,具体结果如下:1)TP53、PTEN和EGFR高表达组的TLD值较高;2)PTEN和EFGR高表达组的CTE/TLD值较高;3)TP53和NEFL高表达组的ELD较高;4)IDH-1高表达组ELD和TLD值较低.
2.3 GBM的MRI形态学指标与特征分子表达的相关性评价图 3表示GBM特征分子表达率与MRI形态学指标相关性分析结果.其中相关系数r值绝对值范围在0.8~1.0为极强相关,在0.6~0.8为强相关,在0.4~0.6为中等程度相关,在0.2~0.4为弱相关,在0.0~0.2为极弱相关或无相关.

|
图 3 有相关性的特征分子表达与MRI形态学指标的相关分析 Figure 3 Correlation analysis between specific molecular expression and imaging index |
TP53表达与TLD(r= 0.188,p<0.05)、ELD(r= 0.001,p<0.05)和CTE/TLD(r= 0.053,p<0.05)均无显著相关性.
NEFL表达与TLD(r= -0.065,p<0.05)、ELD(r= 0.138,p<0.05)、CTE/TLD(r= 0.032,p>0.05)均无显著相关性.
EGFR表达与TLD(r= 0.796,p<0.05)、CTE/TLD(r= 0.622,p<0.05)呈正相关,与ELD(r = -0.142,p<0.05)没有显著相关性.
PTEN表达与ELD(r= -0.081,p<0.05)无显著相关性,与TLD(r= 0.533,p<0.05)和CTE/TLD(r= 0.638,p<0.05)呈正相关.
IDH-1表达与TLD(r= -0.672,p<0.05)和CTE/TLD(r= -0.493,p<0.05)呈负相关,与ELD(r= 0.238,p<0.05)无显著相关性.
2.4 ROC曲线分析选取具有显著相关性的MRI形态学指标与特征分子表达率作ROC分析(图 4).ROC曲线根据一系列不同的二分类方式(分界值或决定阈),以真阳性率(灵敏度)为纵坐标,假阳性率(1-特异度)为横坐标绘制的曲线.
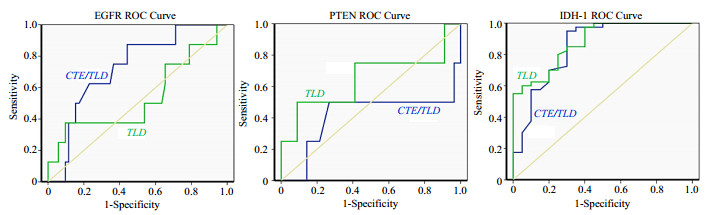
|
图 4 特征分子表达率与MRI形态学指标的ROC曲线分析 Figure 4 ROC curve analysis of specific molecular expression and imaging index |
当TLD取临界值53.25 mm时,其鉴别EGFR阴阳性表达的敏感度为87.5%,特异度为21%,曲线下面积(area under the curve, AUC)为0.535.CTE/TLD取临界值0.77时,其鉴别EGFR阴阳性表达的敏感度为87.5%,特异度均为56%,AUC为0.726.
当TLD取临界值85.45 mm时,其鉴别PTEN阳阴性表达的敏感度为50%,特异度均为99%,AUC为0.647.当CTE/TLD取临界值0.885时,其鉴别PTEN阳阴性表达的敏感度为50%,特异度为73%,AUC为0.413.
当TLD取临界值57.05 mm时,其鉴别IDH-1阳性表达的敏感度为97.5%,特异度为60%,AUC为0.879.当CTE/TLD取临界值0.615时,其鉴别IDH-1阳阴性表达的敏感度为95%,特异度为65%,AUC为0.849.
2.5 GBM的MRI形态学指标与特征分子表达回归分析通过GBM的MRI形态学指标与GBM特征分子表达的相关性分析发现磁共振图像指标(TLD、CTE/TLD)分别与胶质瘤特征分子(EFGR、PTEN、IDH-1)表达率有显著相关性.为深入研究,将EFGR、PTEN和IDH-1表达率分别与TLD、CTE/TLD做多元logistics回归分析.其中EGFR对TLD的回归系数有显著性(β = 0.686,p = 0.000),PTEN对CTE/TLD的回归系数有显著性(β = 0.406,p= 0.002).综上可见,EFGR和PTEN与相应的MRI形态学测量指标较为敏感,在5个指标中有较高的诊断价值.
2.6 信度分析3个不同病理测试专家间MRI形态学指标的ICC值均较高,其TLD、ELD和CTE/TLD的ICC值分别为(0.873±0.084)、(0.816+0.287)和(0.782+0.113).病理测试者间EGFR、IDH-1、TP53、PTEN和NEFL的ICC值分别为(0.799+0.180)、(0.856+0.385)、(0.774+0.147)、(0.714+0.104)和(0.814+0.241).
3 讨论本研究基于回顾性分析我院60例经手术病理证实的GBM常规MRI影像数据,测量计算TLD、ELD、CTE/TLD 3个基于常规磁共振图像的肿瘤形态学指标,同时对患者病理切片分别进行EGFR、IDH-1、TP53、PTEN和NEFL半定量分析(阳性表达率).利用Pearson相关分析获得MRI形态学指标与肿瘤相关分子阳性比率的相关性,并通过ROC分析与多元logistics回归获得各分子亚型敏感的影像学指标(即ROC分析可以获得该指标指征的cut-off值),这些结果对临床GBM诊断治疗具有一定意义.
常规MRI技术(磁共振平扫和增强扫描)为临床胶质瘤诊断常用影像技术.胶质瘤在T2WI为高信号,增强扫描可检测胶质瘤实质区.测量磁共振图像下肿瘤的形态学特征可以获得较为客观的量化指标,这些指标可以反映胶质瘤不同的病理改变.其中TLD为T2WI图像中肿瘤的最长径,反映的是肿瘤的实质部分和占位迁移信息.CTE/TLD是增强扫描和T2WI下图像中肿瘤最长径的比值,反映的是肿瘤的实质部分范围,为评判肿瘤恶性程度的标准之一.ELD值对应于水肿区最长径,其病理基础为肿瘤占位压迫引起的周围组织炎症信息.研究表明脑肿瘤的瘤周水肿的产生以血管源性为主,是血脑屏障(blood brain barrier,BBB)的破坏,除单纯血管源性水肿外还有肿瘤细胞浸润的结果[6].GBM普遍血管异质性强,血管通透性也高,故本实验中的瘤周水肿本身不具备明显的特异性.先进磁共振技术,如动态对比增强磁共振成像(DCE-MRI)、动态磁敏感增强灌注成像(DSC-MRI)及酰胺质子转移(APT-MRI)等存在不足,所获得的参数都是依赖数学模型和缩放因子计算出来的结果,且大多数技术绝对定量存在较大挑战.常规MRI技术与MRI新技术存在特异性不高、参数较少的缺陷,但是测得形态学指标可以与病理结果直接进行比较,且不同测试者间主观误差较小.同时,现行的胶质瘤预后评估标准(如RECIST、RANO等)均推荐使用形态学指标(直径等)来评价肿瘤的治疗反应情况.如伦勃朗视觉感受图像(visually accessible rembrandt images,VASARI)是最常用的标准定量影像学评价.VASARI主要通过分析常规MRI图像得到肿瘤形态体积的具体参数,包括肿瘤对比度强化、瘤内坏死、脑水肿或肿瘤侵袭区域的体积等[7].
按照WHO制定2016年胶质瘤分级指南将EGFR、PTEN、TP53和IDH-1等分子指标作为重要的分级指标[8].这些分子指标的变化可以作为预后评估,如IDH-1突变型的患者预后良好,EGFR突变型患者复发率显著增加[9].研究报道>90%的GBM患者会发生PI3K/AKT/mTOR通路相关蛋白的突变,其中60%突变发生于EGFR蛋白[6].EGFR的表达随着肿瘤的恶性程度增高而升高[10],EGFR是GBM靶向药物重要的靶点.另有学者证实,伴有EGFR扩增的GBM复发时间明显短于无EGFR扩增的GBM患者[11].根据ROC结果可知,CTE/TLD指标对EGFR的提示作用更为合理.IDH是一种在细胞免受氧化性损伤过程中起重要作用的蛋白酶,IDH家族基因突变在胶质瘤的发生、发展过程中起重要作用.据报道5%的GBM伴随着IDH-1/2突变[12],该类型GBM具有良好的预后,因此可以作为是一个独立的预后生物学标志.Parsons等[13]报道在149例胶质母细胞瘤中,IDH-1和IDH-2突变的GBM病人和间变性星形细胞瘤病人的中位生存期明显高于无突变者.此外,有研究报道IDH-1可增加替莫唑胺化疗的敏感性[14].本研究中,IDH-1的表达与TLD、CTE/TLD均呈负相关,同时IDH-1阳性组的TLD、CTE/TLD低于阴性组,即存在IDH-1突变的GBM的肿瘤体积小于未突变者,从影像学层面提示存在IDH-1突变的GBM恶性程度低于未突变者,结合本研究ROC分析,CTE/TLD鉴别IDH-1表达的曲线下面积(AUC=0.849)与鉴别TLD表达的曲线下面积(AUC=0.879)无明显差异,GBM瘤体体积越小、强化范围越小均可能预示着患者对化疗的敏感性越强,对临床治疗方案的选择有一定的指导作用.PTEN主要通过脂质磷酸酶活性发挥抑癌作用,PTEN表达或缺失是人脑胶质瘤发生、发展的重要因素[15].本实验发现TLD、CTE/TLD与PTEN表达呈正相关,表明肿瘤生长活跃,恶性度高的区域PTEN突变表达越多,与文献报道一致.此外,PTEN还可作为预后分子标记,预测个体化治疗之后的效果.当PTEN发生缺失突变时,不使用替莫唑胺而使用其它辅助治疗措施的GBM患者生存期会缩短,使用替莫唑胺化疗的患者生存期会延长[16].结合本实验的结果,通过TLD、CTE/TLD的量化数据可对临床评估化疗效果提供参考依据.NEFL与各MRI影像学指标均无显著相关性,其原因可能是NEFL基因编码一种型中间丝聚合物,维护神经丝的功能[17],而本实验中选取的MRI常规的影像学指标主要反应肿瘤细胞构筑与炎症反应,对胶质瘤的神经丝病变不敏感.此外研究证实,TP53的突变倾向于是胶质瘤的早期事件[18, 19],而本实验中选取被试GBM发展晚期,这可能是导致本实验TP53与影像指标无相关性的原因.
国内外关于常规MRI扫描得到肿瘤形态学特征与胶质瘤的分子生物学特征的关系报道较多,并将其应用于胶质瘤分子生物学诊断.Ellingson等[20]发现IDH-1的突变与肿瘤的位置有相关性,并且常发生于额叶;EGFR扩增与肿瘤位置亦有相关性,常发生于左侧颞叶,O6-甲基鸟嘌呤DNA甲基转移酶(O6-methyl guanine-DNA methultransferase,MGMT)甲基化常发生于左侧大脑.而Drabycz等[21]发现,MGMT甲基化与其解剖位置无明显相关性.Eoli等[22]证实,肿瘤强化的方式及位置可以区别原发型GBM和继发型GBM.Rivka等[23, 24]将代谢和线粒体功能障碍的基因改变与GBM高度恶性和侵袭性的影像学表现(如深部脑白质受侵、室管膜强化、软脑膜的强化、强化/未强化肿瘤是否跨中线、强化/未强化肿瘤边界、肿瘤水肿面积等)做相关性研究,筛选出侵袭性MRI影像生物标记物,并用此标记物发现GBM中代谢异常和线粒体功能障碍,并预测患者的生存期.Colen等[25]研究揭示,多个基因在不完全强化的GBM中的过表达与较长生存时间有关,而在完全强化的GBM中的过表达则与较短生存时间有关.Mut等[18]研究发现,高表达的TP53(>50%)的GBM在T1加权图像上多表现为边界清晰的病灶,增强呈经典的环形强化,而低表达的TP53(≤50%)的GBM则表现为边界不清,增强呈不均匀强化的特点.在2016年WHO发布胶质瘤相关分子分型指南后,寻找相关影像学改变为当前临床胶质瘤研究的热点.本研究在前人研究的基础上,通过对常规MRI形态学指标和分子指标半定量分析相关分析,初步探讨肿瘤不同特征分子表达与常规MRI观察肿瘤形态的相关性.
我们的研究中还存在一些问题.首先,本实验欠缺应用于后期图像分析的先进影像测量分析工具,需要测量者对胶质瘤形态学特征有较高先验知识.其次,分析特征分子病理学指标与MRI常规成像的样本较少,后期需要更大样本的实验进行深入的研究.此外,胶质瘤相关代谢与血管相关MRI指标未有涉及.
4 结论GBM的MRI形态学指标(TLD、CTE/TLD)与GBM特征分子(EGFR、IDH-1和PTEN)病理学指标有一定相关性.常规MRI作为一种临床成熟的诊断手段通过测量获得肿瘤的形态学指标是较为直观的诊断方法,得到肿瘤的这些宏观形态学信息与GBM的微观分子机制的相关性具有重大的临床价值.
| [1] | OMURO A, DEANGELIS L M. Glioblastoma and other malignant gliomas:a clinical review[J]. JAMA, 2013, 310(17): 1842-1850. DOI: 10.1001/jama.2013.280319. |
| [2] | CURTIS C, SHAH S P, CHIN S F, et al. The genomic and transcriptomic architecture of 2, 000 breast tumours reveals novel subgroups[J]. Nature, 2012, 486(7403): 346-352. |
| [3] | VAN LOO P, NORDGARD S H, LINGJAERDE O C, et al. Allele-specific copy number analysis of tumors[J]. Proc Natl Acad Sci U S A, 2010, 107(39): 16910-16915. DOI: 10.1073/pnas.1009843107. |
| [4] | SOTTORIVA A, SPITERI I, PICCIRILLO S G, et al. Intratumor heterogeneity in human glioblastoma reflects cancer evolutionary dynamics[J]. Proc Natl Acad Sci U S A, 2013, 110(10): 4009-4014. DOI: 10.1073/pnas.1219747110. |
| [5] | AMIN M R, KUROSAKI M, WATANABE T, et al. A comparative study of MIB-1 staining indices of gliomas measured by NIH Image analysis program and conventional manual cell counting method[J]. Neurol Res, 2000, 22(5): 495-500. DOI: 10.1080/01616412.2000.11740707. |
| [6] | KAGAN J, STEIN J, BABAIAN R J, et al. Homozygous deletions at 8p22 and 8p21 in prostate cancer implicate these regions as the sites for candidate tumor suppressor genes[J]. Oncogene, 1995, 11(10): 2121-2126. |
| [7] | GEVAERT O, MITCHELL L A, ACHROL A S, et al. Glioblastoma multiforme:exploratory radiogenomic analysis by using quantitative image features[J]. Radiology, 2014, 273(1): 168-174. DOI: 10.1148/radiol.14131731. |
| [8] | LOUIS D N, PERRY A, REIFENBERGER G, et al. The 2016 World Health Organization Classification of Tumors of the Central Nervous System:a summary[J]. Acta Neuropathol, 2016, 131(6): 803-820. DOI: 10.1007/s00401-016-1545-1. |
| [9] | KILLOCK D. CNS cancer:molecular classification of glioma[J]. Nat Rev Clin Oncol, 2015, 12(9): 502. |
| [10] | FISCHER I, ALDAPE K. Molecular tools:biology, prognosis, and therapeutic triage[J]. Neuroimaging Clin N Am, 2010, 20(3): 273-282. DOI: 10.1016/j.nic.2010.05.004. |
| [11] | WANG S, ZHAO Y, RUAN Z, et al. Association between EGF +61 G/A and glioma risk in a Chinese population[J]. BMC Cancer, 2010: 10221. |
| [12] | DUCRAY F, MARIE Y, SANSON M. IDH1 and IDH2 Mutations in Gliomas[J]. New Eng J Med, 2009, 360(21): 2248-2249. DOI: 10.1056/NEJMc090593. |
| [13] | CHAWLA S, KREJZA J, VOSSOUGH A, et al. Differentiation between oligodendroglioma genotypes using dynamic susceptibility contrast perfusion-weighted imaging and proton MR spectroscopy[J]. AJNR Am J Neuroradiol, 2013, 34(8): 1542-1549. DOI: 10.3174/ajnr.A3384. |
| [14] | VERHAAK R G, HOADLEY KA, PURDOM E, et al. Integrated genomic analysis identifies clinically relevant subtypes of glioblastoma characterized by abnormalities in PDGFRA, IDH1, EGFR, and NF1[J]. Cancer Cell, 2010, 17(1): 98-110. DOI: 10.1016/j.ccr.2009.12.020. |
| [15] | HAN F, HU R, YANG H, et al. PTEN gene mutations correlate to poor prognosis in glioma patients:a meta-analysis[J]. Onco Targets Ther, 2016: 93485-3492. |
| [16] | GENTIL B J, COOPER L. Molecular basis of axonal dysfunction and traffic impairments in CMT[J]. Brain Res Bull, 2012, 88(5): 444-453. DOI: 10.1016/j.brainresbull.2012.05.003. |
| [17] | WANG Z, YANG J, XU G, et al. Targeting miR-381-NEFL axis sensitizes glioblastoma cells to temozolomide by regulating stemness factors and multidrug resistance factors[J]. Oncotarget, 2015, 6(5): 3147-3164. |
| [18] | MUT M, TURBA U C, BOTELLA A C, et al. Neuroimaging characteristics in subgroup of GBMs with p53 overexpression[J]. J Neuroimaging, 2007, 17(2): 168-174. DOI: 10.1111/jon.2007.17.issue-2. |
| [19] | ISHII N, TADA M, HAMOU M F, et al. Cells with TP53 mutations in low grade astrocytic tumors evolve clonally to malignancy and are an unfavorable prognostic factor[J]. Oncogene, 1999, 18(43): 5870-5878. DOI: 10.1038/sj.onc.1203241. |
| [20] | ELLINGSON B M, CLOUGHESY T F, POPE W B, et al. Anatomic localization of O6-methylguanine DNA methyltransferase (MGMT) promoter methylated and unmethylated tumors:A radiographic study in 358 de novo human glioblastomas[J]. NeuroImage, 2012, 59(2): 908-916. DOI: 10.1016/j.neuroimage.2011.09.076. |
| [21] | DRABYCZ S, ROLDAN G, DE ROBLES P, et al. An analysis of image texture, tumor location, and MGMT promoter methylation in glioblastoma using magnetic resonance imaging[J]. Neuroimage, 2010, 49(2): 1398-1405. DOI: 10.1016/j.neuroimage.2009.09.049. |
| [22] | EOLI M, MENGHI F, BRUZZONE M G, et al. Methylation of O6-methylguanine DNA methyltransferase and loss of heterozygosity on 19q and/or 17p are overlapping features of secondary glioblastomas with prolonged survival[J]. Clin Cancer Res, 2007, 13(9): 2606-2613. DOI: 10.1158/1078-0432.CCR-06-2184. |
| [23] | JAIN R, POISSON L, NARANG J, et al. Genomic mapping and survival prediction in glioblastoma:molecular subclassification strengthened by hemodynamic imaging biomarkers[J]. Radiology, 2013, 267(1): 212-220. DOI: 10.1148/radiol.12120846. |
| [24] | KICKINGEREDER P, BURTH S, WICK A, et al. Radiomic Profiling of Glioblastoma:Identifying an Imaging Predictor of Patient Survival with Improved Performance over Established Clinical and Radiologic Risk Models[J]. Radiology, 2016, 280(3): 880-889. DOI: 10.1148/radiol.2016160845. |
| [25] | COLEN R R, VANGEL M, WANG J, et al. Imaging genomic mapping of an invasive MRI phenotype predicts patient outcome and metabolic dysfunction:a TCGA glioma phenotype research group project[J]. BMC Med Genomics, 2014: 730. |
 2018, Vol. 35
2018, Vol. 35 
