2. 贵州省中国科学院天然产物化学重点实验室, 贵州 贵阳 550014;
3. 中国科学院 武汉物理与数学研究所, 湖北 武汉 430071
2. The Key Laboratory of Chemistry for Natural Products of Guizhou Province and Chinese Academy of Sciences, Guiyang 550014, China;
3. Wuhan Institute of Physics and Mathematics, Chinese Academy of Sciences, Wuhan 430071, China
中药核磁共振(Nuclear Magnetic Resonance, NMR)指纹图谱是中药指纹图谱中一种鉴定中药成分、测定中药含量和控制中药质量的新方法[1-3],其研究主要集中在核磁共振氢谱(1H NMR)方面,这主要是因为其所需样品量少、扫描时间短、容易获得,故而被广泛应用.1H NMR指纹图谱是指通过选择适当的提取分离程序,获取整株或某部位植物中作为中药特征性化学成分总提取物,采集其1H NMR谱.由于1H NMR谱较为全面准确地反映了药材中的特征化学成分组成,具有唯一性,如同人的指纹,因此称之为1H NMR指纹图谱[3-10].
完整还原振幅频率表(Complete Reduction to Amplitude-Frequency Table,CRAFT)是一种基于贝叶斯解卷积(Bayesian Deconvolution)的分析混合物NMR谱图的工具.它利用贝叶斯解卷积全自动的将一个时域自由感应衰减(Free Induction Decay, FID)信号转化为完整频率振幅表[11],可以提供复杂混合物信号的化学位移、振幅和线宽,消除了由于相位校正和基线校正带来的积分误差,从而用于混合物中化合物的定性和定量分析.它可以将目标化合物的信号从混合物样品中剥离出来,实现不对混合物样品进行化学分离便可分析目标化合物信息的目的,是分析混合物NMR图谱的新工具[12].CRAFT分析的流程(图 1)如下:
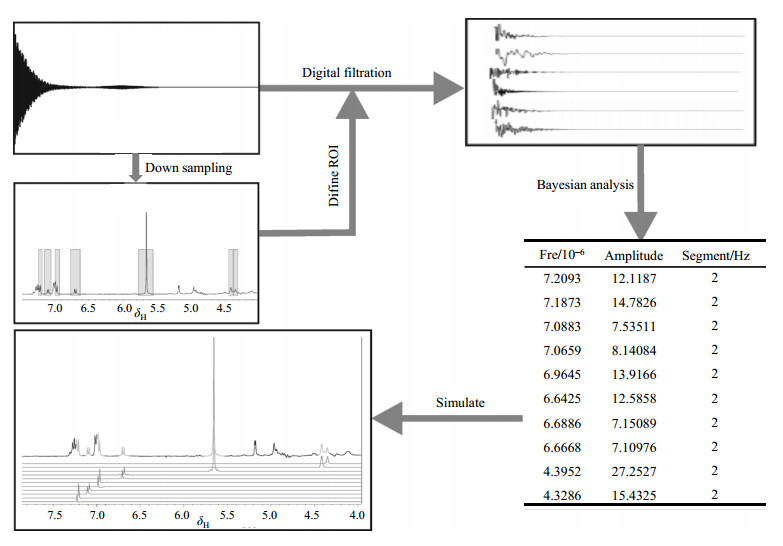
|
图 1 CRAFT基本工作流程图 Figure 1 Basic principles diagram of CRAFT |
第一步:通过数字滤波和定域下采样,把得到的FID分解成多个子FID信号;
第二步:利用贝叶斯解卷积提取信号的频率、振幅和线宽并可拟合成相应的信号模型.
本文将CRAFT技术应用于杜仲指纹图谱的定性和定量分析研究上.首先采用CPMG(Carr-Purcell-Meiboom-Gill)脉冲序列采集了杜仲提取物的1H NMR图谱,通过CRAFT分析技术对杜仲的特征化合物松脂醇二葡萄糖苷(Pinoresinol Glucoside,PDG)进行了定性和定量分析.然后对杜仲1H NMR指纹图谱进行了全指纹分析,获取了杜仲提取物的全指纹数据,建立其药材的指纹数据库,并进行了多元统计学分析,找到不同产地杜仲的差异性[13-17].
1 实验部分 1.1 仪器与试剂美国安捷伦HP-1100液相色谱仪,配备G1311A四元梯度泵、G1322A在线脱气机、G1313A自动进样器、G1314A VWD检测器.色谱柱型号为Kromasil 100-5 C18(250 mm×4.6 mm, 5 μm).
美国Varian INOVA 400型NMR谱仪,NMR测试所用氘代试剂均为美国剑桥同位素实验室产品.
PDG标准品购于成都植标化纯生物技术有限公司.
1.2 样品制备 1.2.1 NMR样品制备准确称取5.0 mg的PDG加入至0.6 mL DMSO-d6[氘代率为99.9%,含体积分数为0.03%的四甲基硅烷(tetramethylsilane,TMS)],超声加热溶解制成用于作对照试验的标准样品,样品编号为PDG-1.
准确称取20.8 mg间苯三酚(C6H6O3·2H2O)标准品溶解至2.0 mL DMSO-d6,制得浓度为10.4 mg/mL的间苯三酚标准溶液,以作为用于NMR定量的内标样品,样品编号为IS-1.
分别取预处理好的的杜仲皮粗粉15.0 g,加入适量氯仿加热回流6 h;过滤,弃去滤液,向滤渣加入适量甲醇,加热回流6 h;回收提取液,将甲醇挥干后制得杜仲皮总提取物浸膏,计算浸膏得率.精密称取30.0 mg杜仲皮浸膏,用0.5 mL DMSO-d6超声充分溶解后过滤;移取滤液0.4 mL至NMR样品管中;然后加入20 μL内标溶液IS-1和0.15 mL的DMSO-d6,待NMR检测.NMR待测杜仲样品共10个,样品编号分别为:贵州市贵阳药用植物园ZWY-1、ZWY-2、ZWY-3、ZWY-4、ZWY-5,湖南郴州HN-1、HN-2、HN-3、HN-4、HN-5.
1.2.2 HPLC样品制备称取杜仲皮总提物浸膏30.6 mg溶于色谱纯甲醇,转移至10 mL容量瓶,加甲醇定容后摇匀,用微孔滤膜(0.22 mm)滤过后取滤液,即得杜仲HPLC待测溶液.
取PDG对照品3.3 mg溶于色谱纯甲醇,转移至5 mL容量瓶,加甲醇定容后摇匀,取1 mL对照品溶液稀释到至10 mL,过滤即得标准品溶液.
1.3 实验条件 1.3.1 1H NMR实验采用PROTON脉冲序列测试1H NMR时的工作频率为399.89 MHz,实验温度为25 ℃,谱宽为4 970.5 Hz.观察道偏置为-368.1 Hz,观察道1H功率为50 db,对应的90°脉冲宽度为11.75 μs,接收增益为20,累加次数64,弛豫延迟为10 s.采用CPMGT2脉冲序列时,1H核的工作频率为399.89 MHz,实验温度为25 ℃,谱宽为4 970.5 Hz,观察道偏置为-368.1 Hz,观察道1H功率为50 db,对应的90°脉冲为11.75 μs,接收增益为20,累加次数64,弛豫延迟为10 s,自旋回波延迟时间(τ)为0.001 s,自旋回波循环时间为0.1 s.1H NMR谱图以TMS定标(δH 0.00).
此外,我们还对杜仲皮提取物样品进行了纵向弛豫时间(T1)的测量,结果显示内标和样品质子的T1均在1~2 s的范围,而实验中弛豫延迟为10 s(≥5T1),因此我们测量得到的是内标和样品质子的全弛豫信号,避免了因弛豫时间不同带来的实验误差.
1.3.2 HPLC实验流动相A相为乙腈,B相为纯水.采用等度洗脱,洗脱相为V乙腈: V水=15: 85.进样量均为10 μL,流速为0.8 mL/min,洗脱时间为12 min,柱温为30 ℃,检测波长为230.8 nm.
2 结果与讨论 2.1 确定CPMGT2图谱为杜仲的NMR指纹图谱杜仲含有大量的大分子杜仲胶,而大分子的横向弛豫时间(T2)短,小分子的T2长.文中采用CPMGT2脉冲序列,通过优化自旋回波相关参数的方法过滤掉大部分大分子的信号而保留小分子的信号,使谱图更加清晰,达到了对谱图进行优化的目的.
以编号为HN-4杜仲待测试溶液为例,分别利用PROTON和CPMGT2脉冲序列采集了其1H NMR谱图,测定结果如图 2(a)所示.可以看出通过CPMGT2技术可以使大分子信号被抑制,小分子信号没有丢失,谱图更加清晰,故而确定CPMGT2图谱作为杜仲1H NMR指纹图谱.图 2(b)为贵州市贵阳药用植物园ZWY-1、ZWY-2、ZWY-3、ZWY-4、ZWY-5,湖南郴州HN-1、HN-2、HN-3、HN-4、HN-5两个不同产地的10株杜仲样品利用相同的测试条件下测得的指纹图谱.
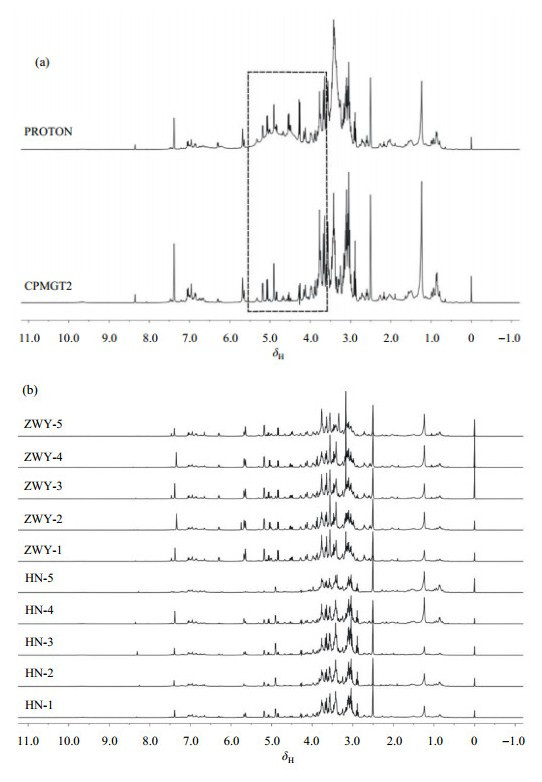
|
图 2 (a) 分别利用PROTON和CPMGT2脉冲序列采集的编号为HN-4的杜仲样品的1H NMR谱;(b)两个不同产地的10株杜仲样品的CPMGT2指纹图谱(400 MHz,DMSO-d6) Figure 2 (a) The 1H NMR spectra of HN-4 Eucommia ulmoides bark extraction acquired with the pulse sequences of PROTON and CPMGT2; (b) CPMGT2 NMR fingerprints spectra of ten Eucommia ulmoides bark extractions from two different areas (400 MHz, DMSO-d6) |
图 3为PDG的结构式,以及编号为HN-4的杜仲样品和PDG标准品的CPMGT2图谱对照,由对照图我们可以看出在HN-4杜仲样品的CPMGT2谱图中可以找到PDG对应的质子信号.PDG标准品和HN-4杜仲样品中PDG的主要质子化学位移如表 1所示.

|
图 3 编号为HN-4杜仲样品和PDG的CPMGT2谱图(400 MHz,DMSO-d6) Figure 3 The CPMGT2 spectra of HN-4 Eucommia ulmoides bark extraction and PDG standard (400 MHz, DMSO-d6) |
| 表 1 PDG特征质子信号在HN-4杜仲样品指纹图谱中的归属 Table 1 The characteristic proton assignment of PDG in fingerprint of HN-4 Eucommia ulmoides bark extraction |
对HN-4样品CPMGT2谱图调整好相位和基线,然后根据归属好的特征化合物PDG的特征峰信号进行CRAFT分析,提取其振幅值(表 2),并利用得到的振幅表对PDG信号进行模拟(图 4).
| 表 2 HN-4杜仲样品中PDG化学指纹和振幅的CRAFT分析 Table 2 Chemical shift and amplitude analysis for PDG in NMR fingerprint of HN-4 Eucommia ulmoides bark extraction using CRAFT |

|
图 4 HN-4杜仲样品中PDG的CRAFT分离模型 Figure 4 CRAFT analysis for PDG in HN-4 Eucommia ulmoides bark extraction |
从图 4中可看出,杜仲样品中PDG的特征峰依据化学指纹能从混合物信号中很好的提取出来,根据CRAFT模型还可还原混合物中PDG的峰形,达到了不分离样品而分离目标化合物信号的目的.
结合表 2和图 4可知,PDG特征峰H-5、H-2、H-6、H-7和Ar-OCH3的振幅值比为1: 1.09: 1.18: 0.89: 2.98.表 2显示CRAFT分离模型中质子振幅之比符合PDG中对应的质子数之比,表明了CRAFT分析方法对杜仲定性分析结果的准确性和有效性.
2.3.2 定量分析(1)定量原理
以编号为ZWY-4的杜仲样品为例,测得CPMGT2谱图.以间苯三酚为内标,用2015版药典绝对定量公式计算待测组分的重量:
| $ Ws = Wr \times (As/Ar) \times (Es/Er) $ | (1) |
Wr为内标物的重量,As和Ar分别为供试品特征峰和内标峰的振幅值,Es和Er分别为供试品和内标物的质子当量重量(以分子量除以特征峰的质子数计算得到),根据Ws和称样量计算组分在样品中的含量.
(2)定量峰的确认
取ZWY-4杜仲样品的CPMGT2谱图与PDG的CPMGT2图谱比较(图 5),选定H-2 [图 5(b)中2号峰]为杜仲的定量峰.内标间苯三酚定量峰为δH 5.64(s, 3H)[图 5(a)中IS].
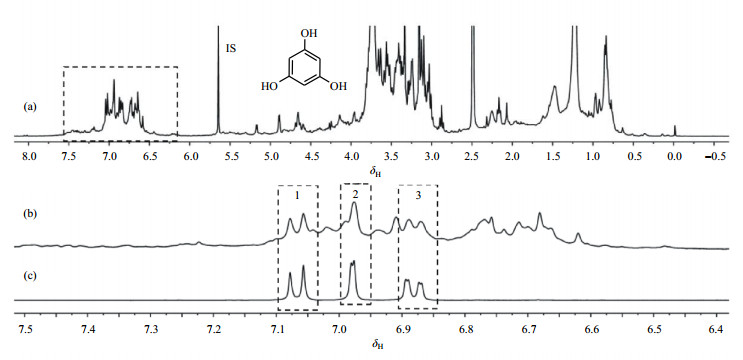
|
图 5 杜仲样品和PDG对照品的CPMGT2谱图. (a) ZWY-4杜仲样品的CPMGT2图;(b) (a)图δH 6.40~7.50段放大图;(c) PDG对照品CPMGT2谱δH 6.40~7.50段放大图 Figure 5 The CPMGT2 spectra of Eucommia ulmoides bark extraction and PDG standard. (a) The CPMGT2 spectra of ZWY-4 Eucommia ulmoides bark extraction; (b) Amplification for δH 6.40~7.50 in fig. (a); (c) Amplification for δH 6.40~7.50 of CPMGT2 spectrum of PDG standard |
(3)特征化合物PDG的定量
样品编号为ZWY-4和HN-3两个杜仲样品根据(1)式计算的NMR定量分析结果和HPLC分析的PDG含量结果如表 3所示,对应的杜仲样品溶液和PDG对照品溶液的HPLC色谱分析如图 6所示.
| 表 3 杜仲皮提取物中PDG定量分析结果 Table 3 Quantitative analysis for PDG in Eucommia ulmoides bar extraction |
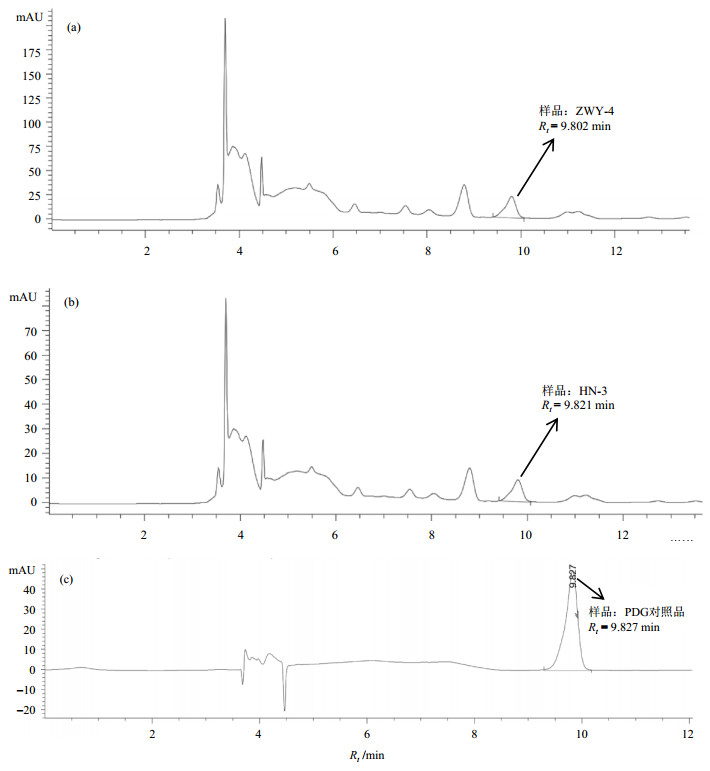
|
图 6 杜仲样品和PDG对照品溶液的HPLC色谱分析. (a) ZWY-4; (b) HN-3; (c) PDG对照品 Figure 6 HPLC chromatogram of Eucommia ulmoides bar extraction and PDG standard. (a) ZWY-4; (b) HN-3; (c) PDG standard |
HPLC外标法定量基本原理如(2)式和(3)式所示:
| $ 校正因子:\ R{F_{({X_i})}} = \frac{{标样浓度C({X_i})}}{{标样响应值R({X_i})}} $ | (2) |
| $ 未知组分浓度:\ {C_i}= R{F_{({X_i})}} \times 样品响应值{A_i} $ | (3) |
式中,由得到的Ci可以计算出杜仲样品溶液中的PDG的百分含量,进一步经过计算可以得出杜仲药材原材料中的PDG的百分含量.
表 3显示NMR定量分析测得贵阳药用植物园的杜仲ZWY-4中PDG的含量为0.275 6%,经过5次计算求的相对标准偏差(RSD)为1.69%,与HPLC定量分析结果(含量为0.269 6%,RSD为0.65%)基本一致;NMR定量分析测得HN-3杜仲样品中含PDG为0.186 7%,RSD为1.56%,与HPLC定量分析结果(含量为0.190 3%,RSD为0.73%)基本一致.
线性关系考察 配制不同浓度的ZWY-4杜仲样品,分别加入内标物间苯三酚,振荡摇匀后测定其CPMGT2谱图.根据NMR定量分析测得的含量计算不同浓度的ZWY-4杜仲样品中PDG的量,以及与内标物质量的比值;对每个样品中PDG和间苯三酚的定量峰进行CRAFT计算提取其振幅值和内标振幅值,得到两个振幅值的比值(表 4).
| 表 4 PDG和间苯三酚的质量比及振幅比 Table 4 Ratio of quality and amplitude of PDG with phloroglucol |
以PDG与间苯三酚定量峰的质量比(Ws/Wr)对PDG与间苯三酚的振幅比(As/Ar)进行线性回归分析(图 7),其线性方程为y = 0.158 3x-3E-6,相关系数(r)为0.999 3.根据(1)式理论计算线性曲线的斜率约为0.158 3,两者一致,说明可用绝对定量模式测定杜仲中松脂醇二葡萄糖苷的含量.

|
图 7 Ws/Wr和As/Ar线性关系图 Figure 7 Linear relation of Ws/Wr and As/Ar |
重复性实验 研究还考察了CRAFT定性分析方法的重复性,选取同一批杜仲样品,制备5份杜仲总提物和内标物间苯三酚的混合样品,测定其CPMGT2谱图,然后进行CRAFT定量分析结果分别为0.188 9%、0.187 6%、0.184 2%、0.190 0%、0.185 2%,表明5次利用CRAFT分析方法对杜仲总提物中PDG含量测定的结果重复性很好,可以得出利用CRAFT作为混合物中特征化合物的定量分析方法其稳定性良好、精密度较高.
2.4 杜仲指纹图谱的全分析本文采集了同一采收期来自贵州省贵阳市药用植物园(ZWY)和湖南郴州(HN)两个产地的杜仲的CPMGT2谱图,利用MestReNova(V 8.1)软件对谱图进行预处理并获得分段积分数据,运用SIMCA-P(V 13.0)软件对积分数据进行多变量分析并建立数学模型,同时也要对模型的有效性进行验证[18].常用的多变量分析包括非监督的主成分分析(Principal Component Analysis,PCA)和监督的偏最小二乘法判别分析(Partial Least Squares-Discriminant Analysis,PLS-DA)[19-21].通常,PCA分析是在最大程度上保留原始信息的基础上对高维复杂数据进行简化和降维,并获得用于描述原始数据的新变量,它一般可以用来初步分析各组样本间的总体分布和聚集状况以及异常点的识别.而PLS-DA分析可以最大程度地展现组间差异,有利于寻找对组间区分有贡献的成分.
由图 8(a)和图 8(b)显示贵阳市药用植物园和湖南郴州杜仲样品在PCA和PLS-DA模型上都有明显的区分.而且,交叉验证和排列验证结果[图 8(c)]确认了PLS-DA模型的有效性,这说明不同产地的杜仲的提取物存在显著差异.该方法对于鉴定不同产地的药材具有一定的实用意义.
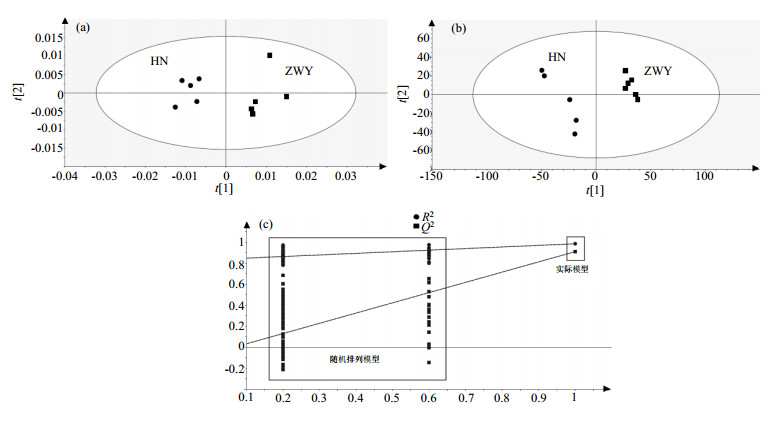
|
图 8 同一采收期来自贵州省贵阳市药用植物园(ZWY)和湖南郴州(HN)两个产地的杜仲皮提取物指纹图谱的PCA和PLS-DA分析. (a) PCA得分图;(b) PLS-DA得分图;(c) PLS-DA排列验证 Figure 8 PCA and PLS-DA analysis for NMR fingerprint of Eucommia ulmoides bark extraction from ZWY and HN. (a) PCA score plot; (b) PLS-DA score plot; (c) Permutation test for PLS-DA |
本文首先运用CRAFT技术对杜仲皮总提物指纹图谱进行了定性分析,提取出的PDG特征峰的振幅比等于其对应的质子数之比.利用CRAFT方法对杜仲皮总提取物的NMR图谱进行分析,PDG定量分析结果与HPLC定量分析结果基本一致.对来自湖南和贵州两个产地的同一采收期杜仲的全指纹数据的多变量分析显示两地的杜仲药材有显著差异.这些结果表明CRAFT技术可消除相位校正和基线校正带来的积分误差,快速简便的进行混合物中目标化合物的定性和定量分析,CRAFT与NMR技术的结合可为中药质量鉴定提供新方法.
| [1] |
FANG D M. Research and application of fingerprint technology of traditional Chinese medicine[J].
Chinese Traditional and Herbal Drugs, 2005, 36(4): 632-634.
房德敏. 中药指纹图谱技术的研究与应用[J]. 中草药, 2005, 36(4): 632-634. |
| [2] | 姚丽娜. 杜仲的化学成分研究[D]. 天津: 天津大学, 2010. http://cdmd.cnki.com.cn/Article/CDMD-10056-1011263432.htm |
| [3] | 吴贵辉. 黔产道地药材杜仲核磁共振指纹图谱研究[D]. 贵阳: 贵州大学, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10657-1015910661.htm |
| [4] | 黄重阳. 喹赛多毒理性质的代谢组学研究[D]. 武汉: 中国科学院武汉物理与数学研究所, 2013. http://cdmd.cnki.com.cn/Article/CDMD-80020-1013293786.htm |
| [5] | 李钊. 基于核磁共振的统计全相关谱的应用[D]. 武汉: 中国科学院武汉物理与数学研究所, 2007. http://cdmd.cnki.com.cn/Article/CDMD-80020-2007137253.htm |
| [6] |
YANG Y W, WANG Y L, SA R N, et al. Chemical fingerprinting of angilica sinensis at different growth stages by 1H NMR[J].
Chinese J Magn Reson, 2013, 30(1): 69-79.
杨应文, 王亚丽, 萨日娜, 等. 不同生长期当归1H NMR指纹图谱的研究[J]. 波谱学杂志, 2013, 30(1): 69-79. |
| [7] |
LUO G A, WANG Y M. Classification and development of TCM fingerprint[J].
Chinese Journal of New Drugs, 2002, 11(1): 46-51.
罗国安, 王义明. 中药指纹图谱的分类和发展[J]. 中国新药杂志, 2002, 11(1): 46-51. |
| [8] |
ZHAO T Z, LIANG X T. Identification of Chinese medicine by 1H-NMR fingerprint[J].
Chinese Traditional and Herbal Drugs, 2000, 31(11): 868-870.
赵天增, 梁晓天. 1H-NMR指纹法鉴定植物中药[J]. 中草药, 2000, 31(11): 868-870. DOI: 10.3321/j.issn:0253-2670.2000.11.037. |
| [9] |
QING H L, ZHAO T Z, DOU H Q, et al. Identification of gastrodia and it's counterfeits by nuclear magnetic resonance spectroscopy[J].
Jorunal of Chinese Medicinal Materials, 1994, 17(6): 23-24.
秦海林, 赵天增, 都恒青, 等. 核磁共振波谱法鉴别天麻及其伪品[J]. 中药材, 1994, 17(6): 23-24. |
| [10] |
QING H L, SHANG Y J, ZHAO W, et al. Study on identification of coptischinensis franch by nuclear magnetic resonance spectroscopy[J].
Chinese Traditional and Herbal Drugs, 2000, 31(1): 48-50.
秦海林, 尚玉俊, 赵伟, 等. 核磁共振氢谱法鉴别黄连的研究[J]. 中草药, 2000, 31(1): 48-50. |
| [11] | KRISHNAMURTHY K. CRAFT (complete reduction to amplitude frequency table)-robust and time-efficient bayesian approach for quantitative mixture analysis by NMR[J]. Magn Reson Chem, 2013, 51(12): 821-829. DOI: 10.1002/mrc.v51.12. |
| [12] | BRADLEY S A, SMITKA T A, RUSSELL D J, et al. Quantitative NMR analysis of complex mixtures using craft (complete reduction to amplitude frequency table) method[J]. Current Metabolomics, 2015, 3(1): 21-31. DOI: 10.2174/2213235X03666150226231519. |
| [13] |
YU S, GUO Q S, WANG H L, et al. Simultaneous determination of resveratrol and polydatin in polygonum cuspidatum by quantitative nuclear magnetic resonance spectroscopy[J].
Chinese Journal of Analytical Chemistry, 2015(1): 69-74.
禹珊, 郭强胜, 王会琳, 等. 定量核磁共振波谱法同时测定中药虎杖中白藜芦醇和虎杖苷的含量[J]. 分析化学, 2015(1): 69-74. |
| [14] | PETER P, ADEK Z A, WILL G. NMR-assignments of n-acylaporphlne alkaloids from Tinospora crispa[J]. Planta Med, 1992, 58(2): 184-187. DOI: 10.1055/s-2006-961425. |
| [15] | LE GALL G, COLQUHOUN I J, DAVIS A L, et al. Metabolite profiling of tomato (lycopersiconesculentum) using 1H NMR spectroscopy as a tool to detect potential unintended effects following a genetic modification[J]. J Agric Food Chem, 2003, 51(9): 2447-2456. DOI: 10.1021/jf0259967. |
| [16] | KOJIMA H, TOMINAGA H, SATO S, et al. Pentacyclic triterpenoids from Prunella Yulgaris[J]. Phytochemistry, 1987, 26(4): 1107-1111. DOI: 10.1016/S0031-9422(00)82359-6. |
| [17] | JUNG Y, LEE J, KWON J, et al. Discrimination of the geographical origin of beef by 1H NMR based metabolomics[J]. J Agric Food Chem, 2010, 58(19): 10458-10466. DOI: 10.1021/jf102194t. |
| [18] | 贾伟. 医学代谢组学[M]. 上海: 上海科学技术出版社, 2011: 193-221. |
| [19] | OKADA T, AFENDI F M, ALTAF-UI-AMIN M, et al. Metabolomics of medicinal plants:the importance of multivariate analysis of analytical chemistry data[J]. Curr Comput Aided Drug Des, 2010, 6(3): 179-196. DOI: 10.2174/157340910791760055. |
| [20] | SACCENTI E, TIMMERMAN E. Approaches to sample size determination for multivariate data:applications to PCA and PLS-DA of omics data[J]. J Proteome Res, 2016, 15(8): 2379-2393. DOI: 10.1021/acs.jproteome.5b01029. |
| [21] | STEWART S, IVY M A, ANSLYN E V. The use of principal component analysis and discriminant analysis in differential sensing routines[J]. Chem Soc Rev, 2014, 43(1): 70-84. DOI: 10.1039/C3CS60183H. |
 2017, Vol. 34
2017, Vol. 34 
