2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
双组分系统是一种在细菌中普遍存在的信号转导系统[1],它调控了细菌绝大多数生理过程,包括细菌的趋化性[2]、感知渗透压[3]、孢子的形成[4]、营养元素的代谢[5]等诸多生理过程.双组分信号转导系统在细菌的生命活动中起到了举足轻重的作用,但是目前为止尚未在人类和其他哺乳动物体内发现它们的存在,因此双组分信号转导系统常被作为抗菌药物的作用靶点[6].由于抗生素的过度使用,细菌的耐药性问题日益严重,通过对双组分信号转导机制进行详细研究,找到抗菌药物新靶位,将为人类疾病提供新的治疗方案[7].
双组分信号转导系统包括组氨酸激酶(Histidine Kinase,HK)和反应调节蛋白(Response Regulator,RR)[8],其信号转导过程首先是由组氨酸激酶膜外的Sensor结构域感知到外界刺激以后,使其DHp结构域上的组氨酸发生磷酸化,随后将磷酸基团转移到反应调节蛋白的天冬氨酸残基上,磷酸化的反应调节蛋白与DNA或者其他配体结合,进而调控相关基因的表达.当反应调节蛋白发生自去磷酸化或者组氨酸激酶发挥磷酸酶活性将反应调节蛋白去磷酸化后,信号转导过程终止[9-11].在整个过程中反应调节蛋白的磷酸化和去磷酸化起到了承前启后的关键作用,最终决定了该系统的信号输出和信号终止,因此其磷酸化和去磷酸化作用位点是控制其功能的关键要素[图 1(a)][12, 13].
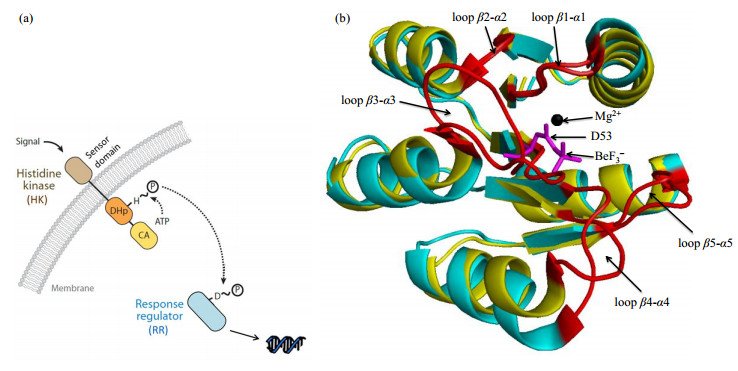
|
图 1 (a) 组氨酸激酶和反应调节蛋白的关系图;(b) apo-RR468(黄色)以及与BeF3-结合的RR468(蓝绿色)结构比对,D53和BeF3-用紫红色棍状结构显示,Mg2+用黑色小球显示,loop区域用红色表示 Figure 1 (a) The relationship of HK and RR; (b) Comparison of the structures of apo-RR468 (yellow) and BeF3--RR468(cyan), D53 and BeF3- are represented by stick mode in magenta, Mg2+ is shown by a black ball. The loops are represented in red |
大多数反应调节蛋白包括两个结构域:Receiver结构域和Effector结构域,其中Receiver结构域结构保守,形成(βα)5拓扑结构[14],不仅可以催化自磷酸化,同时位于该结构域上的天冬氨酸高度保守,用于接受组氨酸激酶传递过来的磷酸基团[15];Effector结构域结构多变,可以与DNA、RNA以及其他配体进行结合,进而产生信号输出[16, 17].反应调节蛋白执行磷酸转移和自去磷酸化功能最主要是由Receiver结构域完成[图 1(b)][18].反应调节蛋白的Receiver结构域磷酸化后会发生构象变化从而调控信号输出,最主要的构象变化体现在3个loop区,分别是loop β3-α3、loop β4-α4以及loop β5-α5[19, 20].其中变化最为明显的是loop β3-α3和loop β4-α4,它们在诱使蛋白产生磷酸化构象过程中发挥了非常重要的作用[21],我们将以这两个loop为出发点,寻找影响磷酸化和去磷酸化作用的关键位点.
有研究表明,分别位于反应调节蛋白CheY两个关键loop:loop β3-α3和loop β4-α4上的残基N59和E89突变后会对其磷酸化和去磷酸化速率产生影响[22, 23],推测二者之间存在相互作用会阻挡水分子的进攻,进而影响去磷酸化速率[24, 25].序列比对发现,这两个残基的保守性较差,该规律对于不同家族的反应调节蛋白是否具有普适性尚不清楚,同时这两个突变体对于蛋白的动力学性质的影响也不明确,而且这两个残基突变后对结构的影响也有待研究[25].
我们将发现于Thermotoga maritima中的反应调节蛋白RR468作为研究对象[26],通过与CheY蛋白进行序列比对,将RR468中与CheY的残基N59和E89对应的关键位点M55和K85分别进行突变,根据蛋白质凝胶电泳功能实验验证这两个突变体对于磷酸化和去磷酸化速率的影响.通过核磁共振(NMR)实验的化学位移扰动分析来解析这两个突变体对蛋白结构的影响[27],再利用弛豫机制对蛋白的动力学性质做研究[28],最终通过结构和动力学信息解释其对于功能影响的本质原因.
1 实验部分 1.1 蛋白样品表达与纯化 1.1.1 野生型RR468及其突变体的表达将野生型RR468或其突变体——M55A和K85E的质粒转化到大肠杆菌BL21(DE3)中,于LB培养基中37 ℃过夜培养;然后接种至15N标记氯化铵的M9培养基中,37 ℃培养至在600 nm波长处的吸光光度值(OD600)达到0.6~0.8,加入终浓度为0.5 mmol/L的异丙基硫代半乳糖苷(IPTG), 于20 ℃诱导表达16~20 h;6 000 rpm离心15 min后去上清,收集沉淀.15N选择性标记的蛋白需在诱导时向M9培养基中加入20种氨基酸,其中甲硫氨酸(Met)上的氮原子为15N标记(15N-Met).
1.1.2 野生型RR468及其突变体的纯化蛋白纯化所用层析柱均购自GE Healthcare公司.将菌液超声破碎后于60 ℃加热30 min,除去杂蛋白,20 000 rpm高速离心后留取上清.蛋白初步纯化采用QFF阴离子交换柱(其中缓冲液Buffer A含20 mmol/L Tris,Buffer B含20 mmol/L Tris及1 mol/L NaCl,pH均为8.0).Buffer A洗平后,用Buffer B从0%~30%进行梯度洗脱,合并目标蛋白[根据十二烷基磺酸钠-聚丙烯酰胺凝胶电泳(SDS-PAGE)进行判断]并浓缩样品体积至10 mL以内;蛋白用丙烯葡聚糖凝胶(Sephacryl S-100,缓冲液含20 mmol/L Tris及150 mmol/L NaCl,pH为8.0)进一步纯化,合并目标蛋白并浓缩样品体积至10 mL以内;最后用脱盐柱对蛋白进行纯化以除去盐离子(缓冲液为无菌双蒸水).分装蛋白并冻干以备用.
1.2 磷酸化及去磷酸化功能实验 1.2.1 磷酸化速率对比实验将冻干的蛋白样品溶于含有20 mmol/L Tris、50 mmol/L NaCl、10 mmol/L MgCl2,pH为8.0的缓冲溶液中,加入氨基甲酰磷酸二锂盐(分子式:NH2COOPO3Li2)作为磷酸供体模拟磷酸化[29],磷酸供体小分子与蛋白的摩尔比为10:1,调节蛋白浓度为0.5 mg/mL,于25 ℃水浴条件下进行磷酸化反应,间隔不同时间取样,时间间隔分别为5、10、15、20、25、30、40、50、60 min,取样后用液氮速冻置于-20 ℃冰箱中.以未加入氨基甲酰磷酸二锂盐的apo态作为空白对照,于90 V电压、4 ℃冰上进行非变性聚丙烯酰胺凝胶电泳(Native-PAGE),时长设为5 h.
1.2.2 自去磷酸化速率对比实验将冻干的蛋白样品溶于含有20 mmol/L Tris、50 mmol/L NaCl、10 mmol/L MgCl2,pH为8.0的缓冲溶液中,加入氨基甲酰磷酸二锂盐作为磷酸供体模拟磷酸化,于25 ℃水浴条件下进行磷酸化反应1 h,用脱盐柱洗脱除去多余的氨基甲酰磷酸二锂盐(洗脱缓冲液与磷酸化反应缓冲液相同),调节蛋白浓度至0.5 mg/mL,于25 ℃水浴条件下进行自去磷酸化反应,间隔不同时间取样,时间间隔分别为0、0.5、1、1.5、2、2.5、3、3.5、4 h,取样后用液氮速冻置于-20 ℃冰箱中.以未加入氨基甲酰磷酸二锂盐的apo态作为空白对照,于90 V电压、4 ℃冰上进行Native-PAGE,时长设为5 h.
1.3 NMR实验 1.3.1 1H-15N HSQC实验1H-15N HSQC实验于Bruker Avance 850 MHz NMR谱仪上完成.将冻干的蛋白样品溶于含有20 mmol/L HEPES、50 mmol/L KCl、10 mmol/L MgCl2和10% D2O的缓冲溶液中,蛋白终浓度为0.4~0.8 mmol/L,pH调为7.0,实验温度为298 K.实验参数为:F2(1H)和F1(15N)维谱宽分别设为10 204.08 Hz和3 015.97 Hz,采样数据点阵t2×t1 = 2 048×256,累加次数(ns)为16,弛豫等待时间(d1)为1.0 s. 15N-Met选择性标记的蛋白样品1H和15N NMR谱宽分别设为8 417.51 Hz和1 419.19 Hz,其余参数同上.化学位移扰动(CSP)由下式[30]算出:
| $ CSP=\sqrt{{{(\mathit{\Delta} {{\delta }_{{}^{1}{{\rm{H}}^{\rm{N}}}}})}^{2}}+{{(0.1\times \mathit{\Delta} {{\delta }_{{}^{\rm{15}}\rm{N}}}\rm{)}}^{\rm{2}}}} $ | (1) |
其中:
BeF3-由BeCl2和NaF的储液混合而成,其中BeCl2和NaF的摩尔比为1: 10[31],以保证溶液中BeF3-为主要配位组分.
BeF3-与RR468蛋白相互作用的1D 19F NMR实验于Bruker 600 MHz NMR谱仪上完成.将冻干的蛋白样品溶于1H-15N HSQC实验时相同的缓冲溶液中,蛋白终浓度为0.3 mmol/L,加入5倍于蛋白物质的量的BeF3-,确保蛋白被完全结合,调节pH为7.0.HK853蛋白的DHp结构域滴定实验中,RR468蛋白样品配备同上,逐渐向RR468样品中加入DHp结构域,同时调节pH为7.0.实验温度为298 K,实验参数为:谱宽为36 764.71 Hz,累加次数(ns)为1 024,弛豫等待时间(d1)为2.0 s.化学位移定标的依据是将含有5.0 mmol/L BeCl2、50.0 mmol/L NaF,pH = 7.0的溶液中的自由氟原子的化学位移定为δ0[31, 32].
1.3.3 CPMG Relaxation Dispersion实验CPMG Relaxation Dispersion实验于Bruker Avance 850 MHz NMR谱仪上完成,将冻干的蛋白样品溶于1H-15N HSQC实验时相同的缓冲溶液中,蛋白终浓度约为0.8 mmol/L,实验温度为298 K,脉冲序列见参考文献文献[33].实验参数为:F2(1H)和F1(15N)维谱宽分别设为10 204.08 Hz和3 015.97 Hz,采样数据点阵t2×t1 = 1 208×216,累加次数(ns)为16,弛豫等待时间(d1)为2.0 s,实验设为一系列不同频率的弛豫谱图,频率分别为40、80、120、160、200、320、480、640、800、960 Hz,CPMG弛豫时间为50 ms.数据处理所用软件为NESSY[34].
2 结果与讨论 2.1 M55A及K85E与野生型RR468磷酸化和自去磷酸化速率比较由于RR468的磷酸化态非常不稳定,磷酸根容易脱落,在文献中常用磷酸类似物BeF3-与RR468相互作用,从而获得稳定的磷酸化态的RR468蛋白.BeF3-能够与RR468的T83、K105残基以及活性位点附近的Mg2+配位,在19F NMR谱上出现3个信号:F1、F2和F3 [35].以此为标记,判断RR468突变体是否能够与BeF3-结合.从图 2(a)和2(b)中我们可以看出M55A或K85E的19F NMR谱中均出现F1、F2、F3的信号,证明二者均可以与BeF3-结合.谱图中δ-37附近有一个宽峰,我们根据文献将其归属为溶液中Mg2+与F-配位形成氟化镁[MgFx(H2O)6-x]的信号,该峰的强度会因蛋白浓度的不同而稍有差异,峰强会随着溶液中未结合蛋白的Mg2+量增加而增强[36].
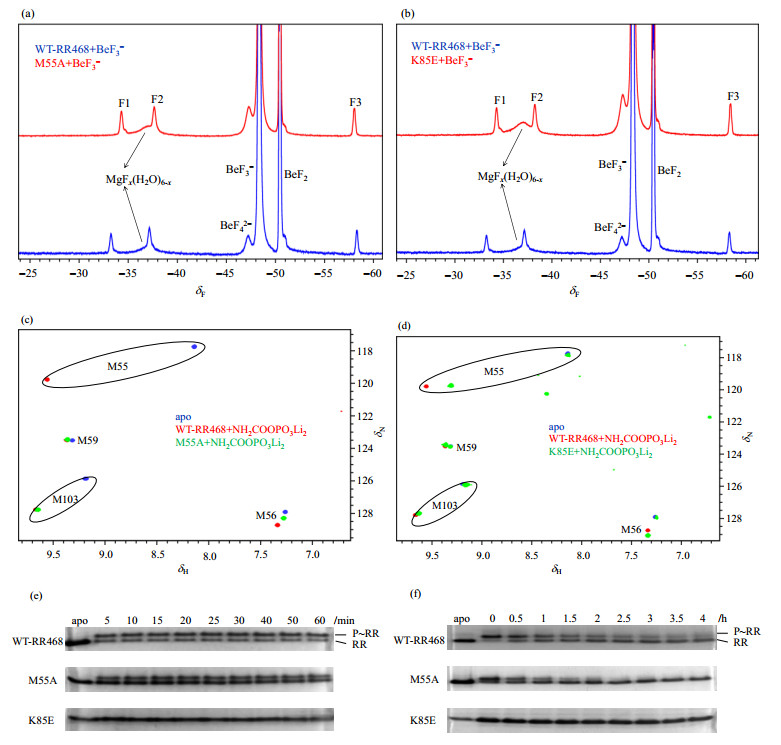
|
图 2 (a) 突变体M55A及野生型反应调节蛋白RR468与BeF3-结合后的19F NMR谱;(b)突变体K85E及野生型反应调节蛋白RR468与BeF3-结合后的19F NMR谱;(c) 15N-Met选择性标记的M55A及野生型反应调节蛋白RR468与NH2COOPO3Li2结合后的1H-15N HSQC谱;(d) 15N-Met选择性标记的K85E及野生型反应蛋白RR468与NH2COOPO3Li2结合后的1H-15N HSQC谱;(e) M55A和K85E与野生型反应调节蛋白RR468的磷酸化速率比较;(f) M55A和K85E与野生型反应调节蛋白RR468的自去磷酸化速率比较.在(e)和(f)中,P~RR代表蛋白的磷酸化状态,RR代表未磷酸化状态 Figure 2 (a) 19F NMR spectra of M55A and WT-RR468 binding with BeF3-, (b) 19F NMR spectra of K85E and WT-RR468 binding with BeF3-, (c) 1H-15N HSQC spectra of 15N-Met labeled M55A and WT-RR468 binding with NH2COOPO3Li2, (d) 1H-15N HSQC spectra of 15N-Met labeled K85E and WT-RR468 binding with NH2COOPO3Li2, (e) Measurement of phosphorylation rate of M55A, K85E and WT-RR468, (f) Measurement of autodephosphorylation rate of M55A, K85E and WT-RR468. In fig. (e) and (f), P~RR represents phosphorylated protein, and RR represents unphosphorylated protein |
RR468可以被小分子氨基甲酰磷酸二锂盐磷酸化,由于15N全部标记的蛋白样品谱峰较多,较难分辨,我们采用了对蛋白进行15N-Met选择性标记的策略.从15N-Met选择性标记蛋白的1H-15N HSQC谱图可以看出,M55A加入氨基甲酰磷酸二锂盐后,谱图发生了扰动,并且与磷酸化后的野生型蛋白的谱峰较为接近,说明M55A可以被氨基甲酰磷酸二锂盐磷酸化[图 2(c)].K85E加入氨基甲酰磷酸二锂盐后,出现两套谱峰,其中一套为apo态,另一套则为磷酸化态,说明了K85E也可以被氨基甲酰磷酸二锂盐磷酸化,但磷酸化不完全[图 2(d)].
我们用氨基甲酰磷酸二锂盐对M55A、K85E及野生型反应调节蛋白分别进行了磷酸化和自去磷酸化速率比较实验,通过Native-PAGE胶图中磷酸化条带出现和消失的时间可以对蛋白的磷酸化和自去磷酸化速率进行比较.从胶图的结果来看,M55A与野生型反应调节蛋白都是从5 min开始出现磷酸化条带并且达到稳定,因而M55A的磷酸化速率与野生型蛋白相比变化并不大[图 2(e)],但是从自去磷酸化速率比较中可以看出,野生型蛋白的磷酸化条带在2.5 h之后逐渐消失,而M55A的磷酸化条带从1 h开始逐渐消失,因而M55A的自去磷酸化速率与野生型蛋白相比有明显加快[图 2(f)].K85E从胶图上并没有看到磷酸化条带,但是根据19F NMR和1H-15N HSQC谱图可以得知其可以被BeF3-和氨基甲酰磷酸二锂盐磷酸化,由于该突变体自去磷酸化太快以至于看不到条带.通过上述实验,我们证实了M55和K85这两个残基突变后均会对蛋白功能产生影响.
2.2 M55A及K85E与HK853的DHp结构域的相互作用组氨酸激酶胞内部分包括DHp结构域和CA结构域,根据文献报道,其执行去磷酸化功能主要由其DHp结构域完成,而与其CA结构域无关[37],因此我们截取了HK853的DHp结构域与野生型RR468及其突变体进行反应.由图 3可以看出,RR468蛋白突变体——M55A和K85E一开始与BeF3-结合,处于非常稳定的磷酸化状态(最下面的19F NMR谱),随着DHp结构域的加入,BeF3--M55A或者BeF3--K85E的F1和F2附近出现新的信号,随DHp浓度的增加新信号逐渐增强,当DHp结构域过量时,旧信号完全消失,只剩下新信号.该现象的产生是由于DHp结构域的加入会逐渐产生BeF3--RR-DHp复合物,使RR468磷酸化位点发生构象变化,因此产生化学位移的变化.
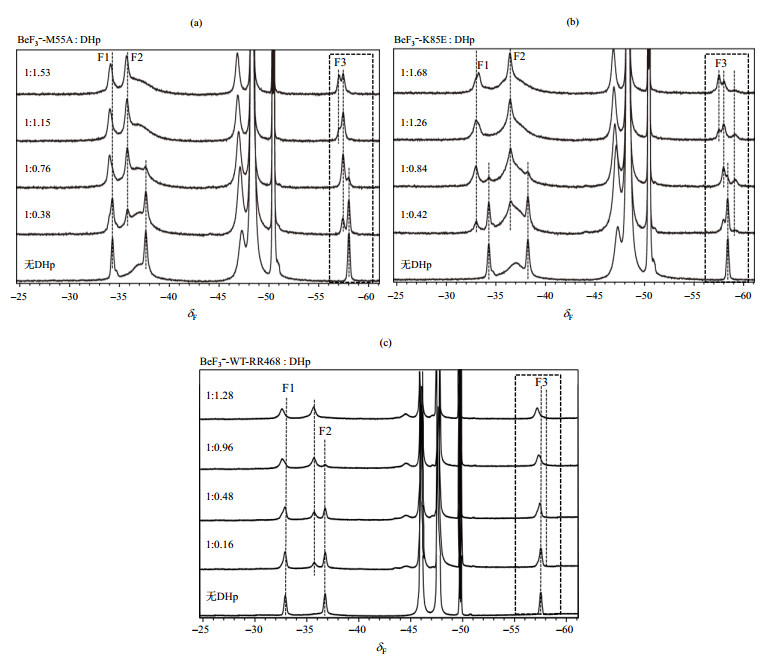
|
图 3 (a) DHp结构域滴定BeF3--M55A的19F NMR谱;(b) DHp结构域滴定BeF3--K85E的19F NMR谱;(c) DHp结构域滴定BeF3--WT-RR468的19F NMR谱 Figure 3 (a) 19F NMR spectra of BeF3--M55A by titration with DHp domain, (b) 19F NMR spectra of BeF3--K85Eby titration with DHp domain, (c) 19F NMR spectra of BeF3--WT-RR468 by titration with DHp domain |
与野生型蛋白滴定DHp结构域[35]不同的是,M55A或者K85E滴定DHp结构域的过程中F3信号附近会出现多个峰,而野生型反应调节蛋白滴定过程中F3处只有化学位移的变化没有谱峰数量的改变.F3是Mg2+的配位位点[20, 38],说明M55A或者K85E改变了野生型反应调节蛋白RR468与DHp的结合方式,并且是通过改变RR468蛋白与Mg2+的配位引起的,进一步证实了这两个残基在DHp结构域催化的去磷酸化过程中发挥了重要的作用.
2.3 M55A及K85E对于RR468三个关键loop区的动力学性质的影响蛋白在溶液中可能存在构象的交换,微秒~毫秒(μs~ms)时间尺度的构象改变往往与蛋白的催化活性或配体结合功能有关,蛋白功能的改变是否由于构象交换速率的变化引起可以通过NMR实验对动力学参数的测定进行验证[39].CPMG Relaxation Dispersion实验可用于研究μs~ms时间尺度的蛋白动力学,该实验为在固定的弛豫时间内施加不同频率(νCPMG)的180°脉冲,进而得到不同频率下的表观横向弛豫时间R2, eff,以νCPMG为横坐标、R2, eff为纵坐标,得到R2, eff衰减曲线,对衰减曲线进行拟合,便可以得到蛋白的构象交换速率kex[40].
我们通过CPMG Relaxation Dispersion实验比较了M55A和K85E这2个突变体对于3个关键loop区——loop β3-α3、loop β4-α4以及loop β5-α5上残基的构象交换速率的影响.从表 1中可以看出,野生型RR468蛋白有ms量级的构象交换现象;残基55-60位于loop β3-α3,交换速率在1 500 Hz左右;残基83-87和103-107分别位于loop β4-α4和loop β5-α5,交换速率在700 Hz左右.M55和K85分别位于loop β3-α3和loop β4-α4,但它们的突变对loop β3-α3上的残基的构象交换速率的影响并不大,而对loop β4-α4的个别残基有影响,K85的突变使loop β5-α5的交换速率稍有加快.考虑到实验误差,认为这两个突变体对于蛋白动力学性质并没有产生本质的影响,进而说明这两个突变体对于蛋白功能的影响并不是主要通过改变动力学性质引起的.
| 表 1 野生型RR468及突体M55A和K85E三个关键loop上残基的构象交换速率比较 Table 1 Comparision of conformational exchange rate for residues on three crucial loops of wild-type RR468 and mutants M55A and K85E |
HSQC谱图能够反映蛋白的总体折叠构象,M55A和K85E的HSQC谱图(图 4)变化不大,说明突变体的结构维持原有的折叠方式,仅有一些残基受到扰动.我们以氨基酸残基的序号为横坐标,M55A和K85E的1H-15N HSQC谱图分别与野生型蛋白的谱图作比较后的化学位移变化为纵坐标作图.通过化学位移扰动分析发现,M55A发生化学位移发生明显变化的结构区域主要位于loop β4-α4和loop β5-α5,而对于其自身突变所在的loop β3-α3影响并不大,仅影响了位于其前后的残基I54和M56.K85E化学位移发生明显变化的结构区域也主要位于loop β4-α4和loop β5-α5,从而说明M55A和K85E这两个突变体对于功能的改变是由于这二者对于RR468 β4-α4以及β5-α5的两个关键loop的结构的改变引起的.同时二者都对D9和D10这两个残基的化学位移产生轻微扰动,而这两个残基也是Mg2+结合位点,因此该现象也与DHp滴定实验的结果相一致.
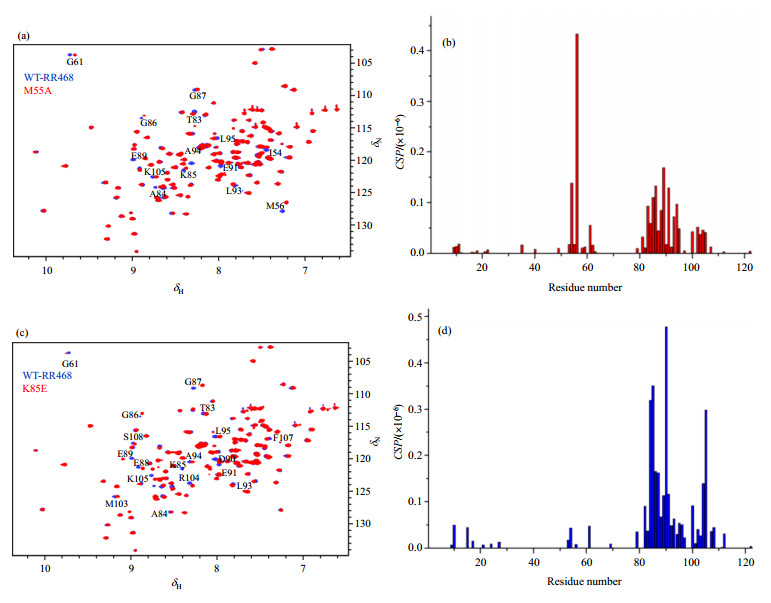
|
图 4 (a) 野生型RR468(蓝色)与M55A(红色)的1H-15N HSQC谱图比较;(b) M55A与野生型RR468相比的化学位移扰动分析;(c)野生型RR468(蓝色)与K85E(红色)的1H-15N HSQC谱图比较;(d) K85E与野生型RR468相比的化学位移扰动分析 Figure 4 (a) 1H-15N HSQC spectra of WT-RR468 (blue) and M55A (red), (b) Chemical shift perturbations analysis of M55A compared to WT-RR468, (c) 1H-15N HSQC spectra of WT-RR468 (blue) and K85E (red), (d) Chemical shift perturbations analysis of K85E compared to WT-RR468 |
我们通过Native-PAGE对细菌反应调节蛋白RR468及其突变体进行了磷酸化和自去磷酸化功能实验,结果表明M55和K85这两个残基是会影响其功能的重要残基,二者分别突变成M55A和K85E后会对RR468的磷酸化产生影响,并加快自去磷酸化速率.用HK853的DHp结构域滴定与BeF3-结合的M55A和K85E将产生蛋白复合物,突变体的蛋白复合物会产生多种构象,证实了这两个突变体会改变RR468与DHp原有的结合模式,从而证明了M55A和K85E在DHp结构域催化的去磷酸化过程中发挥了一定的作用.通过CPMG Relaxation Dispersion和化学位移扰动实验证明了两个突变体对功能的影响并不是由动力学性质的改变引起的,而是由于二者会对loop β4-α4和loop β5-α5这两个loop的结构产生影响,而这两个loop区是参与磷酸化构象转变的关键loop区.loop β3-α3对M55和K85突变的敏感度较低,无论从动力学参数还是化学位移方面受到的影响并不大,由于loop β3-α3更靠近Mg2+结合区loop β2-α2和loop β1-α1,推测它的作用可能更倾向于稳定Mg2+的结合.
综上所述,通过功能实验和NMR实验,我们证实了M55和K85在RR468执行磷酸化和去磷酸化功能中的重要性,也将为磷酸化机制的研究和药物分子的设计提供一定的理论依据.
| [1] | CASINO P, RUBIO V, MARINA A. The mechanism of signal transduction by two-component systems[J]. Curr Opin Struct Biol, 2010, 20(6): 763-771. DOI: 10.1016/j.sbi.2010.09.010. |
| [2] | MOTALEB M A, MILLER M R, LI C H, et al. Phosphorylation assays of chemotaxis two-component system proteins in Borrelia burgdorferi[J]. Methods Enzymol, 2007, 422: 438-447. DOI: 10.1016/S0076-6879(06)22022-6. |
| [3] | TRAN L S P, SHINOZAKI K, YAMAGUCHI SHINOZAKI K. Role of cytokinin responsive two-component system in ABA and osmotic stress signalings[J]. Plant Signal Behav, 2010, 5(2): 148-150. DOI: 10.4161/psb.5.2.10411. |
| [4] | CHEUNG J K, WISNIEWSKI J A, ADAMS V M, et al. Analysis of the virulence-associated RevSR two-component signal transduction system of Clostridium perfringens[J]. Int J Med Microbiol, 2016, 306(6): 429-442. DOI: 10.1016/j.ijmm.2016.05.011. |
| [5] | LEUTHNER B, HEIDER J. A two-component system involved in regulation of anaerobic toluene metabolism in Thauera aromatica[J]. FEMS Microbiol Lett, 1998, 166(1): 35-41. DOI: 10.1111/fml.1998.166.issue-1. |
| [6] | GOTOH Y, EGUCHI Y, WATANABE T, et al. Two-component signal transduction as potential drug targets in pathogenic bacteria[J]. Curr Opin Microbiol, 2010, 13(2): 232-239. DOI: 10.1016/j.mib.2010.01.008. |
| [7] | WANG S S. Bacterial two-component systems:Structures and signaling mechanisms[M]//HUANG C. Protein phosphorylation in human health. USA:InTech, 2012. |
| [8] | HWANG I, CHEN H C, SHEEN J. Two-component signal transduction pathways in Arabidopsis[J]. Plant Physiol, 2002, 129(2): 500-515. DOI: 10.1104/pp.005504. |
| [9] | CHEUNG J, LE-KHAC M, HENDRICKSON W A. Crystal structure of a histidine kinase sensor domain with similarity to periplasmic binding proteins[J]. Proteins, 2009, 77(1): 235-241. DOI: 10.1002/prot.v77:1. |
| [10] | KISHⅡ R, FALZON L, YOSHIDA T, et al. Structural and functional studies of the HAMP domain of EnvZ, an osmosensing transmembrane histidine kinase in Escherichia coli[J]. J Biol Chem, 2007, 282: 26401-26408. DOI: 10.1074/jbc.M701342200. |
| [11] | DUTTA R, QIN L, INOUYE M. Histidine kinases:diversity of domain organization[J]. Mol Microbiol, 1999, 34(4): 633-640. DOI: 10.1046/j.1365-2958.1999.01646.x. |
| [12] | BOURRET R B. Receiver domain structure and function in response regulator proteins[J]. Curr Opin Microbiol, 2010, 13(2): 142-149. DOI: 10.1016/j.mib.2010.01.015. |
| [13] | CAPRA E J, LAUB M T. Evolution of two-component signal transduction systems[J]. Annu Rev Microbiol, 2012, 66(1): 325-347. DOI: 10.1146/annurev-micro-092611-150039. |
| [14] | OCASIO V J, CORREA F, GARDNER K H. Ligand-induced folding of a two-component signaling receiver domain[J]. Biochemistry, 2015, 54(6): 1353-1363. DOI: 10.1021/bi501143b. |
| [15] | SCHNELL R, ÅGREN D, SCHNEIDER G. 1.9 Å structure of the signal receiver domain of the putative response regulator NarL from Mycobacterium tuberculosis[J]. Acta Crystallogr Sect F Struct Biol Cryst Commun, 2008, 64(Pt 12): 1096-1100. |
| [16] | VALVERDE C, HAAS D. Small RNAs controlled by two-component systems[J]. Adv Exp Med Biol, 2008, 631(6): 54-79. |
| [17] | LAGURI C, PHILLIP-SJONES M K, WILLIAMSON M P. Solution structure and DNA binding of the effector domain from the global regulator PrrA (RegA) from Rhodobacter sphaeroides:insights into DNA binding specificity[J]. Nucleic Acids Res, 2004, 31(23): 6778-6787. |
| [18] | CASINO P, RUBIO V, MARINA A. Structural insight into partner specificity and phosphoryl transfer in two-component signal transduction[J]. Cell, 2009, 139(2): 325-336. DOI: 10.1016/j.cell.2009.08.032. |
| [19] | BANERJEE R, YAN H, CUKIER R I. Conformational transition of response regulator RR468 in a two-component system signal transduction process[J]. J Phys Chem B, 2014, 118(18): 4727-4742. DOI: 10.1021/jp4122968. |
| [20] | BANERJEE R, YAN H, CUKIER R I. Conformational transition in signal transduction:Metastable states and transition pathways in the activation of a signaling protein[J]. J Phys Chem B, 2015, 119(22): 6591-6602. DOI: 10.1021/acs.jpcb.5b02582. |
| [21] | BIRCK C, MOUREY L, GOUET P, et al. Conformational changes induced by phosphorylation of the FixJ receiver domain[J]. Structure, 1999, 7(7): 1505-1515. |
| [22] | THOMAS S A, BREWSTER J A, BOURRET R B. Two variable active site residues modulate response regulator phosphoryl group stability[J]. Mol Microbiol, 2008, 69(2): 453-465. DOI: 10.1111/mmi.2008.69.issue-2. |
| [23] | PAGE S C, IMMORMINO R M, MILLER T H, et al. Experimental analysis of functional variation within protein families:receiver domain autodephosphorylation kinetics[J]. J Bacteriol, 2016, 198(18): 2483-2493. DOI: 10.1128/JB.00853-15. |
| [24] | THOMAS S A, IMMORMINO R M, BOURRET R B, et al. Nonconserved active site residues modulate CheY autophosphorylation kinetics and phosphodonor preference[J]. Biochemistry, 2013, 52(13): 2262-2273. DOI: 10.1021/bi301654m. |
| [25] | IMMORMINO R M, STARBIRD C A, SILVERSMITH R E, et al. Probing mechanistic similarities between response regulator signaling proteins and haloacid dehalogenase phosphatases[J]. Biochemistry, 2015, 54(22): 3514-3527. DOI: 10.1021/acs.biochem.5b00286. |
| [26] | CASINO P, FERNANDEZ-ALVAREZ A, ALFONSO C, et al. Identification of a novel two component system in Thermotoga maritima. Complex stoichiometry and crystallization[J]. Biochim Biophys Acta, 2007, 1774(5): 603-609. DOI: 10.1016/j.bbapap.2007.02.005. |
| [27] | DEHNER A, FURRER J, RICHTER K, et al. NMR chemical shift perturbation study of the N-terminal domain of Hsp90 upon binding of ADP, AMP-PNP, geldanamycin, and radicicol[J]. Chembiochem, 2003, 4(9): 870-877. DOI: 10.1002/cbic.v4:9. |
| [28] | MCDONALD L R, BOYER J A, LEE A L. Segmental motions, not a two-state concerted switch, underlie allostery in CheY[J]. Structure, 2012, 20(8): 1363-1373. DOI: 10.1016/j.str.2012.05.008. |
| [29] | SAINZ G, TRICOT C, FORAY M F, et al. Kinetic studies of allosteric catabolic ornithine carbamoyltransferase from Pseudomonas aeruginosa[J]. Eur J Biochem, 1998, 251(1, 2): 528-533. |
| [30] | FARMER B T, CONSTANTINE K L, GOLDFARB V, et al. Localizing the NADP+ binding site on the MurB enzyme by NMR[J]. Nat Struct Biol, 1996, 3(12): 995-997. DOI: 10.1038/nsb1296-995. |
| [31] | LIU Y X, MAO X A, LIU M L, et al. Impact of magnesium(Ⅱ) on beryllium fluorides in solutions studied by 19F NMR spectroscopy[J]. Chinese J Chem, 2014, 32(9): 878-882. DOI: 10.1002/cjoc.201400265. |
| [32] |
DAI C Y, ZHANG Z T, LIU M L, et al. Application of NMR in the studies of structure and interactions of α-synuclein[J].
Chinese J Magn Reson, 2016, 33(1): 153-167.
戴晨晔, 张则婷, 刘买利, 等. NMR在α-synuclein的结构及相互作用研究中的应用[J]. 波谱学杂志, 2016, 33(1): 153-167. DOI: 10.11938/cjmr20160115. |
| [33] | LONG D, LIU M L, YANG D W. Accurately probing slow motions on millisecond timescales with a robust NMR relaxation experiment[J]. J Am Chem Soc, 2008, 130(8): 2432-2433. DOI: 10.1021/ja710477h. |
| [34] | BIERI M, GOOLEY P R. Automated NMR relaxation dispersion data analysis using NESSY[J]. BMC Bioinformatics, 2011, 12(1): 421. DOI: 10.1186/1471-2105-12-421. |
| [35] | 刘乙祥. 细菌双组分信号转导系统蛋白质复合体结构和磷酸转移机制的研究[D]. 武汉: 中国科学院武汉物理与数学研究所, 2015. http://cdmd.cnki.com.cn/Article/CDMD-80020-1016090724.htm |
| [36] | SHIBATA N, SATO H, SAKAKI S, et al. Theoretical study of magnesium fluoride in aqueous solution[J]. J Phys Chem B, 2011, 115(35): 10553-10559. DOI: 10.1021/jp2053647. |
| [37] | HUYNH T N, NORIEGA C E, STEWART V. Missense substitutions reflecting regulatory control of transmitter phosphatase activity in two-component signalling[J]. Mol Microbiol, 2013, 88(3): 459-472. DOI: 10.1111/mmi.12195. |
| [38] |
LIU T, LIU M L, JIANG L. Divalent metal ion binding to the response regulator YycF_N studied by NMR spectroscopy[J].
Chinese J Magn Reson, 2016, 33(1): 77-88.
刘婷, 刘买利, 姜凌. 二价金属离子与YycF_N相互作用的NMR研究[J]. 波谱学杂志, 2016, 33(1): 77-88. DOI: 10.11938/cjmr20160107. |
| [39] | ISHIMA R, TORCHIA D A. Protein dynamics from NMR[J]. Nat Struct Biol, 2000, 7(9): 740-743. DOI: 10.1038/78963. |
| [40] | JIANG B, YU B H, ZHANG X, et al. A (15)N CPMG relaxation dispersion experiment more resistant to resonance offset and pulse imperfection[J]. J Magn Reson, 2015, 257: 1-7. DOI: 10.1016/j.jmr.2015.05.003. |
 2017, Vol. 34
2017, Vol. 34 
