2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
脊椎动物体内含有丰富的白蛋白.在人体血清中,白蛋白约占蛋白总含量的60%,血液中白蛋白的浓度约为50 mg/mL(~600 μmol/L).白蛋白在血液中以单体形式存在[1],其非糖基化单体分子量为66.5 k,含有585个氨基酸,其中包含17对二硫键以及1个游离的半胱氨酸残基[2].其三级结构主要由α-螺旋(α-helix)和环(loop)构成[3, 4],形成了多个结合位点,与金属离子(如Cu2+、Zn2+)、脂肪酸、氨基酸以及药物等具有很强的结合作用.因此,白蛋白最重要的生理作用便是在血液中结合这些内源性或外源性代谢物,并将其运送至靶向机体或组织[1].由于白蛋白能够结合离子,因此还可以调节并维持机体的pH和血浆的渗透压.
血液pH的正常范围基本维持在7.35~7.45之间.正常体温(37℃)下,血清pH为7.4[5].人体生理状态的改变往往会伴随或者引发血液pH的变化.人体的血液在组成和循环上精密的调节维持着机体内环境的稳定,而机体内环境的稳定离不开血清pH的稳定[6].血液中的白蛋白在pH调节中起到了至关重要的作用.在生理环境的pH范围内,白蛋白可以作为一个pH缓冲试剂来调节血液的pH值[7, 8].这主要是由于白蛋白上16个组氨酸的咪唑环以及末端的氨基酸和羧基能够被质子化.
人血清白蛋白(HSA)不仅能调节血液的pH,还是血液循环系统中一些人体必需金属离子(如Cu2+、Zn2+)的重要传输者[9-11].而Zn2+不仅为很多细胞外和细胞内蛋白所必需,还与很多的毒素(如炭疽致死因子和葡萄球菌肠毒素等[12, 13])致病有关.因此对Zn2+和白蛋白结合作用的研究尤为重要.血液中Zn2+的总浓度比较高,约为15~20 μmol/L[14].通常情况下,血浆中的Zn2+浓度会维持在一个相对稳定的水平,而在进食期间Zn2+会有一些消耗.若Zn2+浓度过低,则会引起血浆渗透压的降低或者炎症反应[15].在人体的血浆中,有高达98%的Zn2+与HSA结合[16].小鼠大肠注射实验研究[17]表明,白蛋白在血液中会结合Zn2+,并将其从大肠运输至肝脏.另外,白蛋白还会通过受体介导的细胞吞噬作用,来促进内壁细胞对Zn2+的吸收[18].
X射线(X-ray)晶体衍射和液体核磁共振(NMR)是研究蛋白质结构的主要方法.蛋白质的X-ray晶体衍射可以提供高分辨率的蛋白质空间结构信息[19-21],但并非所有的蛋白质都易于结晶,而且晶体状态并不能反映生理状态下蛋白的结构和动力学.液体NMR技术的一个突出优点是可以研究处于溶液中的蛋白质,并且不需要过多的样品前处理过程,无需破坏样品的完整性,从而大大降低了分离样品所带来的耗时和误差[22-24].利用NMR技术,能够在更接近生理环境的条件中研究生物大分子的空间构象[25].尤其是对难以得到晶体的蛋白质,NMR技术具有不可替代的优越性[26, 27].近年来NMR技术在蛋白-配体相互作用的研究[28]方面得到了越来越广泛的应用.然而天然丰度白蛋白的一维核磁共振氢谱(1D 1H NMR)不仅谱峰严重变宽,而且谱峰之间相互重叠,严重影响我们对谱图的分析.Claudia等人[29]就曾通过1D NMR谱图来研究Zn2+与HSA的结合位点,然而其谱图信噪比低,谱峰信号基线畸变严重.为此我们设计了新的谱编辑技术,通过将WaterLOGSY(water ligand observed via gradient spectroscopy)与横向弛豫加权(T2W)相结合来解决上述问题.
WaterLOGSY[30]技术是常用于检测分子间相互作用的一种谱编辑技术,但其固有灵敏度较低.为提高其灵敏度,本课题组成员对其脉冲序列进行了修改及优化,将水的辐射阻尼效应(radiation damping)应用到了WaterLOGSY的实验中(RD-WaterLOGSY)[31],并结合T2W技术,通过调节自旋回波时间过滤掉白蛋白的宽包信号[32],从而保留了白蛋白分子表面与溶剂水分子具有快速交换或强核Overhauser效应(NOE)的信号.通过T2W-RD-WaterLOGSY技术,我们研究了离子和HSA结合作用,通过观察不同浓度离子与HSA结合之后对谱图的影响,找出了能够指示Zn2+与药物结合的特征信号.
1 实验部分 1.1 仪器及试剂仪器:Bruker Avance Ⅲ 700型NMR谱仪(Bruker).
试剂:HSA固体粉末(去脂肪酸,纯度 > 98%)、重水(D2O)、二水合磷酸二氢钠(NaH2PO4·2H2O).无水磷酸氢二钠(Na2HPO4)和氯化锌(ZnCl2,分析纯)均在sigma公司购买.
1.2 样品配制不同pH的样品配制:配制一系列浓度为0.1 mol/L的磷酸盐缓冲溶液(pH 6.0~8.0,pH间隔为0.2).将HSA固体粉末溶解在pH 7.4的磷酸盐缓冲液中,配制成一定浓度的溶液,分为8份,每份均为350 μL.用已经配好的缓冲溶液100 μL调节HSA溶液pH分别至7.30、7.33、7.36、7.38、7.40、7.42、7.44和7.47.
不同浓度的Zn2+样品配制:将HSA固体粉末溶解在pH 7.4的磷酸盐缓冲液中,配制成一定浓度的溶液,分为8份,每份均为450 μL.用磷酸盐缓冲液配制一系列不同浓度的ZnCl2溶液,取50 μL分别加入到450 μL HSA溶液中,使Zn2+终浓度分别为0、30、60、100、200、250、300和500 μmol/L.
向所有上述配制好的HSA和离子的混合溶液中加入50 μL D2O(用于锁场),0.1%(w/v)的3-(三甲基硅基)氘代丙酸钠(TSP,用于NMR谱图定标δH0.00),以及2.5 μL的二硫苏糖醇(DTT,还原试剂,用于防止HSA在溶液中被氧化,其终浓度为0.5 mmol/L).HSA的终浓度为500 μmol/L.将所有样品装入直径为5 mm的NMR样品管中,在进行NMR实验之前置于4℃的冰箱中保存.
1.3 NMR实验样品的1D 1H NMR、CPMG、T2W-RD-WaterLOGSY和2D 1H-1H TOCSY谱图均在Bruker Avance Ⅲ 700型NMR谱仪上采集,对应的质子共振频率为700.2 MHz,实验温度为298 K.
常规的1D1H NMR实验采用noesypr1d脉冲序列,采用预饱和技术压制水峰,作为参照图谱.采样点数为64 k,累加次数为512,谱宽为14 097.74 Hz,等待时间为2 s,NOE混合时间为50 ms,在等待时间和混合时间均采用预饱和方法进行水峰压制.
T2W实验采用预饱和的CPMG脉冲序列,采样点数为64 k,累加次数为512,谱宽为14 097.74 Hz,等待时间为2 s,总的自旋回波时间为62 ms.
T2W-RD-WaterLOGSY实验在采样之前采用WATERGATE-W5序列来抑制水峰[33, 34],T2W采用CPMG脉冲序列,采样点数为64 k,累加次数为512,谱宽为14 097.74 Hz,等待时间为2 s,极化转移的混合时间为1.5 s,W5脉冲串的间隔时间为60 μs,CPMG总的自旋回波时间为62 ms,辐射阻尼效应调控延迟时间(Δ)为20 ms.
2D 1H-1H TOCSY实验的采样模式为States-TPPI,两维谱宽均为8 402.43 Hz,采样数据点阵t2×t1= 2 048×512,累加次数为16.所有实验的中心频率均保持一致.
1.4 实验数据处理实验数据处理均使用Bruker Topspin V3.2软件完成(Bruker Biospin,Germany).自由感应衰减(FID)信号在进行傅里叶变换之前采用指数窗函数进行处理,线宽因子为1 Hz.
2 结果与讨论HSA含有585个氨基酸,分子量大,因此常规的1D 1H NMR谱图并不能提供有用的谱峰信息.为了能够提取到HSA上的有用信息,通常采用谱编辑技术,而最常用的便是T2W技术,即CPMG自旋回波技术.
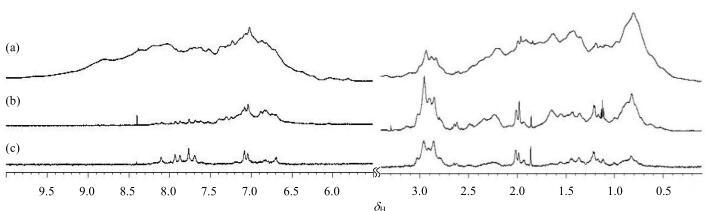
图 1比较了温度为298 K、pH为7.4时,HSA样品的常规1D 1H NMR谱、CPMG谱和T2W-RD-WaterLOGSY谱.
|
图 1 298 K、pH 7.4条件下,HSA的1H NMR谱(δ 5.0~10.0纵向放大了两倍).(a)常规1D 1H NMR谱;(b) CPMG谱;(c) T2W-RD-WaterLOGSY谱 Figure 1 1H NMR spectra of HSA at 298 K with a pH of 7.4. (a) Normal 1D 1H NMR spectrum; (b) CPMG spectrum; (c) T2W-RD-WaterLOGSY spectrum |
由图 1可以看到常规的1D 1H NMR谱图所得到的信号完全被宽峰所掩盖,因此很难得到有用的有关HSA的信息.CPMG谱虽然能够抑制宽峰,但效果并不明显,所有信号均有明显减弱,且基线仍然不平.若进一步增加总的自旋回波时间,则会将HSA结构域表面一些与水中质子有快交换的侧链信号一并抑制;而若总的自旋回波时间较短,则又不能很好地抑制宽峰.这一结果说明该技术对于大小分子混合存在的复杂体系虽然具有较好的效果,但是难以应用于单一的大分子体系.而与之不同的是,我们设计的T2W-RD-WaterLOGSY技术则具有较好的谱线简化效果.该技术主要是通过选择性激发生物样品里水中质子信号来获得大分子信息.水的磁化矢量可以通过HSA表面的活泼质子与水质子的化学交换或者NOE效应被转移到大分子上.由于大分子不同基团与水中质子之间的交换或者NOE效应不同,因此不同基团的信号增强或者传递效果不同.由图 1(c)的谱线可以明显看到,当与传统CPMG技术的自旋回波时间相同时,蛋白质低场区的信号得到了明显的简化:基线较为平整且谱峰分辨率较高.基于WaterLOGSY的原理,我们认为这些信号主要来源于HSA表面与水质子有快交换或者强NOE效应的侧链芳香基团信号.而自旋回波技术的引入,主要是用于抑制蛋白质其它基团的宽包信号.由于T2W-RD-WaterLOGSY谱图在低场区的特征信大大简化了相关蛋白质的谱线,且主要来自于分子表面,因而信号更具有代表性,因此我们后面的研究中都基于低场区信号的改变而进行.
为了明确这些信号的来源,我们通过pH和Zn2+滴定实验,以及多维NMR谱对HSA的这些特征信号进行了初步指认.
2.1 pH变化对HSA的T2W-RD-WaterLOGSY谱峰的影响质子的化学位移会随着其所处的化学环境的不同而发生改变,而在人体环境中,pH会有轻微的变化. HSA会对pH产生一定的缓冲作用,在HSA的氨基酸残基侧链中,组氨酸的咪唑环上具有两个可电离的基团,更容易受到pH变化的影响,这在谱图上表现为低场区特征峰的化学位移改变.图 2是在298 K时,不同pH条件下HSA低场区的T2W-RD-WaterLOGSY谱图.

|
图 2 不同pH条件下,HSA的T2W-RD-WaterLOGSY谱(298 K) Figure 2 T2W-RD-WaterLOGSY spectra of HSA at different pH (298 K) |
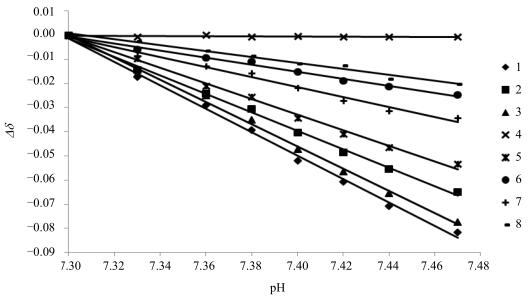
从图中可以看出随着pH值的增加,即溶液的碱性增加时,低场区的信号中除了4号峰的化学位移没有发生任何改变之外,其余峰均在一定程度上向高场方向位移.
在HSA的所有氨基酸残基中,在1H NMR谱图低场区有信号的氨基酸为组氨酸、络氨酸、苯丙氨酸和色氨酸.其中组氨酸的咪唑环最容易被质子化,且咪唑环上的两个质子信号为单峰.当环境的碱性增加时,组氨酸咪唑环上两个氮原子的质子化效应减弱,给电子效应使得环上两个质子的电子云密度增加,化学位移向高场位移.而两个氮原子之间的质子由于受到两边氮原子的影响,因此pH对它的影响比咪唑环上另一个质子大.
图 3是每个峰相对化学位移的变化趋势图.可以看出4号峰的化学位移变化在误差范围内,由此可见4号峰的化学位移对pH不敏感,不来源于组氨酸.前三个峰向高场位移了约49 Hz,而后三个峰向高场位移了约17.5 Hz,这种变化方式与我们前面对组氨酸的分析相符.通过对这种变化趋势的分析,我们认为除了4号峰之外,其他信号均来源于HSA结构域表面的组氨酸.
|
图 3 HSA低场区信号化学位移随pH改变的变化 Figure 3 The relationship between chemical shift change of HSA at low field region and pH |
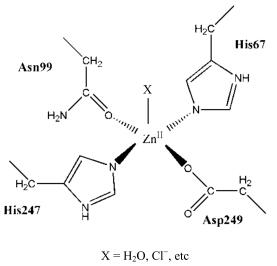
Zn2+在HSA上存在两个结合位点,其中一个是强结合位点,该位点位于由Domain Ⅰ上His67、Asn99,和Domain Ⅱ上His247、Asp249组成的A结合位点[35].
如图 4所示,Zn2+与组氨酸咪唑环上的氮原子以及天冬氨酸(天冬酰胺)上的氧原子形成配位键,从而改变了HSA上A结合位点周围的构象,这将可能导致相关基团运动性减弱、弛豫时间减小,在NMR谱图上表现为峰强度的减弱.
|
图 4 Zn2+在HSA上的结合位点 Figure 4 Zn2+ bingding site on HSA |
图 5是一系列不同浓度的Zn2+与HSA结合后的T2W-RD-WaterLOGSY谱图(HSA浓度为500 μmol/L).体系中的HSA为去离子和去脂肪酸的纯净物,因而在加入外源金属离子前,体系中不含有金属离子.由图中可以看出,随着Zn2+浓度的不断增加,5号峰和8号峰的强度逐渐减弱,而当Zn2+浓度与HSA浓度的比例为1:1(即Zn2+浓度为500 μmol/L)时,5号峰和8号峰刚好消失.

|
图 5 不同浓度的Zn2+结合在HSA上的T2W-RD-WaterLOGSY谱图 Figure 5 T2W-RD-WaterLOGSY spectra of HSA binding with different Zn2+concentration |
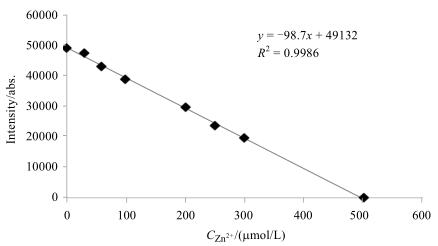
图 6是5号峰的峰强度随着Zn2+浓度的变化曲线,其线性相关性R2= 0.998 6.由图可知,5号峰的变化与Zn2+浓度的变化呈负相关.这一实验结果表明,HSA溶液中加入Zn2+后,Zn2+完全结合在了HSA上的A结合位点上,并且结合比例为1:1.
|
图 6 5号峰的峰强度与Zn2+浓度的线性关系 Figure 6 The relationship between the intensity of signal 5 and the concentration of Zn2+ |
由于5号峰和8号峰变化的一致性,我们认为这两个峰来自同一个组氨酸,而晶体结构表明Zn2+是与His67和His247结合,因此这两个峰可能来源于His67或者His247.
为了进一步对信号进行确认,我们用1H-1H TOCSY谱来对其进行归属.图 7是HSA的1H-1H TOSCY谱图.可以看到,5号峰与8号峰具有交叉峰,因此我们证实了这个两个峰确实来源于同一个组氨酸咪唑环上的两个质子.另外,我们还发现3号峰与7号峰也具有交叉峰.结合之前对pH影响的分析可知,3号峰和5号峰受pH的影响比较大,而7号峰和8号峰的影响很小,这与组氨酸咪唑环的结构完全相符:位于两个氮原子之间的质子受到了双重给电子效应的影响,对pH的敏感程度较咪唑环上另一个质子更大.T2W-RD-WaterLOGSY实验所能检测到的是位于HSA表面与溶剂水质子具有快速交换的氨基酸残基信号,而位于HSA三个结构域表面的组氨酸分别为His67、His146、His242、His247和His288.根据Zn2+与HSA相互作用的T2W-RD-Water-LOGSY谱图,我们判定5号峰和8号峰来源于His67或者His247中的一个,而3号峰和7号峰则来源于另外3个组氨酸中的一个.Blindauer等人[29]在其研究中通过对His247的突变,指出其在2D 1H-1H TOCSY谱图中的信号位于δ 8.48和δ 7.41.因此通过信号比对可以得出5号(δ 7.63)峰和8号峰(δ 6.63)来自于His67.
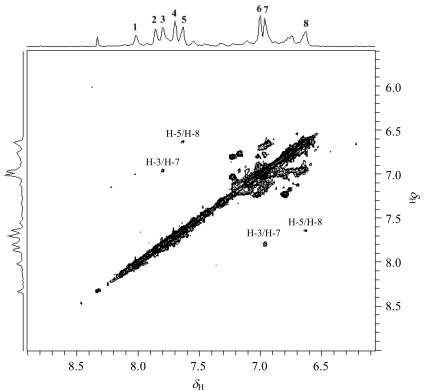
|
图 7 HSA的1H-1H TOCSY谱图 Figure 7 1H-1H TOCSY spectrum of HSA |
我们通过T2W-RD-WaterLOGSY技术观察了pH对HSA谱峰的影响,并结合1H-1H TOCSY谱,初步归属了1D T2W-RD-WaterLOGSY谱图中低场区的一些信号峰:δ 7.63和δ 6.63的谱峰来自于His67;δ 7.79和δ 6.96的谱峰则来自于白蛋白结构域表面的其他的组氨酸.δ 7.69的谱峰对pH不敏感,不是组氨酸上的信号.
通过对Zn2+与HSA结合的研究发现,Zn2+与HSA的结合为强结合,其结合比例为1:1.Zn2+与HSA上His67的结合呈现线性关系,因此δ 7.63和δ 6.63处的谱峰信号可以作为Zn2+在白蛋白上结合的1D NMR探针.该结果表明T2W-RD-WaterLOGSY技术可用于简化天然非突变蛋白的谱线,研究相关蛋白与离子或配体药物的相互作用.
| [1] | PETERS J R T. All about albumin:biochemistry, genetics, and medical applications[M]. America: Academic press, 1995. |
| [2] | DUGAICZYK A, LAW S W, DENNISON O E. Nucleotide sequence and the encoded amino acids of human serum albumin mRNA[J]. P Nat Acad Sci USA, 1982, 79(1): 71-75. DOI: 10.1073/pnas.79.1.71. |
| [3] | SUGIO1 S, KASHIMA A, MOCHIZUKI S, et al. Crystal structure of human serum albumin at 2.5Å resolution[J]. Protein Eng, 1999, 12(6): 439-446. DOI: 10.1093/protein/12.6.439. |
| [4] | HE X M, CARTER D C. Atomic structure and chemistry of human serum albumin[J]. Nature, 1992, 358(6383): 209-215. DOI: 10.1038/358209a0. |
| [5] | STEWART P A. Modern quantitative acid-base chemistry[J]. Can J Physiol Pharm, 1983, 61(12): 1444-1461. DOI: 10.1139/y83-207. |
| [6] |
LI X, LAN W X, ZHU H, et al. Effect of pH on human blood serum studied by 1H NMR spectroscopy[J].
Chinese J Magn Reson, 2008, 25(4): 494-503.
李雪, 蓝文贤, 朱航, 等. pH对血清影响的1H NMR研究[J]. 波谱学杂志, 2008, 25(4): 494-503. |
| [7] | COHN E J, STRONG L E, HUGHES W, et al. Preparation and properties of serum and plasma proteins. Ⅳ. A system for the separation into fractions of the protein and lipoprotein components of biological tissues and fluids1a, b, c, d[J]. J Am Chem Soc, 1946, 68(3): 459-475. DOI: 10.1021/ja01207a034. |
| [8] | STEINHARDT J, KRIJN J, LEIDY J G. Differences between bovine and human serum albumins. Binding isotherms, optical rotatory dispersion, viscosity, hydrogen ion titration, and fluorescence effects[J]. Biochemistry, 1971, 10(22): 4005-4015. DOI: 10.1021/bi00798a001. |
| [9] | BAL W, SOKOLOWSKA M, KUROWSKA E, et al. Binding of transition metal ions to albumin:sites, affinities and rates[J]. Biochim Biophys Acta, 2013, 1830(12): 5444-5455. DOI: 10.1016/j.bbagen.2013.06.018. |
| [10] | FERRARO G, MASSAI L, MESSORI L, et al. Cisplatin binding to human serum albumin:a structural study[J]. Chem Commun, 2015, 51(46): 9436-9439. DOI: 10.1039/C5CC01751C. |
| [11] | TURK B E, WONG T Y, SCHWARZENBACHER R, et al. The structural basis for substrate and inhibitor selectivity of the anthrax lethal factor[J]. Nat Struct Mol Biol, 2004, 11(11): 60-66. |
| [12] | TURK B E, WONG T Y, SCHWARZENBACHER R, et al. The structural basis for substrate and inhibitor selectivity of the anthrax lethal factor[J]. Nat Struct Mol Biol, 2004, 11(1): 60-66. DOI: 10.1038/nsmb708. |
| [13] | PETERSSON K, H KANSSON M, NILSSON H, et al. Crystal structure of a superantigen bound to MHC class Ⅱ displays zinc and peptide dependence[J]. EMBO J, 2001, 20(13): 3306-3312. DOI: 10.1093/emboj/20.13.3306. |
| [14] | CAROLI S, ALIMONTI A, CONI E, et al. The assessment of reference values for elements in human biological tissues and fluids:a systematic review[J]. Crit Rev Anal Chem, 1994, 24(5, 6): 363-398. |
| [15] | COUSINS R J, DUNN M A, LEINART A S, et al. Coordinate regulation of zinc metabolism and metallothionein gene expression in rats[J]. Am J Physiol Endoc M, 1986, 251(6): 688-694. |
| [16] | GIROUX E L, HENKIN R I. Macromolecular ligands of exchangeable copper, zinc and cadmium in human serum[J]. Bioinorg Chem, 1973, 2(2): 125-133. DOI: 10.1016/S0006-3061(00)80156-8. |
| [17] | PATTISON S E, COUSINS R J. Kinetics of zinc uptake and exchange by primary cultures of rat hepatocytes[J]. Am J Physiol Endoc M, 1986, 250(6): 677-685. |
| [18] | ROWE D J, BOBILYA D J. Albumin facilitates zinc acquisition by endothelial cells[J]. P Soc Exp Biol Med, 2000, 224(3): 178-186. DOI: 10.1046/j.1525-1373.2000.22418.x. |
| [19] | GHUMAN J, ZUNSZAIN P A, PETITPAS I, et al. Structural basis of the drug-binding specificity of human serum albumin[J]. J Mol Biol, 2005, 353(1): 38-52. DOI: 10.1016/j.jmb.2005.07.075. |
| [20] | CARTER D C, HE X M, MUNSON S H, et al. Three-dimensional structure of human serum albumin[J]. Science, 1989, 244(4909): 1195-1198. DOI: 10.1126/science.2727704. |
| [21] |
LIU T, LIU M L, JIANG L. Divalent metal ion binding to the response regulator YycFN studied by NMR spectroscopy[J].
Chinese J Magn Reson, 2016, 33(1): 77-88.
刘婷, 刘买利, 姜凌. 二价金属离子与YycFN相互作用的NMR研究[J]. 波谱学杂志, 2016, 33(1): 77-88. DOI: 10.11938/cjmr20160107. |
| [22] | WAGNER G. An account of NMR in structural biology[J]. Nat Struct Biol, 1997, 4: 841-844. |
| [23] | CISTOLA D P, SMALL D, HAMILTON J. Carbon 13 NMR studies of saturated fatty acids bound to bovine serum albumin. Ⅰ. The filling of individual fatty acid binding sites[J]. J Biol Chem, 1987, 262(23): 10971-10979. |
| [24] | CISTOLA D, SMALL D, HAMILTON J. Carbon 13 NMR studies of saturated fatty acids bound to bovine serum albumin. Ⅱ. Electrostatic interactions in individual fatty acid binding sites[J]. J Biol Chem, 1987, 262(23): 10980-10985. |
| [25] |
JIANG X W, SUN P, XIAO N, et al. Sensitivity enhancement in 1H-13C HSQC experiments on aromatic groups in proteins[J].
Chinese J Magn Reson, 2014, 31(1): 61-70.
蒋先旺, 孙鹏, 肖楠, 等. 蛋白质芳香基团的1H-13C HSQC信号增强研究[J]. 波谱学杂志, 2014, 31(1): 61-70. |
| [26] | HENZLER-WILDMAN K, KERN D. Dynamic personalities of proteins[J]. Nature, 2007, 450(7172): 964-972. DOI: 10.1038/nature06522. |
| [27] | SNYDER D A, CHEN Y, DENISSOVA N G, et al. Comparisons of NMR spectral quality and success in crystallization demonstrate that NMR and X-ray crystallography are complementary methods for small protein structure determination[J]. J Am Chem Soc, 2005, 127(47): 16505-16511. DOI: 10.1021/ja053564h. |
| [28] |
LIN D H, HONG J. Mapping protein-ligand interaction by NMR techniques[J].
Chinese J Magn Reson, 2005, 22(3): 321-341.
林东海, 洪晶. 用NMR技术研究蛋白质-配体相互作用[J]. 波谱学杂志, 2005, 22(3): 321-341. |
| [29] | BLINDAUER C A, HARVEY I, BUNYAN K E, et al. Structure, properties, and engineering of the major zinc binding site on human albumin[J]. J Biol Chem, 2009, 284(34): 23116-23124. DOI: 10.1074/jbc.M109.003459. |
| [30] | DALVIT C, FOGLIATTO G, STEWART A, et al. WaterLOGSY as a method for primary NMR screening:practical aspects and range of applicability[J]. J Biomol NMR, 2001, 21(4): 349-359. DOI: 10.1023/A:1013302231549. |
| [31] | SUN P, JIANG X W, JIANG B, et al. Biomolecular ligands screening using radiation damping difference WaterLOGSY spectroscopy[J]. J Biomol NMR, 2013, 56(3): 285-290. DOI: 10.1007/s10858-013-9748-4. |
| [32] | GRZESIEK S, BAX A. Measurement of amide proton exchange rates and NOEs with water in 13C/15N-enriched calcineurin B[J]. J Biomol NMR, 1993, 3(6): 627-638. |
| [33] | LIU M L, MAO X A, YE C H, et al. Improved WATERGATE pulse sequences for solvent suppression in NMR spectroscopy[J]. J Magn Reson, 1998, 132(1): 125-129. DOI: 10.1006/jmre.1998.1405. |
| [34] | WANG J, ZHANG X, SUN P, et al. The impact of pulse duration on composite WATERGATE pulse[J]. J Magn Reson, 2010, 206(2): 205-209. DOI: 10.1016/j.jmr.2010.07.007. |
| [35] | STEWART A J, BLINDAUER C A, BEREZENKO S, et al. Interdomain zinc site on human albumin[J]. P Nat Acad Sci USA, 2003, 100(7): 3701-3706. DOI: 10.1073/pnas.0436576100. |
 2017, Vol. 34
2017, Vol. 34 
