2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
核磁共振(Nuclear Magnetic Resonance, NMR)波谱用于解析蛋白质分子(分子量≤25 000)的三维空间溶液结构已发展成为一种成熟、常规的方法,在难于结晶的蛋白质结构测定和蛋白质弱相互作用界面的刻画方面独具优势.受益于结构基因组的科学家们对蛋白质结构启动计划(Protein Structure Initiative, PSI)的大量研究工作[1],目前存放到国际蛋白质数据库(Protein Data Bank, PDB)的蛋白质NMR结构已多达上万个.应用NMR波谱解析蛋白质分子的液体结构包括以下几个步骤:首先对蛋白质分子进行13C/15N的同位素标记;然后收集和处理异核多维NMR实验数据;进而归属蛋白质分子中的1H、13C和15N等核的化学位移;再获取基于化学位移归属的质子间距离、二面角和氢键等约束条件;最后采用模拟退火(Simulated Annealing, SA)算法得到蛋白质分子的三维空间结构[2, 3].其中,尽可能完全、准确地归属蛋白质分子的NMR谱峰,是解析可信赖、高质量的三维空间结构的重要条件.
大量基于标量偶合或极化传递的异核多维NMR实验,已广泛用于蛋白质分子的主链和侧链原子的化学位移归属(可参考国内外综述文章[4, 5]).蛋白质分子的NMR谱峰信息丰富,对其进行完整和准确地归属是一项费时繁琐的工作.近年来,NMR波谱学家开发了多种自动归属软件,比如Pine Server[6]和AutoAssign[7, 8]等.虽然这些软件能够进行快速的谱峰自动归属,但其归属结果往往不完整,并可能存在错误.对于从事蛋白质NMR波谱研究的研究生和初学者来说,因为缺乏对蛋白质分子的NMR谱峰特性的系统认识,可能会出现对自动归属结果不完整、甚或错误地指认和判断,进而导致蛋白质结构解析的偏差或错误.蛋白质分子的NMR谱峰非常复杂.这种复杂性主要来自以下几个方面:(1)蛋白质分子的动力学特性,包括特定的局部区域(比如N端、C端和loop区域)的动力学问题(比如构象交换)、部分自旋核的快速弛豫、溶质活泼质子与溶剂质子间的快速交换等,导致一些预期可观测的NMR信号消失;(2)蛋白质分子固有的NMR特性,比如谱峰重叠严重和一些原子核的化学位移简并等;(3)NMR实验中的伪峰、噪音以及抑制不完全的溶剂峰等.以上因素会导致蛋白质分子的NMR谱图常常不是初学者所期望的那样完美,为谱峰的准确归属带来不确定性.
本文并不旨在综述蛋白质分子中的原子化学位移归属的基本策略,而是针对蛋白质分子的NMR谱峰特性,作者通过多年的蛋白质NMR结构研究的经验,结合具体的蛋白质分子NMR实验谱图,为进入蛋白质NMR研究领域的新手,介绍蛋白质分子(分子量≤25 000)NMR谱峰归属的一些细节知识,包括二维1H-15N异核单量子相关谱(1H-15N HSQC)中的谱峰信息与15N维的谱宽优化、立体异构、同位素效应、恒时或非恒时实验以及核Overhauser效应谱(Nuclear Overhauser Effect SpectroscopY,NOESY)中的谱峰模式等.本文例举出的NMR谱图数据均来自溶液结构已经解析、且分子量不大于20 000的蛋白质,因为讨论重点在于蛋白质NMR谱峰特性与化学位移归属,而不涉及蛋白质的来源和功能,所以将不提及蛋白质的具体名字.为文中称呼一致,蛋白质分子中的原子命名均遵照国际理论与应用化学联合会(IUPAC)命名法[9],它也是液体蛋白质结构计算软件CYANA所采用的命名法[2].蛋白质分子中氨基酸主链分子式,及本文中将较为详述的11种氨基酸侧链分子式及各个原子命名如图 1所示,供读者阅读参考.
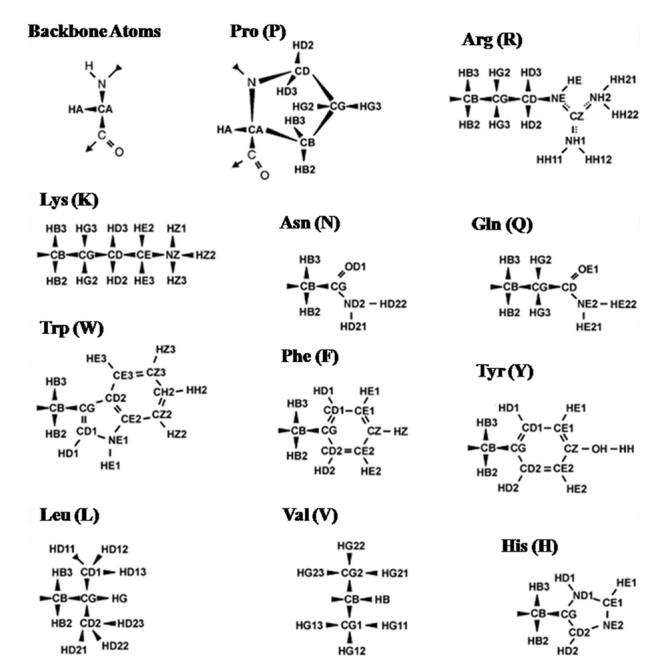
|
图 1 蛋白质分子中的氨基酸主链及本文中将讨论的一些氨基酸的侧链分子式及各个原子的名称[9] Figure 1 Amino acid backbone formula in proteins and side chain formulas of some amino acids discussed in this article, all atoms are indicated as well [9] |
蛋白质分子的二维1H-15N HSQC谱包含了丰富的信息.首先,共振谱峰的信噪比反映蛋白质样品含量;其次,共振谱峰在两维的展宽程度,能够反映蛋白质的折叠程度;此外,二维谱中可观测的共振峰数目,还能够提供蛋白质的动力学信息.比如对于具有不同构象的局部区域,一定速率的构象交换会导致一些NMR谱峰的消失,而慢交换会导致处于不同构象的同一原子的共振峰以不同的化学位移同时出现.
蛋白质分子的二维1H-15N HSQC谱中的交叉峰来自直接相连的H-N偶合.通常,对于具有特定三维空间结构的折叠良好的球形蛋白质来说,主链质子的化学位移覆盖在δ 6~12的范围,而15N的化学位移主要覆盖在δ 100~140的范围.这些共振峰来自氨基酸主链(不含主链氢原子的Pro除外)和一些氨基酸侧链的H-N偶合,包括Asn中的ND2-HD21/HD22、Gln中的NE2-HE21/HE22和Trp中的NE1-HE1[图 2(a),W30HE1和W51HE1].当15N维的谱宽很大时,在15N维高场区还能观测到Arg侧链的NE-HE[图 2(a),R36HE和R49HE].此外,特定环境下能与溶剂质子慢交换的His侧链的ND1-HD1,Arg的NH1-H11/H12和NH2-H21/H22,以及Lys的NZ-HZ1/HZ2/HZ3的信号也可能出现在1H-15N HSQC谱中.
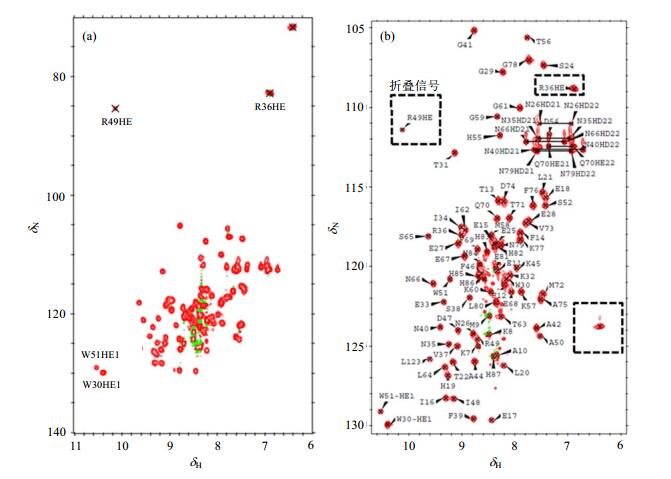
|
图 2 二维1H-15N HSQC谱(850 MHz谱仪, 1H NMR和15N NMR的工作频率分别为850.307 MHz和86.171 MHz).(a) 15N维谱宽为δ 150;(b) 15N维谱宽为δ 26,中心频率位于δ 118.3个方框区为折叠的信号,且不与其他谱峰重叠 Figure 2 2D 1H-15N HSQC spectra (850 MHz, the working frequency of 1H NMR and 15N NMR are 850.307 MHz and 86.171 MHz, respectively). (a) 15N sweep width is δ 150; (b) 15N sweep width is δ 26, and on-resonance frequency is at δ 118. The signals in three resonances in rectangles are folded, but not overlapped with the other resonances |
生物磁共振数据库(Biological Magnetic Resonance Data Bank, BMRB)中,有由统计分析得到的抗磁性蛋白质分子中NMR可观测的各个原子核的化学位移值,为蛋白质分子中各个原子核的化学位移归属提供了参考.虽然15N核的化学位移值主要分布在δ 100~140区域,但Arg侧链的NE(δ 84左右)和NH1/NH2(δ 70~75)却远离主链H-N信号区.在进行常规的蛋白质分子多维异核NMR实验时,为提高分辨率、减少总体采样时间,在观测主链H-N相关信号时,15N维通常会采用较小谱宽.此时,所给谱宽范围外的信号就会产生折叠,不合适的谱宽设置将导致折叠进来的信号与其他谱峰重叠,为谱峰的正确识别带来干扰.因此,15N维谱宽和中心频率需要优化.例如,在图 2(b)中,15N维谱宽为δ 26,中心频率位于δ 118,3个折叠谱峰(方框内)与其它谱峰不产生重叠.因此,正确解读1H-15N HSQC谱,识别不同的H-N共振峰信号,设置合适的15N维谱宽和中心频率,将为蛋白质分子的化学位移准确归属奠定良好的开端.
2 Asn/Gln>侧链NH2的化学位移归属与同位素效应基于标量偶合进行磁化传递的三维异核NMR实验包括CBCA(CO)NH/HNCACB、HNCO/ HN(CA)CO和HNCA/HN(CO)CA等,能够准确归属二维1H-15N HSQC谱中蛋白质分子主链的H-N谱峰.然而,准确归属Asn和Gln中侧链酰氨基团(-CONH2)中的NH2谱峰,还需要注意两点:(1)酰氨基团所具有的平面特性会使-CONH2中两个质子呈现立体异构,而导致化学位移不等价.当它们的化学位移不简并时,如图 3(a)中6条横线所示,已归属的5个Asn和1个Gln的-CONH2中的H-N谱峰是成对出现的.(2)氘(D或2H)同位素效应会对15N NMR化学位移产生影响.以加入10%的锁场重水(D2O)为例,Asn和Gln中侧链的-CONH2通过长时间的氢-氘(H-D)交换后,大约有90%的-CONH2和10%的-CONDH(忽略1%左右的-COND2)存在.如图 3(a)中竖向箭头所示,D会引起一键相连的15N NMR化学位移向高场移动大约δ 0.5,且谱峰的相对强度反映了两者的相对含量.
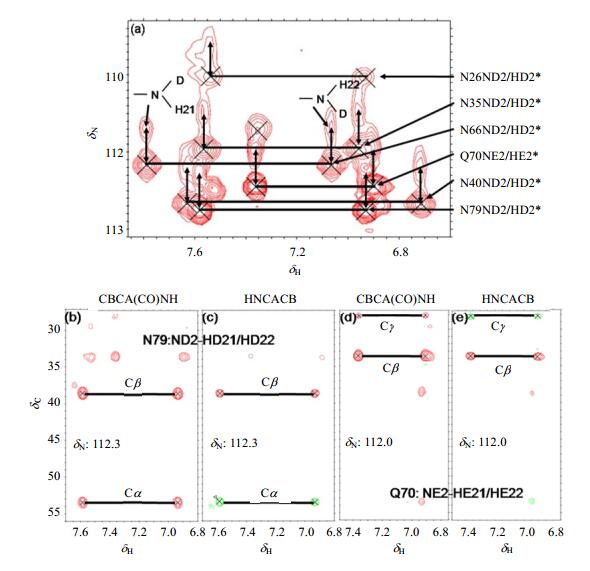
|
图 3 (a) Asn和Gln侧链-NH2信号归属,以及用于归属(b, c) Asn79和(d, e) Gln70中-NH2谱峰的CBCA(CO)NH/HNCACB谱 Figure 3 NH2 assignments for Asn and Gln side-chain (a), and CBCA(CO)NH/HNCACB spectra for side chain NH2 assignments of Asn79 (b, c) and Gln70 (d, e) |
Asn和Gln侧链的-NH2谱峰能够从CBCA(CO)NH/HNCACB谱中归属.需要指出的是,只有Asn和Gln侧链是-CONDH形式时,在CBCA(CO)NH/HNCACB谱中才出现类似Cα/Cβ的信号,这是因为只有-CONDH这种形式,与主链-CONH在三维实验CBCA(CO)NH/HNCACB中的磁化矢量传递才是类似的.此时,15N核的化学位移存在氘同位素效应,在CBCA(CO)NH/HNCACB谱中观测到的Asn79信号来自Cα/Cβ [图(3b)和3(c)],而Gln70信号来自Cγ/Cβ [图(3d)和3(e)].结合Cα/Cβ或者Cγ/Cβ的化学位移值,可以对Asn和Gln侧链-NH2进行准确归属.此外,对于Asn侧链的HD21/HD22和Gln侧链的HE21/HE22的立体异构归属,则要通过15N NOESY谱中这两个质子与自身氨基酸中的Hβ(Asn)或Hγ(Gln)的交叉峰强度来确定它们之间的距离,进而判断其立体构型.
3 蛋白质分子中立体异构原子的化学位移归属蛋白质分子通过大量疏水作用折叠成特定的三维空间结构.因此,某些氨基酸分子中一些单键(比如氨基酸Val和Leu中的两个侧链末端甲基)的自由旋转,可能会受到空间阻碍,从而呈现立体异构和化学位移不等价.Pro侧链CD与主链N原子相连形成五元吡咯烷环,当Pro与前一个氨基酸的羧基形成肽键时(Xaa-Pro),五元吡咯烷环的空间位阻会导致Pro出现顺式(cis-)和反式(trans-)两种不同的构象.此外,His中的咪唑环上存在两个N原子,当H+结合到不同的N原子形成中性分子时,会形成两种不同的互变异构体(δ-或ε-tautomer).以下部分将详述具有立体异构的原子化学位移归属方法.
3.1 Leu和Val侧链末端甲基的立体异构归属与二维恒时/非恒时实验球蛋白中Leu和Val中侧链末端的两个甲基通常因为空间位阻呈现出立体异构,当两者化学位移不简并时,两个立体异构是可以区分的.对蛋白质进行13C同位素标记时,在异丙基硫代半乳糖苷(IPTG)诱导前的M9培养液中加入5%的13C标记的葡萄糖(95%为天然丰度的葡萄糖)[10],蛋白质表达时,Leu的13C同位素标记的甲基表现方式为:13CD1-13CG和13CD2-12CG.类似地,Val中13C同位素标记的甲基表现方式为:13CG1-13CB和13CG2-12CB.因此,Leu中13CD1-13CG和Val中13CG1-13CB存在13C-13C偶合,而相应的13CD2-12CG(Leu)和13CG2-12CB(Val)则没有(忽略天然丰度大约为1%的13C-13C偶合).采用13C演化期间恒时或者非恒时的二维1H-13C HSQC谱,能够区分Leu和Val中具有立体异构效应的两个甲基.以Leu为例,当采用恒时脉冲序列时,通过13C-13C的J-偶合作用,可以让13CD1-13CG信号相位与13CD2-12CG相反.当采用非恒时脉冲序列时,采集13C维高分辨率的二维1H-13C HSQC谱,13CD1-13CG信号会因为13C-13C的J-偶合作用而发生裂分出现两个谱峰;而13CD2-12CG因为没有偶合表现为单峰.例如图 4(a)和4(b)分别为5%的13C标记蛋白质样品的恒时和非恒时的1H-13C HSQC谱图(甲基区域).图 4(a)中来自Leu中的13CD1(绿色)和13CD2(红色)信号相位相反;图 4(b)中的13CD1出现偶合双峰,而13CD2只有单峰.因此,基于实验图谱可以准确归属具有立体异构效应的Leu侧链末端甲基.类似地,Val中两个甲基的立体异构也能够区分和归属.

|
图 4 5% 13C标记蛋白质样品的二维(a)恒时和(b)非恒时1H-13C HSQC(甲基区域) Figure 4 2D constant time (a) and none-constant time; (b) 1H-13C HSQC spectra of 5% 13C-labeled protein (methyl group region) |
类似地,对13C同位素标记的蛋白质分子中的芳香族氨基酸(包括Tyr、Phe、Trp和His),基于与芳香环上13C一键相连的其他13C原子数目的不同,应用二维恒时或非恒时1H-13C HSQC谱能够对这些谱峰进行指认.由图 1中芳香族氨基酸分子式可知,与His中的13CE1一键相连的13C原子数目为0,与His中的13CD2和Trp中的13CD1一键相连的13C原子数目为1,而与Phe、Tyr和Trp中其它芳香环13C一键相连的13C原子数目都为2.芳环区的二维恒时1H-13C HSQC谱[图 5(a)]显示,Trp40的13CD1,Trp51的13CD1和His17的13CD2(1个一键相连的13C),它们的信号(绿色)相位与其他信号(红色)相反;而在非恒时谱中[图 5(b)],它们则因偶合裂分而呈现二重峰.而His17和His20的13CE1因没有一键相连的13C,在非恒时谱中呈现为单峰.其余的芳香环13C,因有两个一键相连的13C发生偶合裂分而出现3重峰.

|
图 5 二维(a)恒时和(b)非恒时1H-13C HSQC谱(芳香环区域) Figure 5 2D constant time (a) and none-constant (b) time1H-13C HSQC spectra (aromatic region) |
Pro在形成肽键时,其自身侧链与主链N原子形成吡咯烷环,不仅主链氢原子会消失,而且其吡咯烷环的空间位阻效应会导致在肽键形成时产生顺式和反式两种立体异构[图 6(a)].蛋白质分子中的Pro主要以反式结构存在,但偶尔也出现顺式的异构体.如图 6(a)所示,对于反式构象,前一个氨基酸的HA与Pro的HD距离会很近,而与HA距离远;反之,则前一个氨基酸的HA与Pro的HA距离会很近,而与HD距离远.质子间的空间距离越近,NOESY谱中两者的交叉峰越强.因此,通过观测NOESY谱中的交叉峰,可以简单快速地判断Pro中吡咯烷环的顺反式异构.参照表 1中某蛋白质中顺序相连的4个氨基酸Ala46、Pro47、Leu48和Pro49的一些化学位移值和NOESY谱[图 6(b)],通过归属NOESY中与Ala46和Leu48的HA产生的交叉峰信号为HA(Ala46)-HD(Pro47) 和HA(Leu48)-HA(Pro49),能够断定Pro47为反式构象而Pro49为顺式构象.同时,NOESY谱中Pro47的HD和Pro49的HA的交叉峰可以验证相同归属.此外,研究报道[11],统计分析发现Pro的顺反式构象可以通过Pro中的13CB与13CG的化学位移值之差△(CB-CG)确定,通常顺式构象的差值较大,而反式构象的差值较小.在本例中,Pro47的差值为δ 4.3,而Pro49的差值为δ 8.6,因此Pro47应该为反式,而Pro49应该为顺式,与前一种基于NOE交叉峰归属方法的结果一致.
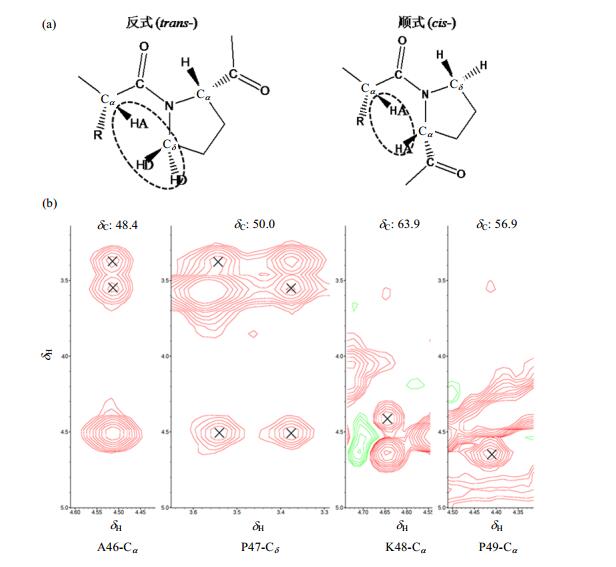
|
图 6 (a) Xaa-Proline肽键的反式和顺式示意图;(b) 13C NOESY谱中Xaa-Proline肽键的反式和顺式识别的NOEs模式 Figure 6 (a) trans- and cis- Xaa-Proline peptide bond; (b) NOEs pattern for trans-/cis- Xaa-Proline peptide bond identification in 13C NOESY spectra |
| 表 1 蛋白质分子中的4个氨基酸(Ala46, Pro47, Leu48和Pro49)的部分化学位移 Table 1 Chemical shifts of some resonances of four amino acids (Ala46, Pro47, Leu48 and Pro49) from one protein sample |
蛋白质分子中的His咪唑环上有两个氮原子,即ND1和NE2(图 1).取决于咪唑环在蛋白质结构中的位置和外部缓冲液的pH值,His可以以带正电荷的质子化(酸性)、中性和带负电荷的去质子化(碱性)等不同形式存在.对于中性His,如果质子与ND1相连称为δ-异构体,与NE2相连称为ε-异构体[图 7(a)].由于与溶液中水分子存在快交换,二维1H-15N HSQC谱中不能观测到咪唑环上的H-N交叉峰.然而,通过偶合常数较小的2JHN和3JHN进行磁化转移,采用合适的磁化转移时间,在二维1H-15N HSQC谱中能够观测到ND1/NE2与HE1、HD2的交叉峰.研究报道[12],图 7(a)的3种不同形式的His咪唑环,ND1/NE2与HE1、HD2的4个交叉峰在二维1H-15N HSQC谱中表现出不同的模式[图 7(b)].因此,通过检测交叉峰模式,可以反过来推测出His咪唑环的形式.
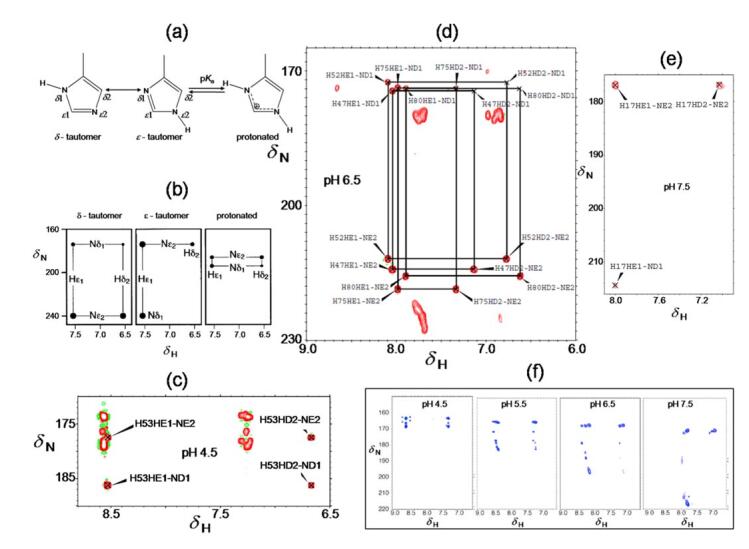
|
图 7 (a) His咪唑环的中性δ-和ε-异构体,以及酸性带正电荷的构象;(b)图(a)中His咪唑环3种不同构象的H-N交叉峰在二维1H-15N HSQC中(通过2JHN和3JHN进行磁化转移)的模式[12];(c~e) 3个不同的蛋白质中His的H-N相关(2JHN和3JHN)的二维1H-15N HSQC谱,3个蛋白质所在的缓冲溶液pH分别为4.5、6.5和7.5;(f)蛋白质中His6-tag的6个His咪唑环上H-N(2JHN和3JHN)交叉峰随pH变化的模式,pH分别为4.5、5.5、6.5和7.5 Figure 7 (a) Three different forms of imidazole ring in histidine: neutral forms (including δ-and ε-tautomer), and positively charged protonated form; (b) Schematic cross resonance patterns of H-N (via 2JHNand3JHN) for the three different imidazole ring forms shown in (a), which were observed in 2D 1H-15N HSQC[12]; (c~e) 2D 1H-15N HSQC spectra of H-N (via 2JHNand 3JHN) in histidine imidazole rings from three different proteins at pH of 4.5, 6.5 and 7.5 as indicated, respectively; (f) 2D 1H-15N HSQC of H-N (via 2JHNand 3JHN) from histidine imidazole rings in protein His6-tag at pH of 4.5, 5.5, 6.5 and 7.5 |
图 7(c)、7(d)和7(e)是来自3个不同蛋白质分子的二维1H-15N HSQC谱(通过2JHN和3JHN进行磁化转移),通过H-N交叉峰模式,不仅可以确定各个His中咪唑环的互变异构形式,还可以归属出His侧链中咪唑环上的ND1、NE2、HE1和HD2的化学位移.另一个有趣的现象,在N-或C-端以His6-tag标记且不再有其他His的蛋白质分子的二维1H-15N HSQC谱中[图 7(f)],当pH为4.5时,咪唑环明确地以质子化带正电荷的形式存在;而当pH分别为5.5、6.5和7.5时,咪唑环变为ε-异构体;同时,NE2和ND1,特别是ND1的化学位移向低场移动,HE1(-CE1) 和HD2(-CD2) 的化学位移向高场移动.因此,根据His6-tag标记的蛋白质样品中His信号的化学位移值,反过来也可以判断样品的pH环境.
4 结合蛋白质的空间结构利用NOESY谱对谱峰进行快速指认蛋白质分子的空间结构信息有助于化学位移的正确归属,但其价值和重要性往往容易被初学者所忽视.Wüthrich[13]在《蛋白质与核酸的核磁共振》(NMR of proteins and nucleic acids)一书中,根据蛋白质分子的二级结构特征,包括α-螺旋、310-螺旋(图 7.11,第122页)、平行和反平行的β-折叠(图 7.13,第126页),总结了在NOESY谱中氨基酸质子间交叉峰信号强度的模式(图 9.1,第166页).譬如,在15N编辑的NOESY谱中,α-螺旋区域中的氨基酸主链质子与前后氨基酸主链质子会出现强的交叉峰,即Hi-Hi-1和Hi-Hi+1,而β-折叠与loop区域中不会出现.反之,如果在15N编辑的NOESY谱中能够观测到强的Hi-Hi-1和Hi-Hi+1交叉峰,一方面表明这些氨基酸具有α-螺旋的二级结构;另一方面,非常有助于自动归属结果的判断.类似地,对于具有反平行的β-折叠区,一条β-折叠中氨基酸的HN(或HA),和与之反平行的β-折叠链上的HN/HA存在特定的NOE交叉峰模式,这类信息非常有助于具有β-折叠的蛋白质二级结构的预测和相关原子的化学位移归属.
应用NOESY谱中1H-1H间的交叉峰可以验证化学位移归属.相对于旨在用主链和侧链原子化学位移归属的多数三维异核NMR实验,NOESY谱往往具有更高的分辨率与灵敏度.譬如,当同一氨基酸中具有两个化学位移值相差较小的HB原子时,通常HBHA(CO)NH实验分辨率低,两个HB原子谱峰可能因为部分重叠而被认为是简并的,但通过高分辨率的NOESY谱,可以更清楚地将两个HB信号分开,从而减少NOE归属的错误或者不能归属.类似地,杨代文研究组[14]提出过一个全新的结构解析策略,不需采用常规的同位素标记和进行主链/侧链原子的化学位移归属,直接通过NOESY实验归属1H-1H NOE交叉峰,进行分子量大的蛋白质NMR结构计算.
部分折叠蛋白质的结构研究是当前的一个研究热点.然而,对于没有或者仅具有少许二级结构的蛋白质,其二维1H-15N HSQC谱中的谱峰展宽有限,特别是分子量大的蛋白质,谱峰重叠非常严重,化学位移归属成为很大的挑战,目前已有报道[15]通过五维甚或七维的异核NMR实验来解决化学位移的归属问题.
5 其它本文结合氨基酸分子的特征,讨论了蛋白质分子化学位移归属,但并未涉及到化学位移归属的所有方面.随着对蛋白质NMR研究的经验积累,初学者对其他的小细节问题会获得更多的认识.比如,通过三维HNCO实验能够归属前一个氨基酸主链CO的化学位移,通过HBHA(CO)NH能够归属前一个氨基酸的HA的化学位移.但对Pro来说,因为没有主链氢原子,由上述两个实验不能归属出前一个氨基酸的CO和HA的化学位移,而是需要通过HN(CA)CO和HNHA实验辅助解决.此外,非常靠近溶剂中水峰的个别氨基酸的HA原子信号,由于实验过程中的溶剂峰抑制常会消失,通过将蛋白质样品溶于重水,实验时无需水峰抑制,会有助于这些谱峰的正确指认.因此,蛋白质分子的NMR谱峰归属,对蛋白质的结构解析和功能研究是一个很基本、很重要但又有些复杂的过程.在此,期望本文的讨论对从事蛋白质NMR的研究者有所帮助.
| [1] | MONTELIONE G T. The protein structure initiative:achievements and visions for the future[J]. F1000 Biol Rep, 2012, 4: 7. DOI: 10.3410/B4-7. |
| [2] | GUNTERT P. Automated NMR structure calculation with CYANA[J]. Methods Mol Biol, 2004, 278: 353-378. |
| [3] | GUNTERT P, BUCHNER L. Combined automated NOE assignment and structure calculation with CYANA[J]. J Biomol NMR, 2015, 62(4): 453-471. DOI: 10.1007/s10858-015-9924-9. |
| [4] |
HU Y F, JIN C W. NMR studies of protein solution structures and dynamics[J].
Chinese J Magn Reson, 2009, 26(2): 151-172.
胡蕴菲, 金长文. 蛋白质溶液结构及动力学的核磁共振研究[J]. 波谱学杂志, 2009, 26(2): 151-172. |
| [5] | SATTLER M, SCHLEUCHER J, GRIESINGER C. Heteronuclear multidimensional NMR experiments for the structure determination of proteins in solution employing pulsed field gradients[J]. Prog Nucl Magn Reson Spectrosc, 1999, 34(2): 93-158. DOI: 10.1016/S0079-6565(98)00025-9. |
| [6] | BAHRAMI A, ASSADI A H, MARKLEY J L, et al. Probabilistic interaction network of evidence algorithm and its application to complete labeling of peak lists from protein NMR spectroscopy[J]. PLoS Comput Biol, 2009, 5(3): e1000307. DOI: 10.1371/journal.pcbi.1000307. |
| [7] | MOSELEY H N, MONLEON D, MONTELIONE G T. Automatic determination of protein backbone resonance assignments from triple resonance nuclear magnetic resonance data[J]. Methods Enzymol, 2001, 339: 91-108. DOI: 10.1016/S0076-6879(01)39311-4. |
| [8] | MOSELEY H N, SAHOTA G, MONTELIONE G T. Assignment validation software suite for the evaluation and presentation of protein resonance assignment data[J]. J Biomol NMR, 2004, 28(4): 341-355. DOI: 10.1023/B:JNMR.0000015420.44364.06. |
| [9] | MARKLEY J L, BAX A, ARATA Y, et al. Recommendations for the presentation of NMR structures of proteins and nucleic acids[J]. J Mol Biol, 1998, 280(5): 933-952. DOI: 10.1006/jmbi.1998.1852. |
| [10] | NERI D, SZYPERSKI T, OTTING G, et al. Stereospecific nuclear magnetic resonance assignments of the methyl groups of valine and leucine in the DNA-binding domain of the 434 repressor by biosynthetically directed fractional 13C labeling[J]. Biochemistry, 1989, 28(19): 7510-7516. DOI: 10.1021/bi00445a003. |
| [11] | SCHUBERT M, LABUDDE D, OSCHKINAT H, et al. A software tool for the prediction of Xaa-Pro peptide bond conformations in proteins based on 13C chemical shift statistics[J]. J Biomol NMR, 2002, 24(2): 149-154. DOI: 10.1023/A:1020997118364. |
| [12] | PELTON J G, TORCHIA D A, MEADOW N D, et al. Tautomeric states of the active-site histidines of phosphorylated and unphosphorylated ⅢGlc, a signal-transducing protein from Escherichia coli, using two-dimensional heteronuclear NMR techniques[J]. Protein Sci, 1993, 2(4): 543-558. |
| [13] | WÜTHRICH K. NMR of proteins and nucleic acids[M]. New York: Wiley-Interscience, 1986. |
| [14] | XU Y, ZHENG Y, FAN J S, et al. A new strategy for structure determination of large proteins in solution without deuteration[J]. Nat Methods, 2006, 3(11): 931-937. DOI: 10.1038/nmeth938. |
| [15] | NARAYANAN R L, DURR U H, BIBOW S, et al. Automatic assignment of the intrinsically disordered protein Tau with 441-residues[J]. J Am Chem Soc, 2010, 132(34): 11906-11907. DOI: 10.1021/ja105657f. |
 2017, Vol. 34
2017, Vol. 34 
