文章信息
- 王华谱, 朱勤俊, 刘买利, 杨运煌, 岳霞丽
- WANG Hua-pu, ZHU Qin-jun, LIU Mai-li, YANG Yun-huang, YUE Xia-li
- 蛋白质INSM1中的锌指结构域ZF (4-5) 的表达、纯化与表征
- Expression, Purification and Characterization of the Zinc-Finger (4-5) Domain in Human Protein INSM1
- 波谱学杂志, 2017, 34(1): 1-7
- Chinese Journal of Magnetic Resonance, 2017, 34(1): 1-7
- http://dx.doi.org/10.11938/cjmr20170101
-
文章历史
收稿日期: 2016-09-26
收修改稿日期: 2017-01-04


DOI:10.11938/cjmr20170101
2. 波谱与原子分子物理国家重点实验室, 武汉磁共振中心(中国科学院 武汉物理与数学研究所), 湖北 武汉 430071
2. State Key Laboratory of Magnetic Resonance and Atomic and Molecular Physics, National Center for Magnetic Resonance in Wuhan (Wuhan Institute of Physics and Mathematics, Chinese Academy of Sciences), Wuhan 430071, China
人体内胰岛素瘤相关蛋白1(INSM1)是一类含有C2H2-型的锌指(zinc finger)结构域,能够识别特定碱基序列的转录调节因子[1].锌指结构域蛋白简称锌指蛋白,由氨基酸残基与Zn2+结合形成具有指状空间构型的结构.锌指蛋白广泛存在于真核生物体中,比如人类基因组中有将近1%的基因编码锌指蛋白.锌指蛋白在细胞分化[2]、神经发育[3]和胚胎发育[4]等生命过程中有非常重要的作用,并与黑色素瘤[5]、神经细胞瘤[6]和糖尿病肾病[7]等疾病相关.INSM1最初从人的胰岛素瘤中分离鉴定,后来发现在神经内分泌系统[8]、胰腺[9]和相关肿瘤[10]中都有表达,对神经内分泌系统和胰岛的正常发育起着重要的调控作用.人体内INSM1含510个氨基酸,分子量为52.9 k.INSM1的C-端包含有5个串联的C2H2-型的锌指蛋白结构域(zinc finger 1-5,图 1).应用PSI-BLAST进行序列比对分析(如图 1所示),发现人体内INSM1的锌指结构域与黑猩猩(Pan troglodytes)的同源蛋白的氨基酸序列相似度高达99.4%,与小鼠(Mus musculus)、非洲爪蟾(Xenopus laevis)和斑马鱼(Danio rerio)的同源性分别为90.6%、55.6%和55.6%,表明INSM1的锌指结构域在物种进化过程中具有高度保守性.
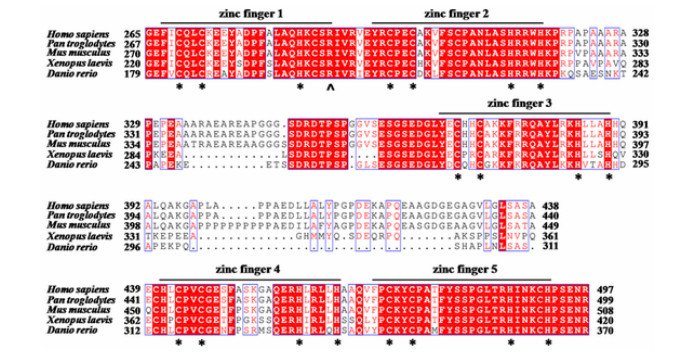
|
| 图 1 PSI-BLAST对来自人(265-497)、黑猩猩、小鼠、非洲爪蟾和斑马鱼的INSM1同源蛋白质的锌指结构域的序列比对.5个锌指区域用横线标记,高度保守的氨基酸用红底白字标记,极性相似的氨基酸蓝框红字表示.可与Zn2+结合的C2H2用*符号标出(除了第一个锌指单元中的末位His被Arg替代,用^符号标出). Fig. 1 Amino acid sequence alignment of zinc finger motifs (265-497) in the human INSM1 with Pan troglodytes, Mus musculus, Xenopus laevis and Danio rerio using PSI-BLAST. Five zinc fingers were designated by lines, highly conserved amino acids marked by white characters in red boxes, the similar amino acids by red characters in blue boxes. All Zn2+-chelating C2H2 in each zinc finger are marked by * (except for the last His in the first zinc finger, which is otherwise replaced by Arg as labeled with ^). |
目前,研究表明含有5个完整锌指结构域ZF (1-5) 的INSM1能结合序列特异性的双链DNA(TG/TC/TC/TT/AGGGGG/TCG/A)[11].然而,各个锌指结构域的结构以及识别特异性DNA的分子机制仍不清楚.为深入理解INSM1中的锌指结构域与序列特异性DNA的相互作用,我们尝试构建了包含不同锌指区间的质粒,比如ZF (1-3)、ZF (2-3) 和ZF (4-5) 等,来制备相应的锌指结构域样品.在成功克隆包含ZF (4-5) 的质粒pET-32m-INSM1(424-497) 的基础上,本文报道了对该蛋白的诱导表达和纯化,以及Zn2+对蛋白质结构域稳定性的影响.15N标记的ZF (4-5) 的核磁共振(NMR)谱图表明该结构域具有折叠的三维空间结构.本工作为后续的ZF (4-5) 结构和功能研究奠定了良好的前期基础.
1 实验部分 1.1 ZF (4-5) 的克隆、表达与纯化采用聚合酶链反应(PCR)方法从HepG2肝癌细胞cDNA扩增出INSM1(424-497) 基因,上游引物为5'-GGATCCGGCGACGGCGAGGGGG-3',下游引物为5'-GGATCCTATCTGTTTTCGGATGG GTGGCAC-3'.扩增条件为98 ℃变性10 s,60 ℃退火15 s,68℃延伸30 s,共30个循环后结束反应.将N端含有His-tag(MHHHHHHSSGLVPRGS)的pET-32m (+) 载体和扩增产物通过琼脂糖凝胶电泳,胶回收后用BamH I酶切.回收酶切产物,用T4连接酶连接后转化至大肠杆菌DH5a,挑取单克隆菌落,筛选阳性克隆,提取重组质粒,进行序列测定.
转化测序正确的重组质粒pET-32m-INSM1(424-497) 到大肠杆菌BL21(DE3) 感受态细胞,挑取单菌落接种到5 mL含氨苄青霉素的LB液体培养基中,在37 ℃、220 r/min的摇床中培养8 h,离心重悬后加入到100 mL M9液体培养基中,在37 ℃、220 r/min的摇床中过夜培养.将100 mL菌液全部转入到1 L的M9培养基中继续在37 ℃、220 r/min的摇床中培养至光密度值(OD)为0.6,向菌液中加入最终浓度为0.1 mmol/L的异丙基-β-D-硫代吡喃半乳糖苷(IPTG)进行诱导,17 ℃诱导20 h收菌.ZF (4-5) 分别在传统的M9培养基和含0.25 mmol/L ZnCl2的M9培养基中进行表达,15N标记的ZF (4-5) 通过加入含0.25 mmol/L ZnCl2和15NH4Cl(购自Cambridge Isotope Laboratories, Inc.)的M9培养基表达.采用缓冲溶液[含200 mmol/L NaCl、20 mmol/L磷酸缓冲盐溶液(PBS)、0.05% β-巯基乙醇,pH 7.0]超声裂解细胞,对表达的蛋白先进行Ni2+柱(GE Healthcare)分离纯化,然后进一步经分子筛层析柱(GE Healthcare)纯化.
1.2 圆二色谱分析室温条件下在Chirascan圆二色谱(CD)仪上进行远紫外圆二色谱(Far-UV CD)实验,氮气吹扫190~260 nm波长范围,石英比色皿光程为2 mm,扫描速度为50 nm/min,扫描3次,累加求平均值.二级结构计算采用软件CDNN Program(version 2.0).
1.3 NMR谱分析 1.3.1 2D 1H-15N HSQC和1H-15N HMQC谱用于NMR实验的缓冲溶液含100 mmol/L NaCl、20 mmol/L 2-(N-吗啉代)乙磺酸(MES)、0.02% NaN3、10 mmol/L二巯基苏糖醇(DTT)、5 mmol/L CaCl2、10% D2O,pH 6.5,15N-ZF (4-5) 的浓度为0.5 mmol/L.2D 1H-15N HSQC直接维(1H)和间接维(15N)谱宽分别为9 000 Hz和18 000 Hz,两维中心分别位于δH 4.70和δN 118.0,采样数据点阵t2×t1=1 024×96,累加次数为4.2D 1H-15N HMQC直接维(1H)和间接维(15N)谱宽分别为9 000 Hz和96 000 Hz,两维中心分别位于δH 4.70和δN 200.0,采样数据点阵t2×t1=1 024×256,累加次数为64.
1.3.2 旋转相关时间(τc)测量采用1D的1H-15N HSQC测定ZF (4-5) 中骨架的15N的纵向弛豫时间(T1)和横向弛豫时间(T2).T1和T2测定实验各选取10个不同的弛豫恢复时间,分别为0.05 s、0.10 s、0.20 s、0.30 s、0.40 s、0.60 s、0.80 s、1.00 s、1.50 s和2.00 s(T1);0.01 s、0.03 s、0.05 s、0.07 s、0.09 s、0.11 s、0.13 s、0.17 s、0.21 s和0.25 s(T2).旋转相关时间(τc)计算公式如下[12]:

|
(1) |
(1) 式中,T1和T2为ZF (4-5) 骨架15N的纵向弛豫时间和横向弛豫时间,νN为NMR实验时15N的进动频率(Hz).
上述所有NMR实验在Bruker Avance Ⅲ 600 MHz NMR谱仪上测定,实验温度为298 K.NMR实验数据用NMRPipe软件进行变换处理[13],采用Sparky软件进行分析[14].
2 结果与讨论 2.1 ZF (4-5) 的诱导表达与纯化图 2为蛋白质ZF (4-5) 在大肠杆菌(E.Coli.)中诱导表达和纯化的十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(SDS-PAGE)胶图.在M9培养基中经过IPTG诱导,仅能观察到非常微弱的蛋白质条带(-ZnCl2),表明ZF (4-5) 基本不表达.当在M9培养基中加入0.25 mmol/L的ZnCl2后,ZF (4-5) 诱导表达明显增强(+ZnCl2).诱导表达的目标蛋白为可溶性蛋白,经过Ni2+柱和分子筛层析柱的先后纯化,蛋白产率可达20.0 mg/L.研究结果表明,外源性的Zn2+对ZF (4-5) 的诱导表达是不可缺少的.
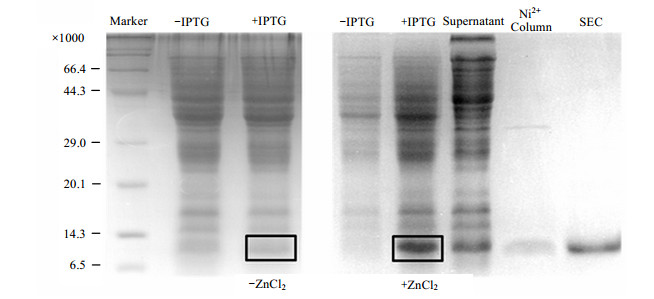
|
| 图 2 ZF (4-5) 在大肠杆菌(E. Coli.)中的诱导、表达和纯化的SDS-PAGE胶图.(左)标准M9培养基,(右)含有0.25 mmol/L ZnCl2的M9培养基 Fig. 2 SDS-PAGE of ZF (4-5) after the IPTG induction, expression and purification. (Left) standard M9 medium, (Right) M9 medium with the addition of 0.25 mmol/L ZnCl2 |
图 3(a)为15N标记的ZF (4-5) 的2D 1H-15N HSQC谱,ZF (4-5) 浓度为0.5 mmol/L,信号累加次数为4.从图中可以观测到:(1)1H-15N交叉峰具有很高的信噪比;(2)骨架1H-15N交叉峰的数目大致与该蛋白的氨基酸数目对应;(3)交叉峰两维的展宽分别为δH6.50~10.00和δN104.0~128.0;(4)各个交叉峰的强度相对均一.实验结果表明ZF (4-5) 具有相对单一的构象和已折叠的二级和三级结构,可应用液体NMR方法解析其三维空间结构.

|
| 图 3 Zn2+对ZF (4-5) 结构稳定性的影响.(a) 0.5 mmol/L ZF (4-5) 的2D 1H-15N HSQC谱;(b) 加入10.0 mmol/L乙二胺四乙酸(EDTA)后的ZF (4-5) 的2D 1H-15N HSQC谱;(c) 在0.003 mmol/L的ZF (4-5) 中加入0.030 mmol/L EDTA前(实线)、后(虚线)的远紫外圆二色谱图 Fig. 3 Structure stability of Zn2+-chelating ZF (4-5). (a) 2D 1H-15N HSQC of 0.5 mmol/L ZF (4-5) in the absence of EDTA; (b) 2D 1H-15N HSQC of 0.5 mmol/L ZF (4-5) in the presence of 10.0 mmol/L EDTA; (c) Far-UV CD spectra of 0.003 mmol/L ZF (4-5) in the absence (solid line) and presence (dash line) of 0.030 mmol/L EDTA |
氨基酸序列相似性分析表明ZF (4-5) 包含两个C2H2-型锌指结构域,电感耦合等离子体质谱(ICP-MS)实验(赛默飞世尔科技上海金桥实验室)进一步证实了实验表达的ZF (4-5) 中存在结合态的Zn2+(未显示的实验数据).然而,Zn2+对ZF (4-5) 结构稳定性的影响尚需验证.首先,在15N-ZF (4-5) 中加入浓度高达10.0 mmol/L乙二胺四乙酸(EDTA),加入过程中样品保持澄清透明,无沉淀出现.2D 1H-15N HSQC谱如图 3(b)所示,发现1H-15N交叉峰的信噪比依然很高,然而在1H和15N两维展宽程度显著降低,呈现出无规则卷曲或仅含有少量二级结构的蛋白质谱峰特征.实验结果表明过量的EDTA虽然从ZF (4-5) 中置换出Zn2+,没有直接导致蛋白的聚集沉淀,但是明显改变了蛋白质的折叠.其次,对加入过量EDTA的ZF (4-5) 样品进一步进行了CD谱分析.实验结果如图 3(c)所示,未加入EDTA的蛋白样品的Far-UV CD曲线(实线),在208 nm和222 nm附近有两个负吸收峰,该信号为α-螺旋的特征吸收峰.过量EDTA加入的样品的Far-UV CD曲线(虚线)发现前述α-螺旋的两个特征吸收峰消失,在203 nm附近出现一个更强的负吸收峰,该信号为蛋白质呈现无规则卷曲时的特征吸收峰.因此,Far-UV CD实验结果表明,当ZF (4-5) 中的Zn2+被置换后,蛋白质的二级结构发生明显改变.因此Zn2+对维持该蛋白的稳定性有着重要的作用.
2.3 ZF4-5在溶液中聚集态的表征蛋白质旋转相关时间(τc)可以通过其骨架15N的横向和纵向弛豫时间估计[12].研究表明,当蛋白质分子量在20 k以下时,分子量与τc呈线性相关[15].图 4(a)和4(b)分别为ZF (4-5) 的骨架15N的横向和纵向弛豫时间测量,最后计算的τc为7.3 ns.图 4(c)的相关性分析表明,ZF (4-5) 的τc所对应的分子量为9.6 k,与ZF (4-5) 单体分子量相吻合,表明该蛋白质样品在研究状态下以单体为主要存在形式.
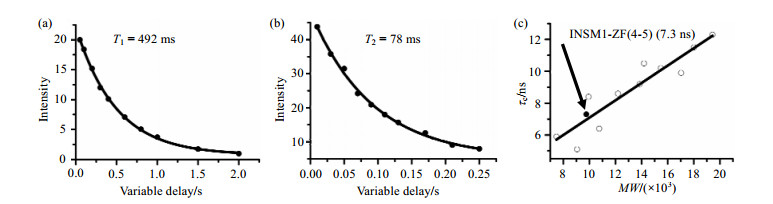
|
| 图 4 ZF (4-5) 的τc测量.骨架15N的T1 (a) 和T2 (b) 测量,τc与蛋白质分子量大小(MW)的相关性分析(c) Fig. 4 T1(a) and T2(b) relaxation measurement for backbone 15N atoms in ZF (4-5), and the correlation between τc and MW (c) |
图 1中氨基酸序列相似性分析表明,ZF (4-5) 含有两个可与Zn2+结合的C2H2-型结构域.已有研究表明,在His咪唑环中的2D 1H-15N HMQC谱中,可观测1H与15N(2JHN或3JHN)的化学位移值的大小与谱峰强度,能够区分组氨酸的两个互变异构体,即δ-/ε-的异构体[16].图 5(a)为15N标记的ZF (4-5) 的2D1H-15N HMQC谱,与Zn2+结合的4个His(H459、H464、H487和H492)咪唑环上通过2JHN或3JHN的1H-15N的交叉峰均已归属.这些交叉峰的信号模式表明这4个His都呈现为δ-异构体,即氢原子结合在δN上[图 5(b),δ1].因此,ZF (4-5) 中的Zn2+是与εN螯合的,图 5(b)展示了C2H2-Zn2+的结合模式.
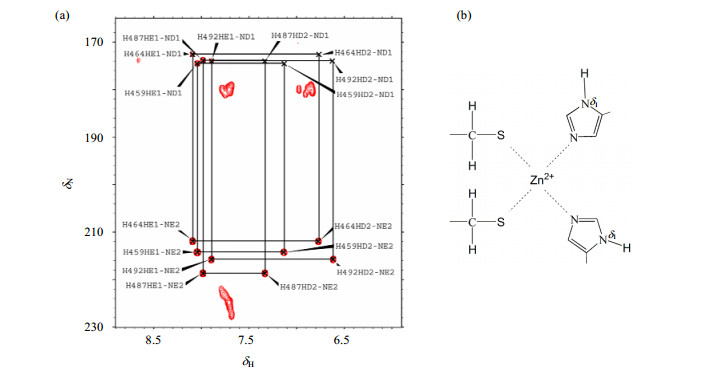
|
| 图 5 (a) 蛋白质ZF (4-5) 的2D1H-15N HMQC谱.4个与Zn2+结合的His(H459、H464、H487和H492)咪唑环上1H-15N的交叉峰均已归属.(b) ZF (4-5) 中的C2H2-Zn2+的结合模式 Fig. 5 (a) 2D 1H-15N HMQC of 0.5 mmol/L 15N-uniformly labeled ZF (4-5). All 1H-15N cross peaks from imidazole rings in H459, H464, H487 and H492 are assigned. (b) The binding scheme of C2H2-Zn2+in ZF (4-5) |
本文通过对锌指蛋白INSM1的ZF (4-5) 的诱导表达和纯化研究,得到了与Zn2+螯合的具有稳定三级结构的蛋白质样品,Zn2+对维持该蛋白结构的稳定性是必不可少的.对蛋白质样品进一步表征分析,该蛋白质在研究条件下主要呈现为单体,适合于NMR结构解析.该锌指结构域的三维空间结构及其与序列特异性的DNA结合的研究正在进行之中.
| [1] | BEERLI R R, BARBAS C F. Engineering polydactyl zinc-finger transcription factors[J]. Nat Biotechnol, 2002, 20(2): 135-141 DOI:10.1038/nbt0202-135 |
| [2] | CHOMETTE D, FRAIN M, CEREGHINI S, et al. Krox20 hindbrain cis-regulatory landscape:interplay between multiple long-range initiation and autoregulatory elements[J]. Development, 2006, 133(7): 1253-1262 DOI:10.1242/dev.02289 |
| [3] | LEE S K, JURATA L W, FUNAHASHI J, et al. Analysis of embryonic motoneuron gene regulation:derepression of general activators function in concert with enhancer factors[J]. Development, 2004, 131(14): 3295-3306 DOI:10.1242/dev.01179 |
| [4] | XUE L, CHEN X, CHANG Y, et al. Regulatory elements of the EKLF gene that direct erythroid cell-specific expression during mammalian development[J]. Blood, 2004, 103(11): 4078-4083 DOI:10.1182/blood-2003-09-3231 |
| [5] | SLUTSKY S G, KAMARAJU A K, LEVY A M, et al. Activation of myelin genes during transdifferentiation from melanoma to glial cell phenotype[J]. J Biol Chem, 2003, 278(11): 8960-8968 DOI:10.1074/jbc.M210569200 |
| [6] | TOMMERUP N, VISSING H. Isolation and fine mapping of 16 novel human zinc finger-encoding cdnas identify putative candidate genes for developmental and malignant disorders[J]. Genomics, 1995, 27(2): 259-264 DOI:10.1006/geno.1995.1040 |
| [7] | HALAMA N, YARD-BREEDIJK A, VARDARLI I, et al. The Kruppel-like zinc-finger gene ZNF236 is alternatively spliced and excluded as susceptibility gene for diabetic nephropathy[J]. Genomics, 2003, 82(3): 406-411 DOI:10.1016/S0888-7543(03)00120-4 |
| [8] | PARLIER D, ARIZA A, CHRISTULIA F, et al. Xenopus zinc finger transcription factor IA1(Insm1) expression marks anteroventral noradrenergic neuron progenitors in Xenopus embryos[J]. Dev Dynam, 2008, 237(8): 2147-2157 DOI:10.1002/dvdy.v237:8 |
| [9] | GIERL M S, KAROULIAS N, WENDE H, et al. The zinc-finger factor Insm1(IA-1) is essential for the development of pancreatic beta cells and intestinal endocrine cells[J]. Gene Dev, 2006, 20(17): 2465-2478 DOI:10.1101/gad.381806 |
| [10] | BRESLIN M B, ZHU M, LAN M S. NeuroD1/E47 regulates the e-box element of a novel zinc finger transcription factor, IA-1, in developing nervous system[J]. J Biol Chem, 2003, 278(40): 38991-38997 DOI:10.1074/jbc.M306795200 |
| [11] | BRESLIN M B, ZHU M, NOTKINS A L, et al. Neuroendocrine differentiation factor, IA-1, is a transcriptional repressor and contains a specific DNA-binding domain:identification of consensus IA-1 binding sequence[J]. Nucleic Acids Res, 2002, 30(4): 1038-1045 DOI:10.1093/nar/30.4.1038 |
| [12] | KAY L E, TORCHIA D A, BAX A. Backbone dynamics of proteins as studied by 15N inverse detected heteronuclear NMR spectroscopy:application to staphylococcal nuclease[J]. Biochemistry, 1989, 28(23): 8972-8979 DOI:10.1021/bi00449a003 |
| [13] | DELAGLIO F, GRZESIEK S, VUISTER G W, et al. NMRPipe:a multidimensional spectral processing system based on UNIX pipes[J]. J Biomol NMR, 1995, 6(3): 277-293 |
| [14] | GODDARD T D, KNELLER D G. SPARKY 3. University of California, San Fransisco.[J]. 2008. |
| [15] | ROSSI P, SWAPNA G V, HUANG Y J, et al. A microscale protein NMR sample screening pipeline[J]. J Biomol NMR, 2010, 46(1): 11-22 DOI:10.1007/s10858-009-9386-z |
| [16] | PELTON J G, TORCHIA D A, MEADOW N D, et al. Tautomeric states of the active-site histidines of phosphorylated and unphosphorylated ⅢGlc, a signal-transducing protein from Escherichia coli, using two-dimensional heteronuclear NMR techniques[J]. Protein Sci, 1993, 2(4): 543-558 |

本作品采用知识共享署名 4.0 国际许可协议进行许可。
 2017, Vol. 34
2017, Vol. 34



