文章信息
- 张雪莹, 王成龙, 谢海滨, 张成秀, 马超, 陆建平, 杨光
- ZHANG Xue-ying, WANG Cheng-long, XIE Hai-bin, ZHANG Cheng-xiu, MA Chao, LU Jian-ping, YANG Guang
- 基于GPU加速的磁共振血管造影图像的并行分割与追踪算法
- Parallel Segmentation and Tracking Algorithm for Magnetic Resonance Angiography Images Based on GPU
- 波谱学杂志, 2016, 33(4): 570-580
- Chinese Journal of Magnetic Resonance, 2016, 33(4): 570-580
- http://dx.doi.org/10.11938/cjmr20160406
-
文章历史
收稿日期: 2016-04-01
收修改稿日期: 2016-11-01


DOI:10.11938/cjmr20160406
2. 上海卡勒幅磁共振技术有限公司, 上海 201614;
3. 上海市第二军医大学附属长海医院 放射科, 上海 200433

 , ZHANG Cheng-xiu2, MA Chao3, LU Jian-ping3, YANG Guang1,2
, ZHANG Cheng-xiu2, MA Chao3, LU Jian-ping3, YANG Guang1,2

2. Shanghai Colorful Magnetic Resonance Technology Corporation Limited, Shanghai 201614, China;
3. Department of Radiology, Changhai Hospital, The Second Military Medical University, Shanghai 200433, China
三维增强磁共振血管造影术(Three Dimensional Contrast Enhanced Magnetic Resonance Angiography,3D CE-MRA)通过在血管内注射造影剂,增加血管与周围组织的对比度,获得高信噪比图像[1, 2].在临床应用3D CE-MRA的过程中,为了避免周围的血管遮挡病灶、更好地观察血管病变的细节,医生常常需要分离出感兴趣区域(Region Of Interest,ROI)的血管.这一过程传统上需要医生手工勾勒出ROI的形状,由于血管造影图像为三维数据,手工勾勒ROI的过程费时费力.
目前,很多常用的分割算法都可以用于自动血管分割[3].移动立方体法[4](Marching Cube,MC)基于三维数据的最小单元体素,建立256种构型,通过查表法构造等值面进行表面重建.Nasrin和Jabbar[5]将MC与轮廓重构方法相结合,减少重构时间、提升视觉效果.区域生长(Region Growing,RG)算法[6, 7]基于迭代的思想实现组织分割,在每一次迭代过程中,判断并聚集与种子点性质相似的邻域内体素,选取新的种子点后进行下一次迭代.由于基于固定阈值以寻找相似体素的RG算法无法满足灰度复杂的图像的要求,因此Rouhi和Jafari等人[8]基于训练的人工神经网络和灰度特征,自适应获得RG算法中判断种子点相似性的局部阈值,能准确地提取乳腺肿瘤,但是该技术应用于不同组织的分割结果存在一定的不确定性.Wang等人[9]提出自适应阈值的区域生长算法,能自动地将血管从周边组织中分离.首先,采用各向异性扩展滤波器进行图像预处理,在抑制噪声的同时保留图像边缘细节信 息.其次,基于移动的、大小变化的局部区域,将计算得出的局部阈值作为种子点相似性的判据,聚集相邻的相似体素,并进行迭代过程.同时,采用形态学方法优化对细小血管分支的检测.该算法可以自动地分割血管并显示ROI,具有较高的准确性和鲁棒性,但缺陷是运算速度不够快,平均需要3 s的时间.
利用现代图形处理器(Graphics Processing Unit,GPU)所具备的大规模并行计算能力可有效的缩短计算时间.中高档GUP可以同时运行1 000~2 000个线程[10],利用这种大规模并行计算能力,图像处理算法常常可以加速2个数量级甚至更多[11],因此目前GPU已被广泛地应用于各种图像处理方法中.然而,该应用虽然能加快部分医学图像分割算法运行时间,但效果仍不理想.局部内区域生长算法对体素的判断是一致的,数据较为并行,然而随着种子点个数的增加,需要重新设置线程数,并再次读取全局内存,影响了并行算法的效率.Harish和Narayanan[12]提出的并行区域生长算法,应用GPU纹理内存、共享内存,加快全局内存的读取速度,能实现1.5 s内分割,但是采用的双缓冲机制无形中增加了显存的使用量.Pan和Gu等人[13]提出通过多种子点生长的方式充分利用GPU资源,实现了2.4 s内6个种子点共同生长的区域生长算法,但运算速度也不高.综上所述,类似区域生长算法这样的血管分割与追踪算法的主要缺点是计算量大、运行时间长.
因此,本文提出了一种新的血管分割与追踪的算法框架,通过采用高度并行化的预处理步骤,使得血管分割与追踪算法的主体可以在GPU上并行实现,从而实现了快速分割血管的目标.基于真实数据的实验结果表明,与基于经典区域生长算法的血管分割算法相比,本文算法的速度更快、分割的结果也更为准确.
1 基本理论 1.1 预处理 1.1.1 模型设计GPU并行计算的特点是每个运算单元之间没有任何的依赖性,具有较高的独立性.为了充分利用GPU的并行计算能力,要求将每个线程处理的数据局部化.为此,我们将原始图像网格化为共面的、紧密排列的立方体,每个立方体中都包含一定数量的体素.每个线程可以对一个立方体进行处理,例如判断立方体内是否包含血管,获得属于血管的体素、血管通过的表面等信息.后续的全局分割以及血管追踪算法,可以利用这一预处理信息快速地进行血管追踪,也可以利用图像全局特性对预处理的结果进行修正.
采用不同大小的立方体对图像进行网格化,对算法效果与执行效率有不同的影响.一方面,若立方体太小,基于单个立方体内体素强度统计的方法会失效;同时,因为单个立方体中的所有体素都在血管内部,可能也无法利用边缘检测算法判断是否存在血管.另一方面,若立方体过大,整体的立方体个数太少,不利于充分利用GPU的大规模并行计算能力;同时,立方体中的血管结构过于复杂,也会给算法预处理步骤带来困难.经实验发现,在目前典型的MRA图像的分辨率下,选择5×5×5个体素大小的立方体,能够取得较好的分割效果,也不会影响算法的并行加速,如图 1所示.
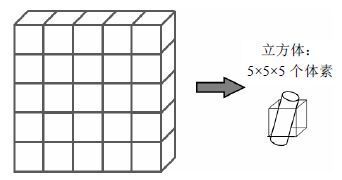
|
| 图 1 模型设计方案 Fig. 1 Model design |
对于每个立方体,算法预处理过程(流程图如图 2所示)利用立方体中的局部数据进行如下处理:(1)判断立方体的各个表面上是否有血管通过;(2)判断立方体内哪些体素属于血管.
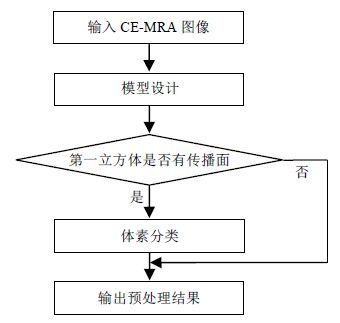
|
| 图 2 算法预处理过程 Fig. 2 Preprocessing step of the algorithm |
有血管通过立方体表面,是立方体包含血管的充要条件.因此,预处理过程首先判断每个立方体的表面是否有血管通过,并将有血管通过的表面称为传播面.很多算法都可以用来判断一个二维平面上是否有血管存在,我们采用的是边缘检测中的SUSAN算子[14-16].SUSAN算子是一种定位精确、抗噪性强、计算速度快的边缘检测算子,它选取圆形掩模,判断某一点周围的灰度值与其自身灰度值的差异:若差异点数足够多,则认为该点位于边界处;反之则认为不是.其算法过程可用以下3个公式表示:
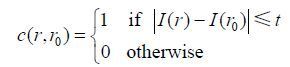
|
(1) |
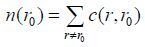
|
(2) |
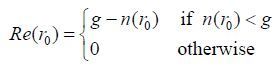
|
(3) |
其中,t为SUSAN算子的阈值,灰度差异大于该阈值,就认为灰度间存在显著差异;为灰度相似点数;为边缘响应函数;
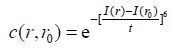
|
(4) |
对于一个特定的表面,如果有体素被SUSAN算子判定位于血管边缘,就可以认为有血管通过该表面,即为传播面.先利用双线性插值将5×5个体素的表面采样为9×9个体素,然后采用SUSAN算子进行边缘检测.SUSAN算子的阈值(t)可以根据每幅MRA图像的全局阈值自适应设定,本文设置为全局阈值的0.3倍.若设置较小的参数t,会有更多立方体的表面被认为位于边缘处,进而提取更多的细节;反之,可以主要观察血管的主干部分.全局阈值可利用大津算法[17]对整个三维图像处理获得.大津算法基于图像归一化后的灰度直方图,将背景与前景的类间方差作为衡量背景与前景差别的标准.当取得拥有最大类间方差的最佳阈值时,能最大程度地体现出图像灰度直方图中左半部分背景与右半部分前景的差异.大津算法也可以利用GPU并行加速实现.
1.1.3 体素分类算法预处理的第二部分工作,则是判断当前立方体中哪些体素属于血管.很多不同的判据可以用于这一步骤:
(1)在CE-MRA图像中,血管为高信号,因此可以利用大津算法[17]进行阈值分割:如果体素的灰度小于大津法确定的局部阈值,则该体素不属于血管;反之则可能属于血管.由于大津算法是基于直方图统计获得的,为了保证算法的鲁棒性,我们在计算局部阈值时,将统计直方图的统计范围扩大到7×7×7个体素的立方体.
(2)属于血管的体素在空间上具有连续性,如果某个体素周围属于血管的体素较多,则该体素属于血管的概率增大.同时属于血管的体素,最终要和传播面上、被判断为属于血管的体素相连.空间上孤立的部分高信号体素,可能是由噪声或伪迹造成的,不应被判断为血管.
要指出的是,基于局部阈值的体素分类结果,可以与前一步骤中基于SUSAN算法的结果相结合,以更好地判断传播面的归属.体素分类完成后,利用属于血管的体素的空间连通性,就可以知道有几条不同解剖结构的血管经过该立方体,每组血管中包括哪些体素,特别是穿过哪些传播面.这些信息都被记录下来,用于后续全局分割与血管追踪处理中.
1.2 预处理并行算法的实现预处理并行算法使用C++编程语言实现,GPU加速部分则使用了NVIDIA公司提供的统一计算设备架构(Compute Unified Device Architecture,CUDA)[10]平台.基于立方体的预处理过程可以很容易地使用GPU并行计算.在算法实现过程中主要考虑以下几点:
(1)每个GPU线程处理的数据是一个5×5×5个体素的立方体,在构建线程模型时,可以采用三维的方式,以便将线程映射到对应的立方体.需要指出的是,在我们的算法中,相邻立方体是共面的,或者说面上的体素被相邻的两个立方体所共享.
(2)优化每个线程的运算方式,减少线程分叉程度,以充分利用计算机并行计算资源.串行处理每个立方体时,当且仅当该立方体有传播面包含血管时,才继续进行体素分类.采用GPU并行计算时,则是先判断完所有立方体是否有传播面后,再基于这些有传播面的立方体,重新设置线程数,只分割包含血管的立方体.这节省了线程计算时容易产生等待、分叉的时间,而在实验中我们也发现这一优化过程节约了一半多的时间.
(3)根据性能分析的结果,合并相关的处理函数,以减少访问原始图像这一全局内存的次数,减少时间消耗.
不同算法,鉴于其内部不同的计算方式,并行化程度也略有不同[18],主要可以从以下6个方面评价本文算法的并行化程度:
(1)数据并行程度高.本文算法是基于单独、局部的立方体进行判断,立方体与立方体之间的没有任何耦合.
(2)设置的线程数多.最佳情况是一个体素一个线程,虽然本文算法没有如此设置,但也达到上万的线程数.
(3)线程分叉程度低.本文算法优化了每个立方体的计算方式,极大地减少了算法遇到分叉的情况.
(4)显存使用量多.最佳情况是超过图像总体素数的5倍,而本文算法达到了近3倍的程度.
(5)本文算法无需同步.
(6)加速程度高.经实验统计发现,基于960核4 096 MB全局可用图形内存的NVIDIA GeForce GTX 660显卡,预处理步骤能达到35倍的加速比,加速程度高,有效地实现了算法的并行化.
1.3 全局分割与血管追踪进行分割预处理后,便可全部获悉三维医学图像中哪些体素属于血管,基于此结果可以对图像进行全局分割与血管追踪.
全局分割可以用于突出显示属于血管的体素,而将背景、噪声的体素弱化或者隐藏.从理论上讲,预处理之后就已经获得了所有的立方体中属于血管的体素,这一结果就可以用于血管的突出显示.具体算法实现中,可以利用立方体传播面的连通信息,将所有包含血管的立方体分组连接.例如,如果某个立方体中的一组血管与某传播面相连,则可以根据与传播面血管体素相连通的相邻立方体中一组血管体素,进而寻找下一个传播面.通过这种方式,可以将所有包含血管的立方体进行分组连接.伪迹或噪声可能造成预处理时将个别立方体误判为包含血管,而在全局分割中,由于这些立方体中包含的血管不与其它立方体中的血管相连,或者与之同组的立方体总数很少,因此不难将这些误判的立方体排除.
血管追踪则是同样利用前面预处理所获得的信息,帮助医生快速地提取ROI的血管.例如,通过指定血管上的两点,本文算法可以利用预处理获得的包含血管的立方体之间的连通信息,快速地找出连通这两点的血管(包含这些血管的立方体),并突出显示这些血管.值得指出的是,本文预处理中获得的关于立方体传播面和体素分组的信息,可以在血管追踪的过程中重复使用,这是基于区域生长的血管追踪方法无法做到的.因此本文算法特别适用于交互式的血管追踪(ROI提取)应用.
2 实验条件本文所用的3幅图像,均是在上海市第二军区大学附属长海医院的Siemens 1.5 T超导磁共振成像(Magnetic Resonance Imaging, MRI)系统上采集的人脑血管3D CE-MRA图像,图像的分辨率均为176×256×80,体素间距均为0.89× 0.89×1.10 mm3,灰阶均为1 120.
程序在Windows 8环境运行;CPU型号为i7-4770,主频为3.40 GHz;内存类型为DDR3,容量为16 GB,工作频率为2 133 MHz;显卡为NVIDIA GeForce GTX 660;基于Visual Studio 2012编译器和CUDA 7.0版本开发;软件交互界面基于微软基础类库(Microsoft Foundation Class, MFC)框架.
3 结果与讨论 3.1 预处理时间分析采用同样算法,比较基于中央处理器(Central Processing Unit, CPU)与GPU的串行、并行预处理所需时间.为了统计准确,两种实现方法各自重复计算100次后,统计各自的平均运算时间(结果如表 1所示).结果显示采用GPU并行计算后,平均时间能控制在0.2 s内,能实现快速分割和用户流畅交互.同时相比CPU,基于GPU的处理平均能加速35倍,体现出本文算法具有高度并行的特点.经典分割算法(如区域生长算法)所需时间平均是在3 s左右[9].因此,本文算法分割的速度要明显快于区域生长算法.
| t(CPU)/s | t(GPU)/s | 加速比* | |
| 图像1 | 6.870 | 0.186 | 36.9 |
| 图像2 | 6.302 | 0.182 | 34.5 |
| 图像3 | 5.747 | 0.167 | 34.4 |
| *加速比= t(CPU)/t(GPU) | |||
算法并行计算过程中,时间主要分为运行kernel函数所需时间及数据在主机端和设备端之间传输所需时间.表 2显示,本文算法将绝大多数时间用于kernel函数运行上,即并行计算,极少时间用于数据传输,说明并行计算效率高.此外,分析kernel函数时间分布发现,判断传播面所需时间占运行kernel函数总时间的19%,体素分类占80%,剩余时间为全局阈值的并行计算,说明并行算法将大部分时间用于立方体分割上,算法效率高.
| t(kernel)/s | t(GPU)/s | 并行计算运行时间比例* | |
| 图像1 | 0.172 | 0.186 | 92.5% |
| 图像2 | 0.170 | 0.182 | 93.1% |
| 图像3 | 0.154 | 0.167 | 92.2% |
| *时间比例= t(kernel)/t(GPU) | |||
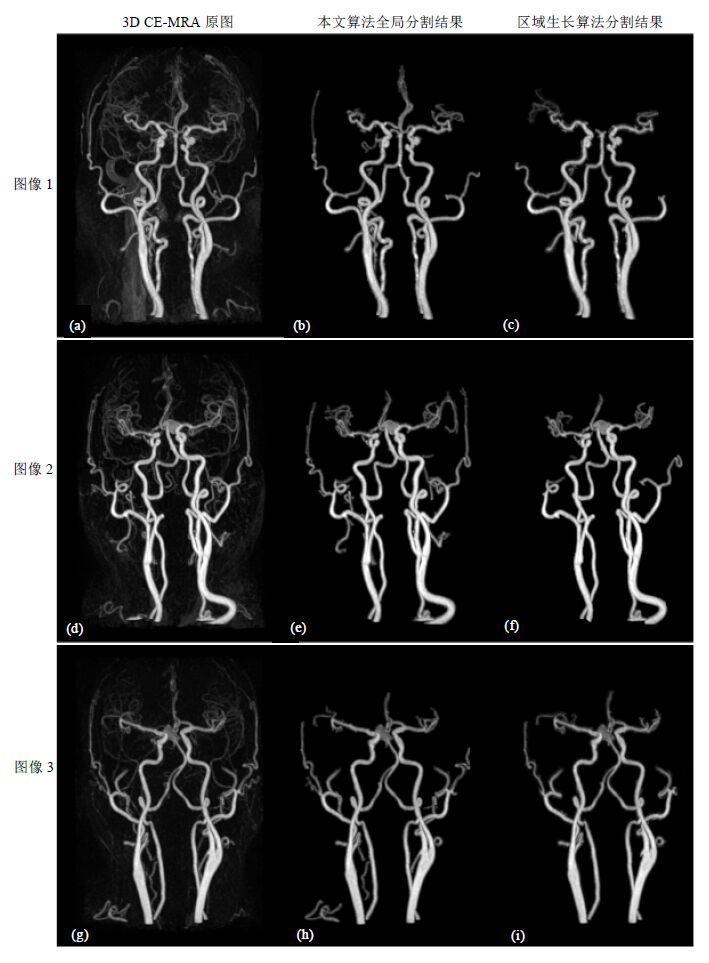
将本文算法全局分割结果与区域生长算法结果进行定性比较,找出两种分割结果的共同点和差异性.结果显示这两种算法都能提取颅内血管动静脉,在提取毛细血管等细节上也各有优劣.然而,总体观察发现本文算法能找出更多的毛细血管,并且观察原图发现,这些也确实是所需细节(如图 3所示).同时,从本文全局分割结果的效果图可发现,全局分割可以滤除图像中的背景和伪迹,突出显示血管.
|
| 图 3 (a), (d), (g)为三幅3D CE-MRA原图;(b), (e), (h)分别是本文算法全局分割结果;(c), (f), (i)为相应区域生长算法分割结果 Fig. 3 (a), (d), (g) Tree 3D CE-MRA original images; (b), (e), (h) The global segmentation results of proposed algorithm; (c), (f), (i) The corresponding results of RG algorithm |
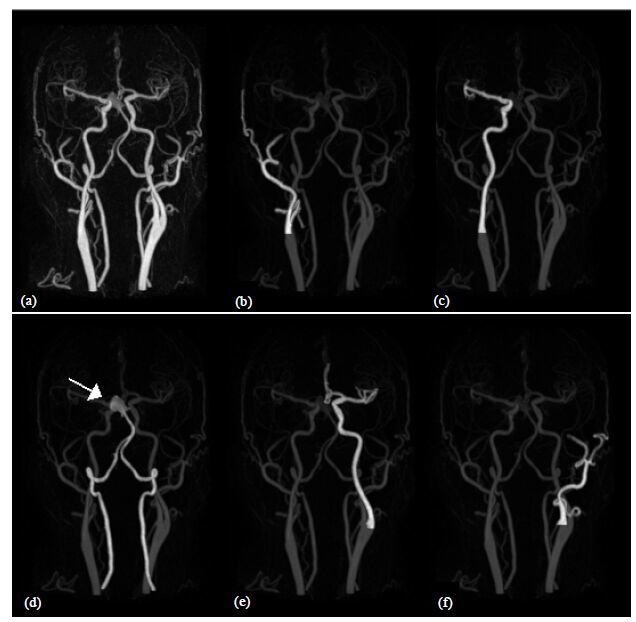
图像3的血管追踪结果如图 4所示,发现本文追踪算法能实现单独提取血管的各个分支,能有针对性地提取用户所需的ROI.为了统计准确,重复同样的算法100次.血管追踪算法的平均运算时间为0.011 s,明显快于区域生长算法所需的3 s.
此外,基于本文算法的全局分割与血管追踪结果都能检测出图像3中的颅内动脉瘤[如图 4(d)中箭头所示],有助于临床医生进行病灶诊断.
|
| 图 4 (a) 图像3的3D CE-MRA原图;(b)~(f) 血管追踪算法运用于ROI提取的结果[图像3中的颅内动脉瘤如(d)图箭头所示] Fig. 4 (a) The third original 3D CE-MRA image; (b)~(f) The results of ROI extraction using vessel tracking algorithm [the intracranial aneurysm location is shown with the white arrow in Fig.(d)] |
本文提出了一种将3D CE-MRA图像网格化为共面立方体后,并行进行分割预处理的算法.基于该预处理的结果,可以方便快速地实现血管全局分割与血管追踪.相比区域生长算法,该算法运行的时间大大缩短,这点在进行交互式血管追踪时尤其有优势,能独立区别各个血管分支;且相比于区域生长算法能提取更多细节.该算法的应用可以为医生利用CE-MRA图像进行临床诊断提供极大的便利.
| [1] | LU Jian-ping(陆建平), LIU Qi(刘崎) . Three dimensional contrast-enhanced magnetic resonance angiography(三维增强磁共振血管成像)[M]. Shanghai(上海): Shanghai Science and Technology Press(上海科学技术出版社), 2005 . |
| [2] | Yang Bao-lian(杨保联) . Future of ultra high field MRI in basic research and clinical applications(超高场磁共振人体成像应用研究和医学前景)[J]. Chinese J Magn Reson(波谱学杂志) , 2015, 32 (4) : 707-714 |
| [3] | Lesage D, Angelini E D, Bloch I et al . A review of 3D vessel lumen segmentation techniques:Models, features and extraction schemes[J]. Med Imag Anal , 2009, 13 (6) : 819-845 DOI:10.1016/j.media.2009.07.011 |
| [4] | Lorensen W E, Cline H E. Marching cubes:A high resolution 3D surface construction algorithm[C]//ACM siggraph computer graphics. ACM, 1987, 21(4):163-169. |
| [5] | Roshnara Nasrin P P, Jabbar S. Efficient 3D visual hull reconstruction based on marching cube algorithm[C]//Innovations in Information, Embedded and Communication Systems (ICⅡECS), 2015 International Conference on IEEE, 2015:1-6. |
| [6] | Adams R, Bischof L . Seeded region growing[J]. IEEE T Pattern Anal , 1994, 16 (6) : 641-647 DOI:10.1109/34.295913 |
| [7] | Hojjatoleslami S A, Kittler J . Region growing:A New Approach[J]. IEEE T Image Process , 1998, 7 (7) : 1079-1084 DOI:10.1109/83.701170 |
| [8] | Rouhi R, Jafari M, Kasaei S et al . Benign and malignant breast tumors classification based on region growing and CNN segmentation[J]. Expert Syst Appl , 2015, 42 (3) : 990-1002 DOI:10.1016/j.eswa.2014.09.020 |
| [9] | Wang Cheng-long(王成龙). A Study on Fast Vessel Segmentation and Tracking Algorithm(快速血管分割追踪算法的研究)[D]. Shanghai(上海): East China Normal University(华东师范大学), 2014. |
| [10] | Farber R . CUDA Application Design and Development[M]. USA: Elsevier, 2011 . |
| [11] | Owens J D, Houston M, Luebke D et al . GPU computing[J]. P IEEE , 2008, 96 (5) : 879-899 DOI:10.1109/JPROC.2008.917757 |
| [12] | Harish P, Narayanan P J . Accelerating Large Graph Algorithms on the GPU Using CUDA[M]. Berlin Heidelberg: Springer, 2007 : 197 -208. |
| [13] | Pan L, Gu L, Xu J. Implementation of medical image segmentation in CUDA[C]//Information Technology and Applications in Biomedicine, 2008. International Conference on IEEE, 2008:82-85. |
| [14] | Smith S M, Brady J M . SUSAN-a new approach to low level image processing[J]. In J Comput Vis , 1997, 23 (1) : 45-78 DOI:10.1023/A:1007963824710 |
| [15] | Qu Z, Wang P, Gao Y et al . Fast SUSAN edge detector by adapting step-size[J]. Optik-International Journal for Light and Electron Optics , 2013, 124 (8) : 747-750 DOI:10.1016/j.ijleo.2012.01.026 |
| [16] | Rezai-Rad G, Aghababaie M. Comparison of SUSAN and sobel edge detection in MRI images for feature extraction[C]//Information and Communication Technologies, 2006. ICTTA'06. 2nd. IEEE, 2006, 1:1103-1107. |
| [17] | Otsu N . A threshold selection method from gray-level histograms[J]. Automatica , 1975, 11 (285-296) : 23-27 |
| [18] | Smistad E, Falch T L, Bozorgi M et al . Medical image segmentation on GPUs-A comprehensive review[J]. Med Imag Anal , 2015, 20 (1) : 1-18 DOI:10.1016/j.media.2014.10.012 |

本作品采用知识共享署名 4.0 国际许可协议进行许可。
 2016, Vol. 33
2016, Vol. 33

