文章信息
- 陈朴, 于燕波, 黄贱英, 李红毅, 董海胜, 陈斌
- CHEN Pu, YU Yan-bo, HUANG Jian-ying, LI Hong-yi, DONG Hai-sheng, CHEN Bin
- 运动员与体力劳动者代谢组学判别模型的建立
- Urinary Metabonome Differentiates Athletes and Labor Workers
- 波谱学杂志, 2016, 33(3): 395-405
- Chinese Journal of Magnetic Resonance, 2016, 33(3): 395-405
- http://dx.doi.org/10.11938/cjmr20160304
-
文章历史
收稿日期: 2015-09-09
收修改稿日期: 2016-07-18


DOI:10.11938/cjmr20160304
21世纪,健康成为全球共同关注的焦点.个体的健康水平受饮食、生活习惯、环境条件和职业等各方面因素的影响.其中职业是每个人必须面临的话题,来自不同职业的压力会对一个人的心理、性格和身体造成不同的影响.比如长期工作抑郁的人患多重人格及精神分裂的可能性更高[1];长期静坐的职业人群患心血管疾病的风险高[2];以快餐为主食并且缺乏运动的职业人群容易发生肥胖和高血脂[3].由职业导致的慢性病具有隐蔽性,常被人们忽视,主要在老年期爆发.慢性病引起的死亡占全世界人类死亡率的2/3,但是经典的传统医学手段无法分析.目前对其研究大部分源自于问卷调查,其结果影响因素较多,而且无法解释其生理学机理并据此制定有效的应对措施[4].
近年来,基因组学及蛋白组学在人体疾病风险因子分析中取得了创新性的进展[5],如单核苷酸多态性研究(SNPs)可找到疾病的靶向基因[6].但由于基因下游仍存在着许多调控及代谢网络,基因及蛋白组学只能预测可能发生了什么[7, 8].相比之下,代谢组学可以从整体上评价生理上已经发生的变化,是基因表达和蛋白调控的最终结果[9],具有综合和非靶向的优点.常用于代谢组学研究的分析技术有核磁共振(NMR)、质谱(MS)等.相比MS,NMR技术具有许多显著的优点,例如:无需样品预处理和预筛选,可以避免由于分离所造成微小成分的丢失;无损伤性,不会破坏样品的结构和性质;可以进行实时和动态检测;没有偏向性,对所有化合物的检测灵敏度一致.而MS则有离子化程度不一致和基质干扰等问题[10].代谢组学模型应用于人群研究主要有两个方面:一是对不同个体进行横向比较,找到各类群体的代谢表型,据此开展个性化的健康管理;二是依据群体在不同生命历程时的代谢特征建立代谢组学模型,进行群体患病风险的预测[11].本文拟从代谢组学的角度出发,对运动员和体力劳动者两类人群进行横向比较,建立二者的预测模型.并对模型进行有效的评价.这为未来个体健康状态评估模型的建立提供了新的思路.
1 实验部分 1.1 仪器及试剂试剂:脲酶(Sigma type Ⅲ,Sigma-Aldrich; cat. No. U1500-100KU);2, 2, 3, 3-氘代三甲基硅烷丙酸钠[3-(trimethylsilyl)propionate-2, 2, 3, 3-d4,BP,98%氘代,CIL];磷酸盐缓冲液(含20% D2O,pH 7.4,Sigma-Aldrich);重水(D2O,99.9%氘代,Waters).
仪器:核磁共振波谱仪(Bruker Avance Ⅲ 600);Peek管(Sigma-Aldrich);低温离心机(德国Eppendorf股份有限公司);-80℃超低温冰箱(中国海尔公司);均质机(Waters).
1.2 实验方法 1.2.1 实验设计随机招募运动员和体力劳动志愿者共73名,并按其从事职业分为两组.其中A组为31名男性运动员(Athlete:A1~A31),年龄在20~40岁之间,工作生活有规律,长期统一饮食;L组为42名男性体力劳动者(Labour:L1~L42),年龄在22~40岁之间,统一饮食2周.分别采取两组志愿者的晨尿,向每份尿液中加入脲酶降解尿液中的尿素(0.4~1.0 U/200 mL),室温下降解30 min后等份分装到灭过菌的离心管中于-80 ℃冻存[12].
1.2.2 尿液1H NMR检测所有尿样室温下解冻后均质1 min,取400 μL尿样至2 mL微量离心管中,于4 ℃、10 000 g下离心10 min;取上清,加入磷酸缓冲液(20% D2O,pH 7.4±0.5)200 μL,平衡10 min后待测. 1H NMR实验都在配有超低温探头的Bruker Avance Ⅲ 600型NMR谱仪上完成,质子共振频率为600.13 MHz,实验温度为298 K.使用Noesygppr1d脉冲序列采集1D 1H NMR谱.实验参数如下:谱宽为12 000 Hz,等待时间(RD)为2 s,混合时间(tm)为100 ms,90°脉宽约为10 μs,采样点数为32 k,累加次数为32.
1.2.3 样本数据预处理将采集到的1H NMR谱通过专业的NMR图谱处理软件Topspin V2.0进行基线(多项式平滑)和相位(解卷积)校正[13].使用TSP对NMR谱的化学位移进行定标(δ0.00).去除NMR谱中的水峰(δ 4.62~5.17)、尿素峰(δ 5.57~6.15),分段积分(积分间隔为2.4 Hz,积分范围为δ0.50~9.50)后得到数据矩阵.尿液NMR图谱化合物的归属通过人类代谢组学数据库(HMBD)及参照文献[12, 14]确定(结果见补充材料图S1).
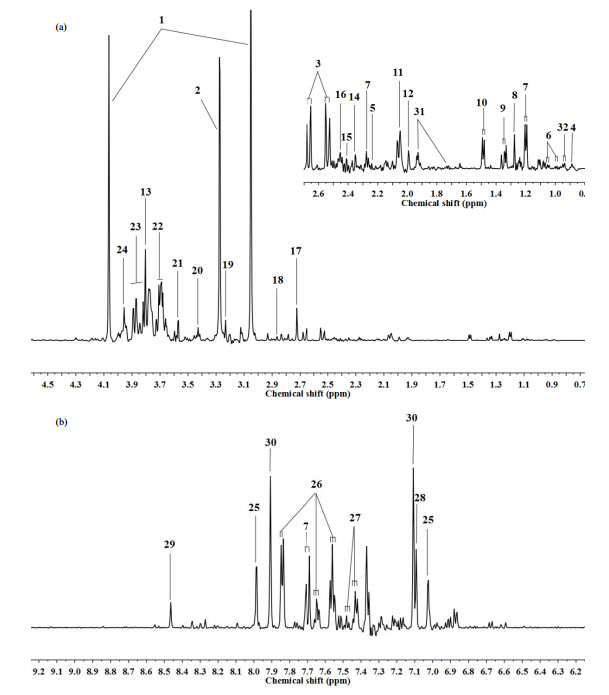
|
| 图 S1 两种体质人群尿样600 Hz一维核磁图谱 注:标号数字标号所对应的化合物如下:1:肌苷酸;2: N-氧基三甲胺;3:柠檬酸;4:丁酸盐;5:丙酮;6:缬氨酸;7: 3-氨基异丁酸;8: 3-羟基异戊酸;9:乳酸盐;10:丙氨酸;11: N-乙酰谷氨酸;12: N-乙酰天冬酰胺;13:胍乙酸;14:丙酮酸;15:琥珀酸盐;16: α-酮戊二酸;17:二甲胺;18:三甲胺;19:甘油磷脂酰胆碱;20:牛磺酸;21:甘氨酸;22:苏氨酸;23:甘露醇;24:肌酐;25: 3-甲基组氨酸;26:马尿酸盐;27:苯乙酰甘氨酸;28:肌肽;29:甲酸盐;30: L-组氨酸;31:赖氨酸;32: 2-羟基异戊酸. Fig. S1 600 Hz 1H NMR spectrum of urine derived from two constitutions Numbers indicates the following metabolites: 1: Creatinine; 2: Trimethylamine N-oxide; 3: Citrate; 4: Butyrate; 5: Acetone; 6: Valine; 7: 3-Aminoisobutyrate; 8: 3-Hydroxyisovalerate; 9: lactate; 10: Alanine; 11: N-Acetylglutamate; 12: N-Acetylaspartate; 13: Guanidoacetate; 14: Pyruvate; 15:Succinate; 16: α-Ketoglutarate; 17: Dimethylamine; 18: Trimethylamine; 19: Glycerophosphorylcholine; 20: Taurine; 21: Glycine; 22: Threonate; 23: Mannitol; 24: Creatine; 25: 3-Methylhistidine; 26: Hippurate; 27: Phenylacetyglycine; 28: Carnosine; 29: Formate; 30: L-Histidine; 31: Lysine; 32: 2-Hydroxyisovalerate. |
将得到的样本数据矩阵导入到SIMCA P+ V14软件中进行多元统计分析.首先对指标(化学位移,1 968个)进行预处理,排除相对标准差(RSD)小于30%的指标(这部分指标在研究过程中未发生明显改变,引入会使后续建模过度拟合);然后,用NMR谱的总面积分别对单个指标进行标准化,降低信号强度差异较大指标间的数量级差;然后对数据进行对数转换(lgχ);最后,对数据进行标准化处理(用平均值中心化,再除相对标准差的平方根).数据预处理结果见补充材料见图S2.利用处理后的数据集建立主成分分析(PCA)模型,对原始数据进行可视化.观察2组样本的聚类趋势及同类样本的离散程度,找出存在于样本中的异常点,排除异常点后重新建立PCA模型.用排除异常点后的数据集建立有监督的模式识别算法——正交偏最小二乘判别分析(OPLS-DA)模型,OPLS-DA使用自适换算(unit variance scaling)的标准化方式.增加引入主成分的个数,直到模型的解释方差(R2)或者模型的预测方差(Q2)增长率不超过2%,建立模型的有效性通过7倍交叉检验及置换检验(200次迭代)来确认.根据建立的OPLS-DA模型的VIP值筛选尿液中和两组人群差异相关的标志代谢物[15].通过ROCCET(http://www.roccet.ca)软件对代谢标志物的数据矩阵进行可接收操作特征曲线(ROC)分析,确认标志代谢物中假阳性的指标,并利用t-test确认代谢标志物的显著性.根据筛选的代谢标志物建立两类人群的偏最小二乘判别分析模型(PLS-DA).并对模型的有效性进行内部交叉检验和内部置换检验.随机选取10个样本作为预测集,对代谢标志物所建立的PLS-DA预测模型进行外部预测检验,以此评价PLS-DA判别模型的精确度及适用范围.
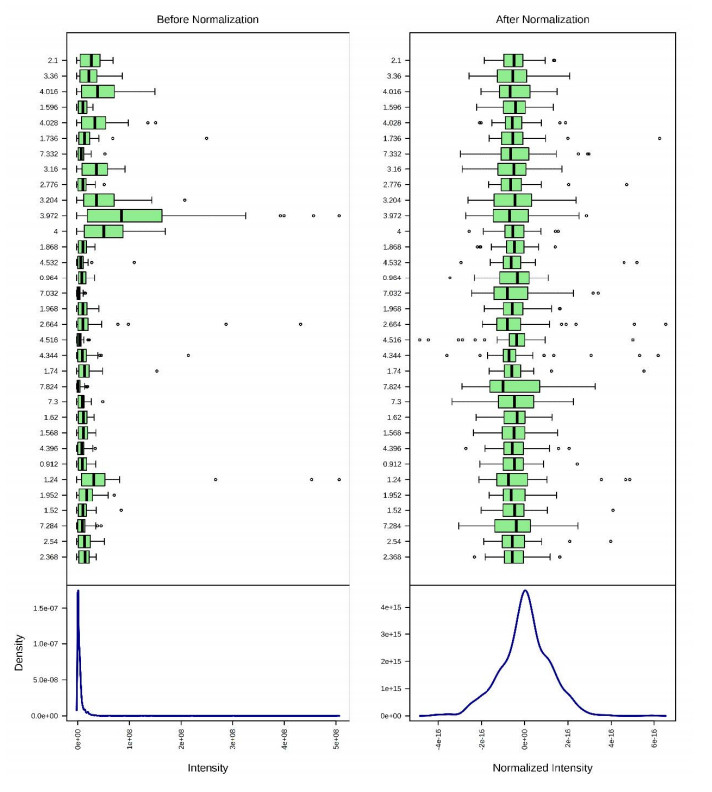
|
| 图 S2 尿液数据未处理(左)及处理后(右)结果对照 Fig. S2 Results of urinary spectral data before (left) and after (right) normalization |
为了观察运动员与体力劳动者样本点间的相似性及差异,首先建立无监督的PCA模型,结果如图 1所示.可以看出两组样本点通过第一主成分(PC1,解释方差R2=15.0%)或第二主成分(PC2,解释方差R2=11.0%)划分时相互覆盖.并且两个主成分的R2值都较小,说明PCA模型对A组和L组间的差异信息提取的不充分.此外,从整体上看两类样本点的离散度都较大,说明样本的个体差异大.综合来看,PCA模型可以最大化的提取样本之间的差异,但是它不能区别该差异是来源于组内还是组间. Motofumi K等人[16]报道PCA等无监督的模式识别算法在提取样本变异来源时忽略了原始样本的分类信息,但它可以观察样本的分布及聚类趋势、排除异常点、提高后续有监督模型的稳健性.

|
| 图 1 73个样本的PCA模型 Fig. 1 PCA score plot of the 73 samples |
为了能够充分提取组间的差异信息,筛选组间差异的标志代谢物.我们建立了OPLS-DA模型,该模型以样本的原始分类信息作为y变量,以样本的1H NMR谱图处理后的数据矩阵作为x变量.该模型可减小组内个体差异对模型的影响,充分提取两组样本间的变异,增强模型的解释能力. OPLS-DA得分散点图如图 2(a)所示.可以看出A组与L组志愿者的样本点分别分布于第一主成分的负半轴和正半轴上. OPLS-DA模型的累计R2=0.959,相比PCA模型(0.712)提高了0.247,说明OPLS-DA模型充分提取了组间的差异信息.此外,OPLS-DA模型的Q2值为0.877,相比PCA提高了0.458. Q2的大小反应了模型的预测精度,可知OPLS-DA的预测能力也同时提高. OPLS-DA模型的Q2大于0.5时,该模型具有良好的预测性能[17].但是在代谢组学研究中,Q2往往会小于0.5;模型过度拟合时,Q2也可大于0.5[18].因此需要对模型的有效性进行验证. 图 2(b)是OPLS-DA模型置换检验(200次迭代)的结果,图中所有Q2均在R2之下,且Q2的回归直线与y轴的交点在负半轴,说明该模型未过度拟合[17]. 表 1是OPLS-DA模型参数方差检验结果,可知p =3.4×10-26 < < 0.01,达极显著水平,说明该模型的预测性能强,充分提取了组间的差异信息,据此判别模型筛选A组和L组间的差异代谢标志物可充分反映两类人群的差异特征.

|
| 图 2 73个样本的OPLS-DA (a)得分图和(b)置换检验散点图 Fig. 2 OPLS-DA (a) score plot and (b) permutation test of the 73 samples |
| 平方和(SS) | 自由度(df) | 均方(MS) | F | pa | 标准差(SD) | |
| 方差总和(Total corr.)b | 72 | 72 | 1 | 1 | ||
| 回归平方和(Regression)c | 63.14 | 8 | 7.89 | 57.02 | 3.4×10–26 | 2.81 |
| 残差(Residual)d | 8.86 | 64 | 0.14 | 0.37 | ||
| a p < 0.05时模型有效;b 方差总和,即模型交叉验证时实际产生的误差平方和;c 回归平方和,即模型交叉验证时预测值与真值之间的误差平方和;d 残差,即方差总和与回归平方和之间的差值. | ||||||
为了评价各指标对两类样本组间差异的贡献大小,特引入各指标OPLS-DA模型的VIP值(图S3).VIP的大小反应了该指标(所对应的化学位移)对两类人群组间差异的贡献能力:VIP越大,表示该指标与样本分类信息的相关性越高;VIP > 1,表明该指标表征的组间差异大于组内误差,可作为代谢标志物的候选指标.据此,筛选出的代谢标志物有24个(如表 2所示).
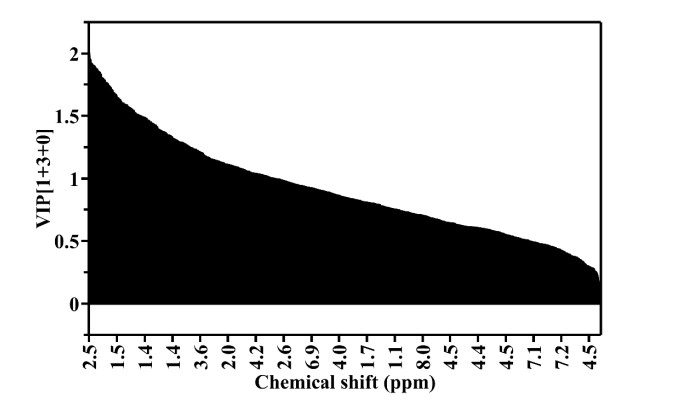
|
| 图 S3 OPLS-DA模型的VIP值 Fig. S3 VIP value derived from OPLS-DA |
| Biomarkers | δHa | VIP b | AUCc | pd |
| Citrate(柠檬酸) | 2.56 (d), 2.66 (d) | 2.103 | 0.929 | 9.28×10–10 |
| 2-Hydroxyisovalerate(2-羟基异戊酸) | 0.94 (d) | 2.014 | 0.702 | 6.16×10–3 |
| Butyrate(丁酸盐) | 0.88 (t) | 1.986 | 0.666 | 3.11×10–2 |
| Mannitol(甘露醇) | 3.80 (d) | 1.687 | 0.571 | 9.40×10–1 |
| Threonine(苏氨酸) | 3.69 (m) | 1.572 | 0.618 | 1.88×10–1 |
| Lactate(乳酸盐) | 1.34 (d) | 1.567 | 0.704 | 4.50×10–3 |
| Succinate(琥珀酸盐) | 2.41 (s) | 1.499 | 0.810 | 3.30×10–6 |
| Taurine(牛磺酸) | 3.43 (t) | 1.470 | 0.840 | 1.98×10–7 |
| N-Acetylaspartate(N-乙酰天冬酰胺) | 1.99 (s) | 1.392 | 0.753 | 1.33×10–4 |
| Glycine(甘氨酸) | 2.57 (s) | 1.370 | 0.823 | 2.97×10–7 |
| Glycerophosphorylcholine(甘油磷脂酰胆碱) | 3.23 (s) | 1.352 | 0.839 | 7.32×10–6 |
| Trimethylamine(三甲胺) | 2.87 (s) | 1.327 | 0.752 | 2.79×10–4 |
| Acetone(丙酮酸) | 2.35 (s) | 1.250 | 0.753 | 1.46×10–4 |
| 3-Hydroxyisovalerate(3-羟基异戊酸) | 1.27 (s) | 1.177 | 0.746 | 2.35×10–4 |
| α-Ketoglutarate(α-酮戊二酸) | 2.45 (t) | 1.165 | 0.786 | 1.97×10–5 |
| Trimethylamine N-oxide(N-氧基三甲胺) | 3.27 (s) | 1.135 | 0.538 | 1.00 |
| Hippurate(马尿酸盐) | 7.56 (t), 7.64 (t), 7.84 (d) | 1.121 | 0.853 | 7.42×10–7 |
| Alanine(丙氨酸) | 1.48 (d) | 1.120 | 0.690 | 4.82×10–3 |
| Dimethylamine(二甲胺) | 2.72 (s) | 1.113 | 0.595 | 1.48×10–1 |
| Lysine(赖氨酸) | 1.92 (m) | 1.096 | 0.749 | 7.89×10–4 |
| 3-Aminoisobutyrate(3-氨基异丁酸) | 1.20 (d) | 1.085 | 0.827 | 4.29×10–4 |
| L-Histidine(L-组氨酸) | 7.90 (s) | 1.034 | 0.826 | 1.15×10–7 |
| N-Acetylglutamate(N-乙酰谷氨酸) | 2.05 (d) | 1.029 | 0.803 | 5.95×10–6 |
| Valine(缬氨酸) | 1.04 (d) | 1.003 | 0.759 | 9.12×10–5 |
| a 1 s(单峰),d(双峰),t(三重峰),m(多重峰);b 由OPLS-DA模型分析获得;c AUC是ROC曲线下的面积;d p < 0.05表示差异显著. | ||||
为了排除OPLS-DA模型所筛选指标中假阳性及假阴性的指标,对VIP值筛选出的候选指标进行t-test及ROC分析.计算每个指标的显著性p值及ROC曲线下面积(area under curve,AUC).表 2结果表明在24个代谢标志物中,有4个代谢物(甘露醇、葡萄糖、二甲胺和N-氧基三甲胺)进行t-test检验时,两组样本间没有显著差异.但这四个代谢物的AUC均大于0.5.由于两类人群中存在着年龄等非实验因素的干扰,而ROC分析适用于仅有实验因素干扰的模型.因此这4个代谢物不作为区分两类人群的代谢标志物.
2.4 两类人群PLS-DA判别模型的建立及检验排除假阳性代谢标志物后,利用剩余代谢标志物的数据矩阵建立有监督PLS-DA分析.随机选取10个样本作为预测集,其余样本作为训练集.建立运动员及体力劳动者两类人群的预测模型,并对模型的有效性进行内部交叉、内部置换及外部预测检验.
2.4.1 内部交叉验证为了确认模型的有效性,采用蒙特卡罗算法(Monte-Carlo cross validation,MCCV)对代谢标志物所建立的PLS-DA预测模型进行交叉验证,每次验证时,2/3的样本用于评价指标的重要性,按重要性等级分别选取2、3、5、10、20和24个指标建立PLS-DA判别模型,余下1/3的样本用于检验这些指标所建判别模型的有效性.按此方法重复100次后,所得的ROC曲线平均,结果如图 3(a)所示.可以看出:当选取20个指标建立PLS-DA模型时,ROC曲线下的面积最大,AUC=0.998(置信区间CI=0.987~1). PLS-DA预测模型内部交叉验证结果如图 3(b)所示.可知20个指标所建立的PLS-DA模型的散点图中,L组与A组样本分布于第一主成分的正负象限内.其中A组有1个样本点判别错误. L组全部样本点判别正确. PLS-DA模型对A组和L组样本的误判率(false discriminating rate,FDR)分别为3.2%和0.
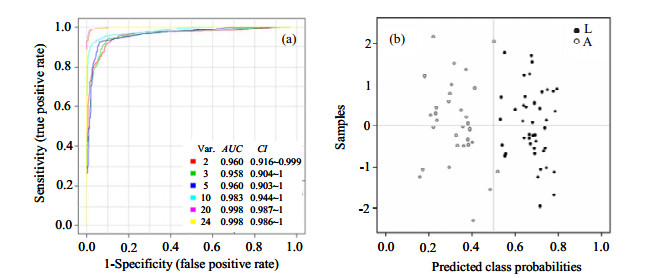
|
| 图 3 代谢标志物PLS-DA模型的(a) ROC分析和(b)内部交叉检验 Fig. 3 (a) ROC analysis of metabolic biomarkers and (b) cross validation of PLS-DA model |
代谢标志物建立PLS-DA预测模型时,假阳性指标的引入会使得模型过度拟合,模型的预测精度下降[19].因此,需对代谢标志物建立的PLS-DA的预测精度进行置换检验.图 4是1 000次置换检验的结果.可以看到其频率近似服从正态分布,且模型检验参数的显著水平p=3.34×10-5 < < 0.001,达极显著水平.说明2.4.1节中代谢标志物所建立的预测模型精度高.也进一步说明,OPLS-DA模型筛选的代谢标志物能体现A组与L组人群间的大部分差异特征.
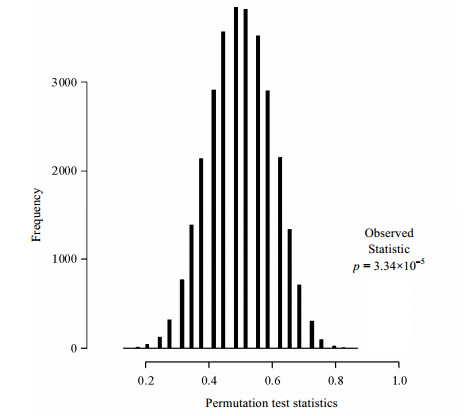
|
| 图 4 代谢标志物所建PLS-DA模型内部置换检验结果 Fig. 4 Permutation test of PLS-DA model built by 20 metabolic biomarkers |
为了检验代谢标志物所建立PLS-DA预测模型的适用范围,特对模型进行外部预测检验.随机选取10个样本(L7、L14、L21、L28、L35、A5、A10、A15、A20、A25)作为预测集,通过PLS-DA预测模型进行预测分析,结果如图 5和表 3所示.可知10个预测样本全部预测正确,误判率为0.说明所建PLS-DA预测模型的预测精度高.也进一步说明:通过ROC分析筛选出的用于建模的20个代谢标志物能够代表两类人群间的代谢差异,假阳性及假阴性指标排除的比较充分.也说明了OPLS-DA模型用于筛选A组与L组间差异的代谢标志物可以降低年龄等非实验因素的干扰.
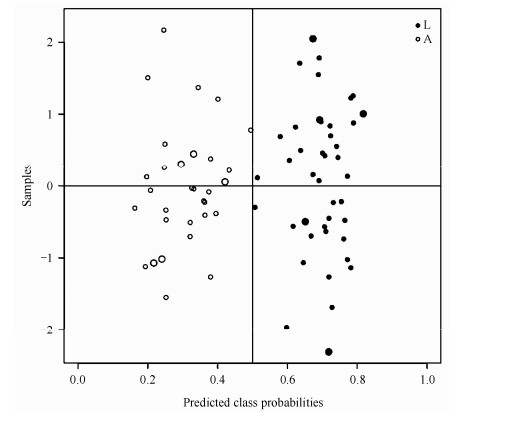
|
| 图 5 代谢标志物所建PLS-DA模型外部预测检验结果.大圆圈和大圆点代表预测集样本,小圆圈和小圆点代表训练集样本;圆圈代表A组样本,圆点代表L组样本. Fig. 5 External prediction tested by PLS-DA model built with metabolic biomarkers. Small circles: training sets of group A; small dots: training sets of group L; big circles: prediction sets of group A; big dots: prediction sets of group L. |
| 样本 | p | Predicted Class | 样本 | p | Predicted Class |
| A5 | 0.588 | A | L7 | 0.697 | L |
| A10 | 0.771 | A | L14 | 0.714 | L |
| A15 | 0.705 | A | L21 | 0.672 | L |
| A20 | 0.659 | A | L28 | 0.737 | L |
| A25 | 0.599 | A | L35 | 0.788 | L |
| 注:A代表运动员,L代表体力劳动者.结果表明10个样本全部预测准确,误判率FDR为0. | |||||
总之,通过20个标志代谢物所建立的运动员和体力劳动者的PLS-DA预测模型通过内部交叉验证、内部置换检验及外部预测检验均表明该模型具有较高的预测精度.在24个代谢标志物中有4个通过独立样本t-test检验不显著,但是通过ROC分析筛选代谢标志物时,有20个标志代谢物加入建模指标.可能原因是:A组或L组人群内存在显著的个体差异(如年龄、饮食、生活习惯等非实验因素),不满足t-test独立检验的条件.因此,不显著的指标中存在假阴性指标.此外,PLS-DA模型只适用于具有线性特征的样本,对于浓度在处理组及对照组间没有高低变化趋势的样本,该预测模型不适合[10].
3 结论本研究通过代谢组学技术分析了运动员、体力劳动者两类人群的代谢特征,结果表明两种工作性质不相近的人群代谢特征具有明显的差别.这说明对于不同职业的人群应根据其代谢特征进行有针对性的健康干预.此外,通过两类人群间差异显著的代谢标志物建立未知职业人群的预测模型,有助于将来对不同职业人群根据其代谢特征进行有针对性的干预提供了新的思路.为了提高预测模型的稳健性,应通过ROC分析确定参与建模的代谢标志物中是否存在假阳性及假阴性指标,并筛选最优的建模指标集合.对PLS-DA预测模型进行内部交叉验证可以反映该模型对组间差异的提取是否完全,信息关联的是否充分;内部置换检验可以反映模型预测精度的大小,观察PLS-DA预测模型是否过度拟合;外部预测检验可以检验模型的稳健性.这3种检验参数达到最佳,才能建立最优的预测模型.本研究通过20个指标所建立的两类人群的预测模型精度比较高(AUC=0.998),对未知样本的预测误判率小(FDR=0).这为今后更多不同职业人群代谢组学模型的建立提供了参考.但是本研究缺乏时序动态分析,即根据不同职业人群代谢特征在不同时间点的变化趋势,预测不同职业人群健康状态发展的方向.
| [1] | Loewenstein R J. An office mental status examination for complex chronic dissociative symptoms and multiple personality disorder[J]. Psychiatric Clinics of North America , 1991, 14 : 567-604 |
| [2] | Pronk N P, Katz A S, Lowry M, et al. Reducing occupational sitting time and improving worker health: The Take-a-Stand Project, 2011[J]. Prev Chronic Dis , 2012, 9 : 110 323 DOI:10.5888/pcd9.110323 |
| [3] | Bauer U E, Briss P A, Goodman R A, et al. Prevention of chronic disease in the 21st century: elimination of the leading preventable causes of premature death and disability in the USA[J]. The Lancet , 2014, 384 (9937) : 45-52 DOI:10.1016/S0140-6736(14)60648-6 |
| [4] | Bartley M, Plewis I. Accumulated labour market disadvantage and limiting long-term illness: Data from the 1971-1991 Office for National Statistics' Longitudinal Study[J]. Int J Epidemiol , 2002, 31 (2) : 336-341 DOI:10.1093/ije/31.2.336 |
| [5] | Merry L L, Manuel M, Aldrin V G, et al. Transformative impact of proteomics on cardiovascular health and disease a scientific statement from the american heart association[J]. Circulation , 2015, 132 (9) : 852-872 DOI:10.1161/CIR.0000000000000226 |
| [6] | Sek W K, In-Hee L, Ignaty L, et al. Summarizing polygenic risks for complex diseases in a clinical whole-genome report[J]. Genet Med , 2014, 17 (7) : 536-544 |
| [7] | Feng W, Themistocles D, Leslie C, et al. Genome-wide gene expression differences in Crohn's disease and ulcerative colitis from endoscopic pinch biopsies: insights into distinctive pathogenesis[J]. Inflamm Bowel Dis , 2007, 13 (7) : 807-821 DOI:10.1002/ibd.20110 |
| [8] | Uta B, Sebastian B, Lars P, et al. Proteomic analysis of the inflamed intestinal mucosa reveals distinctive immune response profiles in Crohn's disease and ulcerative colitis[J]. J Immunol , 2007, 179 (1) : 295-304 DOI:10.4049/jimmunol.179.1.295 |
| [9] | Timothy M D E, Rachel C. Bioinformatic methods in NMR-based metabolic profiling[J]. Prog Nucl Magn Reson Spectrosc , 2009, 55 (4) : 361-374 DOI:10.1016/j.pnmrs.2009.07.003 |
| [10] | 陈波, 康海宁, 韩超, 等. NMR指纹图谱与模式识别方法在食物分析中的应用[J]. 波谱学杂志 , 2006, 23 (3) : 397-407 Chen Bo, Kang Hai-ning, Han Chao, et al. Applications of NMR spectroscopy and pattern recognition in food analysis[J]. Chinese J Magn Reson , 2006, 23 (3) : 397-407 |
| [11] | Nicholson J K, Holmes E, Kinross J M, et al. Metabolic phenotyping in clinical and surgical environments[J]. Nature , 2012, 491 (7424) : 384-392 DOI:10.1038/nature11708 |
| [12] | Beckonert O, Keun H C, Ebbels T M, et al. Metabolic profiling, metabolomic and metabonomic procedures for NMR spectroscopy of urine, plasma, serum and tissue extracts[J]. Nat Protoc , 2007, 2 (11) : 2692-2703 DOI:10.1038/nprot.2007.376 |
| [13] | 刘悦, 高运苓, 程吉, 等. 一种核磁共振波谱谱峰对齐及谱峰提取的方法[J]. 波谱学杂志 , 2015, 32 (2) : 382-392 Liu Yue, Gao Yun-ling, Cheng Ji, et al. A processing method for spectrum alignment and peak extraction for nmr spectra[J]. Chinese J Magn Reson , 2015, 32 (2) : 382-392 |
| [14] | Wishart D S, Tzur D, Knox C, et al. HMDB: The human metabolome database[J]. Nucleic Acids Res , 2007, 35 (S1) : 521-526 |
| [15] | 赵秀举, 王玉兰. 代谢组学数据分析与药物毒理研究[J]. 波谱学杂志 , 2011, 28 (1) : 2-17 Zhao Xiu-ju, Wang Yu-lan. Applications of NMR-based metabonomics approaches in the assessment of drug toxicity[J]. Chinese J Magn Reson , 2011, 28 (1) : 2-17 |
| [16] | Kumazoe M, Fujimura Y, Hidaka S, et al. Metabolic profiling-based data-mining for an effective chemical combination to induce apoptosis of cancer cells[J]. Sci Rep , 2015, 5 : 9474 DOI:10.1038/srep09474 |
| [17] | Eriksson L, Johansson E, Kettaneh-Wold N, et al. Multi-and Megavariate Data Analysis: Principles and Applications[M].3rd ed. Umea: Umetrics Academy, 2001 |
| [18] | Chan E C Y, Pasikanti K K, Nicholson J K. Global urinary metabolic profiling procedures using gas chromatography-mass spectrometry[J]. Nat Protoc , 2011, 6 (10) : 1483-1499 DOI:10.1038/nprot.2011.375 |
| [19] | 徐广通, 袁洪福, 陆婉珍. 近红外光谱定量校正模型适用性研究[J]. 光谱学与光谱分析 , 2001, 21 (4) : 459-463 Xu Guang-tong, Yuan Hong-fu, Lu Wan-zheng. Study of quantitative calibration model suitability in near-infrared spectroscopy analysis[J]. Spectrosc Spect Anal , 2001, 21 (4) : 459-463 |
 2016, Vol. 33
2016, Vol. 33



