文章信息
- 戴晨晔, 张则婷, 刘买利, 李从刚
- DAI Chen-ye, ZHANG Ze-ting, LIU Mai-li, LI Cong-gang
- NMR在α-synuclein的结构及相互作用研究中的应用
- Application of NMR in the Studies of Structure and Interactions of α-Synuclein
- 波谱学杂志, 2016, 33(1): 153-167
- Chinese Journal of Magnetic Resonance, 2016, 33(1): 153-167
- http://dx.doi.org/10.11938/cjmr20160115
-
文章历史
- 收稿日期: 2015-04-20
- 收修改稿日期: 2016-01-25
2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
α-synuclein(AS)是由Maroteaux等人[1]首次在突触前神经末梢中发现,因而被命名为突触核蛋白(synuclein),但它具体的生理功能一直不清楚.1997年,研究者在帕金森病人大脑内患病标志物—路易小体中分离出了AS的一个突变体,AS与帕金森病的相关性由此得到确认[2, 3, 4, 5].路易小体的主要成分是AS蛋白纤维,AS被观察到在体内和体外都可以纤维化,其纤维化过程为:AS由最初的无结构状态到形成部分折叠的中间态,再到寡聚物的形成,进一步形成排列更紧密的纤维.其所经历的过程是成核机制,决速步是形成纤维初核,之后便是快速的装配连接过程[6, 7, 8].
AS包含140个氨基酸残基,由三个部分组成:第1~60位氨基酸残基为N端、含有四个不完全重复的保守片段(KTKEGV);中间的第61~95位氨基酸残基为NAC区域(NAC region),这一区域有三个上述的保守KTKEGV重复片段[9];第96~140位氨基酸残基组成C端,C端富含很多酸性氨基酸和脯氨酸,带有大量负电荷.N端和NAC区域容易形成α-螺旋的二级结构,它是与生物膜相互作用的区域;NAC区域在AS纤维化过程中作为疏水中心,对AS的聚集非常重要,与AS同属一个家族的β-synuclein因为缺少AS NAC区域中的71~82位等12个氨基酸残基而不会聚集;酸性的C端则表现了极强的亲水性,在水溶液、膜环境及纤维中都没有稳定的二级结构[10].
除了AS,与神经退行性疾病相关的tau蛋白、Aβ等都为天然无结构蛋白,常规的蛋白质结构研究方法,如X射线晶体衍射等难以对这类蛋白质进行研究.核磁共振(NMR)方法可以获得原子分辨率的蛋白质结构和动态信息,在天然无折叠蛋白的研究中独具优势.本文以AS为例,综述了NMR技术在天然无结构蛋白AS的结构、聚集机制,以及与生物膜、金属离子和其它蛋白质相互作用中的研究现状及帕金森病发病的可能机制.
1 溶液中AS的系综构象研究近年的研究[11]发现AS并非如早先人们所认为的无结构,在溶液状态下,AS表现出分子内的长程相互作用.
AS的斯托克斯半径(Stokes radius)要远大于相近分子量的球形蛋白,常规的圆二色谱(CD)和傅里叶变换红外光谱(FT IR)数据也都表明AS没有可见的二级结构[12, 13].然而人们发现,通过AS的色谱图所得出的斯托克斯半径要小于完全无结构的相近分子大小的肽链半径[13].
随后Hoyer等人[14]发现在普通的缓冲溶液体系中,高盐条件下AS的纤维化速率要大于低盐条件下,而将AS的C端切除或者加入能和AS的C端作用的精胺(spermine)后,盐浓度对AS纤维化速率的影响不大.因而他们猜测在低盐的情况下,AS的C端和NAC区域有一定的相互作用,阻碍了NAC区域聚集的速率;而高盐条件下,溶液中离子强度高,减弱了分子内的静电相互作用,使得C端打开,释放了NAC区域.因此推测单体AS在溶液中存在着动态涨落的系综构象,部分构象有利于稳定本底水平,自动抑制寡聚,从而防止AS的纤维化[15].之后的研究[11]证实了这样的猜想,同时研究者[11]还发现AS的多个部分之间都存在相互作用.
液体NMR技术中,顺磁弛豫增强(PRE)和残余偶极偶合(RDC)技术的结合对于探测低丰度蛋白质的系综构象、构象中的长程约束、以及精准的偶极朝向具有极大的优势.在溶液条件下,Bertoncini等人[11]通过PRE实验发现了长程的接触:AS的N端(第20位氨基酸残基附近)和C端(第120位氨基酸残基附近)、NAC区域(85~95位)和C端(110~130位)都有接触(图 1).同时15N弛豫速率也显示AS的第20位和第120位氨基酸残基自由度减弱[16].Uversky[7, 17]认为这种结构是一种功能性的错误折叠.
另外,在低pH(pH=3.0)的情况下,AS的CD谱显示出196 nm附近的最小值有所提高,表明AS出现了部分折叠[13].2009年,Wu等人[18]通过测量横向弛豫速率(R2-CPMG)、PRE和RDC等NMR方法证明了这一观点,并认为当pH<4.7(AS等电点pI值为4.7)时,AS被完全质子化,N端和C端的疏水性变强,使得AS的结构变紧密.
因此,AS在溶液中存在着不同的具有部分折叠的系综构象,并且AS容易受到溶剂环境的影响而具有不同的构象.
2 AS与生物膜相互作用研究在生理环境下,约有15%的AS是膜结合态,而其余大部分是游离于细胞质中的自由态[19].AS与膜的结合会导致蛋白质α-螺旋含量的显着增加.不过有研究[20, 21]发现AS和生物膜结合后(人工模拟生物膜)会形成多种构象.
突触细胞膜的主要组成部分包括磷脂酰胆碱(phosphatidylcholine,40.9%)、磷脂酰乙醇胺(phosphatidylethanolamine,24.6%)、缩醛磷脂酰乙醇胺(plasmenylethanolamine,11.5%)、鞘磷脂(sphingomyelin,12%)、磷脂酰丝氨酸(phosphatidylserine,7.3%)以及磷脂酰肌醇(phosphatidylinositol,3.7%),这些成分不均匀地分布于突触细胞膜[22]. 现有研究通常使用上述1~2种磷脂的混合物或者十二烷基磺酸钠(SDS)等表面活性剂制备的胶束、脂双层或多层囊泡来模拟生物膜.
AS与膜结合后会出现不同种类的富含α-螺旋的二级结构,而结构的不同取决于所模拟的生物膜的不同.有实验发现AS和胶束的结合会导致该蛋白形成两条反平行的 α-螺旋,分别为Val3~Val37和Lys45~Thr92,两段α-螺旋由一段有序的结构连接,通常将这样的结构称为断裂型α-螺旋,将中间的连接部分称为螺旋断点(helical break),而AS酸性的C段依旧保持无结构的状态[20, 21].这种断裂型α-螺旋通过液体NMR谱被大量观测到,所用到的方法包括化学位移扰动、15N横向弛豫速率测量等[23, 24, 25, 26].AS和磷脂双分子层的结合只会导致蛋白形成单一的α-螺旋,被称为连续型α-螺旋,而这个α-螺旋的范围为1~90位,很多电子顺磁共振(EPR)实验都能观测到该构象[27].
最近的一项研究[28]表明改变溶液中SDS浓度能够模拟出不同生物膜形态,从而观察到不同类型的富含α-螺旋的二级结构.随着SDS浓度的逐步上升,AS的二级结构出现了一系列的变化.在高浓度SDS条件下(450 mmol/L),AS表现为连续型α-螺旋结构.而当SDS浓度为50 mmol/L时,SDS胶束呈球状,AS与之结合形成了断裂型α-螺旋结构,向这一体系中加入助表面活性剂正己醇将胶束转化为平面结构时,AS的结构会随之转换成连续型的α-螺旋结构.这说明两种结构的出现与胶束的表面弧度有关系(图 2).
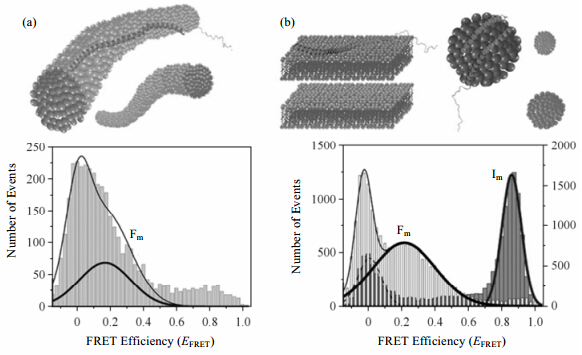
|
| 图 2 不同SDS浓度下AS的形态.(a)450 mmol/L SDS,(b)50 mmol/L SDS时,加入正己醇(左)和没加正己醇(右).下面的两个图为对应的smFRET效率图,Fm为连续型螺旋,Im为断裂型螺旋[28] Fig. 2 AS folding induced by interaction with different lipid membrane mimics.(a)AS in the presence of very high concentration of SDS(450 mmol/L),(b)The addition of the cosurfactant hexanol transforms spherical SDS micelles([SDS]=50 mmol/L)into a stack of flat bilayers,and induces a protein conformational switch from the high-EFRET spherical micelle-bound Imform to the low-EFRET bilayer-bound Fm species[28] |
通过EPR的方法(位点定向自旋标记,SDSL)分析AS与生物膜(POPG LUV)结合后的形态,甚至能够观察到前文所述的两种构象共存,其中断裂型α-螺旋结构占20±5%,连续型α-螺旋结构占80±5% [29].对两种构象进行热力学分析,发现断裂型α-螺旋结构比连续型的自由能高出约1.2 kcal/mol,从而可以猜想在天然情况下,AS与生物膜结合时断裂型α-螺旋结构约占12%[26].
AS与膜结合对膜也会产生影响,通过固体NMR方法对多层囊泡样品(MLV)的2H四极耦合裂分和31P化学位移各向异性的观测,可以得到一些膜表面变化的信息,上述体外实验表明AS倾向于促使磷脂排列成小型囊泡或者非脂双层结构[30].其他的体外实验也发现了AS能改变膜表面弧度,甚至促使大型囊泡变为小型囊泡的现象[31].
由此可见AS与生物膜相互作用后会形成不同构象,这是由膜的性质所决定的.同时,AS也会反过来影响膜的形态.
3 AS的纤维化研究已发现的AS纤维有多种形态,研究者认为这些不溶聚集物的多样化源于纤维化环境不同,导致了蛋白质侧链装配存在差异.环境的不同主要包括不同的pH值、温度、搅拌速率、蛋白质浓度和离子强度等等.
淀粉状纤维(amyloid fibrils)主要有两类:源于天然无结构的蛋白质和源于有结构的蛋白质.天然无结构蛋白质纤维化通常都是从最初的无结构状态折叠为富含β折叠的二级结构,这样形成的纤维通常通过主链上氢键的相互作用装配,所以非常稳定.有结构蛋白质的纤维化则是蛋白质小范围的结构变动使其部分结构暴露出来,所暴露的区域和其他分子相互作用,从而纤维化.这一类蛋白质本身就富含β结构,形成纤维后其结构变化很小[32].
AS是天然无结构蛋白,与之同类的还有tau蛋白和Aβ等等.有研究[33, 34]显示AS纤维是由多个初期的细丝(protofilaments)组成的,这样的细丝中AS的β-str and 平行排列.β-sheet通过侧链紧密装配,所形成的高度有序的结构被称为steric zippers.这些steric zippers上下摆动,将β-sheet界面之间的水赶走[7].这种steric zippers在AS以及其他同类蛋白所形成的淀粉状纤维中很常见.固体NMR技术对于这类淀粉状样品的研究具有一定的优势,有研究者用amylin上20~29位的短肽制备成淀粉状样品,通过固体NMR实验[包括偶极辅助旋转共振(DARR)、多量子核磁共振碳谱(MQ 13C NMR)]观测到了steric zippers的两种形式(β-sheet间的平行和反平行两种排列)[35].
现有实验发现了几种不同构象的AS纤维,氢氘(H/D)交换实验(通过将AS纤维置于重水中将活泼氢置换成氘之后,将AS纤维溶于有机溶剂,如氘代DMSO和氘代TFA)结果显示Ala-27残基的H/D交换曲线显示出一种单调衰减的快交换,而位于纤维中心的Val-52和Ala-90的H/D交换曲线则显示出慢交换和极慢交换两个衰减过程,说明AS纤维存在两种不同构象(图 3)[36],研究者[37]认为这是因为AS经历了不同的成核(nucleation)机制,而成核机制的不同使得b-sheet的排列以及侧链的装配也不尽相同.
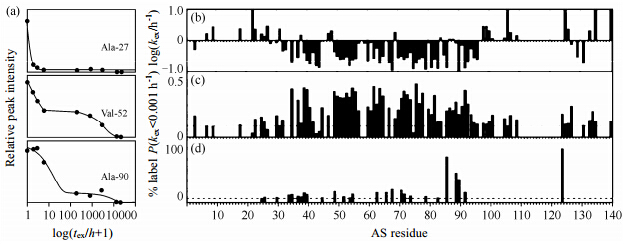
|
| 图 3 AS纤维的H/D交换实验.(a)3个氨基酸残基的H/D交换曲线,(b)各个氨基酸残基快交换和慢交换组分的H/D交换速率,(c)极慢交换的氨基酸残基H/D交换速率,(d)AS纤维Cys侧链溶剂接近度[36] Fig. 3 Detailed H/D exchange analysis and side-chain solvent accessibility of AS fibrils.(a)H/D exchange curves for 3 amide moieties as indicated,(b)Plot of the H/D exchange rates of the fast- and slow-exchanging population against the amino acid sequence,(c)Plot of the fraction of each amide moiety that exhibit extremely slow H/D exchange at rates <0.001 h-1,(d)Side-chain solvent accessibility of Cys in fibrils of AS variants[36] |
由于AS纤维的形态限制,研究者常常选用固体NMR实验对AS纤维进行研究,这方面的研究也有很多有趣的结果.Heise等人[37]通过对比INEPT、CP/MAS、one pulse、T2relaxation filter这4组实验的13C NMR谱,观察到N端从22位开始,结构就比较刚性;同时通过对比5组分别制备的AS纤维样品的NCA谱图(15N-13Cα),以及由几组样品NMR谱通过TALOS所分析得出的扭转角(torsion angles)等数据,发现存在两种不同的AS纤维.两种纤维的差别主要表现在83EGAG86的转角区(turn motif)和随后的β-str and 结构区(S87~V95).进一步分析这些差别发现中间核心区β-sheet区域是非常稳定保守的,而在转角区和之后的β-str and 结构区存在不同(图 4).他们认为这可能是缘于AS纤维化环境的不同,AS纤维化对pH值、温度、搅拌速率和蛋白质浓度都很敏感.之后,Heise等人[38]对AS家族遗传突变体A53T的纤维进行了研究,发现它是不同于上述两种形态的第三种纤维,这种富含β-结构的纤维核心区域(core region)范围更广,覆盖L38~L100,而不是之前在野生型AS纤维中的L38~V95的区域(图 4).最近,Lemkau等人[39]对AS另一种天然条件下的突变体A30P的纤维进行研究,发现虽然A30P的纤维化速率要慢于野生型AS,但是通过谱图比较,其形成的纤维和野生型AS纤维是一样.
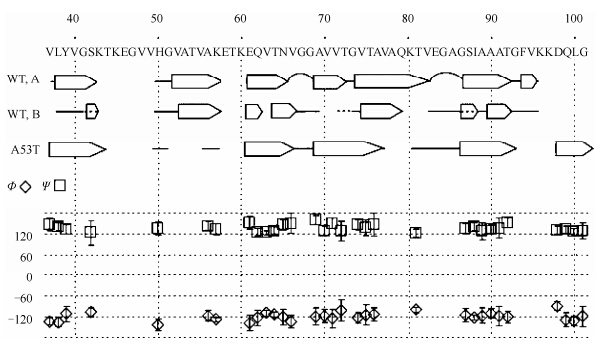
|
| 图 4 根据化学位移,通过TALOS分析得出的主链角度.图中WT,A和WT,B所示的分别是野生型AS所形成的两种纤维,A53T为AS的A53T突变体核心区(core region)的结构示意,箭头表示b-str and ,灰线为未归属的氨基酸,实线为没有算出明确结构的区域,曲线为转角区(turn or loop regions)[38] Fig. 4 Backbone angles obtained with TALOS analysis of the chemical shifts. Only torsion angle pairs where at least eight hits were found are plotted,together with the error bars. For comparison,b-str and s determined earlier for both wild-type forms are given together with the b-str and s of A53T. Nonassigned amino acids are marked with gray bars,and b-sheets as determined by TALOS are indicated by white arrows; assigned residues for which TALOS did not yield conclusive results are marked with a black line[38] |
不同的纤维化环境会诱导形成具有不同形态的AS纤维,这些纤维的装配方式、AS参与区域和纤维化速率都有所不同.
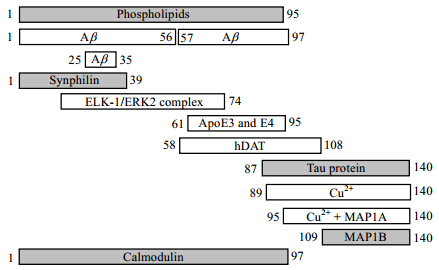
4 AS的聚集机制研究研究发现金属离子、蛋白质、磷脂等都能通过和AS相互作用而影响其聚集.然而这些影响或促进或抑制,所涉及的机制也不尽相同(图 5).
人们对于金属离子与AS作用的关注源于一些流行病学的研究:密西根州工业区域和其他区域相比有更高的PD死亡率[40];PD患者脑组织中黑质(substantia nigra)的金属(铁、锌、铝等)含量比对照组更高[41, 42].以上都表明环境中较高的重金属含量与PD高发病率有一定关联.金属离子对于PD发病率的影响在于其对AS聚集及纤维化的影响.根据已有研究,这种影响可以大致分为两类:一种为金属离子与AS直接作用,从而改变AS的构象[43];另一种为氧化性金属离子和AS发生氧化还原反应,从而影响AS的聚集[44, 45].
利用液体NMR技术研究金属离子和AS的相互作用具有直观方便和快速等优点.通过化学位移扰动、谱峰展宽和消失等情况,可以直接探测相互作用位点和测定结合常数.以下我们选取几种有代表性,且具有生物学意义的金属离子,从分子层面上分析它们对AS纤维化的影响.
4.1.1 铝离子Al3+如今被认为是一种提高PD发病率的金属离子.在中国人的饮食习惯中,Al3+的添加也十分常见.Al3+表现出促进AS聚集的现象.在含有Al3+的环境下,AS的CD谱、紫外吸收光谱(UV)和荧光光谱都显示它出现了部分折叠的中间态[46].这种中间态的变化在低蛋白浓度条件下是快速可逆的[12].
4.1.2 钙离子研究[47]表明AS能够调控Ca2+进入细胞的通路.同时Ca2+、AS和细胞质中的多巴胺三者之间的相互作用会导致黑质中多巴胺能神经元的凋亡[48].研究发现,Ca2+可与AS竞争结合生物膜,导致AS与膜的结合减弱[49].同时有研究发现Ca2+还能够与AS的C端结合,介导AS的C端与生物膜相互作用,促进AS聚集[50].Ca2+存在条件下,AS形成的寡聚物有两种形式,包括环形寡聚物(直径70~90 nm,高4 nm)和球形寡聚物(直径10~20 nm),其中环形寡聚物曾于多系统萎缩(multiple system atrophy)脑组织中提取,因而推测活体中Ca2+可能也具有诱导环形寡聚物形成的作用[51, 52, 53].
4.1.3 铜离子Cu2+与AS有很强的结合作用,其结合位点主要位于AS的N端,分别是1MDVFMKGLS9(kd=0.1±0.01 μmol/L)和48VAHGV52(kd=35.0±4.0 μmol/L)两个区域[54, 55].同时Cu2+与C端119DPDNEA124也有相对较弱的相互作用(kd≈1 mmol/L).但这段序列结合特异性不强,也是其他二价金属离子(Fe2+,Mn2+)与AS的结合区域[55].Cu2+存在条件下,AS单体中“β-like”结构含量增多,而AS纤维化过程中“β-like”结构含量亦显著上升,因而与Cu2+的结合被认为使AS获得了易于聚集的结构[56, 57, 58].同时Cu2+具有氧化能力,可对AS造成氧化性损伤,进一步促进AS的聚集[59].因此,Cu2+被认为是影响AS聚集的重要因素之一.
4.1.4 铁离子
最近的一项研究[60]认为AS是一种Fe2+/Fe3+还原酶,能将Fe3+还原成Fe2+.铁离子对AS聚集的影响主要表现在Fe3+的氧化作用能造成AS氧化损伤,促进AS的聚集;同时,Fe3+也会促使AS聚集形成离子通透性孔状寡聚物[61];此外,Fe2+可以与AS的C端结合,影响AS的聚集.
4.1.5 镁离子研究发现Mg2+摄入不足会提高PD的患病风险,这表明Mg2+对AS的影响有别于其他离子.金属离子普遍表现出促进AS聚集的规律,而Mg2+却相反,甚至与其他离子共存时表现出抵抗其他离子促进AS聚集的作用[62].这暗示Mg2+和AS的相互作用能减缓AS聚集.
Fern and ez等人[63]通过比较金属离子加入前后AS 1H-15N HSQC谱峰强度的改变,发现Mn2+、Co2+、Ni2+和Fe2+等都能和AS的C端相互作用(图 6),且通常优先与Asp121、Asn122和Glu123作用.C端的两个区域(Asp121和Asp135周围)含有很多带电氨基酸残基,是其与金属离子作用的区域.他们通过RDC实验发现AS的C端和金属离子相互作用源于三级作用,而不是人们所认为的电荷驱动.但是只有Cu2+能与AS的N端结合,而且Cu2+与AS相互作用不会诱导蛋白质部分折叠中间态的形成.
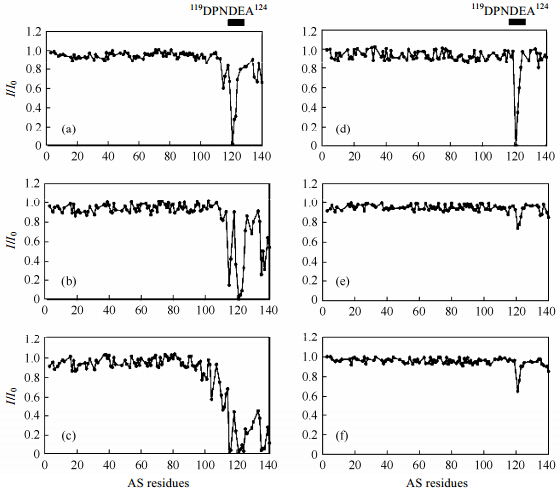
|
| 图 6 15℃下,100 μmol/L AS加入不同金属离子及不同金属离子浓度作用后,主链1H-15N HSQC谱峰强度比较(I/I0).其中各图分别为:(a)15 μmol/L Mn2+,(b)40 μmol/L Mn2+,(c)100 μmol/L Mn2+,(d)100 μmol/L Co2+,(e)100 μmol/L Ni2+,(f)100 μmol/L Fe2+ [63] Fig. 6 I/I0 profiles of the backbone amide groups of AS in the presence of divalent metal ions. 1H-15N HSQC spectra of 100 μmol/L AS in buffer A at 15℃ were registered upon addition of(a)15 μmol/L Mn2+,(b)40 μmol/L Mn2+,(c)100 μmol/L Mn2+,(d)100 μmol/L Co2+,(e)100 μmol/L Ni2+,or(f)100 μmol/L Fe2+. N-Terminal region comprises residues 1~60; NAC region,residues 61~95; and C-terminal region,residues 96~140[63] |
已有研究表明AS和很多蛋白质都有相互作用,而多数的蛋白对于AS、AS的常见变种以及表达后修饰的AS都有作用.通过SILAC技术,人们发现在多巴胺能MES细胞中,能与AS相互作用的蛋白质多达324种[64].自从AS被发现以来,所观测到的与之结合的蛋白质不胜枚举.蛋白质和AS相互作用或抑制、或促进AS的聚集.
4.2.1 AS与synuclein的相互作用β-synuclein和γ-synuclein与AS一样,都是天然无结构蛋白,它们在酸性条件下都显现出部分折叠的构象.AS和γ-synuclein都能够通过体外温育的方法形成纤维,而β-synuclein则不会发生纤维化.研究认为AS中间疏水区的71~82位残基对其纤维化非常关键,β-synuclein不会纤维化的原因就是其缺少这一段区域[65].β-synuclein或γ-synuclein以1:1的摩尔比加入AS溶液中可显著降低AS纤维化的延伸(elongation)速率;而当β-synuclein或γ-synuclein过量(摩尔比为4:1)加入AS溶液时,AS的纤维化被完全抑制[65].研究认为上述两种蛋白质和AS形成混合四聚体是AS聚集被抑制的机制.因此β-synuclein和γ-synuclein被认为是体内AS纤维化的调控因子,在这一过程中表现出分子伴侣的作用[65, 66].
4.2.2 AS与分子伴侣的相互作用热休克蛋白(Hsp)属于分子伴侣的一类,路易小体中发现AS与热休克蛋白共存.其中比较有代表性的是Torsin A,实验发现细胞中过量表达Torsin A会抑制AS聚集;Hsp104(与Torsin A同源)甚至可在ATP驱动下解离AS已经形成的寡聚物[67, 68, 69].哺乳类动物的Hsp70分子伴侣系统有更强的解离AS寡聚物的功能,当在转基因小鼠体内过量表达Hsp70时,AS聚集被抑制[70].进一步实验结果表明Hsp70通过和AS的疏水核心结合,使AS无法形成具有细胞毒性的寡聚物[71].这些实验结果表明分子伴侣通过与AS相互作用抑制AS聚集,并发挥降低AS毒性的重要作用.
4.2.3 AS与tau蛋白的相互作用有些蛋白质在体外实验中表现出促进AS聚集的特性.例如路易小体中的tau蛋白和微管蛋白.而这些蛋白质加速AS聚集的机制被认为是相互做种(cross-seeding)机制[72].这种cross-seeding的机制也同样表现在tau蛋白上,比如AS被发现会导致tau蛋白在细胞环境下聚集[73].
4.2.4 AS与Synphilin-1的相互作用Synphilin-1(Sph1)是可与生物膜结合的突触前蛋白,AS存在条件下,Sph1与膜的结合会受到抑制.酵母双杂交实验结果显示AS和Sph1会发生相互作用[74].研究发现,在酵母细胞中表达Sph-1可促进AS细胞内包含物的形成;同时,在路易小体中也发现Sph1的存在,Sph1被认为有助于AS的聚集[74, 75].三共振实验、滴定实验(titration)和TrNOE(transfer NOE)实验可提供Sph1和AS相互作用的界面信息.结果表明,Sph1通过中间的coiled-coil形成反平行二聚体,二聚界面与AS的N端前部发生相互作用;并且在AS的Asp2、Val3、Phe4、Lys10、Ala11和Lys12上会出现转移的NOE信号[76].
4.3 生物膜及人工生物膜对AS聚集的影响现有的研究[77, 78]发现生物膜加快或减缓AS聚集速率的现象都存在.这一现象被认为是由于AS与细胞膜结合时形成了不同的构象.当体系中存在低浓度SDS(高于SDS的临界胶束浓度)时,只有AS和N端前部与胶束结合,而NAC疏水区依旧暴露,因而AS聚集受到促进;而SDS浓度升高后,39和70位标记19F NMR信号的化学位移出现变化,说明NAC也形成了α-螺旋,参与和胶束的相互作用[79, 80].
19F NMR对环境十分敏感,可用于表征蛋白质的结构变化,该方法对于解析AS和SDS的相互作用起到了重要作用.Wang等人[79]用19F标记AS,通过19F NMR观测AS和胶束相互作用.此外,通过不同位点的标记,在多种SDS浓度下观测19F NMR信号展宽和化学位移变化,证实了AS与胶束结合会产生促进AS聚集的特殊形态(图 7).
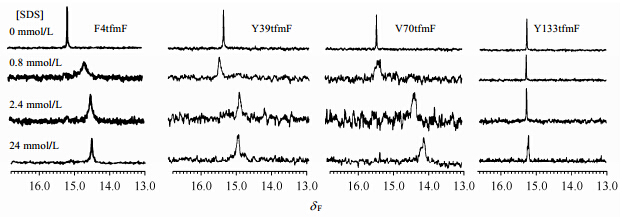
|
| 图 7 不同SDS浓度下,AS各标记位点的19F NMR谱图[79].分别由tfmF特殊标记于4,39,70或133位点处100 μmol/L synucleins在0,0.8,2.4和24 mmol/L SDS条件下的19F NMR谱图.样品溶于磷酸钠缓冲液中(50 mmol/L,pH 7.4).一些明显信噪比较低的谱图是放大y-轴后观察的宽的低灵敏度的共振信号 Fig. 7 19F NMR spectra of labeled 100 μmol/L AS with a tfmF residue at position 4,39,70 or 133 in 0,0.8,2.4 and 24 mmol/L SDS. Samples were prepared in sodium phosphate buffer(50 mmol/L,pH 7.4). The apparently lower signal-to-noise ratio of some spectra is the result of exp and ing the y-axis to facilitate observation of broad,low-sensitivity resonances[79] |
本文简要地介绍了近年来在AS的结构研究方面取得的进展,希望以AS为例,归纳出适合于AS结构研究的NMR方法,提出研究天然无结构蛋白结构的可行方案.
虽然如今已有很多关于AS的研究,不过AS的生理机制和生理功能等很多方面仍不清楚.比如Mg2+能够阻碍AS因受其他金属离子影响而加速聚集的过程;AS在经过泛素化、磷酸化和氧化等表达后修饰后会出现新的生理功能;AS能够和多种多样的蛋白质发生相互作用等等.上述这些过程都预示着AS结构上的未知变化,而这些结构变化以及动态过程的研究会有助于我们更好地理解AS与PD.传统的NMR技术在解决这些问题的过程中发挥了很大作用,但同时也需要发展一些新方法和新技术.
| [1] | Maroteaux L, Campanelli J T, Scheller R H. Synuclein: A neuron-specific protein localized to the nucleus and presynaptic nerve terminal[J]. J neurosci, 1988, 8(8): 2804-2815. |
| [2] | Polymeropoulos M H, Lavedan C, Leroy E, et al. Mutation in the α-synuclein gene identified in families with Parkinson's disease[J]. Science, 1997, 276(5321): 2045-2047. |
| [3] | Spillantini M G, Schmidt M L, Lee V M Y, et al. α-synuclein in Lewy bodies[J]. Nature, 1997, 388(6645): 839-840. |
| [4] | Masliah E, Rockenstein E, Veinbergs I, et al. Dopaminergic loss and inclusion body formation in α-synuclein mice: implications for neurodegenerative disorders[J]. Science, 2000, 287(5456): 1265-1269. |
| [5] | Betarbet R, Sherer T B, Mackenzie G, et al. Chronic systemic pesticide exposure reproduces features of Parkinson's disease[J]. Nat Neurosci, 2000, 3(12): 1301-1306. |
| [6] | Esler W P, Stimson E R, Jennings J M, et al. Alzheimer's disease amyloid propagation by a template-dependent dock-lock mechanism[J]. Biochemistry, 2000, 39(21): 6288-6295. |
| [7] | Breydo L, Wu J W, Uversky V N. α-synuclein misfolding and Parkinson's disease[J]. BBA-Mol Basis of Dis, 2012, 1822(2): 261-285. |
| [8] | Wood S J, Wypych J, Steavenson S, et al. α-synuclein fibrillogenesis is nucleation-dependent implications for the pathogenesis of parkinson's disease[J]. J Biol Chem, 1999, 274(28): 19509-19512. |
| [9] | Kessler J C, Rochet J C, Lansbury P T. The N-terminal repeat domain of α-synuclein inhibits b-sheet and amyloid fibril formation[J]. Biochemistry, 2002, 42(3): 672-678. |
| [10] | Giasson B I, Murray I V J, Trojanowski J Q, et al. A hydrophobic stretch of 12 amino acid residues in the middle of α-synuclein is essential for filament assembly[J]. J Biol Chem, 2001, 276(4): 2380-2386. |
| [11] | Bertoncini C W, Jung Y S, Fernandez C O, et al. Release of long-range tertiary interactions potentiates aggregation of natively unstructured α-synuclein[J]. Proc Nat Acad Sci USA, 2005, 102(5): 1430-1435. |
| [12] | Weinreb P H, Zhen W, Poon A W, et al. NACP, a protein implicated in Alzheimer's disease and learning, is natively unfolded[J]. Biochemistry, 1996, 35(43): 13709-13715. |
| [13] | Uversky V N, Li J, Fink A L. Evidence for a partially folded intermediate in α-synuclein fibril formation[J]. J Biol Chem, 2001, 276(14): 10737-10744. |
| [14] | Hoyer W, Cherny D, Subramaniam V, et al. Impact of the acidic C-terminal region comprising amino acids 109-140 on α-synuclein aggregation in vitro[J]. Biochemistry, 2004, 43(51): 16233-16242. |
| [15] | Fernández C O, Hoyer W, Zweckstetter M, et al. NMR of α-synuclein-polyamine complexes elucidates the mechanism and kinetics of induced aggregation [J]. EMBO J, 2004, 23(10): 2039-2046. |
| [16] | Bussell R, Eliezer D. Residual structure and dynamics in parkinson's disease-associated mutants of α-synuclein[J]. J Biol Chem, 2001, 276(49): 45996-46003. |
| [17] | Uversky V N. Intrinsically disordered proteins may escape unwanted interactions via functional misfolding[J]. BBA-Proteins Proteom, 2011, 1814(5): 693-712. |
| [18] | Wu K P, Weinstock D S, Narayanan C, et al. Structural reorganization of α-synuclein at low pH observed by NMR and REMD simulations[J]. J Mol Biol, 2009, 391(4): 784-796. |
| [19] | Lee H J, Choi C, Lee S J. Membrane-bound α-synuclein has a high aggregation propensity and the ability to seed the aggregation of the cytosolic form[J]. J Biol Chem, 2002, 277(1): 671-678. |
| [20] | Ulmer T S, Bax A, Cole N B, et al. Structure and dynamics of micelle-bound human α-synuclein[J]. J Biol Chem, 2005, 280(10): 9595-9603. |
| [21] | Ulmer T S, Bax A. Comparison of structure and dynamics of micelle-bound human α-synuclein and Parkinson disease variants[J]. J Biol Chem, 2005, 280(52): 43179-43187. |
| [22] | Michaelson D M, Barkai G, Barenholz Y. Asymmetry of lipid organization in cholinergic synaptic vesicle membranes[J]. Biochem J, 1983, 211(1): 155-162. |
| [23] | Chandra S, Chen X, Rizo J, et al. A broken α-helix in folded α-synuclein[J]. J Biol Chem, 2003, 278(17): 15313- 15318. |
| [24] | Perrin R J, Woods W S, Clayton D F, et al. Interaction of human α-synuclein and Parkinson's disease variants with phospholipids structural analysis using site-directed mutagenesis[J]. J Biol Chem, 2000, 275(44): 34393-34398. |
| [25] | Bisaglia M, Tessari I, Pinato L, et al. A topological model of the interaction between α-synuclein and sodium dodecyl sulfate micelles[J]. Biochemistry, 2005, 44(1): 329-339. |
| [26] | Lokappa S B, Ulmer T S. α-synuclein populates both elongated and broken helix states on small unilamellar vesicles[J]. J Biol Chem, 2011, 286(24): 21450-21457. |
| [27] | Jao C C, Hegde B G, Chen J, et al. Structure of membrane-bound α-synuclein from site-directed spin labeling and computational refinement[J]. Proc Nat Acad Sci USA, 2008, 105(50): 19666-19671. |
| [28] | Ferreon A C M, Gambin Y, Lemke E A, et al. Interplay of α-synuclein binding and conformational switching probed by single-molecule fluorescence[J]. Proc Nat Acad Sci USA, 2009, 106(14): 5645-5650. |
| [29] | Robotta M, Braun P, Van Rooijen B, et al. Direct evidence of coexisting horseshoe and extended helix conformations of membrane-bound α-synuclein[J]. Chem Phys Chem, 2011, 12(2): 267-269. |
| [30] | Madine J, Doig A J, Middleton D A. A study of the regional effects of α-synuclein on the organization and stability of phospholipid bilayers[J]. Biochemistry, 2006, 45(18): 5783-5792. |
| [31] | Pandey A P, Haque F, Rochet J-C, et al. α-synuclein-induced tubule formation in lipid bilayers[J]. J Phys Chem B, 2011, 115(19): 5886-5893. |
| [32] | Nelson R, Eisenberg D. Recent atomic models of amyloid fibril structure[J]. Curr Opin Struc Biol, 2006, 16(2): 260-265. |
| [33] | Serpell L C, Berriman J, Jakes R, et al. Fiber diffraction of synthetic α-synuclein filaments shows amyloid-like cross-b conformation[J]. Proc Nat Acad Sci USA, 2000, 97(9): 4897-4902. |
| [34] | Sawaya M R, Sambashivan S, Nelson R, et al. Atomic structures of amyloid cross-b spines reveal varied steric zippers[J]. Nature, 2007, 447(7143): 453-457. |
| [35] | Madine J, Jack E, Stockley P G, et al. Structural insights into the polymorphism of amyloid-like fibrils formed by region 20-29 of amylin revealed by solid-state NMR and X-ray fiber diffraction[J]. J Am Chem Soc, 2008, 130(45): 14990- 15001. |
| [36] | Vilar M, Chou H T, Lührs T, et al. The fold of α-synuclein fibrils[J]. Proc Nat Acad Sci USA, 2008, 105(25): 8637- 8642. |
| [37] | Heise H, Hoyer W, Becker S, et al. Molecular-level secondary structure, polymorphism, and dynamics of full-length α-synuclein fibrils studied by solid-state NMR[J]. Proc Nat Acad Sci USA, 2005, 102(44): 15871-15876. |
| [38] | Heise H, Celej M S, Becker S, et al. Solid-state NMR reveals structural differences between fibrils of wild-type and disease-related A53T mutant α-synuclein [J]. J Mol Biol, 2008, 380(3): 444-450. |
| [39] | Lemkau L R, Comellas G, Kloepper K D, et al. Mutant protein A30P α-synuclein adopts wild-type fibril structure, despite slower fibrillation kinetics[J]. J Biol Chem, 2012, 287(14): 11526-11532. |
| [40] | Rybicki B A, Johnson C C, Uman J, et al. Parkinson's disease mortality and the industrial use of heavy metals in Michigan[J]. Movement Disorders, 1993, 8(1): 87-92. |
| [41] | Dexter D T, Carayon A, Javoy-Agid F, et al. Alterations in the levels of iron, ferritin and other trace metals in Parkinson's disease and other neurodegenerative diseases affecting the basal ganglia[J]. Brain, 1991, 114(4): 1953-1975. |
| [42] | Hirsch E, Brandel J P, Galle P, et al. Iron and aluminum increase in the substantia nigra of patients with parkinson's disease: An X-ray microanalysis[J]. J Neurochem, 1991, 56(2): 446-451. |
| [43] | Uversky V N, Li J, Fink A L. Metal-triggered structural transformations, aggregation, and fibrillation of human α-synuclein: A possible molecular link between parkinson′s disease and heavy metal exposure[J]. J Biol Chem, 2001, 276(47): 44284-44296. |
| [44] | Ostrerova-Golts N, Petrucelli L, Hardy J, et al. The A53T α-synuclein mutation increases iron-dependent aggregation and toxicity[J]. J Neurosci, 2000, 20(16): 6048-6054. |
| [45] | Paik S R, Shin H J, Lee J H. Metal-catalyzed oxidation of α-synuclein in the presence of copper (II) and hydrogen peroxide[J]. Arch Biochem Biophys, 2000, 378(2): 269-277. |
| [46] | Paik S R, Lee J H, Kim D H, et al. Aluminum-induced structural alterations of the precursor of the non-Ab component of alzheimer's disease amyloid[J]. Arch Biochem Biophys, 1997, 344(2): 325-334. |
| [47] | Hettiarachchi N T, Parker A, Dallas M L, et al. α-synuclein modulation of Ca2+ signaling in human neuroblastoma (SH-SY5Y) cells[J]. J Neurochem, 2009, 111(5): 1192-1201. |
| [48] | Mosharov E V, Larsen K E, Kanter E, et al. Interplay between cytosolic dopamine, calcium, and α-synuclein causes selective death of substantia nigra neurons[J]. Neuron, 2009, 62(2): 218-229. |
| [49] | Zhang Z, Dai C, Bai J, et al. Ca2+ modulating α-synuclein membrane transient interactions revealed by solution NMR spectroscopy[J]. BBA-Biomembranes, 2014, 1838(3): 853-858. |
| [50] | Tamamizu-Kato S, Kosaraju M G, Kato H, et al. Calcium-triggered membrane interaction of the α-synuclein acidic tail[J]. Biochemistry, 2006, 45(36): 10947-10956. |
| [51] | Lowe R, Pountney D L, Jensen P H, et al. Calcium(II) selectively induces α-synuclein annular oligomers via interaction with the C-terminal domain[J]. Protein Sci, 2004, 13(12): 3245-3252. |
| [52] | Nath S, Goodwin J, Engelborghs Y, et al. Raised calcium promotes α-synuclein aggregate formation[J]. Mol Cell Neurosci, 2011, 46(2): 516-526. |
| [53] | Pountney D L, Lowe R, Quilty M, et al. Annular a-synuclein species from purified multiple system atrophy inclusions[J]. J Neurochem, 2004, 90(2): 502-512. |
| [54] | Lee J C, Gray H B, Winkler J R. Copper(II) binding to α-synuclein, the parkinson's protein[J]. J Am Chem Soc, 2008, 130(22): 6898-6899. |
| [55] | Binolfi A, Valiente-Gabioud A A, Duran R, et al. Exploring the structural details of Cu(I) binding to α-synuclein by NMR spectroscopy[J]. J Am Chem Soc, 2010, 133(2): 194-196. |
| [56] | Sandal M, Valle F, Tessari I, et al. Conformational equilibria in monomeric α-synuclein at the single-molecule level[J]. PLoS Biol, 2008, 6(1): e6. |
| [57] | Dudzik C G, Walter E D, Millhauser G L. Coordination features and affinity of the Cu2+ site in the α-synuclein protein of Parkinson’s disease[J]. Biochemistry, 2011, 50(11): 1771-1777. |
| [58] | Sung Y H, Rospigliosi C, Eliezer D. NMR mapping of copper binding sites in α-synuclein[J]. BBA-Proteins Proteom, 2006, 1764(1): 5-12. |
| [59] | Lucas H R, Debeer S, Hong M S, et al. Evidence for copper-dioxygen reactivity during α-synuclein fibril formation[J]. J Am Chem Soc, 2010, 132(19): 6636-6637. |
| [60] | Davies P, Moualla D, Brown D R. α-synuclein is a cellular ferrireductase[J]. PLoS ONE, 2011, 6(1): e15814. |
| [61] | Kostka M, Högen T, Danzer K M, et al. Single particle characterization of iron-induced pore-forming α-synuclein oligomers[J]. J Biol Chem, 2008, 283(16): 10992-11003. |
| [62] | André C, Truong T T, Robert J F, et al. Effect of metals on herbicides-α-synuclein association: A possible factor in neurodegenerative disease studied by capillary electrophoresis[J]. Electrophoresis, 2005, 26(17): 3256-3264. |
| [63] | Binolfi A, Rasia R M, Bertoncini C W, et al. Interaction of α-synuclein with divalent metal ions reveals key differences: A link between structure, binding specificity and fibrillation enhancement[J]. J Am Chem Soc, 2006, 128(30): 9893-9901. |
| [64] | Jin J, Li G J, Davis J, et al. Identification of novel proteins associated with both α-synuclein and DJ-1[J]. Mol Cell Proteomics, 2007, 6(5): 845-859. |
| [65] | Uversky V N, Li J, Souillac P, et al. Biophysical properties of the synucleins and their propensities to fibrillate: Inhibition of α-synuclein assembly by b- and g-synucleins[J]. J Biol Chem, 2002, 277(14): 11970-11978. |
| [66] | Israeli E, Sharon R. b-Synuclein occurs in vivo in lipid-associated oligomers and forms hetero-oligomers with a-synuclein[J]. J Neurochem, 2009, 108(2): 465-474. |
| [67] | Shashidharan P, Kramer B C, Walker R H, et al. Immunohistochemical localization and distribution of torsinA in normal human and rat brain[J]. Brain Res, 2000, 853(2): 197-206. |
| [68] | Mclean P J, Kawamata H, Shariff S, et al. Torsina and heat shock proteins act as molecular chaperones: Suppression of α-synuclein aggregation[J]. J Neurochem, 2002, 83(4): 846-854. |
| [69] | Lo Bianco C, Shorter J, Xe, et al. Hsp104 antagonizes α-synuclein aggregation and reduces dopaminergic degeneration in a rat model of Parkinson disease[J]. J Clin Invest, 2008, 118(9): 3087-3097. |
| [70] | Klucken J, Shin Y, Masliah E, et al. Hsp70 Reduces α-synuclein aggregation and toxicity[J]. J Biol Chem, 2004, 279(24): 25497-25502. |
| [71] | Luk K C, Mills I P, Trojanowski J Q, et al. Interactions between Hsp70 and the hydrophobic core of α-synuclein inhibit fibril assembly[J]. Biochemistry, 2008, 47(47): 12614-12625. |
| [72] | Morales R, Estrada L D, Diaz-Espinoza R, et al. Molecular cross talk between misfolded proteins in animal models of Alzheimer's and Prion diseases[J]. J Neurosci, 2010, 30(13): 4528-4535. |
| [73] | Waxman E A, Giasson B I. Induction of intracellular tau aggregation is promoted by α-synuclein seeds and provides novel insights into the hyperphosphorylation of tau[J]. J Neurosci, 2011, 31(21): 7604-7618. |
| [74] | Engelender S, Kaminsky Z, Guo X, et al. Synphilin-1 associates with α-synuclein and promotes the formation of cytosolic inclusions[J]. Nat Genet, 1999, 22(1): 110-114. |
| [75] | Büttne S, Delay C, Franssens V, et al. Synphilin-1 enhances α-synuclein aggregation in yeast and contributes to cellular stress and cell death in a Sir2-dependent manner[J]. 2010, 5(10): e13700. |
| [76] | Xie Y Y, Zhou C J, Zhou Z R, et al. Interaction with synphilin-1 promotes inclusion formation of α-synuclein: mechanistic insights and pathological implication[J]. FASEB J, 2010, 24(1): 196-205. |
| [77] | Necula M, Chirita C N, Kuret J. Rapid anionic micelle-mediated α-synuclein fibrillization in vitro[J]. J Biol Chem, 2003, 278(47): 46674-46680. |
| [78] | Zhu M, Fink A L. Lipid binding inhibits α-synuclein fibril formation[J]. J Biol Chem, 2003, 278(19): 16873-16877. |
| [79] | Wang G F, Li C, Pielak G J. Probing the micelle-bound aggregation-prone state of α-synuclein with 19F NMR spectroscopy[J]. Chem Bio Chem, 2010, 11(14): 1993-1996. |
| [80] | Ahmad M F, Ramakrishna T, Raman B, et al. Fibrillogenic and non-fibrillogenic ensembles of SDS-bound human α-synuclein[J]. J Mol Biol, 2006, 364(5): 1061-1072 |
 2016, Vol. 33
2016, Vol. 33