文章信息
- 刘婷, 刘买利, 姜凌
- LIU Ting, LIU Mai-li, JIANG Ling
- 二价金属离子与YycFN相互作用的NMR研究
- Divalent Metal Ion Binding to the Response Regulator YycFN Studied by NMR Spectroscopy
- 波谱学杂志, 2016, 33(1): 77-88
- Chinese Journal of Magnetic Resonance, 2016, 33(1): 77-88
- http://dx.doi.org/10.11938/cjmr20160107
-
文章历史
- 收稿日期: 2015-03-30
- 收修改稿日期: 2016-01-22
2. 中国科学院大学, 北京 10049
2. University of Chinese Academy of Sciences, Beijing 100049, China
双组分信号转导系统(Two-Component signal transduction System,TCS)普遍存在于原核生物中,真核生物如酵母和高等植物中也有少量存在.它是细胞感受外界环境变化和调控内部相关基因表达的“分子开关”[1],在细胞适应不同外部环境、新陈代谢和致病机制等方面起重要作用.TCS主要包括膜相关组氨酸激酶(histidine kinase,HK)和反应调节蛋白(response regulator,RR):HK蛋白受激后自磷酸化,将磷酸根传递给RR蛋白;RR蛋白活化后改变构象继而调控基因表达,以实现特定的细胞应答[2].TCS由信号输入(input)、HK自磷酸化活化、RR磷酸化活化以及信号输出(output)等多个环节构成[3].RR蛋白的空间结构非常保守,一般形成(βα)5折叠桶形结构,活性位点附近的氨基酸序列十分相似[图 1(a)].研究发现,二价金属离子在反应调节蛋白的磷酸化反应过程中起到非常关键的作用.无论是自由态还是磷酸化态,金属离子与RR蛋白主要以1:1配位,而且配位模式一致.如图 1(b)所示,Mg2+的六个配位轨道分别被三个保守的天冬氨酸(Asp)残基(其中一个通过水分子配位)、一个来自主链的羰基基团和两个水分子占据.由于Mg2+的部分配位轨道是由水分子占据的,因此结合位点可以适应不同半径的金属离子,不受空间位阻的限制[4].对于大部分反应调节蛋白的来说,最理想的金属离子参与者为Mg2+,但是其他金属离子(如Ca2+、Mn2+)也可以与其发生相互作用[5, 6].目前金属离子与RR蛋白相互作用的动力学研究较少,解离常数Kd范围还不确定.一些典型的RR蛋白,如CheY与Mg2+的Kd值为0.5~1.0 mmol/L(推测CheY在细胞内主要以离子结合态存在)[7];而Spo0F与Mg2+的Kd值为20±5 mmol/L,与Ca2+的Kd值为3.5 mmol/L.它们的结合强度可能与蛋白磷酸化效率有关[8].

|
| 图 1 不同菌株反应调节蛋白N端调节域的氨基酸序列和金属配位模式.(a)蛋白序列比对图(CLUSTALW程序[9]),YycFN的二级结构在序列上方显示,“*”代表完全相同的氨基酸,用深灰色显示,“:”和“.”代表高度保守和保守的氨基酸,用浅灰色显示;(b)反应调节蛋白与Mg2+的配位模式示意图[4] Fig. 1 Amino acid sequences of response regulators from different species.(a)Sequence alignment was achieved by the program CLUSTALW[9] and manually adjusted based on structural alignments. The secondary structural elements of YycFN are shown above the sequences. Identical residues are highlighted in dark gray boxes,while residues with high similarity as defined by CLUSTALW are highlighted in gray boxes.(b)Coordination geometry scheme of Mg2+ with response regulator[4] |
YycGF是与细胞存活密切相关的TCS之一,最早发现于枯草芽孢杆菌(Bacillus subtilis)[10],随后通过序列同源性查找,在葡萄球菌[11]、链球菌[12]和肠球菌[13]等低鸟嘌呤和胞嘧啶(G+C)含量的革兰氏阳性菌中被发现,这其中包括重要的人类致病菌,例如金黄色葡萄球菌(Staphylococcus aureus)[11]和肺炎链球菌(Streptococcus pneumoniae)[12]等.YycGF参与肽聚糖合成和分解肽酶类自溶素、胆碱结合蛋白和一些水解酶的基因表达[14],在细胞壁代谢和细胞分裂中起关键调节作用[15, 16].该系统异常会直接导致细菌的生存力严重下降,甚至死亡,因此成为防治该类病原菌的重要药物靶标[17, 18].在YycGF系统中,YycG和YycF分别行使HK和RR蛋白功能.YycF属于OmpR/PhoB家族[15],它包括N端调节域和C端效应域,N端调节域从YycG接受磷酸根,活化后构象改变,并促使C端效应域与DNA相结合以调控基因表达[19, 20].YycF的N端调节域(YycFN)由120个氨基酸组成,专一性接受来自磷酸化YycG上的磷酸根,转移至保守的Asp53残基上,引发自身的变构活化.YycF也可以非特异性地被磷酸铵、乙酰磷酸和磷酸酯等小分子磷酸化[21].来源于链球菌和枯草芽孢杆菌的YycFN晶体结构分别于2004和2009年被解析[22, 23].但已知的YycFN是自由态晶体结构,不含磷酸根和金属离子,YycFN与金属离子的相互作用研究尚未见报道,因此研究金属离子与YycFN的相互作用具有重要的生物学意义.
基于核磁共振技术(NMR)的化学位移微扰法是研究小分子与蛋白质相互作用的重要手段[24, 25, 26, 27, 28, 29].相互作用会导致蛋白质结合区域的残基化学位移发生变化,整体构象变化会导致非结合区域的残基化学位移产生移动[30].本文用两种不同的金属离子(Mg2+和Ca2+)分别对YycFN进行滴定,发现金属离子对其构象改变有非常明显的作用.文中用化学位移扰动的方法确定了相互作用的区域,并计算了YycFN与Mg2+、Ca2+的解离常数.研究结果对阐明二价金属离子在YycFN信号传导过程中所起的作用提供了重要线索,并为研究其他反应调节蛋白的磷酸化机制提供了参考数据.
1 实验部分 1.1 样品准备
YycFN质粒(1~120)的构建与蛋白表达纯化方法在文献[23]基础上进行了优化:将含有YycFN质粒的LB菌液37℃过夜培养后,接种至M9培养基中;待OD600值达到0.8时加入终浓度为1 mmol/L的异丙基-β-D-硫代吡喃半乳糖苷(Isopropyl β-D-1-Thiogalactopyranoside,IPTG);然后低温、16℃过夜表达约16 h,离心收集菌体.
蛋白纯化所用的层析柱均购自GE Healthcare公司.将菌体用缓冲液Buffer A(含20 mmol/L Tris-HCl、1 mmol/L EDTA和1 mmol/L DTT,pH 7.5)重悬;超声破碎菌液后高速离心取上清液,载入用Buffer A平衡的HiTrap QFF柱;用Buffer B(Buffer A中加入1 mol/L NaCl)梯度洗脱;SDS-PAGE鉴定在50% Buffer B洗脱液中含有较高纯度目标蛋白;合并浓缩目标蛋白进一步用凝胶柱Sephacryl S-100纯化,缓冲液为Buffer C(含20 mmol/L Tris、1 mmol/L EDTA、1 mmol/L DTT和250 mmol/L NaCl,pH 7.5);将纯化得到的样品通过脱盐柱(desalting column)置换成缓冲液Buffer D(含20 mmol/L Tris-HCl、1 mmol/L EDTA和1 mmol/L DTT,pH 6.5),并载入Source Q柱;用Buffer E(Buffer D中加入1 mol/L NaCl)梯度洗脱;收集目标蛋白,经SDS-PAGE鉴定纯度达95%以上;最后,用脱盐柱将缓冲液置换为无菌双蒸水,超滤浓缩;分装冻干后置于 -80 ℃冰箱保存.纯化后的蛋白用质谱验证分子量为13 768.7,和计算分子量一致.15N标记样品和15N、13C双标记样品通过加入15NH4Cl或13C-glucose同位素来源的M9培养基获得,纯化步骤同上.同位素样品购自Cambridge Isotope Laboratories公司.
NMR实验样品浓度均为0.6~0.8 mmol/L.样品缓冲溶液含20 mmol/L HEPES(pH 7.0)、1 mmol/L DTT和10% D2O.
1.2 NMR实验和数据处理NMR实验均在Bruker AvanceⅢ 600和800 MHz核磁共振谱仪上完成,仪器配有Z-梯度场的三共振超低温探头,实验温度为310 K.NMR数据都用NMRpipe/NMRDraw软件包[31]处理,数据处理后谱图用软件CARA进行分析.
1.2.1 蛋白质主链原子归属为归属酰胺质子1HN和酰胺N的化学位移,用15N和13C双标记样品采集了HNCO、HN(CO)CA、HNCA、CBCA(CO)NH和HNCACB等三共振实验进行主链归属.直接检测维先采用去溶剂峰处理,之后与间接维相同:依次进行基线校正、加正弦窗函数、线性预测、充零、傅里叶变换和相位校正等处理.
1.2.2 化学位移微扰实验向浓度均为0.6 mmol/L的15N标记的YycFN蛋白样品中逐步滴入MgCl2或CaCl2溶液,金属离子浓度范围各为:Ca2+ 0~20 mmol/L;Mg2+ 0~20 mmol/L.滴定过程中通过1H-15N HSQC谱监测酰胺基团的化学位移变化.1H-15N HSQC F2维(1H)和F1维(15N)谱宽分别为8 064.5和1 986.8 Hz,数据采集点数t2×t1 = 2 048×256.对1H-15N HSQC谱常用加权平均的1H/15N化学位移来描述酰胺基团的化学位移扰动,由(1)式[32]计算得到:
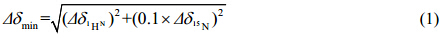
为了定量分析相互作用的结合程度,我们通过化学位移的变化计算了平衡解离常数Kd.由于反应调节蛋白和金属离子主要以1:1配位[5, 8, 33],我们以单配位模式进行数据拟合.对于可逆反应,

YycFN是指自由态蛋白,M2+是指二价金属离子,则解离常数Kd定义为:

一般认为,蛋白质与配体之间的特异性结合符合化学计量关系.滴定过程中,结合位点处或发生构象变化部位的核自旋化学位移发生了变化.由于在滴定过程中只观察到一组共振峰,说明两者的相互作用是快交换过程[34].对于一个观察核,所观察到的化学位移是自由态和结合态两种状态下化学位移值的平均值:

Xb、Xf分别是指结合态、自由态在总态中所占的摩尔分数,${{\delta }_{\text{b}}}$、${{\delta }_{\text{f}}}$和${{\delta }_{\text{obs}}}$分别是结合态、自由态及观察到的化学位移值.
同时,由质量作用定律,我们有:
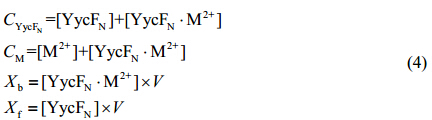
${{C}_{\text{Yyc}{{\text{F}}_{\text{N}}}}}$和CM分别是YycFN蛋白和二价金属离子的总浓度,$\text{ }\!\![\!\!\text{ Yyc}{{\text{F}}_{\text{N}}}\text{ }\!\!]\!\!\text{ }$和$\text{ }\!\![\!\!\text{ }{{\text{M}}^{\text{2+}}}\text{ }\!\!]\!\!\text{ }$分别是YycFN和二价金属离子在自由态下的平衡浓度,$\text{ }\!\![\!\!\text{ Yyc}{{\text{F}}_{\text{N}}}\cdot {{\text{M}}^{\text{2+}}}\text{ }\!\!]\!\!\text{ }$是指YycFN与二价金属离子所形成的配合物复合体的平衡浓度,V指样品溶液的体积.由(2)和(4)式得出:
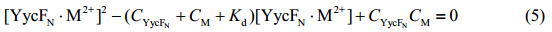
结合(3)和(5)式,可得出最终我们观察到的化学位移${{\delta }_{\text{obs}}}$与二价金属离子配体浓度CM之间的关系式:
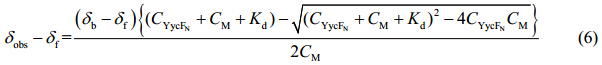
${{C}_{\text{Yyc}{{\text{F}}_{\text{N}}}}}$及${{\delta }_{\text{f}}}$均已知,按(6)式以${{\delta }_{\text{obs}}}$对CM进行拟合,利用模拟回归可算出Kd和${{\delta }_{\text{b}}}$[35].
2 结果与讨论 2.1 YycFN主链NMR信号归属
YycFN含有120个氨基酸,从二维1H-15N HSQC谱上可以看出,该蛋白主链酰胺基团的谱峰分散性较好,蛋白形成了较稳定的空间构象.通过HNCO、HN(CO)CA、HNCA、CBCA(CO)NH和HNCACB等三共振实验对蛋白进行了主链归属,除了1、54、55、56、58、59、60和106几个残基外,其他所有非脯氨酸(Pro)的主链酰胺基团的信号都可以在二维1H-15N HSQC谱上得到很好的辨认[图 2(a)].未归属的残基大都位于磷酸根结合位点Asp53附近,它们在自由态YycFN蛋白的晶体结构中处于loop区域,构象不稳定,可能造成信号展宽而没有被观察到.

|
| 图 2 YycFN(a)与YycFN-Mg2+(b)的二维1H-15N HSQC谱. 图中氨基酸残基均用单字母及其序号表示 Fig. 2 1H-15N HSQC spectra of response regulator YycFN(a) and YycFN with Mg2+(b),Assigned residues are labeled by the 1-letter amino acid codes with their sequential numbers |
我们选取了两种不同的二价金属离子对YycFN进行滴定,发现Mg2+和Ca2+离子的加入导致YycFN主链信号产生了非常明显的化学位移扰动,且滴定过程在NMR时间尺度上表现为快交换过程.由于信号重叠,无法分析离子加入导致YycFN信号化学位移发生变化的轨迹.为了确定相互作用界面,我们对YycFN与Mg2+的复合物也采集了三共振实验,并完成了主链信号归属[图 2(b)].复合物的归属结果与YycFN类似,93%的信号得以确认,而在磷酸化位点附近的多个氨基酸没有获得归属.
如图 3所示,Mg2+和Ca2+的加入引起了1H-15N HSQC谱图非常明显的改变.两者的化学位移变化非常相似[图 3(c)],而Ca2+滴定的谱图比Mg2+滴定的谱图信号强度更加均一,预示了该空间构象更加稳定.
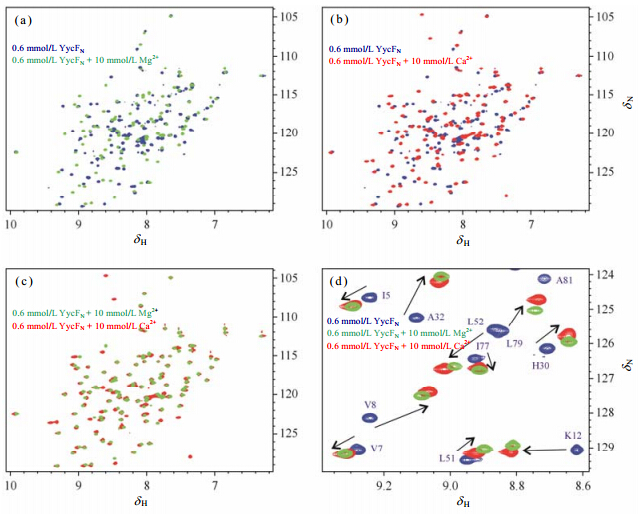
|
| 图 3 不同金属离子滴定YycFN的1H-15N HSQC谱图.(a)自由态YycFN(蓝色)和Mg2+结合YycFN蛋白(绿色);(b)自由态YycFN(蓝色)和Ca2+结合YycFN蛋白(红色);(c)Mg2+和Ca2+结合YycFN蛋白的叠加谱图;(d)滴定前后的局部叠加谱图 Fig. 3 1H-15N HSQC spectra of YycFN in complex with different metal ions.(a)free YycFN(blue) and YycFN-Mg2+(green),(b)free YycFN(blue) and YycFN-Ca2+(red),(c)overlayed spectra of Mg2+ and Ca2+ binding protein,(d)exp and ed and overlayed spectra |
图 4的柱状图显示了根据(1)式计算出的化学位移变化与氨基酸序号的对应关系.将$\Delta {{\delta }_{\text{min}}}>0.20$的残基用红色或蓝色小球在YycFN晶体结构(PDB:3F6P)上标注出来(图 4),我们发现受Mg2+和Ca2+影响比较明显的残基主要集中在Asp9和Asp53这些保守位点附近.在晶体结构中位于b1片段的Asp9、Asp10与位于b3片段的磷酸化位点Asp53构成了负电荷的“酸性口袋”[36],容易与带正电荷的金属离子结合,这与其他RR蛋白的金属离子结合位点是十分一致的[37].除该结合位点外,金属离子的加入还使蛋白的整体构象发生了变化.Ala15、Asp16、Leu18、Arg23位于a1螺旋沿线,除Ala15外,均远离“酸性口袋”,但它们的化学位移仍然受到了较大影响,说明Mg2+和Ca2+的结合改变了蛋白a1螺旋的轴向,使之更利于金属离子的结合.图 4(a)中Mg2+结合还造成了His33和Gly61的显著位移,这两个位点距离Asp9和Asp53较近,产生变化在预料之中;Ca2+的结合也引起了这两个位点附近的变化,但幅度较小.Thr80则正好相反,它在Mg2+滴定中引起的化学位移变化没有Ca2+明显.Thr80位于b4a5的loop区,对其他RR蛋白的研究证实这一位点非常保守[38],可以与磷酸基团配位,参与信号转导.Tyr99是一个中等保守的残基,位于b5片段的中间[39],空间上与Thr80相近,也是与信号转导密切相关的位点.另一个与磷酸化相关的保守位点是Lys102,虽然没有在晶体结构中被标注,但其化学位移变化也比较明显.位于序列末端的Arg118化学位移变化也非常明显,可能源于溶液离子强度的变化导致侧链基团与水的交换作用产生了变化.

|
| 图 4 Mg2+、Ca2+滴定引起的YycFN化学位移变化,横坐标代表氨基酸序列,纵坐标代表(a)Mg2+(b)Ca2+滴定前后化学位移值的变化.Δδmin > 0.20的残基用红色或蓝色小球标注在YycFN的晶体结构(PDB:3F6P)上. 图中氨基酸残基均用单字母及其序号表示 Fig. 4 Chemical shift perturbations of YycFN caused by titration of Mg2+ and Ca2+. The x-axis represents the amino acid number,while the y-axis represents the chemical shift perturbations of YycFN after(a)Mg2+ and (b)Ca2+ binding. The residues with Δδmin > 0.20 were plotted on the ribbon structure of YycFN(PDB:3F6P)with red or blue balls. The residues are labeled by single letters with their sequential numbers |
根据化学位移变化可以计算Ca2+、Mg2+与YycFN相互作用的Kd值.我们选取了几个变化比较明显的残基如Val8、Ile17、Cys31、Ala32、His33、Glu37和Thr80,计算得到各残基与Ca2+和Mg2+的Kd值.虽然通过NMR滴定实验计算得到的Kd值包含一定误差,但从表 1可以看出,各残基与Ca2+和Mg2+的Kd值均为毫摩(mmol/L)级别,说明结合反应属于典型的弱结合作用.对Mg2+而言,Kd值在2.0~3.5 mmol/L范围内,而Ca2+滴定的Kd值为0.5~1.0 mmol/L,比Mg2+滴定小3~5倍,由此进一步推测Ca2+与蛋白结合力比Mg2+更强(图 5).它们的结合位点都是位于酸性口袋及b4a4界面,对阐明二价金属离子在RR蛋白信号转导过程中所起的作用机制提供了重要的线索.
| Residue No. | V8 | I17 | C31 | A32 | H33 | E37 | T80 |
| Kd(Mg2+)/(mmol/L) | 2.53 | 2.88 | 2.65 | 3.23 | 2.09 | 2.00 | 2.74 |
| Kd(Ca2+)/(mmol/L) | 0.57 | 0.85 | 0.84 | 0.71 | 0.55 | 0.63 | 0.48 |
| *Mg2+滴定拟合计算Kd时标准偏差约为±0.10(mmol/L),Ca2+滴定拟合计算Kd时标准偏差约为±0.50(mmol/L).表中氨基酸残基均用单字母及其序号表示 | |||||||

|
| 图 5 Mg2+(a)、Ca2+(b)滴定YycFN的化学位移变化和金属离子配体浓度CM拟合曲线. 图中氨基酸残基均用单字母及其序号表示 Fig. 5 Chemical shift perturbations and CM fitting curves for YycFN after the binding of(a)Mg2+ and (b)Ca2+. The residues are labeled by single letters with their sequential numbers |
我们采用基于NMR技术的化学位移微扰法研究了不同金属离子与YycFN的相互作用,确定了相互作用的位点和解离常数.研究发现,Mg2+和Ca2+与YycFN的相互作用区域相同,但Ca2+与之结合得更加紧密,体现在1H-15N HSQC谱图的化学位移扰动相似、Ca2+滴定的谱图信号强度比Mg2+更加均一以及更小的Kd值.Mg2+、Ca2+的加入造成了YycFN信号发生移动,主要集中于a1b2片段的酸性口袋及其附近区域,并同时影响了b4a4界面上与磷酸化信号转导密切相关的Thr80、Tyr99和Lys102位点.因此,从整体来看Mg2+、Ca2+引起的蛋白构象转变是一致的,而Ca2+-YycFN复合物相较于Mg2+-YycFN而言,对潜在的磷酸化位点的局部构象产生了更为显著的影响.
实验证明YycFN蛋白对二价金属离子的尺寸大小并没有很高的选择性.这是由于二价金属离子与反应调节区蛋白的配位区域是有水分子占据的,结合位点可以适应不同半径的金属离子,不受空间位阻的限制.由于细胞中存在一定浓度的游离二价金属离子,比如大肠杆菌的细胞质中Mg2+的浓度为0.5~1.0 mmol/L,而YycFN与Mg2+的Kd值为2.0~3.5 mmol/L,在细胞中是以金属离子结合态和自由态两种构象共存的.研究结果为探索不同金属离子对RR蛋白N端调节域的相互作用提供了原子水平的基础数据.
| [1] | Stock A M, Robinson V L, Goudreau P N. Two-component signal transduction[J]. Annu Rev Biochem, 2000, 69: 183-215. |
| [2] | Galperin M Y. Structural classification of bacterial response regulators: Diversity of output domains and domain combinations[J]. J Bacteriol, 2006, 188(12): 4 169-4 182. |
| [3] | Fabret C, Feher V A, Hoch J A. Two-component signal transduction in Bacillus subtilis: How one organism sees its world[J]. J Bacteriol, 1999, 181(7): 1 975-1 983. |
| [4] | Bourret R B. Receiver domain structure and function in response regulator proteins[J]. Curr Opin Microbiol, 2010, 13(2): 142-149. |
| [5] | Lukat G S, Stock A M, Stock J B. Divalent metal ion binding to the CheY protein and its significance to phosphotransfer in bacterial chemotaxis[J]. Biochemistry, 1990, 29(23): 5 436-5 442. |
| [6] | Needham J V, Chen T Y, Falke J J. Novel ion specificity of a carboxylate cluster Mg(II) binding site: strong charge selectivity and weak size selectivity[J]. Biochemistry, 1993, 32(13): 3 363-3 367. |
| [7] | Hubbard J A, MacLachlan L K, King G W, et al. Nuclear magnetic resonance spectroscopy reveals the functional state of the signalling protein CheY in vivo in Escherichia coli[J]. Mol Microbiol, 2003, 49(5): 1 191-1 200. |
| [8] | Kojetin D J, Thompson R J, Benson L M, et al. Structural analysis of divalent metals binding to the Bacillus subtilis response regulator Spo0F: the possibility for in vitro metalloregulation in the initiation of sporulation[J]. Biometals, 2005, 18(5): 449-466. |
| [9] | Thompson J D, Higgins D G, Gibson T J. Clustal W: Improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice[J]. Nucleic Acids Res, 1994, 22(22): 4 673-4 680. |
| [10] | Bisicchia P, Noone D, Lioliou E, et al. The essential YycFG two-component system controls cell wall metabolism in Bacillus subtilis[J]. Mol Microbiol, 2007, 65(1): 180-200. |
| [11] | Fukushima T, Szurmant H, Kim E J, et al. A sensor histidine kinase co-ordinates cell wall architecture with cell division in Bacillus subtilis[J]. Mol Microbiol, 2008, 69(3): 621-632. |
| [12] | Ahn S J, Burne R A. Effects of oxygen on biofilm formation and the AtlA autolysin of Streptococcus mutans[J]. J Bacteriol, 2007, 189(17): 6 293-6 302. |
| [13] | Hancock L, Perego M. Two-component signal transduction in Enterococcus faecalis[J]. J Bacteriol, 2002, 184(21): 5 819-5 825. |
| [14] | Dubrac S, Boneca I G, Poupel O, et al. New insights into the WalK/WalR (YycG/YycF) essential signal transduction pathway reveal a major role in controlling cell wall metabolism and biofilm formation in Staphylococcus aureus[J]. J Bacteriol, 2007, 189(22): 8 257-8 269. |
| [15] | Dubrac S, Msadek T. Identification of genes controlled by the essential YycG/YycF two-component system of Staphylococcus aureus[J]. J Bacteriol, 2004, 186(4): 1 175-1 181. |
| [16] | Howell A, Dubrac S, Andersen K K, et al. Genes controlled by the essential YycG/YycF two-component system of Bacillus subtilis revealed through a novel hybrid regulator approach[J]. Mol Microbiol, 2003, 49(6): 1 639-1 655. |
| [17] | Watanabe T, Okada A, Gotoh Y, et al. Inhibitors targeting two-component signal transduction[J]. Adv Exp Med Biol, 2008, 631: 229-236. |
| [18] | Qin Z Q, Zhang J, Xu B, et al. Structure-based discovery of inhibitors of the YycG histidine kinase: New chemical leads to combat Staphylococcus epidermidis infections[J]. BMC Microbiol, 2006, 6: 96-113. |
| [19] | Schnell R, Agren D, Schneider G. 1.9 Å structure of the signal receiver domain of the putative response regulator NarL from Mycobacterium tuberculosis[J]. Acta Crystallogr F, 2008, 64(12): 1 096-1 100. |
| [20] | Gao R, Mack T R, Stock A M. Bacterial response regulators: Versatile regulatory strategies from common domains[J]. Trends Biochem Sci, 2007, 32(5): 225-234. |
| [21] | Lukat G S, McCleary W R, Stock A M, et al. Phosphorylation of bacterial response regulator proteins by low molecular weight phospho-donors[J]. Proc Natl Acad Sci U S A, 1992, 89(2): 718-722. |
| [22] | Bent C J, Isaacs N W, Mitchell T J, et al. Crystal structure of the response regulator 02 receiver domain, the essential YycF two-component system of Streptococcus pneumoniae in both complexed and native states[J]. J Bacteriol, 2004, 186(9): 2 872-2 879. |
| [23] | Zhao H, Heroux A, Sequeira R D, et al. Preliminary crystallographic studies of the regulatory domain of response regulator YycF from an essential two-component signal transduction system[J]. Acta Crystallogr F, 2009, 65(7): 719-722. |
| [24] | Pellecchia M, Montgomery D L, Stevens S Y, et al. Structural insights into substrate binding by the molecular chaperone DnaK[J]. Nat Struct Biol, 2000, 7(4): 298-303. |
| [25] | Stevens S Y, Sanker S, Kent C, et al. Delineation of the allosteric mechanism of a cytidylyltransferase exhibiting negative cooperativity[J]. Nat Struct Biol, 2001, 8(11): 947-952. |
| [26] | Zuiderweg E R. Mapping protein-protein interactions in solution by NMR spectroscopy[J]. Biochemistry, 2002, 41(1): 1-7. |
| [27] | Chang D K, Chien W J, Arunkumar A I. Conformation of a protein kinase C substrate NG(28-43), and its analog in aqueous and sodium dodecyl sulfate micelle solutions[J]. Biophys J, 1997, 72(2 Pt 1): 554-566. |
| [28] | Chen A, Shapiro M J. Affinity NMR[J]. Anal Chem, 1999, 71(19): 669A-675A. |
| [29] | Shuker S B, Hajduk P J, Meadows R P, et al. Discovering high-affinity ligands for proteins: SAR by NMR[J]. Science, 1996, 274(5 292): 1 531-1 534. |
| [30] | van Nuland N A, Kroon G J, Dijkstra K, et al. The NMR determination of the IIA(mtl) binding site on HPr of the Escherichia coli phosphoenol pyruvate-dependent phosphotransferase system[J]. FEBS Lett, 1993, 315(1): 11-15. |
| [31] | Delaglio F, Grzesiek S, Vuister G W, et al. NMRPipe: A multidimensional spectral processing system based on UNIX pipes[J]. J Biomol NMR, 1995, 6(3): 277-293. |
| [32] | Farmer B T, 2nd, Constantine K L, Goldfarb V, et al. Localizing the NADP+ binding site on the MurB enzyme by NMR[J]. Nat Structl Biol, 1996, 3(12): 995-997. |
| [33] | Stock A M, Martinez-Hackert E, Rasmussen B F, et al. Structure of the Mg(2+)-bound form of CheY and mechanism of phosphoryl transfer in bacterial chemotaxis[J]. Biochemistry, 1993, 32(49): 13 375-13 380. |
| [34] | Clore G M, Gronenborn A M, Birdsall B, et al. 19F-n.m.r. studies of 3',5'-difluoromethotrexate binding to Lactobacillus casei dihydrofolate reductase. Molecular motion and coenzyme-induced conformational changes[J]. Biochem J, 1984, 217(3): 659-666. |
| [35] | Lennon A J, Scott N R, Chapman B E, et al. Hemoglobin affinity for 2,3-bisphosphoglycerate in solutions and intact erythrocytes: Studies using pulsed-field gradient nuclear magnetic resonance and Monte Carlo simulations[J]. Biophys J, 1994, 67(5): 2 096-2 109. |
| [36] | Birck C, Chen Y, Hulett F M, et al. The crystal structure of the phosphorylation domain in PhoP reveals a functional tandem association mediated by an asymmetric interface[J]. J Bacteriol, 2003, 185(1): 254-261. |
| [37] | Davies K M, Lowe E D, Venien-Bryan C, et al. The HupR receiver domain crystal structure in its nonphospho and inhibitory phospho states[J]. J Mol Biol, 2009, 385(1): 51-64. |
| [38] | Appleby J L, Bourret R B. Proposed signal transduction role for conserved CheY residue Thr87, a member of the response regulator active-site quintet[J]. J Bacteriol, 1998, 180(14): 3 563-3 569. |
| [39] | Lukat G S, Lee B H, Mottonen J M, et al. Roles of the highly conserved aspartate and lysine residues in the response regulator of bacterial chemotaxis[J]. J Biol Chem, 1991, 266(13): 8 348-8 354. |
 2016, Vol. 33
2016, Vol. 33




