文章信息
- 李华, Yuji O. KAMATARI, Ryo KITAHARA, Kazuyuki AKASAKA
- LI Hua, Yuji O. KAMATARI, Ryo KITAHARA, Kazuyuki AKASAKA
- 高压NMR在蛋白质结构和动力学研究中的应用
- High-Pressure NMR for Studying Protein Structure and Dynamics
- 波谱学杂志, 2016, 33(1): 1-26
- Chinese Journal of Magnetic Resonance, 2016, 33(1): 1-26
- http://dx.doi.org/10.11938/cjmr20160101
-
文章历史
- 收稿日期: 2015-07-22
- 收修改稿日期: 2016-01-21
2. Life Science Research Center, Gifu University, Gifu 501-1194, Japan;
3. College of Pharmaceutical Sciences, Ritsumeikan University, Kusatsu 525-8577, Japan;
4. Graduate School of Life and Environmental Sciences, Kyoto Prefectural University, Kyoto 606-8522, Japan
2. Life Science Research Center, Gifu University, Gifu 501-1194, Japan;
3. Collage of Pharmaceutical Sciences, Ritsumeikan University, Kusatsu 525-8577, Japan;
4. Graduate School of Life and Environmental Sciences, Kyoto Prefectural University, Kyoto 606-8522, Japan
压力,对于我们来说其实并不陌生,只是生活在常压下的我们时常忽略了压力的存在.高山上,空气稀薄压力低;深海里,海水越深水压越大,但众所周知,高山上和深海里都有生命的存在.蛋白质是生命的物质基础,是生命活动的主要承担者.早在100多年前的1914年,Bridgman[1]就发现在7 000个大气压的压力下,蛋清能完全凝固.此后,人们开始探讨压力导致蛋白质变性之谜.1960年,Suzuki[2]绘制了第一张温度-压力相图.1970年代早期,Brandts[3]、Hawley[4]以及Kauzmann[5]等人使用紫外吸收光谱分别探讨了核糖核酸酶(ribonuclease)A、胰凝乳蛋白酶原(chymotrypsinogen)和正铁肌红蛋白(metmyoglobin)去折叠(unfolding)的温度-压力相图特征.到了1980年代,相继出现了压力振动光谱、高压荧光光谱(fluorescence spectroscopy)、高压X射线小角散射(small-angle X-ray scattering,SAXS)和高压核磁共振波谱法(nuclear magnetic resonance spectroscopy,NMR spectroscopy)等技术.近来,Royer和Winter[6]利用高压荧光光谱、高压傅里叶变换红外光谱(Fourier transform infrared spectroscopy, FTIR spectroscopy)和高压SAXS,而Lassalle等人[7]利用一维核磁共振氢谱(1D 1H NMR)研究了葡萄球菌核酸酶(Staphylococcal nuclease)在温度-压力轴的热力学稳定性,并测定了热力学参数.除了热力学方面的研究之外,在过去的几十年里,利用位置特异性探针如色氨酸(tryptophan,Trp)的高压荧光光谱[8]、高压FTIR[9]、高压NMR[10, 11, 12, 13, 14]等方法对蛋白质结构和动力学进行研究的 例子开始逐渐增多.在高压NMR研究方面,上世纪80年代,Wagner等人[10]利用一维核磁共振氢谱研究了蛋白质内部芳香环基团的翻转运动(flip-flop motion)速率在高压下的变化;Morishima等人[11]则利用一维核磁共振氢谱研究了肌红蛋白(myoglobin)和血红蛋白(hemoglobin)中血红素(heme)周围的结构变化.90年代,继Wagner和Morishima的先驱性工作之后,Jonas等人[15, 16]利用更加耐压的高压装置,大大拓展了高压NMR的研究对象和应用范围,他们的研究涉及蛋白质变性、中间体结构以及低温变性等方面.但由于他们所用的高压装置中核磁信号检测器灵敏度的局限性,使得他们的研究手段仍止步于一维核磁共振氢谱.到了 90年代后期,Akasaka等人研制了独特的耐压样品管,并将其用于蛋白质科学研究中. 这种样品管可直接插入NMR谱仪的探头中而无需对核磁谱仪的硬件进行任何改造,因此,这种加压方式可以充分发挥多维NMR的长处.90年代正值蛋白质同位素标记技术以及多维NMR技术的迅猛发展时期,借助这两项技术,高压NMR研究也得到了长足的发展,研究方法从一维核磁共振氢谱拓展到更加丰富的多维NMR,使得在原子水平详尽地研究蛋白质的结构和动力学的压力效应成为可能[17, 18, 19, 20, 21, 22, 23].
本篇综述首先介绍利用高压NMR技术研究蛋白质结构和动力学的有关概念和高压NMR技术;其次,结合实例,阐述高压NMR在蛋白质结构和动力学研究中的应用;最后,对高压NMR在蛋白质研究中的应用前景进行展望.
本文中所涉及的压力单位及它们之间的换算关系说明如下:压力的国际单位是帕(Pa),常用单位有兆帕(MPa)、千帕(kPa),惯用单位是巴(bar)、标准大气压(atm),它们之间的换算关系是:1 bar =105 Pa= 0.1 MPa = 100 kPa = 0.9869 atm. 1 高压NMR研究中的有关概念
压力是定义蛋白质热力学状态的基本变量.在基本热力学变量中,为人熟知的还有温度.熵是温度的共轭变量,体积是压力的共轭变量.与熵相比,体积与蛋白质结构的关系更加密切.蛋白质在溶液中的有效体积(effective volume)通常用偏摩尔体积(partial molar volume,PMV)来表示.蛋白质的偏摩尔体积由三部分组成,如(1)式所示:

蛋白质在溶液中是多种构象的平衡体,各种构象处于各自相应的能量状态.对于处于不同能量状态的构象,它们的偏摩尔体积也不同,而不同的偏摩尔体积之间存在着体积波动(volume fluctuation).加压时,平衡体系将向蛋白质有效体积变小的方向移动.在蛋白质溶液体系中,主要有两种方式获得较小的有效体积:(a)同种构象群(ensemble)内的压缩(例如同属于折叠构象的构象群内的压缩),以及(b)不同构象亚群(subensemble)之间的平衡移动(图1(a) )[24].
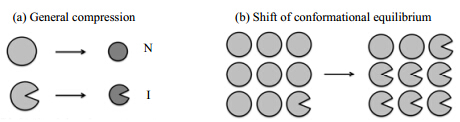
|
| 图 1 蛋白质溶液中加压改变体积的两种主要方式:(a) 在同种构象群内的压缩;(b) 不同构象亚群之间的平衡移动.经许可摘自文献[24],版权2003 American Chemical Society Fig. 1 Schematic representation showing two categories of volume changes by pressure for a protein system in solution: (a) the general compression within a subensemble of a conformer; (b) the shift of conformational equilibrium between different subensembles of conformers characterized with different free energy levels. Adapted with permission from ref. [24]. Copyright 2003 American Chemical Society |
如图1(a) 所示,蛋白质可通过同种构象群内压缩的方式获得较小的有效体积.蛋白质的同种构象群内存在着构象波动;而构象波动可由体积波动来表征.蛋白质体积波动的均方值(mean square)与蛋白质的等温压缩率(isothermal compressibility, βT)密切相关[见(2)式],而βT[定义式如(3)式所示]即等温条件下体积随压力(p)的变化率.βT通常由绝热压缩率(adiabatic compressibility,βS)换算得出[(4)式],而βS则可通过实验获得,即对透过蛋白质溶液的声速及其密度进行测定之后计算获得[(5)式].




等温压缩率和绝热压缩率都是描述溶液中蛋白质的宏观量,而这些宏观压缩率必定源自蛋白质原子间距离或角度等三维结构上的微观变化(相当于微观压缩率).80年代,高压晶体学先于高压NMR在原子水平成功地检测到蛋白质在加压后的结构变化.对溶菌酶(lysozyme)[26]和肌红蛋白[27]的高压晶体学研究发现,高压下蛋白质的压缩是不均一的,局部还有膨胀现象.目前,在高压下如何得到稳定的晶体仍是一个技术难题,但这种情况正在迅速地得到改进[28].后来,高分辨高压NMR技术的开发使得微观压缩率的检测在溶液中成为可能.例如,利用高压NMR技术检测到了化学位移和核奥弗豪塞尔效应(nuclear Overhauser effect,NOE)在加压前后的变化[29, 30],而化学位移和NOE都是与蛋白质结构密切相关的NMR参数.高压NMR的研究结果也表明,蛋白在高压下的压缩是不均一的,甚至伴随着局部的膨胀. 1.2 不同构象亚群之间的平衡移动
蛋白质在溶液中也可通过不同构象亚群之间的平衡移动来获得较小的有效体积[图1(b)].溶液中,蛋白质的不同构象亚群之间处于热力学平衡,这些亚群(例如天然构象亚群N和部分折叠构象亚群I)具有不同的构象、不同的热力学稳定性以(ΔG=GI-GN)及不同的偏摩尔体积(ΔV=VI-VN).在加压的情况下,构象N和I之间的平衡常数K会随压力作如下变化.
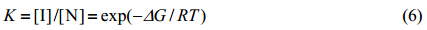
根据对蛋白质样品的加压方式,高压NMR技术可分为两种:一种是耐压容器方式[16],另一种是耐压样品管方式[17].耐压容器方式是指将包括NMR检测线圈在内的整个NMR探头置于高压釜内,然后对整个高压釜进行加压;耐压样品管方式则只将压力加到样品管内,之后将加压中的样品管插入到一般商用的NMR探头内即可,此时NMR探头本身处于常压环境,无需对其进行任何改造和加工.耐压容器方式的优点是耐压性强,工作压力可高达103 MPa左右;缺点是由于需要特制的NMR探头和线圈,因此磁场均一性、检测性能以及所能使用的NMR脉冲程序都受到限制.耐压样品管方式的优点是便捷、灵敏度高.由于只要将耐压样品管插入常规NMR探头即可开始测定,不需要对NMR谱仪以及探头进行任何改造和加工,因此原则上这种方式可适用于任何一台商用NMR谱仪,操作上非常便捷.对NMR谱仪不进行任何改造带来的优势是可以充分享用丰富的二维及多维脉冲程序进行二维或多维高分辨谱图测定.耐压样品管方式的缺点是样品管体积小(内径通常<1 mm)、工作压力较低(几百MPa以内).用于耐压样品管的耐压材料多种多样,包括玻璃、石英[41]、蓝宝石(sapphire)[42, 43]和氧化锆(zirconia)[44, 45],所能达到的压力极限在100~400 MPa之间,其中,应用较多的是石英耐压管.最近,出现了内径较大(3 mm)的氧化锆制耐压管[44, 45],它可以保证更好的信噪比.
20世纪90年代初,Akasaka和Yamada等人[17, 41]独创性地研发了耐压样品管方式的高压NMR技术.他们手工制作石英耐压管,然后将它组装到750 MHz的现代核磁共振谱仪上,开始了系统性的蛋白质高压研究.本综述介绍的高压NMR实例大多是Akasaka研究组研发的这种高压NMR装置完成的.石英制耐压样品管方式的高压NMR装置示意图见图 2.样品装在石英耐压样品管中,石英管内径约为1 mm,外径约为3 mm,石英管外有聚四氟乙烯保护套以防加压时样品管炸裂,石英管和保护套构成样品管体,插入5 mm的NMR探头中.蛋白质样品和传压媒介煤油(kerosene)之间用无摩擦聚四氟乙烯制活塞来隔离,活塞则置于铍铜(BeCu)制的圆柱形分离器内.压力通过放在远处的手动压力泵来调节,压力大小由Heise Bourdon gauge压力计测定.压力通过内装有煤油的不锈钢管传递至石英耐压管内的样品溶液.当压力调至所需压力后,即可进行NMR测定,这样就可以获得各压力下蛋白质化学位移、峰宽、信号强度、自旋耦合常数、NOE以及弛豫参数等NMR参数.原则上,一维、二维和三维脉冲程序都可以使用.该高压装置可用于平衡状态下的测定,也可用于压力突降(pressure-jump)实验(改变压力所需时间约为1 min)[46, 47].最近,对快速压力突降的应用也有报导(改变压力所需时间约为30 ms)[48].这一装置将改变压力所需时间融进脉冲程序编程,用于测定蛋白质不同构象状态之间的相关谱图.同样的原理也曾用于状态相关二维温度突降(temperature-jump)实验[49],但是,由于压力突降过程中构象跃迁的可逆性质,压力突降对蛋白质研究的应用范围会更广.
利用高压NMR装置对蛋白质溶液加压进行NMR测定时发现,当压力较低(几十~几百MPa以下)时,许多蛋白质并不变性,NMR谱图上并不出现信号峰分布狭窄等变性蛋白特有的现象,但是却观测到了明显的化学位移变化,这说明,加压导致了蛋白质结构的变化.作为宏观量的等温压缩率βT 与蛋白质的体积波动有关[见(2)式],压缩率越大,体积波动越大;而宏观压缩率应源于微观压缩率,即单位压力下原子间距离或角度的变化,而这些变化则反映在化学位移等NMR参数的变化上.本节将从氢键、二面角、时间尺度等方面阐述蛋白质的压力效应.
3.1.1 压力对蛋白质氢键的影响利用高压NMR装置进行实验时,90年代之前使用的多是非同位素标记的蛋白样品,之后随着同位素蛋白标记技术的进步,逐步开始使用标记蛋白.图 3(a)表示的是碱性胰蛋白酶抑制剂(basic pancreatic trypsin inhibitor,BPTI)在0.1 MPa(深灰色)和200 MPa(浅灰色)下的二维1H-1H NOESY谱图.如图 3(b)所示,随着压力的增加,大多数残基的酰胺基质子(1HN)的化学位移向低场方向移动[29],即化学位移值变大.众所周知,1HN的化学位移与其所参与的氢键距离存在很好的相关性[50, 51, 52]:氢键距离越短,1HN的化学位移值越大.因此,高压下1HN的化学位移低场移动这一倾向表明压力缩短了1HN所参与的氢键距离.具体而言,压力缩短了蛋白质分子内的氢键(N-H…O=C),同时更缩短了蛋白质与溶剂水分子之间的氢键(N-H…O-H),因为不参与分子内氢键形成的1HN,大多与溶剂水形成氢键,加压使这些1HN的化学位移低场移动得更多[图 3(c)].根据1HN的化学位移与其所参与的氢键距离的相关性以及加压下1HN的化学位移变化值可以预测氢键距离缩短的程度,分子内氢键在200 MPa时平均缩短约1%.此外,压力引起的化学位移对整个蛋白而言是非常不均一的,这揭示了每一个氢键压缩率的不均一性.继BPTI之后,我们迄今为止利用高压NMR研究过许多大小、形状各异的天然折叠蛋白,得知加压后1HN的化学位移低场移动以及变化程度的不均一性是蛋白的通性.有趣的是,与天然状态的卵清溶菌酶(hen lysozyme)[图 4(a),图 4(c)~(d)]相比,对于在8 mol/L尿素(urea)溶液中完全变性的hen lysozyme [图 4(b),图 4(e)~(f)]而言,这种高压下结构变化的不均一性则完全消失了[53].
|
| 图 3 压力对蛋白质氢键的影响.(a)BPTI的二维1H-1H NOESY谱图的1HN…Ha区域,深灰色和淡灰色信号分别表示36℃时0.1 MPa和200 MPa压力下的谱图.(b)压力引起的BPTI酰胺基质子1HN和Ha化学位移变化柱状图.(c)BPTI的20个主链结构立体示意图,图中表示出了主链HN键的位置和方向,而HN键的颜色则表示压力引起的1HN化学位移变化的程度,颜色越深,化学位移变化越大.经许可摘自文献[29].版权2001 American Chemical Society Fig. 3 Pressure effects on individual hydrogen bonds in proteins.(a)Fingerprint(1HN…Ha)regions of the 2D 1H-1H NOESY spectra of BPTI measured at 36℃ and 0.1 MPa(dark grey)or 200 MPa(light grey),respectively.(b)Histograms of pressure-induced chemical shift changes for individual peptide 1HN and Haprotons of BPTI.(c)Cross-eyed stereoview of the backbone atoms with position and directions of the HN bonds,derived from 20 NMR structures of BPTI. The colors of the HN bonds represent the extent of the pressure-induced chemical shift changes,the darker the color,the larger the chemical shift changes. Adapted with permission from ref. [29]. Copyright 2001 American Chemical Society |
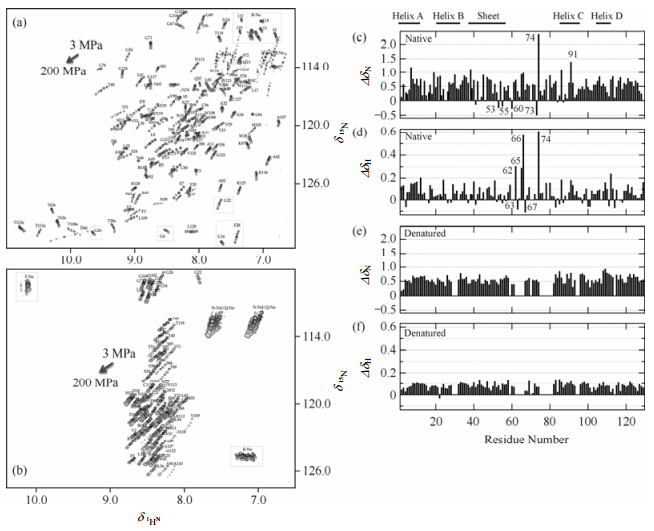
|
| 图 4 压力对天然状态以及变性状态的lysozyme的化学位移影响.(a)天然状态和(b)变性状态(变性条件为8 mol/L urea和pH 2)条件下lysozyme在3、50、100、200 MPa下的1H-15N HSQC谱图重叠.压力引起的天然状态条件下lysozyme的酰基15N(c)和1HN(d)的化学位移变化.压力引起的变性状态条件下lysozyme的酰基15N(e)和1HN(f)的化学位移变化.经许可摘自文献[53].版权2001 John Wiley and Sons Fig. 4 Pressure effects on the chemical shifts of the native and denatured lysozyme. Overlay of 1H-15N HSQC spectra of(a)native and (b)denatured(8 mol/L urea and pH 2)lysozyme recorded at high pressure at 3,50,100,200 MPa. 15N and 1HN pressure shifts of the main chain amide groups of native(c and d) and denatured(e and f)lysozyme. Adapted with permission from ref. [53]. Copyright 2001 John Wiley and Sons |
对于分子内氢键N-H…O=C,可以测到由氢键介导的酰胺基氮15N和羰基碳C'之间的三键耦合常数3JNC',这也证实了氢键具有部分共价键的特性[54, 55, 56, 57].在200 MPa时对G蛋白(protein G)的3JNC'进行测定的结果显示,同常压相比,高压下3JNC'的数值有增加也有减少,变化是不均一的[58].该结果首次揭示了压力可以改变氢键的电子轨道重叠.
3.1.2 压力对蛋白质二面角的影响酰胺基氮15N的化学位移不仅与氢键有关,而且与蛋白质的二面角也有相关性[59],因此,压力引起的15N化学位移的变化可以给出更丰富的信息.如图 5所示,常压下,BPTI的15N的化学位移与氢键距离仅有一定的相关性[图 5(a)],但是与主链的二面角f和y却表现出很好的相关性[图 5(b)和图 5(c)],与位于β-sheet前一残基的二面角yi-1尤其相关.据此预测在200 MPa时,BPTI Ψi-1的变化值为1~2°[59].另外,对hen lysozyme的研究发现,那些在加压下显示异常大的15N化学位移变化的残基大多分布在分子内部水分子附近[53].这一结果表明,在有内部水分子分布的区域,蛋白质的主链会显示异常大的构象波动.
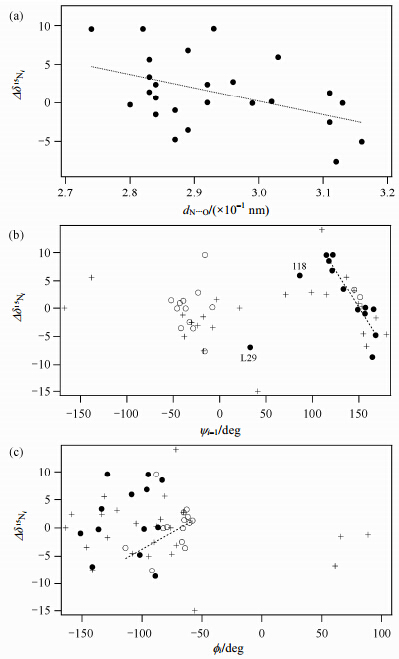
|
| 图 5 压力对蛋白质主链二面角的影响.(a)主链15N的折叠化学位移[folding shift $\Delta {{\delta }_{{}^{15}{{\text{N}}_{i}}}}$,是指主链15N天然状态和r and om coil状态之间的化学位移差]与距离dN…O之间的相关图.(b)主链15N的折叠化学位移与前一个残基的二面角以Ψi-1及(c)自身残基的二面角Øi之间的相关图.图中,实心圆、空心圆和十字符号分别代表位于β-sheet、α-helix和loop的残基.经许可摘自文献[59].版权1999 John Wiley and Sons Fig. 5 Pressure response of the protein backbone torsion angles. Folding shifts of BPTI backbone amide 15N,$\Delta {{\delta }_{{}^{15}{{\text{N}}_{i}}}}$,plotted against(a)N…O distance(dN…O);(b)dihedral angle Ψi-1; and (c)fi. Filled circles,open circles and cross symbols represent the residues located at β-sheet,α-helix and loop,respectively. Adapted with permission from ref. [59]. Copyright 1999 John Wiley and Sons |
压力除了导致主链的化学位移变化以外,还导致侧链的化学位移变化,这预示着蛋白质的三级结构也发生了变化[30, 60].Williamson等人开发了一种基于化学位移的结构计算方法,即输入1H和/或13C化学位移的变化量,计算得到蛋白质三维结构的变化.Williamson等人[61, 62, 63, 64, 65, 66]将这种方法应用到高压NMR研究中,利用加压所导致的1H和/或13C化学位移变化计算得到加压所导致的蛋白质结构变化,即将“化学位移的变化”转换为“结构坐标的变化”.该计算方法已成功地应用到hen lysozyme[61]、BPTI[62]、蜂毒素[63]、DNA发卡结构(DNA hairpin)[64]、protein G[65]、barnase/barnase-nucleotide复合物[66]等的高压研究中.结果表明,总体说来,这些蛋白的三维结构在在高压下所受到的压缩是非常不均一的.例如,在200 MPa下,hen lysozyme的a结构域受到了压缩,而b 结构域却没有受到压缩,这一结果与高压晶体学的结果非常一致[26].此外,那些发生较大体积变化的残基恰好位于含有内部水分子的空洞附近[61, 62],与分析高压所致15N化学位移变化时的预测是吻合的[53].
3.1.4 压力对不同时间尺度上的构象波动的影响构象波动有不同的时间尺度,有趣的是,压力对不同时间尺度上的构象波动表现出不同的效应.
(一)压力对快时间尺度的构象波动的影响蛋白质分子在快时间尺度的构象波动表现在键的转动和振动等方面.蛋白质酰胺基的15N自旋弛豫解析是研究主链在快时间尺度上[皮秒~纳秒(ps~ns)]运动的常用方法.我们在3 MPa和200 MPa时分别进行了BPTI的主链动力学测定.图 6表示的是蛋白质BPTI在3 MPa和200 MPa时酰胺基15N的纵向自旋弛豫速率(R1)、横向自旋弛豫速率(R2),以及根据Lipari-Szabo模型计算得到的序参量(order parameter,S2)[67].由图可见,S2在两个压力下几乎保持不变,都在0.8~1范围内,这说明每一个NH矢量的快速内部运动基本上不受压力的影响.另外,对源自菌视紫红质(bacterial rhodopsin)的一个α-螺旋(α-helix)片段也进行了不同压力下的动力学测定,分析结果表明,S2在两个压力下几乎保持不变[68].因此,压力几乎不影响蛋白质在快时间尺度上的运动.

|
| 图 6 压力对蛋白质的快时间尺度动力学.3 MPa(实线)和200 MPa(点线)时BPTI的主链动力学参数,包括(a)纵向弛豫速率R1,(b)横向弛豫速率R2以及(c)序参量S2.经许可摘自文献[67].版权2000 Elsevier Fig. 6 Pressure is insensitive to the protein dynamics on the fast time scale. Plot of experimentally determined(a)longitudinal(R1),(b)transverse(R2)15N relaxation rates and (c)order parameters(S2)for backbone amide groups at 3 MPa(solid lines) and at 200 MPa(dotted lines). Adapted with permission from ref. [67]. Copyright 2000 Elsevier |
蛋白质的结构内部存在着构象波动,例如,我们会观测到蛋白质分子内部酰胺基氢与重水溶剂之间的氢氘交换、内部Trp的荧光被外部离子淬灭、内部芳香环的翻转运动等现象.有时,蛋白质的这些构象波动会进行得很慢,时间尺度在微秒~毫秒(μs~ms).下面以芳香环的翻转运动为例,阐述压力对慢时间尺度构象波动的影响.
蛋白质芳香环残基的环平面常以Cβ-C1为轴进行左右翻转,这一现象称为芳香环的翻转运动[图 7(a)].芳香环翻转运动的速度通常很快,慢速翻转的芳香环在蛋白中很少观测到.自从1976年发现至今,见于文献报道并详尽解析的芳香环慢速翻转的例子仅见于细胞色素c(cytochrome c)[69]、BPTI[10, 30]和含组氨酸的磷酸载体蛋白(histidine-containing phosphocarrier protein,HPr)[70]等蛋白.Wagner等人[10]首先利用一维核磁共振氢谱研究了压力对BPTI的芳香环翻转速率的影响.BPTI中有4个酪氨酸(tyrosine,Tyr)和4个苯丙氨酸(phenylalanine,Phe);其中,Tyr35和Phe45的翻转速率比较慢[图 7(b)],常压下的时间尺度为ms~ms,这种翻转受压力影响而明显减速,同常压时相比,在117 MPa时速度降低了1~2个数量级.此后,我们利用二维核磁共振氢谱对此现象进行了证实,通过线形模拟方法计算了各压力下Tyr35和Phe45的翻转速率,并计算了57 ℃时二者的活化体积,分别是0.085±0.02 nm3(85±20 Å3)(或51.2 mL/mol)和0.046±0.009 nm3(46±9 Å3)(或27.7 mL/mol)[图 7(c)~(e)][30].因此,压力虽然对快时间尺度的运动几乎没有影响,却能显著地降低慢时间尺度的运动速度.此外,对来源于肉葡萄球菌(Staphylococcus carnosus)的HPr蛋白也进行了类似研究,并观测到HPr的Tyr6的翻转运动也随压力的升高而减慢;而且,不同温度下的Tyr6翻转运动的活化体积在28~51 mL/mol范围内,恰好与BPTI芳香环的活化体积范围相吻合[70].不同蛋白之间活化体积相吻合的现象说明,芳香环进行翻转运动需要额外的空间,而活化体积相当于这种额外空间的最小共通体积.额外空间的产生可能是芳香环附近的原子协同进行热波动(thermal fluctuation)的结果,这一解释与蛋白质内部空洞形成的动力学特性或长期以来的移动缺陷模型(mobile defect model)的说法一致[71, 72].Mobile defect model模型曾用于解释内部酰胺基团能与重水溶剂进行氢氘交换、以及内部Trp的荧光被外部离子淬灭的现象.
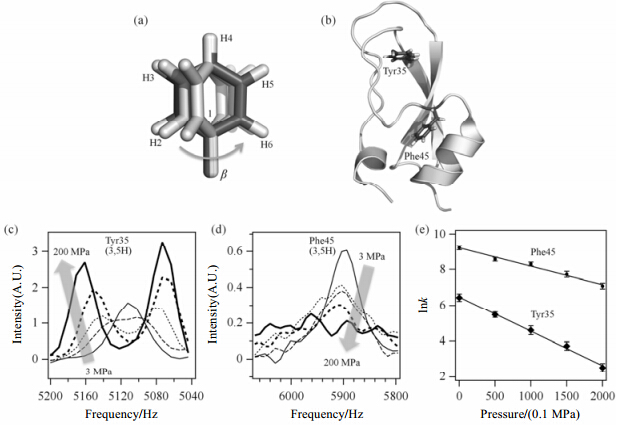
|
| 图 7 压力对慢时间尺度芳香环翻转运动速率的影响.(a)芳香环翻转运动示意图.例如,Phe的苯环可围绕Cβ-C1轴做翻转运动.(b)BPTI三维结构图,图中示有Tyr35和Phe45的位置.五个压力下(3,50,100,150,200 MPa)在(c)Tyr35-2H和(d)Phe45-2H位置从二维1H-1H TOCSY做横切得到的切片谱重叠,切片谱的信号分别来自Tyr35-3H和Tyr35-5H以及Phe45-3H和Phe45-5H,箭头方向表示压力升高方向.(e)翻转速率k的ln值与压力的相关图,二者拟合可计算得到翻转运动所需的活化体积.经许可摘自文献[30].版权1999 Elsevier. Fig. 7 Effect of pressure on the flip-flop motions of the aromatic residues on the slow timescale.(a)Schematic plot showing the flip-flop motion of the aromatic residue. For example,the benzene ring of Phe undergoes flip-flop motion around Cβ-C1 axis.(b)A ribbon model for BPTI with locations of Tyr35 and Phe45. Superposition of the 3H and 5H region of the 1H-1H TOCSY slice spectra taken at 2H resonance position of(c)Tyr35-2H and (d)Phe45-2H measured at five different pressures(3,50,100,150,200 MPa). The arrow indicates the increase direction of pressure.(e)Correlation of ln k(the flip-flop rate of the ring),for Tyr35 and Phe45 against pressure. The activation volume of the flip-flop motion is calculated accordingly. Adapted with permission from ref. [30]. Copyright 1999 Elsevier |
化学位移对蛋白质的构象波动非常敏感.观测蛋白质酰胺基1HN和15N的化学位移随压力变化时发现,有些残基的化学位移随压力呈直线变化;而有些残基的化学位移随压力呈曲线(或非线性)变化(图 8).化学位移的非线性变化这一现象表明,随着压力的升高,在蛋白质天然折叠构象群N内,混入了其它种类的构象群N'.二者具有不同的压缩率,而且N'的偏摩尔体积要小于N,因而二者的相对存在频率([N']/[N])能够随压力的增加而增加.在相对较低的压力范围内(50~100 MPa),位移随压力的变化就表现出明显的非线性,这意味着N和N'之间的自由能差$(\Delta {{G}^{0}})$不是很大,于是我们将N'称为蛋白质的低激发态(low-lying excited states)[73].一般而言,N'包含了一群构象,这些构象群具有不同的$\Delta {{G}^{0}}$和$\Delta {{V}^{0}}$值.在大多数蛋白中都发现了非线性压力位移的现象,这说明蛋白质的低激发态是普遍存在的,但低激发态的存在频率却因蛋白质种类的不同而迥然不同.
|
| 图 8 压力对主链酰胺基氢1HN化学位移的影响.(a)BPTI,(b)DHFR和(c)RalGDS-RBD的1HN的化学位移随压力变化的示意图.经许可摘自文献[73].版权2001 American Chemical Society Fig. 8 Pressure effects on the chemical shifts of the amide protons. Plot of chemical shifts of selected amide protons of(a)BPTI,(b)DHFR and (c)RalGDS-RBD as a function of pressure. Adapted with permission from ref. [73]. Copyright 2001 American Chemical Society |
为揭示化学位移对压力呈非线性变化的原因,我们利用最小二乘法对每一个残基酰胺基团的1HN和15N的化学位移随压力变化的线性项和非线性项进行了定量分析,所用的公式如(8)式所示:

|
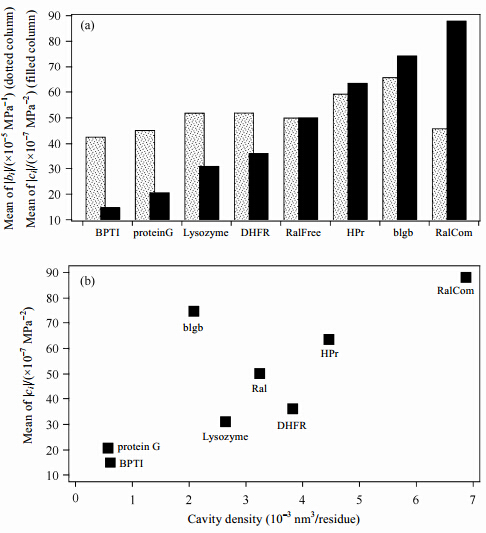
| 图 9 压力引起的化学位移线性变化和非线性变化分别体现了蛋白质的普遍性和特异性.(a)8个蛋白质主链酰胺基1HN的化学位移随压力变化的线性系数(点心柱)和非线性系数(实心柱)的绝对平均值的柱状图.(b)主链酰胺基1HN的非线性系数的绝对平均值与蛋白质空洞密度(可由蛋白质的空洞总体积除以其氨基酸残基个数计算得到)的相关图.计算空洞总体积时仅采用了体积>0.02 nm3的空洞.经许可摘自文献[73].版权2001 American Chemical Society Fig. 9 The linear and nonlinear pressure response of the chemical shifts reflects the generality and uniqueness of proteins,respectively.(a)Histograms of the mean of the absolute values of the linear coefficient bi(dotted columns) and the nonlinear coefficient ci(filled columns)for 1HN in eight globular proteins.(b)Plot of the mean of the absolute values of the nonlinear coefficient ci of 1HN pressure shifts vs the density of cavities(the total cavity volume divided by the number of amino acid residues)for the eight proteins. Cavity volumes >0.02 nm3 are employed. Adapted with permission from ref. [73]. Copyright 2001 American Chemical Society |
对ci的详细分析还可以了解N'的结构特点.如图 9(b)所示,蛋白的ci绝对值的平均值与各蛋白相应的空洞密度具有很好的相关性.这说明,N'与蛋白质的空洞分布关系密切.这里的空洞密度是指蛋白质内部空洞(>0.02 nm3)的总体积与残基总数的比值.此外,众所周知,蛋白质分子内部的空洞里有时会观测到水分子,这些水分子(又称为分子内水分子或可接近水分子)可与溶剂水交换.对ci的详细分析发现,在BPTI、hen lysozyme、RalGDS-RBD[74]、b-lactoglobulin[75]以及GB1的折叠结构中,具有较大的非线性压力位移的残基多出现在水分子可接近的空洞附近.这些发现表明,非线性位移与蛋白质空洞的水合作用有关,即N'比N处于更加水合的状态.高压晶体学的研究也证实了这一点.L99A是T4 lysozyme的空洞突变体,对L99A的X线结晶学研究发现,与野生型相比,在高压下空洞内的内部水分子的个数明显增加[76].
3.3 利用高压NMR技术可研究更广阔的蛋白质构象空间对处于天然折叠状态的蛋白逐步加压测定1H-15N HSQC谱时发现,信号峰会发生一系列的变化,包括化学位移的变化以及信号强度的变化,这说明在蛋白到达完全变性之前,途经一系列的部分折叠中间态.以脱辅基肌红蛋白(apomyoglobin)为例,随着压力的增加,apomyoglobin会经历从天然状态N到中间态I、再到熔球(molten globule,MG)状态、最后到变性状态U等一系列的构象变化[77].N→I→MG→U的顺序也是构象秩序(conformational order)降低的顺序,即构象从有序到无序.这一结果还说明,在相对较窄的压力范围内(0.1~几百MPa),高压NMR就可以“扫描”并“捕获”蛋白质在其能级地貌图(energy l and scape)中所有可能存在的构象.此外,随着压力的增加,蛋白质的偏摩尔体积也在变小.因此,蛋白质偏摩尔体积的减少与构象秩序降低的方向是一致的.这种一致性已从许多蛋白的实验中得到了证实,我们将之称为“蛋白质的体积原理(volume theorem of protein)”[24].该原理为以压力为扰动研究蛋白质多种构象特征提供了指导原则和理论基础.
高压NMR技术已应用到很多蛋白,包括二氢叶酸还原酶(dihydrofolate reductase)[78]、RalGDS-RBD[74]、b-lactoglobulin[75]、泛素(ubiquitin)[79]、仓鼠朊病毒(hamster prion)[80]、apomyoglobhin[77]、p13MTCP1[46]、外膜蛋白A(outer surface protein A,OspA)[81]等等.实验发现,这些蛋白质在常压和高压下的构象变化是可逆的,这些构象之间处于平衡状态.换而言之,在高压下检测到的构象其实在常压下也存在,只是在常压下它们的存在频率很低,仿佛“隐藏”在存在频率很高的天然构象群中,不易被检测到;而压力改变了这种平衡,于是在高压下这些构象的存在频率升高,就“暴露”出来从而得以检测.很多情况下,在常压下因存在频率很低而“隐藏”着的构象对蛋白质的功能研究至关重要.下面以OspA蛋白的高压实验为例进行说明[81].OspA由N端和C端两个结构域组成,在导致莱姆病(Lyme disease)的伯氏疏螺旋体(Borrelia burgdorferi)传染中起着关键的作用.在250 MPa下,OspA的N端结构域处于天然构象,而C端结构域则表现出局部去折叠构象[81].由于高压实验的整个过程是可逆的,因此通过热力学平衡计算可以推出该局部去折叠构象在常压下的存在频率,结果显示该构象仅占全体构象的0.001%左右.OspA的结合抗原表位(binding epitope)位于C端结构域,在常压下的天然构象中,C端结构域深埋在蛋白疏水核内;而参与感染时,抗原表位有必要暴露出来与抗原结合,因此OspA行使功能时伴随着很大的构象变化,而高压下检测到的构象很可能就是这种功能性构象.所以利用高压NMR技术,可以通过加压改变构象平衡,提高功能性构象的存在频率,从而对其进行深入研究.
在高压下,蛋白质构象的秩序降低和偏摩尔体积减少主要归结于两个原因:一是分子内空洞的体积变小,二是蛋白质表面溶剂层的压缩.近年来,高压下蛋白质变性机制在原子水平上的研究取得了令人瞩目的进展[82, 83].Royer研究组以Staphylococcalnuclease为模型蛋白,通过残基突变制备了许多含有不同空洞体积的突变体,然后对这些突变体进行高压变性实验,计算了各个突变体从天然构象至高压变性时的体积变化.结果发现,高压变性时的体积变化与天然构象的空洞体积之间呈现了很好的相关性,因此推断,蛋白质分子内空洞对高压变性的体积变化起到了主要作用.
此外,高压与低温的并用也是扩展蛋白质构象空间研究的一种物理手段.在200 MPa时,水的冰点是-21 ℃,在这样的低温条件下,可以相对容易地观察到蛋白质的低温变性.自从Jonas在高压下观测到了hen lysozyme的低温变性以来,在许多蛋白中同样也观测到了高压低温下的变性构象或接近变性时的构象,包括hamster prion[80]、HPr[70]、ubiquitin[84]、hen lysozyme[85]、c-Myb R2结构域[86]等,这些探讨为研究蛋白质的构象空间给予了很大启发.
3.4 高压下的淀粉样纤维(amyloid fibril)形成与解离的动力学淀粉样纤维的形成与诸多疾病有关,如阿尔茨海默病(Alzheimer's disease)、疯牛病、II型糖尿病等,是蛋白质多聚体形成的特殊现象.而众所周知,压力可导致蛋白质多聚体的解离,一个最好的例子是纤维状肌动蛋白(F-actin)的高压解离[87].因此,近年来高压技术在这些方面的应用格外受到关注.Akasaka研究组所做的平衡状态下的高压实验[88]以及压力突降[47]实验清楚地表明,淀粉样原纤维(amyloid protofibril)在高压作用下会解离,而且其形成与解离在高压下是一个可逆的过程.下面以lysozyme的突变体为例,简述高压对淀粉样纤维形成与解离的影响.
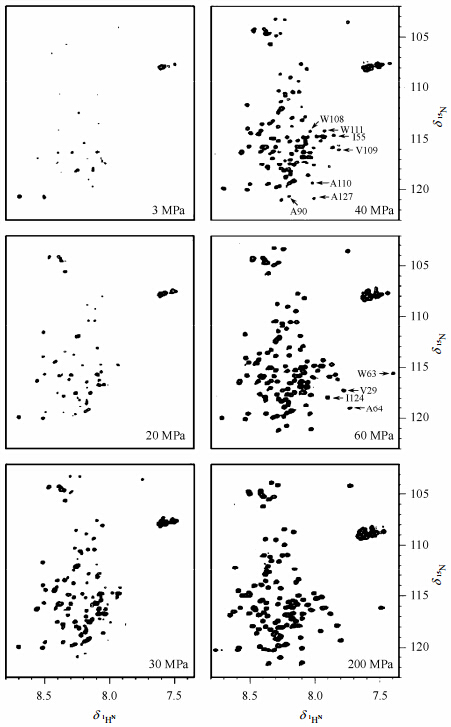
天然lysozyme由a和b两个结构域组成,含有四个二硫键,形成这四个二硫键的八个半胱氨酸(cysteine,Cys)分别突变为丙氨酸(alanine,Ala)或丝氨酸(serine,Ser)之后的突变体(称为0SS)不含三级结构,在柔和的酸性且含有NaCl的条件下,可寡聚成淀粉样原纤维,放置几个月后会长成很长的纤维.对这一体系,利用高压NMR技术分别进行了平衡条件下的高压NMR[88]以及压力突降实验[47].如图 10所示的平衡条件下的高压NMR实验显示,淀粉样原纤维在几百MPa下可完全解离,而当压力回到常压后又会重新聚合,这表明多聚体与单体之间存在热力学平衡[88].从200 MPa减压至60 MPa和40 MPa时,有些残基的信号消失了,而这些残基多为疏水性残基,这说明多聚体的分子间相互作用具有疏水性质.在广义上,寡聚-解离现象也属于蛋白质构象波动.对这一平衡体系进行热力学参数计算的结果表明,以单体为单位时的聚合自由能是负值(-23.3±0.8 kJ/mol),体积变化是正值(52.7±11.3 mL/mol),即多聚体的体积大于单体的体积.上述结果表明,看似不可逆的聚集(aggregation)反应在加压下是可逆的,而且还通过压力扰动实验求得了该平衡体系的热力学参数[88].另外,Akasaka等人[89]使用高精度的密度仪和超声波测速仪,直接测定了0SS的偏摩尔体积和压缩率.结果表明,0SS非常“蓬松”,具有很好的压缩性,这与高压NMR实验中聚合的体积变化是正值这一结论非常一致.此外,通过压力突降实验对0SS的解离和寡聚的动力学(kinetics)也进行了研究,包括由低压至高压的解离过程以及由高压至低压的再聚合(reassociation)过程的动力学[47].结果表明,纤维的解离速度在高压下会加速,解离时的活化体积是负值.
|
| 图 10 高压可使淀粉样原纤维完全解离.Lysozyme 0SS突变体在各压力(3,20,30,40,60,200 MPa)时的1H-15N HSQC.在40 MPa和60 MPa谱图中标示的残基信号随压力的降低而消失,这些残基多为疏水性残基,参与分子间的相互作用.经许可摘自文献[88].版权2004 National Academy of Sciences of the United States of America Fig. 10 Dissociation of amyloid protofibrils by pressure. 1H-15N HSQC spectra of uniformly 15N-labeled lysozyme 0SS variant at various pressures(3,20,30,40,60,200 MPa)as indicated. The cross peaks labeled in the spectrum at 40 MPa or 60 MPa diminished conspicuously with decreasing pressure,most of these residues being hydrophobic ones involved in intermolecular interactions. Adapted with permission from ref. [88]. Copyright 2004 National Academy of Sciences of the United States of America |
蛋白质在从折叠构象(N)变成去折叠构象(U)的过程中,常伴随着折叠中间体的出现,中间体是蛋白质折叠动力学研究的重要对象.而如上述的高压NMR研究所指出的,蛋白质在溶液中多种构象平衡存在,其中包括N构象和U构象;换句话说,蛋白质即使在0.1 MPa的生理条件下,也在不断地重复着折叠和去折叠过程.于是,我们提出的问题是:动力学中间体与压力平衡实验中检测到的中间体有何关联?
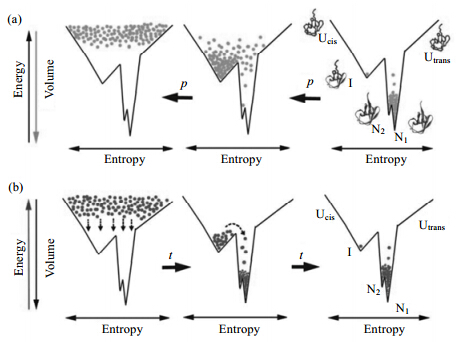
在蛋白质的折叠研究中经常使用折叠漏斗假设(folding funnel hypothesis)来描绘蛋白质构象的能量地貌图.图 11给出的是ubiquitin在pH 4.5和20 ℃条件下的平衡压力实验与折叠动力学实验的比较示意图[90].图 11(b)折叠动力学实验漏斗示意图中漏斗顶端表示的是蛋白质所有可能采取的构象.向体系施予折叠条件,让蛋白质开始折叠时,位于顶部的构象便沿着漏斗形状的能量地貌图开始重新分布,这些构象最终达到新的平衡.在这一过程中,某些构象可能短暂地陷入漏斗旁边的凹槽内而形成折叠中间体.
|
| 图 11 平衡压力实验与折叠动力学实验的概念比较示意图.(a)平衡状态下的压力实验.(b)折叠动力学实验.详情见文中叙述.经许可摘自文献[90].版权2003 National Academy of Sciences of the United States of America Fig. 11 Concept comparison of the equilibrium pressure experiment vs. the kinetic folding experiment.(a)The equilibrium pressure experiment.(b)The kinetic folding experiment. See the main text for details. Adapted with permission from ref. [90]. Copyright 2003 National Academy of Sciences of the United States of America |
设想一下,在构象分布已达到平衡的状态下开始加压会发生什么情况?在等温条件下加压会使得平衡向具有较小体积构象的方向移动[参见(6)式],直至达到新的构象分布平衡.由于蛋白质的体积与其构象秩序的对应性,即体积越小,构象秩序越低,因此,所加压力越高,新增加的构象会出现在构象秩序较低的漏斗上端,于是,新的平衡分布最终可以达到图 11(a)左图所示的样子.由此可见,动力学中间体与压力平衡中间体有可能非常相似,但是,与动力学实验中出现的瞬间即逝的中间体相比,在平衡状态的压力实验中检测到的中间体比较稳定、寿命长(几个小时或几天),因此可用二维或多维NMR实验详细地研究中间体的结构和各种性质.另外,应该强调的是,温度和化学试剂(如pH、变性剂等)等扰动方式有可能改变能级地貌图,但是压力扰动对能级地貌图的改变则微乎其微,这使得高压NMR成为详细研究蛋白质中间体结构的很有用的技术.
3.6 构象波动中蛋白质结构的NMR“快照(snapshot)”在溶液中,蛋白质的天然构象与其它非天然构象处于动态平衡.众所周知,利用X射线晶体学或NMR方法,可以在原子水平解析存在频率很高的蛋白质天然构象的三维结构.那么,是否也有方法在原子水平对与蛋白质天然构象共存的其它构象进行三维结构进行解析呢?高压NMR技术以ubiquitin为例进行了这一方面的尝试.
在3~350 MPa的压力范围内对ubiquitin进行1H-15N HSQC测定时发现,一些残基的主链酰胺基团表现出了可逆的、非线性的化学位移变化,而且很多信号随压力呈S形变化,这些特征表明ubiquitin处于二状态跃迁(two-state transition).对化学位移变化的分析表明,在ubiquitin体系中,平衡地存在着两种主要折叠构象N1和N2(见下式),它们之间存在着快速构象交换(即k12,k21>>103 s-1,k12和k21分别为构象N1至N2以及构象N2至N1的构象交换速率)[79].

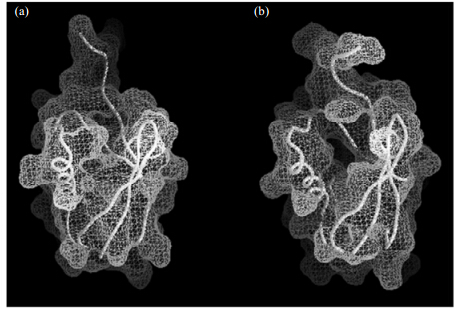
为了进一步得到N1和N2的结构信息,在3 MPa和300 MPa的条件下,对ubiquitin分别进行了基于NOESY的结构计算,求出了两个压力下的结构坐标(图 12)[91].结构解析的结果清楚地展示了N1和N2这两个结构的不同之处:C末端的a-helix在常压下是摆进的(swinging-in)[图 12(a)],而在高压下则是摆出的(swinging-out)[图 12(b)],即N1和N2构象之间存在着swinging in- and -out的动态平衡.因此,高压下的结构所展示的相当于波动中的蛋白质结构的NMR“快照”,这是第一次在原子分辨率下所“拍到的”蛋白质构象波动.用GRASP[92]计算得到的ubiquitin在3 MPa和300 MPa时的分子表面积发现,相比N1而言,N2显示了部分张开的结构,其分子表面积相应地增加了5%.张开的结构使更多的分子表面得以暴露,这样会导致蛋白质与溶剂水的接触面积增加,从而使得偏摩尔体积减小.因此,高压所致的结构变化与热力学解析所得到的偏摩尔体积减小的结论是一致的.
|
| 图 12 蛋白质波动结构的NMR“快照”.(a)3 MPa和(b)300 MPa时ubiquitin立体结构的分子表面示意图.分子表面由GRASP软件计算得到.经许可摘自文献[91].版权2005 Elsevier Fig. 12 NMR “snapshot” of the fluctuating protein structure. Illustration of the molecular surface of ubiquitin at(a)3 MPa and at(b)300 MPa calculated using the program GRASP. Adapted with permission from ref. [91]. Copyright 2005 Elsevier |
在高压条件下以及1 ms的时间尺度上,对ubiquitin进行了分子动力学(molecular dynamics,MD)模拟计算[93].结果表明,高压下ubiquitin从“关闭”结构过渡(transition)到“张开(open)”结构,这一MD模拟计算再现了高压NMR的结构计算结果.此外,MD模拟计算中还观察到了溶剂水分子向“张开”的疏水性空洞的钻入(penetration)现象,这一现象揭示了高压下ubiquitin的结构呈“张开”构象的意义.水分子的钻入通常认为是导致高压下蛋白质变性的主要驱动力,这一观点从模拟计算中得到了验证.此外,关于这一观点,Kamatari等人[85]在以lysozyme为对象的压力研究中发现,蛋白质空洞的水合现象是蛋白质去折叠的必经之路.
虽然蛋白质的构象波动与其功能密切相关,但是对波动时实际发生的结构变化,很少有实验能对其进行可视化,而高压NMR可以实现这个目标.高压NMR方法具有普适性,原则上,只要谱图分辨率足够好、信噪比足够高,这个方法可以应用于任何一个蛋白.从原理上说,加压可以改变平衡构象的存在频率,使蛋白质从具有较大体积的构象移动到具有较小体积的构象.因此在高压NMR方法中,压力的作用是放大并捕捉波动的构象,而NMR的作用是在原子水平解析“捕捉到的”蛋白质的结构.
4 总结与展望与温度和化学试剂这两种扰动相比,压力属于一种柔和的扰动,它通过改变平衡体系中各构象的存在频率以增大蛋白质固有的构象波动.压力扰动可从结构生物学、热力学和反应动力学(kinetics)等方面提供有关蛋白质构象波动的新信息,这些信息综合起来将构成现代蛋白质动力学研究的核心.压力扰动与现代多维NMR技术以及丰富多样的NMR脉冲程序的有机结合使得原子水平的蛋白质高压研究成为可能.另一方面,耐压NMR管的开发也从未停步.近年来,Daedalus Innovations LLC公司开发了一款内径为3 mm、样品体积高达120 mL的新型陶瓷高压样品管,这大大提高了高压下谱图采集的灵敏度.采用这种新型的陶瓷高压样品管,Grizesiek研究组[44]在250 MPa时测定了ubiquitin中由氢键介导的耦合常数3JNC'.与之前Akasaka研究组[58]以protein G为样品所测得的耦合常数3JNC'相比,灵敏度有了很大提高.采用同样的新型陶瓷高压样品管,Bax研究组[45]研究了天然变性蛋白a-突触核蛋白(a-synuclein)在高压下的构象特性,并首次在高压下测定了三键耦合常数3JHN-Ha;W and 研究组[94]则首次测定了高压下的残余偶极耦合(residue dipolar coupling,RDC)值,并研究了高压下5种配向物质的特性.
压力扰动的重要性在于它能够研究更广阔的构象空间,以及时间尺度上比较慢的、存在频率比较稀少的和运动幅度比较大的构象波动,而这些运动在其它扰动下却常被忽略、或检测不到.高压NMR的诸多应用实例表明,这些稀少的运动对理解蛋白质的功能至关重要.高压NMR是一种在平衡体系下研究蛋白质以及其他生物大分子的高分辨结构和动力学的技术,可用于研究用其他方法“看不到”的、但在功能机制中至关重要的构象,从而为蛋白质的动力学和功能研究提供新的思路.
| [1] | Bridgman P W. The coagulation of albumen by pressure[J]. J Biol Chem, 1914, 19(4): 511-512. |
| [2] | Suzuki K. Studies on the kinetics of protein denaturation under high pressure[J]. Rev Phys Chem Japan, 1960, 29(2): 91-98. |
| [3] | Brandts J F, Oliveira R J, Westort C. Thermodynamics of protein denaturation. Effect of pressure on the denaturation on ribonuclease A[J]. Biochemistry, 1970, 9(4): 1 038-1 047. |
| [4] | Hawley S A. Reversible pressure-temperature denaturation of chymotrypsinogen[J]. Biochemistry, 1971, 10(13): 2 436-2 442. |
| [5] | Zipp A, Kauzmann W. Pressure denaturation of metmyoglobin[J]. Biochemistry, 1973, 12(21): 4 217-4 228. |
| [6] | Panick G, Vidugiris G, Malessa R, et al. Exploring the temperature-pressure phase diagram of Staphylococcal nuclease[J]. Biochemistry, 1999, 38(13): 4 157-4 164. |
| [7] | Lassalle M W, Yamada H, Akasaka K. The pressure-temperature free energy-landscape of Staphylococcal nuclease monitored by 1H NMR[J]. J Mol Biol, 2000, 298(2): 293-302. |
| [8] | Weber G, Drickamer H G. The effect of high pressure upon proteins and other biomolecules[J]. Q Rev Biophys, 1983, 16(1): 89-112. |
| [9] | Frauenfelder H, Alberding N A, Ansari A, et al. Proteins and pressure[J]. J Phys Chem 1990, 94(3): 1 024-1 037. |
| [10] | Wagner G. Activation volumes for the rotational motion of interior aromatic rings in globular proteins determined by high resolution 1H NMR at variable pressure[J]. FEBS Lett, 1980, 112(2): 280-284. |
| [11] | Morishima I, Ogawa S, Yamada H. High-pressure proton nuclear magnetic resonance studies of hemoproteins. Pressure-induced structural change in heme environments of myoglobin, hemoglobin, and horseradish peroxidase[J]. Biochemistry, 1980, 19(8): 1 569-1 575. |
| [12] | Jonas J, Jonas A. High-pressure NMR spectroscopy of proteins and membranes[J]. Rev Biophys Biomol Struct, 1994, 23: 287-318. |
| [13] | Royer C A, Hinck A P, Loh S N, et al. Effects of amino acid substitutions on the pressure denaturation of Staphylococcal nuclease as monitored by fluorescence and nuclear magnetic resonance spectroscopy[J]. Biochemistry, 1993, 32(19): 5 222-5 232. |
| [14] | Fuentes E J, Wand A J. Local stability and dynamics of apocytochrome b562 examined by the dependence of hydrogen exchange on hydrostatic pressure[J]. Biochemistry, 1998, 37(28): 9 877-9 883. |
| [15] | Jonas J, Ballard L, Nash D. High-resolution, high-pressure NMR studies of proteins[J]. Biophys J, 1998, 75(1): 445-452. |
| [16] | Jonas J. High-resolution nuclear magnetic resonance studies of proteins[J]. Biochim Biophys Acta, 2002, 1 595(12): 145-159. |
| [17] | Akasaka K, Yamada H. On-line cell high-pressure nuclear magnetic resonance technique: Application to protein studies[J]. Methods Enzymol, 2002, 338: 134-146. |
| [18] | Kamatari Y O, Kitahara R, Yamada H, et al. High-pressure NMR spectroscopy for characterizing folding intermediates and denatured states of proteins[J]. Methods, 2004, 34(1): 133-143. |
| [19] | Akasaka K. Probing conformational fluctuation of proteins by pressure perturbation[J]. Chem Rev, 2006, 106 (5): 1 814-1 835. |
| [20] | Li H, Akasaka K. Conformational fluctuations of proteins revealed by variable pressure NMR[J]. Biochim Biophys Acta, 2006, 1 764(3): 331-345. |
| [21] | Lassalle M W, Akasaka K. The use of high-pressure nuclear magnetic resonance to study protein folding[J]. Methods Mol Biol, 2007, 350: 21-38. |
| [22] | Akasaka K, Kitahara R, Kamatari Y O. Exploring the folding energy landscape with pressure[J]. Arch Biochem Biophys, 2013, 531(1, 2): 110-115. |
| [23] | Kitahara R, Hata K, Li H, et al. Pressure-induced chemical shifts as probes for conformational fluctuations in proteins[J]. Prog Nucl Magn Reson Spectrosc, 2013, 71: 35-58. |
| [24] | Akasaka K. Highly fluctuating protein structures revealed by variable-pressure nuclear magnetic resonance[J]. Biochemistry, 2003, 42(37): 10 875-10 885. |
| [25] | Gekko K, Hasegawa Y. Compressibility-structure relationship of globular proteins[J]. Biochemistry, 1986, 25(21): 6 563-6 571. |
| [26] | Kundrot C E, Richards F M. Crystal structure of hen egg-white lysozyme at a hydrostatic pressure of 1000 atmospheres[J]. J Mol Biol, 1987, 193(1): 157-170.. |
| [27] | Urayama P, Phillips G N, Gruner S M. Probing substates in sperm whale myoglobin using high-pressure crystallography[J]. Structure, 2002, 10(1): 51-60. |
| [28] | Fourme R, Girard E, and Akasaka K. High-pressure macromolecular crystallography and NMR: Status, achievements and prospects[J]. Curr Opin Struct Biol, 2012, 22(5): 636-642. |
| [29] | Li H, Yamada H, Akasaka K. Effect of pressure on individual hydrogen bonds in proteins. Basic Pancreatic Trypsin Inhibitor[J]. Biochemistry, 1998, 37(5): 1 167-1 173. |
| [30] | Li H, Yamada H, Akasaka K. Effect of pressure on the tertiary structure and dynamics of folded basic pancreatic trypsin inhibitor[J]. Biophys J, 1999, 77(5): 2 801-2 812. |
| [31] | Woodward C K, Hilton B D. Hydrogen exchange kinetics and internal motions in proteins and nucleic acids[J]. Annu Rev Biophys Bioeng, 1979, 8: 99-127. |
| [32] | Lee Y H, Goto Y. Kinetic intermediates of amyloid fibrillation studied by hydrogen exchange methods with nuclear magnetic resonance[J]. Biochim Biophys Acta, 2012, 1 824(12): 1 307-1 323. |
| [33] | Korzhnev D M, Kay L E. Probing invisible, low-populated States of protein molecules by relaxation dispersion NMR spectroscopy: an application to protein folding[J]. Acc Chem Res, 2008, 41(3): 442-451. |
| [34] | Bouvignies G, Vallurupalli P, Hansen D F, et al. Solution structure of a minor and transiently formed state of a T4 lysozyme mutant[J]. Nature, 2011, 477(7 362): 111-114. |
| [35] | Meinhold D W, Wright P E. Measurement of protein unfolding/refolding kinetics and structural characterization of hidden intermediates by NMR relaxation dispersion[J]. Proc Natl Acad Sci U S A, 2011, 108(22): 9 078-9 083. |
| [36] | Wen Yi(文祎), Lin Dong-hai(林东海). Protein dynamics studied by NMR spin relaxation(基于NMR自旋弛豫技术的蛋白质动力学研究)[J]. Chinese J Magn Reson(波谱学杂志), 2012, 29(2): 288-306. |
| [37] | Clore G M, Tang C, Iwahara J. Elucidating transient macromolecular interactions using paramagnetic relaxation enhancement[J]. Curr Opin Struct Biol, 2007, 17(5): 603-616. |
| [38] | Tang C, Louis J M, Aniana A, et al. Visualizing transient events in amino-terminal autoprocessing of HIV-1 protease[J]. Nature. 2008, 455(7 213): 693-696. |
| [39] | Liu Zhu(刘主), Tang Chun(唐淳). Paramagnetic relaxation enhancement—A tool for visualizing transient protein structures(顺磁弛豫增强技术与蛋白质瞬态结构)[J]. Chinese J Magn Reson(波谱学杂志), 2011, 28(3): 301-316. |
| [40] | Atkins P W. The Elements of Physical Chemistry[M]. 3rd ed. Oxford: Oxford University Press, 1993. |
| [41] | Yamada H, Nishikawa K, Honda M, et al. Pressure-resisting cell for high-pressure, high-resolution nuclear magnetic resonance measurements at very high magnetic fields[J]. Rev Sci Instrum, 2001, 72(2): 1 463-1 471. |
| [42] | Urbauer J L, Ehnhardt M R, Bieber R J, et al. High-resolution triple-resonance NMR spectroscopy of a novel calmodulin·peptide complex at kilobar pressures[J]. J Am Chem Soc, 1996, 118(45): 11 329-11 330. |
| [43] | Arnold M R, Kremer W, Luedemann H D, et al. 1H-NMR parameters of common amino acid residues measured in aqueous solutions of the linear tetrapeptides Gly-Gly-X-Ala at pressures between 0.1 and 200 MPa[J]. Biophys Chem, 2002, 96(2-3): 129-140. |
| [44] | Nisius L, Grzesiek S. Key stabilizing elements of protein structure identified through pressure and temperature perturbation of its hydrogen bond network[J]. Nat Chem, 2012 4(9): 711-717. |
| [45] | Roche J, Ying J, Maltsev A S, et al. Impact of hydrostatic pressure on an intrinsically disordered protein: A high-pressure NMR study of a-synuclein[J]. Chembiochem, 2013, 14(14): 1 754-1 761. |
| [46] | Kitahara R, Royer C, Yamada H, et al. Equilibrium and pressure-jump relaxation studies of the conformational transitions of P13MTCP1[J]. J Mol Biol, 2002, 320(3): 609-628. |
| [47] | Kamatari Y O, Yokoyama S, Tachibana H, et al. Pressure-jump NMR study of dissociation and association of amyloid protofibrils[J]. J Mol Biol, 2005, 349(5): 916-921. |
| [48] | Kremer W, Arnold M, Munte C E, et al. Pulsed pressure perturbations, an extra dimension in NMR spectroscopy of proteins[J]. J Am Chem Soc, 2011, 133(34): 13 646-13 651. |
| [49] | Akasaka K, Naito A, Nakatani H. Temperature-jump NMR study of protein folding: Ribonuclease A at low pH[J]. J Biomol NMR, 1991, 1(1): 65-70. |
| [50] | Wagner G, Pardi A, Wuthrich K. Hydrogen bond length and proton NMR chemical shifts in proteins[J]. J Am Chem Soc, 1983, 105(18): 5 948-5 949. |
| [51] | Pardi A, Wagner G, Wuthrich K. Protein conformation and proton nuclear-magnetic-resonance chemical shifts[J]. Eur J Biochem, 1983, 137(3): 445-454. |
| [52] | Asakura T, Taoka K, Demura M, et al. The relationship between amide proton chemical shifts and secondary structure in proteins[J]. J Biomol NMR, 1995, 6(3): 227-236. |
| [53] | Kamatari Y O, Yamada H, Akasaka K, et al. Response of native and denatured hen lysozyme to high pressure studied by 15N/1H NMR spectroscopy[J]. Eur J Biochem, 2001, 268(6): 1 782-1 793. |
| [54] | Dingley A J, Grzesiek S. Direct observation of hydrogen bonds in nucleic acid base pairs by internucleotide 2JNN couplings[J]. J Am Chem Soc, 1998, 120(33): 8 293-8 297. |
| [55] | Cordier F, Grzesiek S. Direct observation of hydrogen bonds in proteins by interresidue 3hJNC' scalar couplings[J]. J Am Chem Soc, 1999, 121(7): 1 601-1 602. |
| [56] | Cornilescu G, Hu J S, Bax A. Identification of the hydrogen bonding network in a protein by scalar couplings[J]. J Am Chem Soc, 1999, 121(12): 2 949-2 950. |
| [57] | Cornilescu G, Ramirez B E, Frank M K, et al. Correlation between 3hJNC' and hydrogen bond length in proteins[J]. J Am Chem Soc, 1999, 121(26): 6 275-6 279. |
| [58] | Li H, Yamada H, Akasaka K, et al. Pressure alters electronic orbital overlap in hydrogen bonds[J]. J Biomol NMR, 2000, 18(3): 207-216. |
| [59] | Akasaka K, Li H, Yamada H, et al. Pressure response of protein backbone structure. Pressure-induced amide 15N chemical shifts in BPTI[J]. Protein Sci, 1999, 8(10): 1 946–1 953. |
| [60] | Akasaka K, Tezuka T, Yamada H. Pressure-induced changes in the folded structure of lysozyme[J]. J Mol Biol, 1997, 271(5): 671-678. |
| [61] | Refaee M, Tezuka T, Akasaka K, et al. Pressure-dependent changes in the solution structure of hen egg-white lysozyme[J]. J Mol Biol, 2003, 327(4): 857-865. |
| [62] | Williamson M P, Akasaka K, Refaee M. The solution structure of bovine pancreatic trypsin inhibitor at high pressure[J]. Protein Sci, 2003, 12(9): 1 971-1 979. |
| [63] | Iwadate M, Asakura T, Dubovskii P V, et al. Pressure-dependent changes in the structure of the melittin a-helix determined by NMR[J]. J Biomol NMR, 2001, 19(2): 115-124. |
| [64] | Wilton D J, Ghosh M, Chary K V, et al. Structural change in a B-DNA helix with hydrostatic pressure[J]. Nucleic Acids Res, 2008, 36(12): 4 032-4 037. |
| [65] | Wilton D J, Kitahara R, Akasaka K, et al. Pressure-dependent 13C chemical shifts in proteins: Origins and applications[J]. J Biomol NMR, 2009, 44(1): 25-33. |
| [66] | Wilton D J, Kitahara R, Akasaka K, et al. Pressure-dependent structure changes in barnase on ligand binding reveal intermediate rate fluctuations[J]. Biophys J, 2009, 97(5): 1 482-1 490. |
| [67] | Sareth S, Li H, Yamada H, et al. Rapid internal dynamics of BPTI is insensitive to pressure. 15N spin relaxation at 2 kbar[J]. FEBS Lett, 2000, 470(1): 11-14. |
| [68] | Orekhov V Y, Dubovskii P V, Yamada H, et al. Pressure effect on the dynamics of an isolated a-helix studied by 15N-1H NMR relaxation[J]. J Biomol NMR, 2000, 17(3): 257-263. |
| [69] | Campbell I D, Dobson C M, Moore G R, et al. Temperature dependent molecular motion of a tyrosine residue of ferrocytochrome C[J]. FEBS Lett, 1976, 70(1): 96-100. |
| [70] | Hattori M, Li H, Yamada H, et al. Infrequent cavity-forming fluctuations in HPr from Staphylococcus carnosus revealed by pressure- and temperature-dependent tyrosine ring flips[J]. Protein Sci, 2004, 13(12): 3 104-3 114. |
| [71] | Lumry R, Rosenberg A. Mobile-defect hypothesis of protein function[J]. Coll Int CNRS L'Eau Syst Biol, 1975, 246: 55-63. |
| [72] | Pain R H. New light on old defects[J]. Nature, 1987, 326(6 110): 247-247. |
| [73] | Akasaka K, Li H. Low-lying excited states of proteins revealed from nonlinear pressure shifts in 1H and 15N NMR[J]. Biochemistry, 2001, 40(30): 8 665-8 671. |
| [74] | Inoue K, Maurer T, Yamada H, et al. High-pressure NMR study of the complex of a GTPase Rap1A with its effector RalGDS. A conformational switch in RalGDS revealed from non-linear pressure shifts[J]. FEBS Lett, 2001, 506(3): 180-184. |
| [75] | Kuwata K, Li H, Yamada H, et al. High pressure NMR reveals a variety of fluctuating conformers in b-lactoglobulin[J]. J Mol Biol, 2001, 305(5): 1 073-1 083. |
| [76] | Collins M D, Hummer G, Quillin M L, et al. (2005) Cooperative water filling of a nonpolar protein cavity observed by high pressure crystallography and simulation[J]. Proc Natl Acad Sci U S A, 102(46): 16 668-16 671. |
| [77] | Kitahara R, Yamada H, Akasaka K, et al. High pressure NMR reveals that apomyoglobin is an equilibrium mixture from the native to the unfolded[J]. J Mol Biol, 2002, 320(2): 311-319. |
| [78] | Kitahara R, Sareth S, Yamada H, et al. High pressure NMR reveals active-site hinge motion of folate-bound Escherichia coli dihydrofolate reductase[J]. Biochemistry, 2000, 39(42): 12 789-12 795. |
| [79] | Kitahara R, Yamada H, Akasaka K. Two folded conformers of ubiquitin revealed by high-pressure NMR[J]. Biochemistry, 2001, 40(45): 13 556-13 563. |
| [80] | Kuwata K, Li H, Yamada H, et al. Locally disordered conformer of the hamster prion protein: A crucial intermediate to PrPSc? [J] Biochemistry, 2002, 41(41): 12 277-12 283. |
| [81] | Kitahara R, Simorellis A K, Hata K, et al. A delicate interplay of structure, dynamics, and thermodynamics for function: a high pressure NMR study of outer surface protein A[J]. Biophys J, 2012, 102(4): 916-926. |
| [82] | Roche J, Caro J A, Norberto D R, et al. Cavities determine the pressure unfolding of proteins[J]. Proc Natl Acad Sci U S A, 2012, 109(18): 6 945-6 950. |
| [83] | Lassalle M W, Yamada H, Morii H, et al. Filling a cavity dramatically increases pressure stability of the c-Myb R2 subdomain[J]. Proteins, 2001, 45(1): 96-101. |
| [84] | Kitahara R, Okuno A, Kato M, et al. Cold denaturation of ubiquitin at high pressure[J]. Magn Reson Chem, 2006, 44(S1): S108-S113. |
| [85] | Kamatari Y O, Smith L J, Dobson C M, et al. Cavity hydration as a gateway to unfolding: An NMR study of hen lysozyme at high pressure and low temperature[J]. Biophys Chem, 2011, 156(1): 24-30. |
| [86] | Narayanan S P, Maeno A, Matsuo H, et al. Extensively hydrated but folded: A novel state of globular proteins stabilized at high pressure and low temperature[J]. Biophys J, 2012, 102(2): 8-10. |
| [87] | Garcia C R, Amaral J A, Abrahamsohn P, et al. Dissociation of F-actin induced by hydrostatic pressure[J]. Eur J Biochem, 1992, 209(3): 1 005-1 011. |
| [88] | Niraula T N, Konno T, Li H, et al. Pressure-dissociable reversible assembly of intrinsically denatured lysozyme is a precursor for amyloid fibrils[J]. Proc Natl Acad Sci U S A, 2004, 101(12): 4 089-4 093. |
| [89] | Akasaka K, Latif A R, Nakamura A, et al. Amyloid protofibril is highly voluminous and compressible[J]. Biochemistry, 2007, 46(37): 10 444-10 450. |
| [90] | Kitahara R, Akasaka K. Close identity of a pressure-stabilized intermediate with a kinetic intermediate in protein folding[J]. Proc Natl Acad Sci U S A, 2003, 100(6): 3 167-3 172. |
| [91] | Kitahara R, Yokoyama S, Akasaka K. NMR snapshots of a fluctuating protein structure: ubiquitin at 30 bar-3 kbar[J]. J Mol Biol, 2005, 347(2): 277-285. |
| [92] | Nicholls A, Sharp K A, Honig B. Protein folding and association: Insights from the interfacial and thermodynamic properties of hydrocarbons[J]. Proteins: Struct Funct Genet, 1991, 11(4): 281-296. |
| [93] | Imai T, Sugita Y. Dynamic correlation between pressure-induced protein structural transition and water penetration[J]. J Phys Chem B, 2010, 114(6): 2 281-2 286. |
| [94] | Fu Y, Wand A J. Partial alignment and measurement of residual dipolar couplings of proteins under high hydrostatic pressure[J]. J Biomol NMR, 2013, 56(4): 353-357. |
 2016, Vol. 33
2016, Vol. 33





