文章信息
- 杜天鹏, 张子微, 曾丹云, 刘买利, 姜凌
- DU Tian-peng, ZHANG Zi-wei, ZENG Dan-yun, LIU Mai-li, JIANG Ling
- 流感病毒融合肽磁共振研究进展
- Structure and Function of Influenza Virus Fusion Peptide Studied by Nuclear Magnetic Resonance Spectroscopy
- 波谱学杂志, 2014, 31(4): 596-607
- Chinese Journal of Magnetic Resonance, 2014, 31(4): 596-607
-
文章历史
收稿日期: 2014-01-17
收修改稿日期: 2014-10-28


2. 中国科学院大学,北京 100049
2. University of Chinese Academy of Science, Beijing 100049, China
流感病毒(influenza virus)属于包膜病毒(enveloped viruses),对人类健康危害很大,2009年爆发的猪流感H1N1 (swine flu H1N1)以及近段时间出现的禽流感H7N9 (avian virus H7N9)使流感病毒再度成为公众健康问题的焦点.包膜病毒侵染细胞的生命周期经历吸附、侵入、脱壳、病毒成分的复制、病毒粒子的装配与释放等一系列步骤[1].其中侵入过程是病毒生命周期的第一个关键步骤,通过病毒囊膜与宿主细胞膜的融合从而使病毒进入细胞质中.由于两个生物膜间的水化层存在斥力,脂质双分子层的融合过程不会自发发生,需要额外的能量用于克服斥力并改变膜结构.包膜病毒利用病毒表面的融合蛋白(fusion protein)介导融合,融合蛋白都存在一段高度保守的疏水序列,称为融合肽(fusion peptide, FP).在融合过程中,融合肽靠近并插入宿主细胞膜,把宿主细胞膜跟病毒包膜拉近,从外层至内层逐步融合形成融合孔,最终将两个独立的膜结构融合成一个更大的膜结构[2].
甲型流感病毒命名中的H代表流感病毒的融合蛋白--血凝素(hemagglutinin,HA).HA是流感病毒的主要抗原,共有16个亚型,可以引起宿主强烈的免疫反应.为逃避宿主的免疫系统监视,HA不断突变,如1968年香港型流感病毒(Hong Kong Flu)的HA每年以3.4个氨基酸的速度突变[3],但是融合肽却高度保守,若将这段保守序列切除后病毒无法与宿主细胞膜融合,即使做保守的点突变也会使病毒丧失融合功能.因此在原子分子水平上研究膜融合蛋白的融合肽结构域既有助于我们认识病毒侵染过程的本质, 又有利于有针对性地设计药物, 达到预防与治疗病毒病的目的.血凝素亦是现阶段了解较为透彻的融合蛋白,对其他种类病毒融合蛋白的研究具有非常强的借鉴意义.下面将详细介绍流感病毒融合肽的结构特征和融合机制.
1 流感病毒融合蛋白的空间结构血凝素HA是流感病毒表面分布最多的膜蛋白,在病毒表面呈刺突状且有糖基化修饰,又被称为刺突状糖蛋白[4].HA前体(precursor) HA0通常以三聚体存在,每个单体由受体结合亚基HA1(328个氨基酸)和融合蛋白亚基HA2(221个氨基酸)组成,两个亚基经二硫键相连[5-7].每个HA1分子形成球状的“头部”(head)区域,与唾液酸受体结合启动内吞途径;主干由3个HA2拥簇形成“茎”(stem)状的三链α-螺旋之卷曲结构(α-helical coiled-coil).HA0经酶切后结构基本未变,但在HA2的N端产生一段疏水序列,即融合肽[8][图 1(a), (b)],包埋在HA2亚基的疏水环境中.切割后的HA处于亚稳态(metastable)[9],当被低pH诱导时,HA1球状亚基解聚,暴露出HA2亚基[10].HA2构象发生重排[图 1(c), (d)],一段环结构(loop)变成α-螺旋,将原有的α-螺旋结构延长,使融合肽暴露并前移约10 nm,插入到宿主细胞膜中,这个过程是不可逆的[11, 12].HA2的另一段螺旋则转变为环结构,使C端翻转180°并与N端靠近,迫使宿主膜(由N端融合肽牵拉)和病毒膜(C端跨膜区牵拉)的外层膜融合(hemifusion),进一步内层膜融合,最后形成融合孔洞(fusion pore).融合后的HA是三聚体发夹结构,被认为是低能量的稳定结构[9, 13].HA的融合效率与HA的密度有关,未与宿主细胞接触的HA有助于融合孔洞的扩大,提示在融合过程中HA之间的存在协同作用[14-16].

|
| 图 1 (a)血凝素前体HA0的一个单体;(b) HA0单体经宿主酶切割后形成HA1,HA2两个亚基,融合肽用黑色标示;(c)中性环境下的HA2单体;(d)融合后HA2单体的构象(根据PDB结构重新作图) Fig. 1 (a) Monomer of hemagglutinin HA0; (b) monomer of HA0 cleavaged by human protease resulting in HA1 and HA2 subunit and fusion peptide colored black; (c) the conformation of HA2 in the nentral pH condition; (d) the conformation of HA2 in the acidic condition |
流感病毒融合肽位于HA2结构域的N末端,是一段大约20多个氨基酸的疏水序列,实验证实这段序列是HA中唯一插入宿主细胞膜的序列[12, 17, 18].融合肽序列十分保守,尽管某些关键氨基酸的突变不影响流感病毒HA的表达和酶切反应及低pH下诱导的构象变化,但是会影响HA的融合功能[19-22].例如把Gly1突变为Ser只有半融合(hemifusion)功能(即外层膜融合,内层膜仍保持独立状态),Gly1突变为Val,Glu,Arg,Gln,Phe或Leu时失去融合功能.融合肽的长度对融合也很重要,把Gly1或Leu2删除都会失去融合活性.综述文献[23]对这些突变与融合功能的关系做了全面的介绍.
流感病毒融合肽通过一个柔性的铰链区与HA2其它部分相连,且在膜中的折叠过程独立于其它部分[24],因此有人把融合肽称为融合结构域(fusion domain).实验证实单独的一段融合肽就可以诱导脂质体融合,表明融合肽的确参与了融合,并且可以独立地执行融合功能[18, 25, 26].既然融合肽不仅仅是将HA“锚定”到宿主细胞膜上,它具有明确的融合功能,解析融合肽的空间结构是了解其融合机制的关键.
3 流感病毒融合肽的空间结构由于融合肽序列疏水性很强,为了制备融合蛋白的晶体,通常要把融合肽切除,因此用X-ray晶体衍射法研究融合肽结构是不完整的.相比之下,一些谱学的方法,如电子顺磁共振(Electron paramagnanetic Resonance, EPR),傅里叶转换红外线光谱(Fourier Transform infrared spectroscopy, FTIR)和核磁共振技术(Nuclear Magnetic Resonance, NMR)等更适合研究膜环境中的融合蛋白的结构.在这些方法中,核磁共振方法可以获得融合肽的精细构象与动力学性质,已成为融合肽研究的主要手段.
流感病毒融合肽富含甘氨酸.甘氨酸之间一般间隔3个其它氨基酸,暗示可能存在螺旋结构[27, 28].2000年Chang等人解析了酸性条件下H3亚型25个氨基酸长度的融合肽在SDS(Sodium dodecyl sulfate)胶束中的NMR结构,融合肽呈直棒状α螺旋,N端入膜,16~18位氨基酸位于胶束和溶液界面上[29].同年Dubovsk Ⅱ解析了DPC胶束中20个氨基酸的E5融合肽NMR结构(将第4,8,15,18位氨基酸突变为Glu,因为序列里含有5个Glu被称为E5,有融合活性),发现在酸性环境下该融合肽呈近似直棒状的两亲性α-螺旋结构,疏水面朝向胶束,亲水面朝向水溶液[30].2002年Chang等人解析了酸性条件下SDS胶束中E5和E5的两个功能减弱的突变体G13L和L17A的结构,发现E5呈螺旋-转角-螺旋结构,而两个突变体都呈线型结构[33].随着NMR实验技术的发展,融合肽的结构研究受到重视,相关文献十分丰富,其中Tamm和Bax两个小组的研究结果有系统性,他们也各自在NMR结构解析的基础上提出了不同的融合模型.
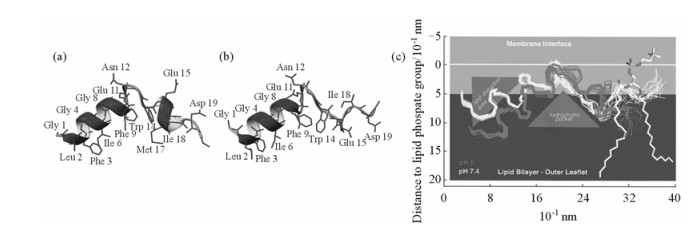
3.1 Boomerang构象融合模型Tamm研究组在H3亚型的20个氨基酸的融合肽C端加上一段亲水序列GCGKKK K,获得了具有诱导红细胞溶血和脂质体融合活性的融合肽(命名为H3-HAfp20)[26].他们用同核谱解析了pH=7.4/5.0条件下DPC胶束中的融合肽结构,发现两个pH条件下融合肽都呈倒“V”状结构,pH=7.4结构的N端α-螺旋终止于Ⅱe10,随后Asn12和Gly13形成转角,Trp14-Gly20是一段无规的卷曲结构[图 2(a)];而pH=5.0条件下融合肽N端α-螺旋延长到Glu11,C端Glu15-IIe18形成了310-螺旋,Glu15和Asp19从一侧旋转到另一侧,使得310-螺旋呈两亲性[图 2(b)].结果显示在低pH条件下由于Glu15和Asp19的旋转,产生一段两亲性螺旋,在倒“V”状结构的底部形成了一个疏水“口袋”.
|
| 图 2 H3-P20H7在DPC中的结构与在脂双层膜中对接的结构.(a) pH=5.0的结构;(b) pH=7.4的结构,残基侧链用短线表示;(c) H3-P20H7在脂双层膜中对接图,白色线表示pH=5.0结构;灰色线表示pH=7.0结构,右侧白色结构是磷脂分子[32] Fig. 2 Structures of the H3-P20H7 in DPC micelles and docking of the NMR structures to the lipid bilayers; (a) pH=5.0 structure with side chain shown as stick models; (b) pH=7.4 structure with side chain shown as stick models; (c) the 20 lowest energy conformers of the H3-P20H7 positioned into lipid bilayers by the best fits to EPR data at pH=5.0(gray) and pH=7.4(white), a conformer of a phospholipid is shown on the right[32] |
倒“V”状融合肽结构的下侧形成了疏水区域,可能与DPC的疏水核心有相互作用.虽然DPC胶束可以模拟生物膜环境,但是DPC胶束的曲率很大且组成与真实的生物膜不同,为了探究在DPC胶束中得到的结构能否真实反映融合肽在生物膜中的结构,作者用电子顺磁共振实验(electron paramagnetic resonance,EPR)进行了验证[32].EPR可以给出大分子体系如生物膜中蛋白质结构的低分辨率信息,验证NMR解析的结构在生物膜中是否发生改变.由于EPR测定未成对电子在磁场中对微波的共振吸收信号,因此需要在融合肽中引入半胱氨酸残基将顺磁探针连接到特定位置.甲硫代磺酸(MTSSL)是最常见的自旋标记物,对样品结构基本没干扰.通过功率饱和电子顺磁共振实验(power-saturation EPR)可以获得探针在膜中深度的信息,实验误差在±0.2 nm[31].作者将融合肽的每个氨基酸逐一进行标记,除了两个位点连接MTSSL导致活性丧失,其余的18个位点均可以诱导红细胞溶血且融合肽结构未改变.EPR实验数据证实,两个pH条件下融合肽在脂双层中都保持倒“V”状结构,pH=5.0时融合肽N端和C端比pH=7.4时插膜更深,最深处可达脂双层的中部(脂双层的厚度在4 nm左右),两种pH条件下融合肽转角部位Glu11和Asn12在脂双层膜中的位置基本一致.EPR方法提供了融合肽在脂双层中的低分辨率结构,验证了在DPC胶束中解析的NMR结构可以真实反映在生物膜环境中的状态,此外还提供了融合肽在生物膜中的插膜深度信息[图 2(c)],证实低pH条件下融合肽入膜更深,与膜作用更强[32].
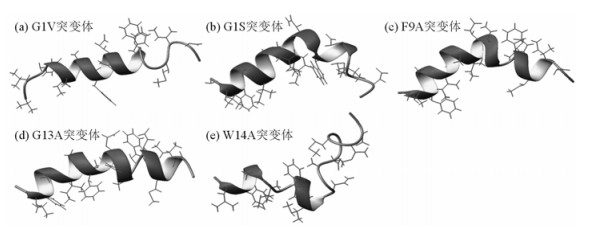
通过对突变体的研究可以更加全面深入地了解融合肽结构与功能的关系.Tamm小组相继解析了一系列突变体结构[34-38],其中突变体G1V没有融合能力,NMR结果显示融合肽的倒“V”状结构消失,取而代之的是一个直棒状结构,Gly4-Trp14形成规则的α-螺旋[图 3(a)].G1S突变体只能使细胞膜形成半融合状态,其NMR结构基本未变,仍保持螺旋-转角-螺旋的两亲性结构[图 3(b)].在这些结构的基础上,Tamm提出融合肽的倒“V”状结构可能是膜融合过程的必要条件[34].为了验证这个设想,Tamm解析了倒“V”状结构转角处几个关键氨基酸的突变体结构.W14A突变体没有融合功能,结构呈倒“V”状[图 3(e)],但收敛性很差,而且EPR实验发现其N端平行于膜界面插入膜中,C端指向膜外,与野生型的插膜方向不同[35].F9A突变体有融合功能,其结构与野生型极为类似[图 3(c)],入膜较深[36].G13A突变体在融合过程中会导致脂质体内溶物的泄露,其结构呈倒“V”状,但是倒“V”的夹角较野生型更大[图 3(d)][38].这些数据都极好地支持了Boomerang构象的融合模型.
Tamm小组还通过连续波电子顺磁共振实验(continuous wave EPR)和脉冲双电子电子共振(pulsed double electron electron resonance,DEER)实验测量了野生型,F9A,W14A和F9A/I10A突变体第3和8位氨基酸距离的分布.发现野生型和F9A突变体的两个探针间的距离在2.4 nm左右,而F9A/I10A和W14A突变体的距离分布在2.1~3.3 nm,显示有融合功能的融合肽比没有融合功能的突变体运动更为受限,提示倒“V”状转角的柔性可能会影响活性[37].热力学计算发现融合过程中熵(enthalpy)和自由能(free energy)的变化与融合活性都有很好的相关性[39].
|
| 图 3 H3-P20H7突变体在酸性条件下的结构,N末端在左边,侧链用短线表示. (a) G1V突变体;(b) G1S突变体;(c) F9A突变体;(d) G13A突变体;(e) W14A突变体 Fig. 3 A. Structures of H3-P20H7 mutants at acidic condition and N termini are on the left with side chain shown as stick models. (a) G1V mutant; (b) G1S mutant; (c) F9A mutant; (d) G13A mutant; (e) W14A mutant |
融合肽行使功能的一个重要结果就是对细胞膜的扰动.Tamm等人通过衰减全反射傅里叶红外光谱(FTIR-ATR)分析脂肪酸链的序参数(order parameter),发现有融合活性的融合肽会导致脂质的序参数变大,即运动受限;含有丧失融合活性的融合肽的脂质的序参数较小,说明脂质分子柔性比较大[35, 36, 38].2009年Ge等人通过EPR实验也发现有融合活性的融合肽会导致脂双层的外层脂质序参数变大,突变体则没有变化,验证了脂质运动受限对融合活性的重要性[40].
3.2 螺旋型发夹构象融合模型最近Bax小组对Tamm小组的工作提出质疑:认为倒“V”状结构无法说明融合肽在低pH值条件下带正电荷的N端如何克服在疏水的膜环境中的能量上的不利.他们发现融合肽的C端Trp21-Tyr22-Gly23序列很保守,用重组蛋白表达的方法制备了来自H1亚型的23个氨基酸的融合肽,C端加了SGKKKD亲水标签(命名为H1-HAfp23).2010年Bax小组发表了流感病毒融合肽H1-HAfp23的NMR研究结果[41].1H-13C/1H-15NHSQC和RDC数据显示pH=4.0和pH=7.4条件下融合肽的结构高度相似.作者用异核谱解析了pH=7.4的结构,发现融合肽由两个α-螺旋反平行形成发夹(helical hairpin)结构[图 4(a)],两个螺旋的CαH和CO之间形成多个氢键来稳定发夹构象,N端Gly1的NH3+与Gly20的CO有相互作用,维持了发夹的稳定性.整个螺旋呈两亲性分布,氢氘交换实验得知融合肽的疏水面朝向DPC胶束内核,亲水面朝向溶液中[图 4(b)].

|
| 图 4 (a) H1-HAfp23在pH=7.4的结构,主链用飘带表示,侧链用短线表示,Gly用白色范德华半径表示;(b) H1-HAfp23与DPC间NOE强度关系,浅灰色表示存在强的NOE,深灰色表示存在弱的NOE[41];(c) H1-HAfp23在膜中的位置和运动示意图.球棒模型表示H1-HAfp23的主链结构及各残基的NH键.平面表示膜-水界面.右方的坐标系指示定义的xyz轴方向,x轴为螺旋NH键的平均方向,y轴为Gly13的NH方向,z轴垂直于膜-水界面[44] Fig. 4 (a) H1-HAfp23 structure at pH=7.4 with mainchain represented by ribbon and sidechain by stick model, and Gly residues shown in white van der Waals representation; (b) NOE intensity between the DPC methylene protons and the backbone HN protons of the fusion peptide mapped on the structure, with light grey corresponding to strongest NOEs[41]; (c) Cα backbone representation (including backbone amide 15N-1H pairs) for H1-HAfp23 and its approximate location relative to the plane of the water-lipid interface. The axis system shown has its x axis parallel to the average N-H vector orientation of residues 3~12 and 14~22 and its z axis orthogonal to the plane. The Gly13 N-H vector is nearly parallel to the y axis[44] |
在后续的研究中,Bax小组通过15N化学位移拟合出融合肽Gly1的NH3+基团的pKa值为8.80,比普通溶液中蛋白质或多肽N端NH3+基团的pKa (7.5~8.0)大很多,而pH滴定实验发现融合肽C端的羰基碳原子的化学位移有变化,认为融合肽的N端与C端形成了电荷-偶极相互作用,合理地解释了N端Gly1保守的原因[43].他们改变bicelle的大小,用15N弛豫[44]测定实验研究了融合肽的运动特性,发现融合肽整体发生大幅度摇摆运动,幅度大约为绕x轴摆幅16°,绕y轴的摆幅20°[45] [图 4(c)],时间尺度在纳秒量级.在测量H1-HAfp23融合肽的主链15N弛豫速率时发现两个pH条件下融合肽的结构都很刚性,但是pH=4.0时横向弛豫速率(R2)较快,提示在低pH条件下融合肽还有其它低丰度构象存在,于是设计了G8A突变体来研究不同构象的特征及分布情况.通过化学位移比对,RDC实验[46]和NOE强度分析等手段,Bax等人发现G8A突变体存在两个以上的构象,其中高丰度构象[图 5(a), (b)]呈伸展或“L”状结构,约占85%左右,低丰度构象[图 5(c)]呈螺旋型发夹结构,约占15%左右.野生型H1-HAfp23在12℃酸性条件下的T1ρ-CPMG实验发现有20%构象是打开的结构,与G8A突变体的高丰度构象相似,由此间接证明了在酸性条件下H1-HAfp23处于亚毫秒量级的构象交换状态,打开和闭合的发夹结构同时存在[25].这一点亦被最近发表的H3亚型20氨基酸融合肽的固体NMR实验所证实[42].

|
| 图 5 H3-HAfp23-G8A的3种构像.(a)伸展构象;(b) “L”构象;(c)螺旋发夹结构构象;(d) 3种构象的叠加,主链3~10位氨基酸用于叠加[25] Fig. 5 Structures for the three-conformer ensembles of the H3-HAfp23-G8A, and ribbon model representations of the three conformers taken from the ensemble with lowest total energy. (a) The extended conformations; (b) the L-shaped conformations; (c) helical hairpin conformations; (d) the structures have been aligned to yield minimal coordinate rmsd for the backbone heavy atoms of residues 3~10 for each conformer[25] |
H1-HAfp23和H3-HAfp20在体外都可以将脂质体融合,都是独立的功能片段,但三维结构有很大区别,为了了解这两个结构之间的关联,Bax小组设计了以下不同长度的融合肽序列进行比较:
H3-HAfp20:GLFGAIAGFIENGWEGMIDGSGKKKKD
H1-HAfp14: GLFGAIAGFIEGGWKKKKD
H1-HAfp20:GLFGAIAGFIEGGWTGMIDGSGKKKKD
H1-HAfp23:GLFGAIAGFIEGGWTGMIDGWYGSGKKKKD
H1-HAfp28:GLFGAIAGFIEGGWTGMIDGWYGYHHQNSGKKKKD
1H-15NHSQC谱图显示H3-HAfp20和H1-HAfp20的化学位移很接近,而H1-HAfp20和H1-HAfp14,H1-HAfp23与H1-HAfp28的化学位移分别近似.表明两个短序列(H1-HAfp20/H1-HAfp14)结构很相似,两个长序列(H1-HAfp23/H1-HAfp28)结构很相似,但是短序列结构与长序列结构差别很大,这个结论也得到15N弛豫数据的支持.通过比较H1-HAfp23和H1-HAfp20的NOE谱交叉峰,发现H1-HAfp20存在低丰度的发夹结构.由于在快交换体系下化学位移信息可以反映构象的百分比,作者比较了这些不同长度融合肽的化学位移值.H1-HAfp14是打开结构(即螺旋结构),H1-HAfp23是闭合结构(发夹结构),而H1-HAfp20化学位移在H1-HAfp14和H1-HAfp23化学位移的加权平均处,拟合得到打开结构约占90%左右,闭合结构占10%左右[47].这个结果具有重要意义,它指出流感病毒融合肽同时具有打开和闭合的发夹构象,不同构象之间不断交换.不同长度的融合肽反映的构象交换比例不同,而NMR捕捉到的是占主导地位的平均构象.这种结构之间的内在联系,合理地解释了前期不同研究者发表的融合肽结构有巨大差别的现象,也提示C端Trp21-Tyr22-Gly23保守序列对稳定发夹结构的重要性.
我们注意到,Bax与Tamm小组研究的流感病毒融合肽分属不同的亚型,H1和H3亚型的融合肽一级序列高度相似,区别是H1亚型的第12位氨基酸是Gly12,第15位是Thr15,而H3亚型的相应位置是Asn12和Glu15.无论在Boomerang结构还是发夹结构中这两个氨基酸均位于螺旋的转角处,其中Thr15和Glu15的侧链朝向溶液不会对结构有影响,而Asn12侧链面向C端螺旋,可能会对整体结构产生影响.由于16种亚型一级序列中Gly12/Asn12突变非常保守,研究H3亚型23个氨基酸的长链融合肽的结构,将有助于进一步理解融合肽结构的保守性.我们表达并制备了H3亚型的23个氨基酸的融合肽,在C端加了一段GKKKKD的亲水标签(命名为H3-HAfp23).通过异核谱解析了该融合肽在DPC胶束中的结构,H3-HAfp23呈典型的发夹构象,反平行的两个螺旋间形成了多个氢键,其N端与C端残基间也有明显的NOE相互作用.Asn12位于螺旋转角处,由于空间位阻,与H1-HAfp23的发夹结构相比,H3-HAfp23的第二个α-螺旋整体下移,导致Asn12和Gly16的Cα间距扩大(0.482 nm),而在H1-HAfp23结构中Gly12和Gly16的Cα间距仅为0.370 nm [图 6(a)].由此可见,螺旋型发夹结构在HA所有亚型中是十分保守的.为了进一步了解融合肽C端Trp21-Tyr22-Gly23序列在结构中的作用,我们解析了H3-HAfp23的突变体G1S和G1V在DPC胶束中的NMR结构,发现半融合活性的G1S突变体其N末端和C末端有较弱的氢键相互作用[图 6(b)],而没有融合活性的G1V突变体的N末端和C末端距离较远,没有相互作用,发夹结构被打开[图 6(c)].H3-HAfp23、G1S和G1V三个结构的N端C端之间的距离依次为0.52 nm、0.90 nm和1.40 nm.Mn2+滴定实验显示H3-HAfp23和G1S突变体入膜较G1V突变体更深.这些实验结果揭示了稳定的螺旋型发夹结构对融合肽的活性起到了关键性的作用[48].

|
| 图 6 H3-HAfp23及其突变体在pH=5.0条件下的NMR结构.(a) H1-HAfp23(深灰色)和H3-HAfp23(浅灰色)的主链叠加图(Leu2-Glu11用于叠加);(b) G1S突变体,侧链用短线标示;(c) G1V突变体,侧链用短线标示 Fig. 6 NMR structures of H3-HAfp23 and its mutants at pH=5.0. (a) Superimposed backbone stick structure of HAfp23 from H1-subtype (light grey) and H3-subtype (dark grey). The Leu2-Glu11 fragment is used for fitting and the sidechains of Asn12 and Glu15are highlighted. (b) The ribbon structure of G1S mutant with sidechains by stick. (c) The ribbon structure of G1V mutant with sidechains by stick |
流感病毒融合肽在流感病毒的感染阶段起重要作用,研究融合肽的结构与功能的关系有重要的生物学意义.Tamm和Bax等研究小组运用NMR实验技术解析了融合肽的结构,比较了不同pH条件、不同溶剂介质、不同肽链长度、以及各关键位点的突变对结构和功能的影响.在融合肽的研究过程中充分体现了核磁共振技术的逐步推进,从简单的同核实验到借助同位素标记的异核实验解析融合肽结构,现阶段更是采用了先进的弛豫和RDC技术对融合肽的动力学性质给予了充分的阐释,展示了核磁共振方法在这一领域的强大技术优势.研究揭示了流感病毒融合肽三维结构的保守性,结构的动态变化对融合功能的重要影响,但是仍有很多问题有待解决.比如融合肽的打开构象如何扰动细胞膜?融合肽的三聚结构在融合过程中起什么作用?膜的结构在与融合肽的相互作用中发生了怎样的变化?全长融合蛋白中融合肽以外的其他部分在融合过程中与融合肽是否有相互作用等一系列问题都有待探索解答.相信随着实验技术的不断发展和完善,这些问题终将被破译和诠释.
| [1] | Webster R G, Granoff A . Encyclopedia of Virology[M]. London: Academic Press Ltd, 1994 . |
| [2] | Harrison S C . Viral membrane fusion[J]. Nat Struct Mol Biol , 2008, 15 (7) : 690-698 DOI:10.1038/nsmb.1456 |
| [3] | Bean W, Schell M, Katz J et al . Evolution of the H3influenza virus hemagglutinin from human and nonhuman hosts[J]. J Virol , 1992, 66 (2) : 1129-1138 |
| [4] | White J, Helenius A, Gething M . Haemagglutinin of influenza virus expressed from a cloned gene promotes membrane fusion[J]. Nature , 1982, 300 (5893) : 658-659 DOI:10.1038/300658a0 |
| [5] | Klenk H D, Rott R, Orlich M et al . Activation of influenza a viruses by trypsin treatment[J]. Virology , 1975, 68 (2) : 426-439 DOI:10.1016/0042-6822(75)90284-6 |
| [6] | Huang R, Wahn K, Klenk H D et al . Fusion between cell membrane and liposomes containing the glycoproteins of influenza virus[J]. Virology , 1980, 104 (2) : 294-302 DOI:10.1016/0042-6822(80)90334-7 |
| [7] | Huang R T, Rott R, Klenk H D . Influenza viruses cause hemolysis and fusion of cells[J]. Virology , 1981, 110 (1) : 243-247 DOI:10.1016/0042-6822(81)90030-1 |
| [8] | Skehel J J, Waterfield M D . Studies on the primary structure of the influenza virus hemagglutinin[J]. Proc Natl Acad Sci USA , 1975, 72 (1) : 93-97 DOI:10.1073/pnas.72.1.93 |
| [9] | Chen J, Wharton S A, Weissenhorn W et al . A soluble domain of the membrane-anchoring chain of influenza virus hemagglutinin (HA2) folds in Escherichia coli into the low-pH-induced conformation[J]. Proc Natl Acad Sci USA , 1995, 92 (26) : 12205-12209 DOI:10.1073/pnas.92.26.12205 |
| [10] | Stegmann T, Delfino J, Richards F et al . The HA2subunit of influenza hemagglutinin inserts into the target membrane prior to fusion[J]. J Biol Chem , 1991, 266 (27) : 18404-18410 |
| [11] | Carr C M, Kim P S . A spring-loaded mechanism for the conformational change of influenza hemagglutinin[J]. Cell , 1993, 73 (4) : 823-832 DOI:10.1016/0092-8674(93)90260-W |
| [12] | Durrer P, Galli C, Hoenke S et al . H+-induced membrane insertion of influenza virus hemagglutinin involves the HA2amino-terminal fusion peptide but not the coiled coil region[J]. J Biol Chem , 1996, 271 (23) : 13417-13421 DOI:10.1074/jbc.271.23.13417 |
| [13] | Carr C M, Chaudhry C, Kim P S . Influenza hemagglutinin is spring-loaded by a metastable native conformation[J]. Proc Natl Acad Sci USA , 1997, 94 (26) : 14306-14313 DOI:10.1073/pnas.94.26.14306 |
| [14] | Leikina E, Mittal A, Cho M S et al . Influenza hemagglutinins outside of the contact zone are necessary for fusion pore expansion[J]. J Biol Chem , 2004, 279 (25) : 26526-26532 DOI:10.1074/jbc.M401883200 |
| [15] | Blumenthal R, Sarkar D P, Durell S et al . Dilation of the influenza hemagglutinin fusion pore revealed by the kinetics of individual cell-cell fusion events[J]. J Cell Biol , 1996, 135 (1) : 63-71 DOI:10.1083/jcb.135.1.63 |
| [16] | Floyd D L, Ragains J R, Skehel J J et al . Single-particle kinetics of influenza virus membrane fusion[J]. Proc Natl Acad Sci USA , 2008, 105 (40) : 15382-15387 DOI:10.1073/pnas.0807771105 |
| [17] | Harter C, James P, B?chi T et al . Hydrophobic binding of the ectodomain of influenza hemagglutinin to membranes occurs through the "fusion peptide"[J]. J Biol Chem , 1989, 264 (11) : 6459-6464 |
| [18] | Lear J D, DeGrado W F . Membrane binding and conformational properties of peptides representing the NH2terminus of influenza HA-2[J]. J Biol Chem , 1987, 262 (14) : 6500-6505 |
| [19] | Qiao H, Armstrong R T, Melikyan G B et al . A specific point mutant at position 1of the influenza hemagglutinin fusion peptide displays a hemifusion phenotype[J]. Mol Biol Cell , 1999, 10 (8) : 2759-2769 DOI:10.1091/mbc.10.8.2759 |
| [20] | Gething M J, Doms R W, York D et al . Studies on the mechanism of membrane fusion: site-specific mutagenesis of the hemagglutinin of influenza virus[J]. J Cell Biol , 1986, 102 (1) : 11-23 DOI:10.1083/jcb.102.1.11 |
| [21] | Steinhauer D A, Wharton S A, Skehel J J et al . Studies of the membrane fusion activities of fusion peptide mutants of influenza virus hemagglutinin[J]. J Virol , 1995, 69 (11) : 6643-6651 |
| [22] | Cross K J, Wharton S A, Skehel J J et al . Studies on influenza haemagglutinin fusion peptide mutants generated by reverse genetics[J]. EMBO J , 2001, 20 (16) : 4432-4442 DOI:10.1093/emboj/20.16.4432 |
| [23] | Cross K J, Langley W A, Russell R J et al . Composition and functions of the influenza fusion peptide[J]. Protein Peptide Lett , 2009, 16 (7) : 766-778 DOI:10.2174/092986609788681715 |
| [24] | Chen J, Skehel J J, Wiley D C . N-and C-terminal residues combine in the fusion-pH influenza hemagglutinin HA2subunit to form an N cap that terminates the triple-stranded coiled coil[J]. Proc Natl Acad Sci USA , 1999, 96 (16) : 8967-8972 DOI:10.1073/pnas.96.16.8967 |
| [25] | Lorieau J L, Louis J M, Schwieters C D et al . pH-triggered, activated-state conformations of the influenza hemagglutinin fusion peptide revealed by NMR[J]. Proc Natl Acad Sci USA , 2012, 109 (49) : 19994-19999 DOI:10.1073/pnas.1213801109 |
| [26] | Han X, Tamm L K . A host-guest system to study structure-function relationships of membrane fusion peptides[J]. Proc Natl Acad Sci USA , 2000, 97 (24) : 13097-13102 DOI:10.1073/pnas.230212097 |
| [27] | Russ W P, Engelman D M . The GxxxG motif: a framework for transmembrane helix-helix association[J]. J Mol Biol , 2000, 296 (3) : 911-919 DOI:10.1006/jmbi.1999.3489 |
| [28] | Kleiger G, Grothe R, Mallick P et al . GXXXG and AXXXA: common a-helical interaction motifs in proteins, particularly in extremophiles[J]. Biochemistry , 2002, 41 (19) : 5990-5997 DOI:10.1021/bi0200763 |
| [29] | Chang D, Cheng S, Yang S . The amino-terminal region of the fusion peptide of influenza virus hemagglutinin HA2inserts into sodium dodecyl sulfate micelle with residues 16-18at the aqueous boundary at acidic pH. Oligomerization and the conformational flexibility[J]. J Biol Chem , 2000, 275 (25) : 19150-19158 DOI:10.1074/jbc.M907148199 |
| [30] | DubovskII P V, Li H, Takahashi S et al . Structure of an analog of fusion peptide from hemagglutinin[J]. Protein Sci , 2000, 9 (4) : 786-798 |
| [31] | Altenbach C, Greenhalgh D A, Khorana H G et al . A collision gradient method to determine the immersion depth of nitroxides in lipid bilayers: application to spin-labeled mutants of bacteriorhodopsin[J]. Proc Natl Acad Sci USA , 1994, 91 (5) : 1667-1671 DOI:10.1073/pnas.91.5.1667 |
| [32] | Han X, Bushweller J H, Cafiso D S et al . Membrane structure and fusion-triggering conformational change of the fusion domain from influenza hemagglutinin[J]. Nat Struct Mol Biol , 2001, 8 (8) : 715-720 DOI:10.1038/90434 |
| [33] | Hsu C H, Wu S H, Chang D K et al . Structural characterizations of fusion peptide analogs of influenza virus hemagglutinin Implication of the necessity of a helix-hinge-helix motif in fusion activity[J]. J Biol Chem , 2002, 277 (25) : 22725-22733 DOI:10.1074/jbc.M200089200 |
| [34] | Li Y, Han X, Lai A L et al . Membrane structures of the hemifusion-inducing fusion peptide mutant G1S and the fusion-blocking mutant G1V of influenza virus hemagglutinin suggest a mechanism for pore opening in membrane fusion[J]. J Virol , 2005, 79 (18) : 12065-12076 DOI:10.1128/JVI.79.18.12065-12076.2005 |
| [35] | Lai A L, Park H, White J M et al . Fusion peptide of influenza hemagglutinin requires a fixed angle boomerang structure for activity[J]. J Biol Chem , 2006, 281 (9) : 5760-5770 |
| [36] | Lai A L, Tamm L K . Locking the kink in the influenza hemagglutinin fusion domain structure[J]. J Biol Chem , 2007, 282 (33) : 23946-23956 DOI:10.1074/jbc.M704008200 |
| [37] | Tamm L K, Lai A L, Li Y . Combined NMR and EPR spectroscopy to determine structures of viral fusion domains in membranes[J]. Biochimica et Biophysica Acta (BBA)-Biomembranes , 2007, 1768 (12) : 3052-3060 DOI:10.1016/j.bbamem.2007.09.010 |
| [38] | Lai A L, Tamm L K . Shallow boomerang-shaped influenza hemagglutinin G13A mutant structure promotes leaky membrane fusion[J]. J Biol Chem , 2010, 285 (48) : 37467-37475 DOI:10.1074/jbc.M110.153700 |
| [39] | Tamm L K . Hypothesis: spring-loaded boomerang mechanism of influenza hemagglutinin-mediated membrane fusion[J]. Biochimica et Biophysica Acta (BBA)-Biomembranes , 2003, 1614 (1) : 14-23 DOI:10.1016/S0005-2736(03)00159-7 |
| [40] | Ge M, Freed J H . Fusion peptide from influenza hemagglutinin increases membrane surface order: an electron-spin resonance study[J]. Biophys J , 2009, 96 (12) : 4925-4934 DOI:10.1016/j.bpj.2009.04.015 |
| [41] | Lorieau J L, Louis J M, Bax A . The complete influenza hemagglutinin fusion domain adopts a tight helical hairpin arrangement at the lipid: water interface[J]. Proc Natl Acad Sci USA , 2010, 107 (25) : 11341-11346 DOI:10.1073/pnas.1006142107 |
| [42] | Ghosh U, Xie L, Weliky D P . Detection of closed influenza virus hemagglutinin fusion peptide structures in membranes by backbone 13CO-15N rotational-echo double-resonance solid-state NMR[J]. J Biomol NMR , 2013, 55 (2) : 139-146 DOI:10.1007/s10858-013-9709-y |
| [43] | Lorieau J L, Louis J M, Bax A . Helical hairpin structure of influenza hemagglutinin fusion peptide stabilized by charge-dipole interactions between the N-terminal amino group and the second helix[J]. J Am Chem Soc , 2011, 133 (9) : 2824-2827 DOI:10.1021/ja1099775 |
| [44] | Hu Yun-fei(胡蕴菲), Jin Chang-wen(金长文) . NMR studies of protein structures and dynamics(蛋白质溶液结构及动力学的核磁共振研究)[J]. Chinese J Magn Reson(波谱学杂志) , 2009, 26 (2) : 151-172 |
| [45] | Lorieau J L, Louis J M, Bax A . Whole-body rocking motion of a fusion peptide in lipid bilayers from size-dispersed 15N NMR relaxation[J]. J Am Chem Soc , 2011, 133 (36) : 14184-14187 DOI:10.1021/ja2045309 |
| [46] | Lin Dong-hai(林东海), Liu Xue-hui(刘雪辉) . Residual dipolar couplings and their applications in determination of protein structures(残留偶极耦合及其在蛋白质结构研究中的应用)[J]. Chinese J Magn Reson(波谱学杂志) , 2005, 22 (1) : 85-98 |
| [47] | Lorieau J L, Louis J M, Bax A . The impact of influenza hemagglutinin fusion peptide length and viral subtype on its structure and dynamics[J]. Biopolymers , 2013, 99 (3) : 189-195 DOI:10.1002/bip.v99.3 |
| [48] | Du T P, Jiang L, Liu M L . NMR structures of fusion peptide from influenza hemagglutinin H3-subtype and its mutants[J]. Journal of Peptide Science , 2014, 20 (4) : 292-297 DOI:10.1002/psc.v20.4 |
 2014, Vol. 31
2014, Vol. 31



