核磁共振(Nuclear Magnetic Resonance,NMR)纯化学位移(又称“同核宽带去偶”)技术能够明显提高NMR重叠谱图的分辨率.在过去的几年里,研究人员对这一技术的发展和应用产生了浓厚兴趣,提出了许多新的纯化学位移方法,并进行了应用[1-4].本文主要讨论了三个问题:纯化学位移技术是如何发展的,它目前发展到了什么程度,以及在接下来的发展和应用中它将会遇到怎样的挑战.
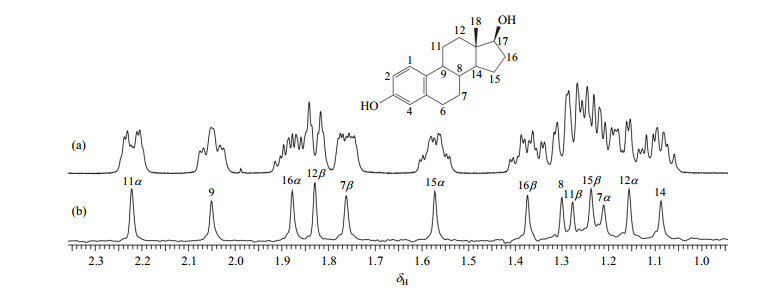
在NMR波谱技术中,标量偶合(J couplings)能够为分子结构解析提供关键信息,但由它引起的谱峰裂分往往会使谱图信息复杂化.在1H NMR实验中,氢核有限的化学位移分布范围(约为10 ppm),以及氢核之间相互的标量偶合(JHH couplings)引起的谱峰裂分往往使得复杂样品的1H NMR谱图存在谱峰拥挤重叠和谱图分辨率低的问题.纯化学位移技术的主要目的就是消除标量偶合对谱图所产生的影响.通过这一实验技术,谱图中由于标量偶合裂分产生的多重峰可转化为单峰,从而简化谱图信息,降低谱图解析的难度.纯化学位移1H NMR谱图的优势如图 1所示,去除了复杂的标量偶合的影响后,每组磁等价质子的共振峰均为单峰,谱峰重叠现象消除,谱图重叠区域的分辨率大幅提高,对谱图中复杂区域的解析也因此得以简化.
|
图 1 溶于DMSO-d6的雌二醇样品的(a)常规1H NMR谱(局部)和(b)纯化学位移1H NMR谱(局部).实验在Varian 500 MHz谱仪下完成,配备了含有xyz三维自屏蔽梯度线圈的5 mm间接检测探头,实验温度为295 K Fig. 1 The partial (a) conventional and (b) pure shift 1H NMR spectra of an estradiol sample in DMSO-d6. The experiment was performed at 295 K using a Varian 500 MHz spectrometer, equipped with a 5 mm xyz indirect detection probe |
在NMR发展初期,纯化学位移方法就被提出.1963年,Ernst和Primas提到:“如果能够一次性去除谱图中所有的偶合信息就太完美了[5].”在过去的50多年里,研究人员们陆续提出了一系列纯化学位移波谱方法,这些方法能够有效解决1H NMR谱中谱峰拥挤重叠的问题.
最早提出的用于去除标量偶合的波谱方法,是通过在采样期对单个信号连续进行频选波照射来实现的,但这种方法只能对部分信号实现去偶,而无法获得宽带同核去偶谱[6].此后,第一代纯化学位移方法诞生,研究人员通过对二维谱图进行数据后处理来获得一维纯化学位移谱.1976年,Ernst小组首次提出了基于二维J分解谱获取高分辨率纯化学位移谱的方法,该方法先将二维J分解绝对值谱进行45°旋转,再将其沿间接维进行投影,以获取纯化学位移谱[7].整个操作过程并没有专门的自旋去偶作用,而是通过对二维谱图数据处理后获得纯化学位移谱.这样的纯化学位移一维谱还可通过投影二维谱的间接维来获得(例如恒时演化实验[8]和时间反演实验[9]),或通过投影反z-COSY实验[10]的对角线来获得.
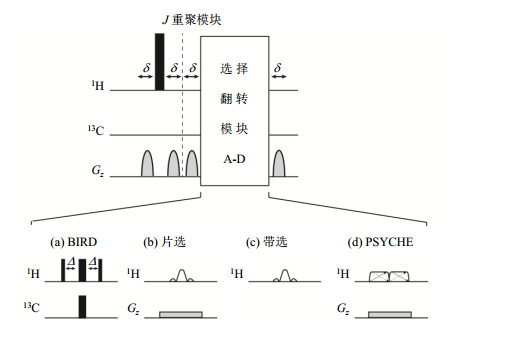
第二代纯化学位移方法通过选择一组核(激发核)并对其进行独立操作,消除激发核与相邻核(偶合核)之间的偶合作用关系,这一过程省去了二维谱复杂的实验操作.1982年,Garbow等[11]基于这一想法首次提出双线性自旋去偶(BIlinear Rotation Decoupling,BIRD)模块[如图 2(a)],该模块对激发核与偶合核分别进行操作,将与13C核相连的1H核从与12C核相连的1H核中区分出来.BIRD模块是选择翻转模块的一种,选择翻转模块与180°硬脉冲组合后称为J重聚模块.J重聚模块能够重聚JHH偶合并保留化学位移信息,是用于设计现代纯化学位移实验最为重要的模块.在实验中,它位于演化时间t1的中心,t1按一定规律不断增加,进行类似于二维采样的实验,从而获得一组二维数据.取这组二维数据的每个自由感应衰减信号(Free-Induction Decay,FID)的前端数据点进行拼接,以产生一个新的FID信号,再对新的FID信号进行傅里叶变换,即可得到宽带同核去偶一维纯化学位移谱.这种采样方式被称为伪二维采样方法(又称干涉图采样方法).在所获得的谱图中只能观察到与13C核相连的1H(激发核)的信号.
|
图 2 基础的J重聚模块,由非选择性180°脉冲和选择性自旋翻转模块组成,可以抑制同核偶合的演化.其中选择翻转模块可以是(a) BIRD模块、(b)片选模块、(c)带选模块和(d) PSYCHE模块(根据文献[3]重制) Fig. 2 Basic J-refocusing elements consisting of a nonselective 180° pulse followed by a selective spin inversion element to suppress the homonuclear J coupling evolution. This selective inversion can be implemented by using (a) a BIRD filter, (b) a slice-selective element, (c) a band-selective pulse or (d) a PSYCHE module (This figure is redrawn according to literature [3]) |
1997年,Zangger和Sterk[12]提出了一种新的选择翻转模块.该模块通过使用空间编码(又称“片选”)的方法来选择性翻转激发核[如图 2(b)].选择性180°脉冲在结合了弱脉冲梯度场(Pulsed Field Gradient,PFG)后,可以实现对样品管不同层所对应的不同信号进行选择性去偶.与BIRD方法类似,为获得一维宽带同核去偶谱,片选实验也需要进行伪二维采样,采样后使用的是每个FID信号的前端小部分的数据块(数据块长度为间接维谱宽的倒数,1/SW1),并通过对脉冲序列的设计使得JHH偶合在该数据块的中点处被重聚[13].在对各个FID信号的前端数据块进行拼接后,可获得新的同核去偶FID信号,经傅里叶变换后即可得一维纯化学位移谱.
2012年,Lupulescu等[14]提出了一个新的采样方法,此采样方法在采样期实时使用J重聚模块(这里使用BIRD模块作为选择翻转模块)来获得新的同核去偶FID信号[如图 3(b)].在采样过程中,JHH偶合被周期性重聚.不同于干涉图采样,此方法的最大优势在于它缩短了纯化学位移方法的实验时间,并且在单位时间内能获得较高的灵敏度.不仅如此,此方法也不需要特殊的数据后处理方法,简单操作就能将其应用于标准一维谱和多维谱的直接维.2013年,Zangger和Meyer[15]在此基础上提出了一种将片选方法和实时采样相结合的新方法,以实现单扫描采样纯化学位移谱.

|
图 3 获得同核去偶FID信号的一般采样框架.(a)二维采样模式的干涉图方法,通过将每个FID信号的前端数据块进行拼接,重构出同核去偶的FID;(b)实时采样模式,将采样模块重复多次以得到完整的FID(根据文献[3]重制) Fig. 3 General acquisition schemes used to obtain homo-decoupled FID. (a) 2D interferogram-mode acquisition where the final homo-decoupled FID is obtained after a reconstruction process where the initial chunks of each increment are concatenated to reconstruct a new FID without J couplings; (b) Real-time-mode acquisition where the acquisition element is repeated n times to collect a complete homo-decoupled FID (This figure is redrawn according to literature [3]) |
不久后,将实时采样与同核带选(HOmonuclear Band-Selective,HOBS)相结合的去偶方法也被提出[16, 17].在此方法中,带选脉冲被用作选择翻转模块[如图 2(c)],对在该范围内不存在偶合关系的共振信号进行选择,从而获得仅包含这些共振信号的纯化学位移谱.HOBS在研究多肽物质和蛋白质上有着很好的效果,这是因为其激发核和偶合核的化学位移出现在不同的区域,因而能被很好的区分开.同时,HOBS也适用于同分异构体的混合物.与上述片选纯化学位移谱和BIRD实时纯化学位移谱不同的是,HOBS不存在灵敏度损失的问题.相反,由于HOBS方法可以将谱图中的多重峰简化为单峰,该方法甚至具有比常规一维1H NMR谱图更高的信噪比(Signal-to-Noise Ratio,SNR).
2014年,Foroozandeh等[18]又提出了一种称为“PSYCHE”(Pure Shift Yielded by Chirp Excitation)的新纯化学位移方法,此方法将两个小角度扫频脉冲(Chirp脉冲)与弱PFG结合,达到选择性翻转激发核的目的[如图 2(d)].这种使用Chirp脉冲产生纯化学位移谱的新方法,可以获得灵敏度较好的宽带同核去偶谱,且该方法相比于其他纯化学位移方法更能克服强偶合作用的影响.
2 纯化学位移存在的问题及其解决方法目前,上述的纯化学位移方法已被成功应用到一维和多维NMR实验中,由此产生的新方法也具有一定的应用潜力,其实用性也已被证实.然而,没有一种NMR实验是完美的,纯化学位移方法也不例外.我们将进一步讨论纯化学位移实验中存在的一些问题与局限,并提出一些解决方法.
2.1 灵敏度灵敏度损失是获得宽带同核去偶谱所需要付出的主要代价之一.一般来说,大多数纯化学位移实验都有低信噪比的缺陷,这是使用选择翻转模块的固有问题.使用BIRD模块[如图 2(a)]时,只有与13C核相连的1H核被选择.由于同位素13C核的自然丰度为1.1%,此方法得到的谱图灵敏度也只有常规1H NMR谱信噪比的1.1%.然而,异核相关谱实验存在特殊情况,当脉冲通道选择的是1H核与13C核时,最终的纯化学位移谱相较于通过常规单脉冲获得的1H NMR谱来说,不存在灵敏度额外损失的问题[19].
在片选方法[如图 2(b)]中,每个信号只来自一小片区域(层)的样品,因而检测到的信号强度取决于片选脉冲的激发带宽,这也导致片选方法的信噪比与常规氢谱相比会产生大幅度的减弱.目前已经有一些技术通过使用等距[20]或非等距[21]相位调制的多色脉冲(多层激发方法),来提高其单位时间灵敏度.而HOBS方法[如图 2(c)]只使用带选脉冲而不使用PFG,没有进行片层选择,可以在去除标量偶合的同时保持信噪比不受影响,因此,理论上使用HOBS方法可以得到一张高灵敏度谱图.
PSYCHE模块[如图 2(d)]的灵敏度主要取决于Chirp脉冲的翻转角度.翻转角度越大,信噪比越高,但同时谱图中也会产生更多的伪峰.因此,在对谱图的质量和灵敏度进行折衷考虑后,所采用的翻转角度一般为10°~30°,对应所得谱图的信噪比是常规单脉冲一维谱图的3%~20%.PSYCHE方法的灵敏度,尽管与常规一维谱相比有所下降,但比起其他伪二维采样的纯化学位移方法,如片选方法和BIRD方法,却有一定程度的提高.表 1是对上述几种选择翻转模块所得到的谱图灵敏度进行的总结性比较.
| 表 1 使用不同选择翻转模块获得的1H NMR谱图相对于常规单脉冲1H NMR谱图的灵敏度(根据文献[3]重制) Table 1 Relative sensitivity 1H NMR spectra acquired using different selective inversion elements compared with that obtained by using a hard pulse (This table is remade according to literature [3]) |
在纯化学位移实验中,选择翻转模块对谱图的灵敏度起到了关键性作用.而另一个影响单位时间信噪比的因素是采样模式.使用伪二维采样方法[如图 3(a)],需要获取二维采样所采集到的每个FID信号的前端数据块,将其拼接起来以获得去偶的FID信号,因此,其实验时间会比常规一维谱更长.在实时采样方法[如图 3(b)]中,每采样2t1/n的时间长度后对J偶合进行重聚,通过拼接可以一次性获得所需FID信号.需要注意的是,尽管PSYCHE模块能够获得具有最高灵敏度的宽带去偶全谱,但它目前无法在实时采样模块中使用.实时方法由于只需要进行一维采样,因此相对于需要进行二维采样的干涉图方法,能够大大缩短实验时间.
将不同的选择翻转模块和不同的采样模式进行组合,可得到不同级别灵敏度的谱图.使用HOBS重聚模块和实时采样模式相组合进行实验,能获得具有最高单位时间信噪比的谱图,其灵敏度与常规单脉冲一维谱相近甚至更高.其主要局限在于它只能得到部分频宽的纯化学位移谱.在宽带去偶实验中,简化信号的代价是灵敏度的大幅损失.目前,PSYCHE方法是获取未知样品宽带去偶谱的最有应用前景的方法,它易于操作,且谱图灵敏度相对较高[18].
目前只有一种纯化学位移实验在去偶之后没有额外的灵敏度损失,那就是基于BIRD的二维实时1H-13C HSQC实验[19].它使用了实时BIRD去偶方法,BIRD模块只能对与13C核相连的1H核进行去偶,尽管它有效抑制了与12C核相连的1H核的磁化矢量对谱图产生的强信号干扰,但也使信噪比有所损失.HSQC实验中的INEPT模块进行了极化转移,增强了信号强度,弥补了因自然丰度导致的信噪比损失,因此其灵敏度在去偶前后不会有所损失.与常规HSQC实验相比,这里的去偶方法实际上实现了灵敏度和分辨率二者的同时提高.然而这一实验无法测出同碳非对映1H核间的2JHH,这是因为BIRD模块不能区分与同一个13C核相连的非对映1H核.在最终得到的纯化学位移谱中,同碳1H核之间的相互关系被保存下来,并且非等价亚甲基上的1H核所产生的信号表现为双重峰.这一局限可以通过使用恒时BIRD[22]或完美BIRD[23]模块重聚同碳偶合的影响来克服.然而,这些模块只能与干涉图采样模式结合使用(伪三维实验),与采用了实时采样模式的方法相比,其信噪比有所下降.
多维纯化学位移实验的灵敏度存在更多问题,同时其间接维也对分辨率有更高的要求.为了保证实验时间处于合理范围,目前已经提出谱峰重叠[24-26]和非均匀采样(NonUniform Sampling,NUS)[25, 27, 28]两种技术与纯化学位移方法相结合以缩短实验时间.在多维1H-1H同核NMR谱图中,通过在直接维和间接维都使用J重聚模块进行去偶,可以获得多个维度的超高分辨率纯化学位移谱,但同时却会导致极低的信号灵敏度和极长的实验时间.为了避免这些问题,目前的一些方法只对直接维[29]或间接维[30]中的某一个维度进行了宽带去偶,并对另一维度进行协方差后处理.
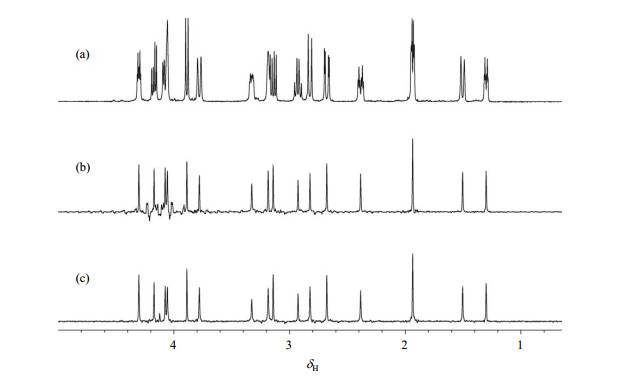
2.2 谱图质量大多数的纯化学位移NMR实验都能获得高分辨率去偶谱,但强偶合效应或数据块拼接产生的边带伪峰等因素会对谱图质量产生影响.当两个氢核之间的偶合常数(JHH)与它们之间的频率差(∆v)处于相同数量级(∆v≈JHH)时,它们被认为存在强偶合关系.一般情况下,1H NMR谱的强偶合会造成信号幅度和位置的畸变.一般而言,所有的纯化学位移技术都能很好地应用于弱偶合体系,但对于强偶合体系而言,所得到的谱图常常会因此产生伪峰而变得复杂.目前,已有一些方法能有效地克服强偶合,将其负面影响降到最低.例如,如果将具有强选择性的脉冲(激发带宽很窄)用于激发不同片层里的每个信号,那么片选方法就能够很好地克服强偶合作用.然而,脉冲的选择性越强,所得谱图的灵敏度就越低,而且脉冲时间超过20 ms的选择性脉冲通常只能用于伪二维采样方法.BIRD方法在大部分情况下都能有较好的谱图质量[31],但当与13C核相连的1H核之间存在强偶合时,这种方法便会失效.PSYCHE方法与其它纯化学位移方法相比,对强偶合具有更好的鲁棒性.小角度Chirp脉冲和脉冲梯度场的结合使用,可削弱强偶合效应产生的相干转移路径(Coherence Transfer Pathways,CTP).在三自旋回波PSYCHE(Triple Spin Echo PSYCHE,TSE-PSYCHE)实验中,将PSYCHE的J重聚模块与两个额外的Chirp脉冲组合使用,能够得到更好的实验效结果(如图 4)[32].然而PSYCHE方法及TSE-PSYCHE方法需要在灵敏度和去偶效果上进行折衷.后来提出的完美自旋回波纯化学位移方法(Perfect Echo Pure Shift Improved Experiment,PEPSIE)则将片选方法与完美回波方法相结合,去除强偶合的同时,缓解了片选方法固有的灵敏度损失问题[33].
|
图 4 溶于CDCl3的士的宁样品的(a)常规1H NMR谱、(b) PSYCHE谱和(c) TSE-PSYCHE谱(摘自文献[32]) Fig. 4 (a) Conventional, (b) PSYCHE and (c) TSE-PSYCHE 1H NMR spectra for a strychnine sample dissolved in CDCl3 (This figure is excerpted from literature [32]) |
另外,纯化学位移实验的谱图质量也会受到数据块拼接所产生的边带效应的影响,这些边带与分段采样方式(“数据分块”)有关,而该方式在伪二维采样[如图 3(a)]和实时采样[如图 3(b)]中均有使用.在这两种采样模式中,JHH在每个数据块的中心被重聚,而且要保证每个数据块的持续时间(tchunk≡1/SW1≡2τ)足够短,使得JHH演化在数据块边缘的值都很小(tchunk≪1/JHH).然而残余的JHH调制仍然会使每个数据块边缘的信号稍弱于中间的信号,而这周期性的强度变化在进行傅里叶变换后,会导致在每个去偶信号两边产生弱的边带,这些边带具有一定的频率间隔(数据块时长的倒数,1/tchunk),且其强度在去偶信号两边快速衰减.总的来说,数据块的时长越短,与准确J重聚的偏离越小,纯化学位移谱就越“干净”.由于数据块边带的强度近似随(JHH·tchunk)2变化,因而数据块的时间一般选定为10~20 ms.在大多数情况下,数据块边带的强度与13C核的卫星峰在同一水平.2017年,Moutzouri等[34]提出一种被称为“SAPPHIRE”(Sideband Averaging by Periodic PHase Incrementation of Residual J Evolution)的新方法,此方法在原有的纯化学位移伪二维序列基础上额外增加了一个时间可变的短回波,以调节J偶合重聚点向前或向后移动,采样后的谱图经过平均后,得到能够抑制边带的谱图.
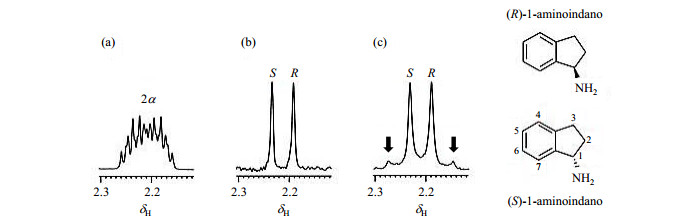
在实时纯化学位移实验[如图 3(b)]中,除了数据块边缘的J调制会产生数据边带外,每个J重聚模块间存在的横向弛豫也会对谱图质量产生影响.采样中断期的额外横向弛豫使得FID信号受到阶梯状调制[如图 3(b)中的FID信号],导致谱图中的去偶信号两边出现弱的伪峰,且以数据块时长倒数为频率间隔等距出现[如图 5(c)].一般而言,J重聚模块的持续时间越短,伪峰的强度就越弱.最近,一种降低实时实验中边带伪峰的方法被提出,该方法通过改变每次独立扫描时的FID信号数据块的长度来实现[35].由于边带伪峰的位置取决于数据块的持续时间,因此通过改变数据块时长可以削弱边带伪峰,使得谱图中的伪峰强度可以降低到13C核卫星峰的信号强度水平之下.
|
图 5 扩展区域表示了等摩尔(R, S)-1-氨基茚满盐酸盐对映体的混合物与4.5当量塔木德乙醇的(溶于CDCl3)的(a)常规1H NMR谱、(b)干涉图片层选择谱和(c)实时片层选择谱.两张纯化学位移谱均使用了20 ms的选择180°高斯脉冲.(c)图中的箭头标注了实时谱中的伪峰.在干涉图采样的谱和实时采样的谱中可以观察到不同的线宽.(摘自文献[3]) Fig. 5 Expanded region showing proton 2α from the equimolar enantiomeric mixture of (R, S)-1-aminoindan with 4.5 equivalents of Pirkel alcohol in CDCl3. Comparison of the spectral quality of (a) standard 1H NMR, (b) interferogram slice-selective and (c) real-time slice-selective spectra. A 20 ms selective 180° Gaussian pulse was used in both pure shift spectra. The arrows in (c) mark the artifacts observed in the real-time spectrum. Different linewidths are also observed in interferogram and real-time spectra. (This figure is excerpted from literature [3]) |
当使用J重聚模块来获得纯化学位移谱时,选择一个合适的相干转移路径是十分重要的,它可以避免不需要的磁化矢量(即没有受到J重聚影响的磁化矢量)对最终信号造成影响.否则,在谱图中也将会因此产生伪峰.这些伪峰出现在与数据块边带相同的频率位置上,然而它们在去偶信号的两侧衰减缓慢.对相干转移路径进行有效选择可以通过在180°硬脉冲或J重聚模块的选择翻转模块(如图 2)的任意一侧使用脉冲梯度场来实现.不仅如此,相干转移路径的选择还能与选择翻转模块(BIRD,选择性脉冲和Chirp脉冲)的相位循环相结合.
除了强偶合效应和边带伪峰之外,纯化学位移实验的谱图质量还取决于同核去偶信号的分辨率.伪二维采样方法的信号分辨率直接与间接维的时间增量个数有关.而去偶FID信号由每次采样的前端数据块重构而成,因此增量数目决定了数据块数目,也决定了重构谱图的分辨率.总的来说,增量数目(数据块数目)越多,重构的FID信号越长,信号分辨率越高.正常来说,在对信号分辨率、谱图质量和实验时间三者进行折衷考虑后,间接维一般需要16~32个采样点.
实时方法获得的去偶信号的分辨率取决于重构的FID信号长度和施加J重聚模块时的额外T2横向弛豫.在实时方法中,采样到的各个数据块之间被插入了J重聚模块,导致获得的FID信号比常规的FID信号更短,造成谱图信号被轻微展宽.对于快速弛豫(T2较短)的分子,FID信号中的各数据块间的横向弛豫衰减更加严重,导致获得FID信号更短且谱图信号更宽.如果在实时实验中使用BIRD模块,那么J重聚模块的持续时间应设为2∆ [∆ = 1/(2·1JCH)],因而通常将其设置为5~10 ms.如果使用片选方法,那么J重聚模块的持续时间将由180°选择性脉冲的宽度来决定,而其脉冲宽度的选择则取决于被解析的自旋系统.对于两个化学位移相近的信号互相偶合的系统,一般需要使用一个脉冲宽度较长(更强选择性)的脉冲,而这会造成数据块之间的间隔变长,从而导致在获得的纯化学位移谱中出现更宽的信号和更强的伪峰.最近,一种叫做“EXACT”(Extended Acquisition Time)的核磁共振新方法被提出,该方法无需去除无法采集到的数据块,而是在采样后用算法对其进行重建[36].通过这种方法,FID信号恢复了全长,避免了FID信号人为缩短,从而使谱图的线宽变窄.半实时方法(semi-real-time method)[37]也是一种解决方法.相比于实时方法,这一方法需要两次或多次的实验,并将得到的数据进行交互拼接.尽管采样时间有所增加,但得到的谱图信噪比优于实时采样方法.在实际实验中,如果要获得高质量的谱图,一般不推荐使用时长超过15 ms的选择性脉冲.因此,如果横向弛豫时间较短或需要高分辨率,干涉图方法比起实时去偶方法(如图 5)能提供更好的分辨率.
3 纯化学位移发展的机遇与挑战在纯化学位移NMR研究领域,存在一个关于“下一步应该怎么走”的有趣探讨.一些研究人员认为该领域方法上的巨大发展十分困难,因而未来的工作将会集中在纯化学位移方法的应用上.而另一些研究人员则认为纯化学位移在技术上还有许多令人期待的发展空间.
3.1 新方法的发展目前纯化学位移新方法的发展仍是热点课题,大部分的新方法都集中在提高纯化学位移谱的灵敏度和减少其边带伪峰上.其主要目的是,在与常规NMR实验时间相当的情况下,获得高灵敏度、高质量的宽带同核去偶谱.遗憾的是,现有的方法均不能实现这一目的,唯一例外的是实时BIRD-HSQC[19]方法,但它只能用于含有13C核的分子,然而13C核自然丰度较低.
在纯化学位移新方法的发展中,最大的挑战在于如何提高谱图灵敏度.尽管近些年来,一些提高谱图灵敏度的方法已被提出,但是,大部分纯化学位移方法获得的同核宽带去偶谱的灵敏度仍远低于常规谱.正如前面提到的,灵敏度损失的主要原因在于J重聚模块中的选择翻转模块以及采样模式的选择.因此,在不久的将来,如何设计更好的J重聚方法以提高谱图灵敏度和鲁棒性将成为一个最大的挑战.
另一方面,强偶合效应仍是一个很难攻克的问题.抑制同核去偶谱图中强偶合体系的影响常常会用到BIRD[31]或TSE-PSYCHE[32]模块,但是至今没有一个能适用于所有情况的方法.至于伪二维采样获得的纯化学位移谱中存在的数据块边带,则可以使用一些已经提出的方法进行减弱和去除.例如,在实时实验中通过改变FID信号数据块重构长度来减弱边带[35]的想法也可被用于伪二维方法中.另一种可能用于移除数据块边带的方法是,对伪二维谱进行逐点构建(例如在Garbow的方法[11]),这样可以避免任何残余的J调制,但此方法的缺点是,为了使得到的去偶FID信号具有可接受的分辨率,需要增加间接维采样点数,从而导致实验时间大幅增加.而这可能需要通过结合非均匀采样技术来加速采样.
未来几年,最具探索前景的领域可能在于通过使用数据后处理方法或深度学习方法来提升现有的宽带同核去偶技术,甚至创建合成的纯化学位移谱.这种方法可以缩短实验时间,解决灵敏度较低的问题,甚至可能获得没有伪峰的纯化学位移谱.协方差处理已经被用于获得半合成的1H-1H同核二维纯化学位移谱,此方法使用了一张F1维或F2维的同核去偶谱,并将去偶维提取出来的信息映射到偶合维[29, 30].根据这一想法,一种广义间接协方差后处理方法被提出,它以1H-1H的F1维去偶谱(只包含对角峰)为参考,生成合成的二维同核或异核纯化学位移谱[38, 39].
3.2 应用前景纯化学位移技术在简化谱图和获取高分辨率谱图方面已经展现了很大的应用潜力.现有的纯化学位移技术已被成功应用在不同大小分子[27, 40, 41]的结构解析,对称结构[24, 42, 43]、非对称结构[44]和复杂混合物[25, 28, 45, 46]的研究,以及扩散[47-50]、动力学[51, 52]和非均匀场下[53]的研究等方面.纯化学位移技术的一个重要的实际应用在于,通过获取高分辨率的纯化学位移谱来测量标量偶合和残余的偶极偶合常数的大小和符号[22, 23, 26, 54-60].更多具有广阔前景的应用也已被提出,例如将纯化学位移技术与动态核极化技术[61]相结合,或使用纯化学位移技术进行代谢组学的定量分析[62].目前,纯化学位移技术在其他核(例如19F、31P或2H)[63]上的应用范围可能会进一步扩展.在质量控制过程中,药物降解研究和快速反应监测中的应用也可能会成为应用探索的新方向.另一方面,低场NMR中由于J偶合引起的谱图重叠问题也有望通过应用纯化学位移技术来解决.不仅如此,纯化学位移技术的发展,使得通过自动化方式,如计算机辅助结构鉴定程序,来进行结构鉴定和分子结构判定成为可能[64, 65].
过去几年里,纯化学位移方法上的改进使得实验数据越来越易于处理,比如使用数据后处理代码对通过干涉图采样方法得到的数据进行处理,或是将常规的傅里叶变换用在实时方法中.NMR领域的研究人员们已经完成了这些实验的设计工作,以确保便于普通NMR用户操作,省去繁琐的实验设置环节.大多数的脉冲程序、参数设置和数据都可以在线获得,这有助于纯化学位移方法在商用NMR设备中的应用得以实现.
4 总结与展望获得高分辨率纯化学位移波谱的方法及其应用越来越受到人们的关注,本文归纳总结了现有获得纯化学位移波谱的主要方法,对其存在的问题进行了分析,并讨论了其在发展过程中遇到的机遇与挑战.纯化学位移波谱方法可以保留化学位移信息、去除标量偶合、提高谱图信号的分辨率,但其灵敏度和谱图质量还有待提高.如何通过优化脉冲序列和数据后处理方法来提高纯化学位移波谱方法的谱图质量,以及如何将这些方法应用于实际生物化学体系(如在NMR定域谱的应用研究),还值得进一步深入探讨和研究.
| [1] | ZANGGER K. Pure shift NMR[J]. Prog Nucl Magn Reson Spectrosc, 2015, 86-87: 1-20. DOI: 10.1016/j.pnmrs.2015.02.002. |
| [2] | CASTAÑAR L, PARELLA T. Broadband 1H homodecoupled NMR experiments: Recent developments, methods and applications[J]. Magn Reson Chem, 2015, 53(6): 399-426. DOI: 10.1002/mrc.4238. |
| [3] | LAURA C. Pure shift 1H NMR: What's next?[J]. Magn Reson Chem, 2017, 55: 47-53. DOI: 10.1002/mrc.4545. |
| [4] |
ZHOU Q J, XIANG J F, TANG Y L, et al. Pure shift proton NMR spectroscopy and its applications[J].
Chinese J Magn Reson, 2016, 33(3): 502-513.
周秋菊, 向俊锋, 唐亚林, 等. 纯位移核磁共振氢谱及其应用[J]. 波谱学杂志, 2016, 33(3): 502-513. |
| [5] | ERNST R R, PRIMAS H. Nuclear magnetic resonance with stochastic high-frequency fields[J]. Helv Phys Acta, 1963, 36(5): 583-600. |
| [6] | ANDERSON W A, FREEMAN R. Influence of a second radiofrequency field on high-resolution nuclear magnetic resonance spectra[J]. J Chem Phys, 1962, 37(1): 85-103. |
| [7] | AUE W P, KARHAN J, ERNST R R. Homonuclear broad band decoupling and two-dimensional J-resolved NMR spectroscopy[J]. J Chem Phys, 1976, 64(10): 4226-4227. DOI: 10.1063/1.431994. |
| [8] | BAX A, MEHLKOPF A F, SMIDT J. Homonuclear broadband-decoupled absorption-spectra, with linewidths which are independent of the transverse relaxation rate[J]. J Magn Reson, 1979, 35(1): 167-169. |
| [9] | SØRENSEN O W, GRIESINGER C, ERNST R R. Time-reversal of the evolution under scalar spin spin interactions in NMR-application for ω1 decoupling in two-dimensional NOE spectroscopy[J]. J Am Chem Soc, 1985, 107(25): 7778-7779. DOI: 10.1021/ja00311a101. |
| [10] | PELL A J, EDDEN R A E, KEELER J. Broadband proton-decoupled proton spectra[J]. Magn Reson Chem, 2007, 45(4): 296-316. |
| [11] | GARBOW J R, WEITEKAMP D P, PINES A. Bilinear rotation decoupling of homonuclear scalar interactions[J]. Chem Phys Lett, 1982, 93(5): 504-509. DOI: 10.1016/0009-2614(82)83229-6. |
| [12] | ZANGGER K, STERK H. Homonuclear broadband-decoupled NMR spectra[J]. J Magn Reson, 1997, 124(2): 486-489. |
| [13] | AGUILAR J A, FAULKNER S, NILSSON M, et al. Pure shift 1H NMR: A resolution of the resolution problem?[J]. Angew Chem Int Ed, 2010, 49(23): 3901-3903. DOI: 10.1002/anie.201001107. |
| [14] | LUPULESCU A, OLSEN G L, FRYDMAN L. Toward single-shot pure-shift solution 1H NMR by trains of BIRD-based homonuclear decoupling[J]. J Magn Reson, 2012, 218: 141-146. DOI: 10.1016/j.jmr.2012.02.018. |
| [15] | MEYER N H, ZANGGER K. Simplifying proton NMR spectra by instant homonuclear broadband decoupling[J]. Angew Chem Int Ed, 2013, 52(28): 7143-7146. DOI: 10.1002/anie.201300129. |
| [16] | CASTAÑAR L, NOLIS P, VIRGILI A, et al. Full sensitivity and enhanced resolution in homodecoupled band-selective NMR[J]. Chem Eur J, 2013, 19(51): 17283-17286. DOI: 10.1002/chem.201303235. |
| [17] | YING J, ROCHE J, BAX A. Homonuclear decoupling for enhancing resolution and sensitivity in NOE and RDC measurements of peptides and proteins[J]. J Magn Reson, 2014, 241: 97-102. DOI: 10.1016/j.jmr.2013.11.006. |
| [18] | FOROOZANDEH M, ADAMS R W, MEHARRY N J, et al. Ultrahigh-resolution NMR spectroscopy[J]. Angew Chem Int Ed, 2014, 53(27): 6990-6992. DOI: 10.1002/anie.201404111. |
| [19] | PAUDEL L, ADAMS R W, KIRALY P, et al. Simultaneously enhancing spectral resolution and sensitivity in heteronuclear correlation NMR spectroscopy[J]. Angew Chem Int Ed, 2013, 52(44): 11616-11619. DOI: 10.1002/anie.201305709. |
| [20] | CASTAÑAR L, NOLIS P, VIRGILI A, et al. Experiments simultaneous multi-slice excitation in spatially encoded NMR experiments[J]. Chem Eur J, 2013, 19(46): 15472-15475. DOI: 10.1002/chem.201303272. |
| [21] | COTTE A, JEANNERAT D. 1D NMR homodecoupled 1H spectra with scalar coupling constants from 2D NemoZS-DIAG experiments[J]. Angew Chem Int Ed, 2015, 54(20): 6016-6018. DOI: 10.1002/anie.201500831. |
| [22] | REINSPERGER T, LUY B. Homonuclear BIRD-decoupled spectra for measuring one-bond couplings with highest resolution: CLIP/CLAP-RESET and constant-time-CLIP/CLAP-RESET[J]. J Magn Reson, 2014, 239: 110-120. DOI: 10.1016/j.jmr.2013.11.015. |
| [23] | KALTSCHNEE L, KOLMER A, TIMARI I, et al. "Perfecting" pure shift HSQC: Full homodecoupling for accurate and precise determination of heteronuclear couplings[J]. Chem Commun, 2014, 50(99): 15702-15705. DOI: 10.1039/C4CC04217D. |
| [24] | PEREZ-TRUJILLO M, CASTAÑAR L, MONTEAGUDO E, et al. Simultaneous 1H and 13C NMR enantiodifferentiation from highly-resolved pure shift HSQC spectra[J]. Chem Commun, 2014, 50(71): 10214-10217. DOI: 10.1039/C4CC04077E. |
| [25] | CASTAÑAR L, ROLDAN R, CLAPES P, et al. Disentangling complex mixtures of compounds with near-identical 1H and 13C NMR spectra using pure shift NMR spectroscopy[J]. Chem Eur J, 2015, 21(21): 7682-7685. DOI: 10.1002/chem.201500521. |
| [26] | MARCÓ N, FREDI A, PARELLA T. Ultra high-resolution HSQC: Application to the efficient and accurate measurement of heteronuclear coupling constants[J]. Chem Commun, 2015, 51(15): 3262-3265. DOI: 10.1039/C4CC10279G. |
| [27] | HANSEN S U, MILLER G J, CLIFF M J, et al. Making the longest sugars: A chemical synthesis of heparin-related[J]. Chem Sci, 2015, 6(11): 6158-6164. DOI: 10.1039/C5SC02091C. |
| [28] | KAKITA V M, HOSUR R V. Non-uniform-sampling ultrahigh resolution TOCSY NMR: Analysis of complex mixtures at microgram levels[J]. ChemPhysChem, 2016, 17(15): 2304-2308. DOI: 10.1002/cphc.201600255. |
| [29] | MORRIS G A, AGUILAR J A, EVANS R, et al. True chemical shift correlation maps: A TOCSY experiment with pure shifts in both dimensions[J]. J Am Chem Soc, 2010, 132(37): 12770-12772. DOI: 10.1021/ja1039715. |
| [30] | FOROOZANDEH M, ADAMS R W, NILSSON M, et al. Ultrahigh-resolution total correlation NMR spectroscopy[J]. J Am Chem Soc, 2014, 136(34): 11867-11869. DOI: 10.1021/ja507201t. |
| [31] | AGUILAR J A, NILSSON M, MORRIS G A. Simple proton spectra from complex spin systems: Pure shift NMR spectroscopy using BIRD[J]. Angew Chem Int Ed, 2011, 50(41): 9716-9717. DOI: 10.1002/anie.201103789. |
| [32] | FOROOZANDEH M, ADAMS R W, KIRALY P, et al. Measuring couplings in crowded NMR spectra: Pure shift NMR with multiplet analysis[J]. Chem Commun, 2015, 51(84): 15410-15413. DOI: 10.1039/C5CC06293D. |
| [33] | ILGEN J, KALTSCHNEE L, THIELE C M. A pure shift experiment with increased sensitivity and superior performance for strongly coupled systems[J]. J Magn Reson, 2018, 286: 18-29. DOI: 10.1016/j.jmr.2017.11.001. |
| [34] | MOUTZOURI P, CHEN Y X, FOROOZANDEH M, et al. Ultraclean pure shift NMR[J]. Chem. Commun, 2017, 53: 10188. DOI: 10.1039/C7CC04423B. |
| [35] | MAUHART J, GLANZER S, SAKHAⅡ P, et al. Faster and cleaner real-time pure shift NMR experiments[J]. J Magn Reson, 2015, 259: 207-215. DOI: 10.1016/j.jmr.2015.08.011. |
| [36] | NDUKWE I E, SHCHUKINA A, KAZIMIERCZUK K, et al. EXtended ACquisition Time (EXACT) NMR-a case for 'burst' non-uniform sampling[J]. ChemPhysChem, 2016, 17(18): 2799-2803. DOI: 10.1002/cphc.201600541. |
| [37] | KIRALY P, NILSSON M, MORRIS GA. Semi-real-time acquisition for fast pure shift NMR at maximum resolution[J]. J Magn Reson, 2018, 293: 19-27. DOI: 10.1016/j.jmr.2018.05.012. |
| [38] | FREDI A, NOLIS P, COBAS C, et al. Exploring the use of generalized Indirect covariance to reconstruct pure shift NMR spectra: Current Pros and Cons[J]. J Magn Reson, 2016, 266: 16-22. DOI: 10.1016/j.jmr.2016.03.003. |
| [39] | FREDI A, NOLIS P, COBAS C, et al. Access to experimentally infeasible spectra by pure-shift NMR covariance[J]. J Magn Reson, 2016, 270: 161-168. DOI: 10.1016/j.jmr.2016.07.010. |
| [40] | LIU Y, GREEN M D, MARGUES R, et al. Using pure shift HSQC to characterize microgram samples of drug metabolites[J]. Tetrahedron Lett, 2014, 55(40): 5450-5453. DOI: 10.1016/j.tetlet.2014.06.067. |
| [41] | KALTSCHNEE L, KNOLL K, SCHMIDTS V, et al. Extraction of distance restraints from pure shift NOE experiments[J]. J Magn Reson, 2016, 271: 99-109. DOI: 10.1016/j.jmr.2016.08.007. |
| [42] | CASTAÑAR L, PEREZ-TRUJILLO M, NOLIS P, et al. Enantiodifferentiation through frequency- selective pure shift 1H nuclear magnetic resonance spectroscopy[J]. ChemPhysChem, 2014, 15(5): 854-857. DOI: 10.1002/cphc.201301130. |
| [43] | LAKSHMIPRIYA A, CHAUDHARI S R, SURYAPRAKASH N. Enantio-differentiation of molecules with diverse functionalities using a single probe[J]. Chem Commun, 2015, 51(70): 13492-13495. DOI: 10.1039/C5CC05290D. |
| [44] | ADAMS R W, BYRNE L, KIRALY P, et al. Diastereomeric ratio determination by high sensitivity band-selective pure shift NMR spectroscopy[J]. Chem Commun, 2014, 50(19): 2512-2514. DOI: 10.1039/C3CC49659G. |
| [45] | KAKITA V M R, VEMULAPALLI S P B, BHARATAM J. Band-selective excited ultrahigh resolution PSYCHE-TOCSY: Fast screening of organic molecules and complex mixtures[J]. Magn Reson Chem, 2016, 54(4): 308-314. DOI: 10.1002/mrc.4376. |
| [46] | POGGETTO G D, CASTANAR L, MORRIS G A, et al. A new tool for NMR analysis of complex systems: Selective pure shift TOCSY[J]. RSC Adv, 2016, 6(102): 100063-100066. DOI: 10.1039/C6RA22807K. |
| [47] | NILSSON M, MORRIS G A. Pure shift proton DOSY: Diffusion-ordered 1H spectra without multiplet structure[J]. Chem Commun, 2007(9): 933-935. DOI: 10.1039/b617761a. |
| [48] | HAMDOUN G, SEBBAN M, COSSOUL E, et al. 1H pure shift DOSY: A handy tool to evaluate the aggregation and solvation of organolithium derivatives[J]. Chem Commun, 2014, 50(31): 4073-4075. DOI: 10.1039/C4CC00111G. |
| [49] | GLANZER S, ZANGGER K. Directly decoupled diffusion-ordered NMR spectroscopy for the analysis of compound mixtures[J]. Chem Eur J, 2014, 20(35): 11171-11175. DOI: 10.1002/chem.201402920. |
| [50] | LI C, ZHAN H L, YAN J, et al. A pure shift and spin echo based approach for high-resolution diffusion-ordered NMR spectroscopy[J]. J Magn Reson, 2019, 305: 209-218. DOI: 10.1016/j.jmr.2019.07.021. |
| [51] | CASTAÑAR L, NOLIS P, VIRGILI A, et al. Measurement of T1/T2 relaxation times in overlapped regions from homodecoupled 1H singlet signals[J]. J Magn Reson, 2014, 244: 30-35. DOI: 10.1016/j.jmr.2014.04.003. |
| [52] | HUANG Y Q, ZHAN H L, YOU X Q, et al. A pure shift-based NMR method for transverse relaxation measurements on complex samples[J]. IEEE Transactions on Instrumentation and Measurement, publishing, 2019. |
| [53] | HUANG Y Q, CAO S H, YANG Y, et al. Ultrahigh-resolution NMR spectroscopy for rapid chemical and biological applications inhomogeneous magnetic fields[J]. Anal Chem, 2017, 89: 7115-7122. DOI: 10.1021/acs.analchem.7b01036. |
| [54] | CHEN Z, HUANG Y Q, LIN Y Q, et al. Accurate measurement of small J Couplings[J]. Annual Reports on NMR Spectroscopy, 2010, 72: 157-183. |
| [55] | LIN Y Q, ZENG Q, LIN L J, et al. High-resolution methods for the measurement of scalar coupling constants[J]. Prog Nucl Magn Reson Spectrosc, 2018, 109: 135-159. DOI: 10.1016/j.pnmrs.2018.08.003. |
| [56] | CASTAÑAR L, GARCÍA M, HELLEMANN E, et al. One-shot determination of residual dipolar couplings: Application to the structural discrimination of small molecules containing multiple stereocenters[J]. J Org Chem, 2016, 81(22): 11126-11131. DOI: 10.1021/acs.joc.6b02103. |
| [57] | TIMÁRI I, KALTSCHNEE L, RAICS MH, et al. Real-time broadband proton-homodecoupled CLIP/CLAP-HSQC for automated measurement of heteronuclear one-bond coupling constants[J]. RSC Adv, 2016, 6(91): 87848-87855. DOI: 10.1039/C6RA14329F. |
| [58] | ZENG Q, LIN L J, CHEN J Y, et al. A simultaneous multi-slice selective J-resolved experiment for fully resolved scalar coupling information[J]. J Magn Reson, 2017, 282: 27-31. DOI: 10.1016/j.jmr.2017.07.001. |
| [59] | LIN Y L, GUAN Q S, SU J W, et al. Combining Fourier phase encoding and broadband inversion toward J-edited spectra[J]. J Magn Reson, 2018, 291: 1-7. DOI: 10.1016/j.jmr.2018.02.021. |
| [60] | ZENG Q, LIN Y Q, CHEN Z. Pushing resolution limits for extracting 1H-1H scalar coupling constants by a resolution-enhanced selective refocusing method[J]. J Chem Phys, 2019, 150: 184-202. |
| [61] | DONOVAN K J, FRYDMAN L. HyperBIRD: A sensitivity-enhanced approach to collecting homonuclear-decoupled proton NMR spectra[J]. Angew Chem Int Ed, 2015, 54(2): 594-598. |
| [62] | MAUVE C, KHLIFI S, GILARD F, et al. Sensitive, highly resolved, and quantitative 1H-13C NMR data in one go for tracking metabolites in vegetal extracts[J]. Chem Commun, 2016, 52(36): 6142-6145. DOI: 10.1039/C6CC01783E. |
| [63] | ARIANA B J, GUY C L, DUŠAN U. SHARPER reaction monitoring: Generation of a narrow linewidth NMR singlet, without X-pulses, in an inhomogeneous magnetic field[J]. Anal Chem, 2017, 89: 10013-10021. DOI: 10.1021/acs.analchem.7b02437. |
| [64] | ELYASHBERG M E, WILLIAMS A J, MARTIN G E. Computer-assisted structure verification and elucidation tools in NMR-based structure elucidation[J]. Prog Nucl Magn Reson Spectrosc, 2008, 53(1-2): 1-104. DOI: 10.1016/j.pnmrs.2007.04.003. |
| [65] | ELYASHBERG M E, WILLIAMS A, BLINOV K. Contemporary computer-assisted approaches to molecular structure elucidation[M]. Britain: RSC Publishing, 2012. |
 2019, Vol. 36
2019, Vol. 36 
