文章信息
- miR-9500靶向SMAD2调控肺腺癌细胞的迁移和侵袭
- miR-9500 Regulates Migration and Invasion of Lung Adenocarcinoma Cells by Targeting SMAD2
- 肿瘤防治研究, 2023, 50(3): 236-242
- Cancer Research on Prevention and Treatment, 2023, 50(3): 236-242
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2023.22.0826
- 收稿日期: 2022-07-26
- 修回日期: 2022-11-24
世界卫生组织国际癌症研究机构(IARC)发布的全球癌症报告显示,2020年全球肺癌新发病例约220万例,死亡约180万例,位居全球癌症死亡人数第一[1]。肺癌起病隐匿,大多肺癌患者在确诊时已发生远端转移,使得肺癌患者5年平均生存率仅约20%[2-3]。肺腺癌作为肺癌的主要类型,其发病率近年来持续上升[4]。MicroRNAs(miRNAs)是真核生物中一类由21~25个核苷酸组成的短链非编码RNA,它可通过结合靶基因3’UTR序列来抑制靶基因翻译[5]。越来越多的研究证据表明miRNAs在肺腺癌转移中发挥重要调控作用[6-8]。目前关于miR-9500在肺腺癌细胞迁移侵袭中的作用机制相关报道较少。本研究基于生物信息学筛选miR-9500的靶基因,进一步作蛋白互作网络分析并筛选其核心靶基因,进行GO和KEGG富集分析及生存分析。通过体外实验探究miR-9500靶向SMAD2调控肺腺癌细胞迁移侵袭的相关分子机制,从而为肺腺癌的进展机制研究及其靶向治疗提供新的思路。
1 材料与方法 1.1 miR-9500共同靶基因的筛选利用TargetScan、miRDB、miRWalk数据库分别预测miR-9500的靶基因,并使用在线韦恩图工具Draw Venn Diagram制作韦恩图取交集,筛选miR-9500的共同靶基因。
1.2 蛋白互作网络分析及miR-9500核心靶基因筛选利用STRING在线数据库中的Multiple proteins工具对miR-9500共同靶基因进行蛋白互作网络分析。操作步骤如下:(1)在List Of Names中输入miR-9500的共同靶基因;(2)Organisms选择Homo sapiens;(3)点击SEARCH;(4)在Settings中minimum required interaction score选择high confidence(0.700),并在network display options选择hide disconnected nodes in the network;(5)导出蛋白互作表格及网络图。运用Cytoscape3.9.1软件中的cytoHubba插件进行核心基因筛选,采用度值(Degree)拓扑分析方法,将排名前20的基因视为miR-9500的核心靶基因。
1.3 GO功能分析与KEGG信号通路富集分析将cytoHubba得到的核心靶基因导入DAVID在线数据库,进行GO功能分析和KEGG信号通路富集分析。使用DAVID在线数据库中Functional Annotation分析工具,设置条件如下:(1)Enter Gene List输入miR-9500核心靶基因;(2)Select Identifier里选择OFFICIAL_GENE_SYMBOL,并在Select species中填写Homo sapiens;(3)List Type选择Gene List;(4)Submit List。提交列表后在Gene Ontology中进行GO功能分析,在Pathways中进行KEGG信号通路富集分析,并利用微生信网站将DAVID在线数据库得到的靶基因富集结果进行可视化。
1.4 miR-9500关键靶基因生存分析借助UALCAN数据库分析miR-9500关键靶基因(排名前10的核心靶基因)的表达水平与肺腺癌患者总生存期的关系。设置条件如下:(1)在Enter gene symbol输入miR-9500关键靶基因;(2)TCGA dataset中选择Lung adenocarcinoma,点击Explore;(3)Links for analysis下选择Survival进行生存分析。
1.5 细胞培养与处理人肺腺癌细胞系A549购自中国科学院典型培养物保藏委员会细胞库。培养基为含10%FBS的F12K培养基,于37℃、5%CO2培养箱内培养。将A549细胞铺于6孔板内,细胞密度达到50%~60% 时,按照所用jetPRIME转染试剂(Polyplus)说明书进行转染;NC对照和miR-9500 mimics作用的终浓度均为50 nmol/L,并分别于24 h和48 h后提取肺腺癌细胞A549的总RNA和总蛋白进行后续实验。
1.6 miR-9500与SMAD2的靶向关系验证借助TargetScan数据库预测miR-9500与SMAD2的结合位点。通过双荧光素酶报告实验验证miR-9500与SMAD2之间的靶向关系,即:合成含有miR-9500结合位点的SMAD2 3’UTR序列及miR-9500结合位点突变的SMAD2 3’UTR序列,分别构建SMAD2-wt与SMAD2-mut双荧光素酶报告重组质粒。肺腺癌细胞A549密度达到60%左右时进行转染,实验共分为4组,分别为SMAD2- wt+NC、SMAD2-mut+NC、SMAD2-wt+miR-9500 mimics和SMAD2-mut+miR-9500 mimics。利用双荧光素酶报告基因检测试剂盒检测各处理组酶活性的变化,以海肾荧光素酶活性作为内参。
1.7 细胞迁移和侵袭能力的检测划痕实验:将A549细胞接种于6孔板中,转染,用10 μl枪头垂直沿6孔板中线轻轻划线,PBS清洗除去悬浮细胞,换1%血清培养基,分别于0、24、48和72 h拍照,以检测分析各处理组细胞的迁移能力。Transwell实验:将转染后的A549细胞计数并接种于上室中,培养48 h后用0.1%结晶紫染色,计数膜背面穿膜细胞数并拍照,后用DMSO洗脱下室滤膜上着色细胞,酶标仪检测光密度OD值,以检测分析各处理组细胞的迁移能力。基质胶侵袭实验:在滤膜上平铺一层基质胶,将转染后的A549细胞计数并接种于上室中,培养48 h后用0.1% 结晶紫染色,计数膜背面穿膜细胞数并拍照,洗脱并测定OD值,以分析各处理组细胞的侵袭能力。
1.8 Western blot与qRT-PCR检测SMAD2蛋白和mRNA的表达Western blot:按常规步骤操作。RIPA裂解液提取NC对照组与miR-9500 mimics处理组细胞总蛋白,BCA蛋白定量、电泳、转膜、封闭、抗体孵育、ECL曝光,分析各处理组细胞中MMP2、MMP9及SMAD2的蛋白表达水平差异。所用一抗均来自于Proteintech(中国武汉),稀释比例如下:MMP2(1:1 000)、MMP9(1:1 000)、SMAD2(1:1 000)、β-actin(1:4 000)。qRTPCR:TRIzol法分别提取NC对照组与miR-9500 mimi cs处理组细胞总RNA,用反转录试剂盒(TransGen Biotech)将所提RNA反转录为cDNA,并用qRT-PCR试剂盒(TargetMol)检测各处理组细胞中SMAD2 mRNA相对表达水平的差异。
1.9 统计学方法采用SPSS21.0软件进行统计学分析,两组间比较采用独立样本t检验,P < 0.05为差异有统计学意义。
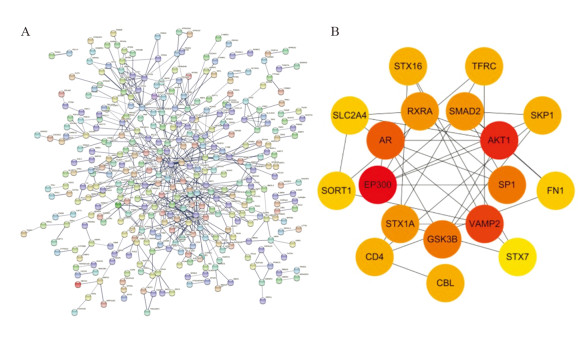
2 结果 2.1 miR-9500靶基因预测及其蛋白互作网络分析通过筛选共获得698个miR-9500的共同靶基因,见图 1。借助STRING在线数据库构建了这些靶基因的蛋白互作网络,见图 2A。进一步利用Cytoscape软件中的cytoHubba插件进行核心靶基因筛选,将Degree排名前20的基因作为miR-9500的核心靶基因,结果见图 2B。

|
| 图 1 韦恩图取交集获得miR-9500的靶基因 Figure 1 Venn diagram of miR-9500 target genes |
|
| 图 2 蛋白互作网络分析(A) 及miR-9500核心靶基因筛选(B) Figure 2 Protein interaction network analysis(A) and screening of miR-9500 core target genes(B) |
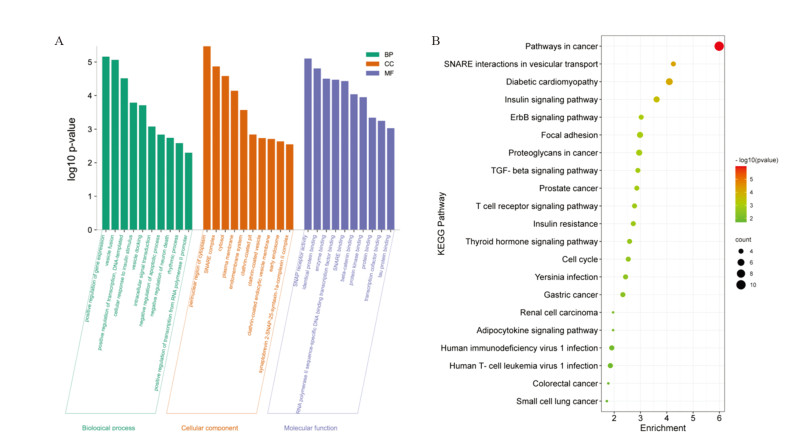
GO功能分析结果表明这些基因在生物学过程(BP)方面主要集中于细胞内信号转导和凋亡抑制等;这些基因的产物在执行功能时所处细胞成分的结构位置(CC)主要集中于细胞质的核周区域和细胞质等;这些基因及其产物的分子功能(MF)主要集中于β-catenin结合和蛋白激酶结合等,见图 3A。KEGG信号通路富集分析结果表明,miR-9500核心靶基因相关通路主要富集于癌症通路(如:前列腺癌、胃癌、肾细胞癌、结直肠癌、小细胞肺癌等)、ErbB信号通路、黏着斑、TGF-β信号通路、细胞周期等,见图 3B。
|
| 图 3 miR-9500核心靶基因的GO功能分析(A)和KEGG信号通路富集分析(B) Figure 3 GO analysis(A) and KEGG signaling pathway enrichment analysis(B) of miR-9500 core target genes |
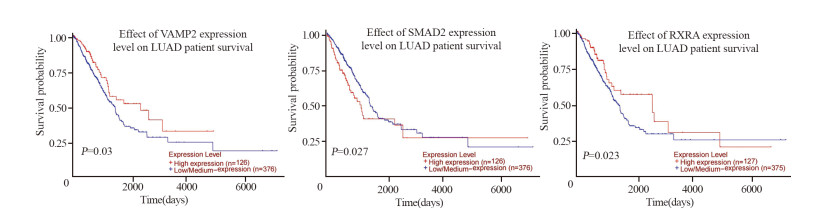
将排名前1 0的m i R - 9 5 0 0核心靶基因作为miR- 9 5 0 0关键靶基因,分别为:EP 3 0 0、AKT1、VAMP2、AR、SP1、GSK3B、STX1A、SMAD2、RXRA、STX16。其中只有VAMP2、SMAD2及RXRA的表达水平与肺腺癌患者的总生存期显著相关;且相比于低表达组,VAMP2和RXRA高表达组肺腺癌患者的总生存期均显著延长,而SMAD2高表达组肺腺癌患者的总生存期显著缩短,见图 4。
|
| 图 4 miR-9500关键靶基因与肺腺癌患者总生存期之间的关系 Figure 4 Relationship between miR-9500 key target genes and overall survival of patients with lung adenocarcinoma |
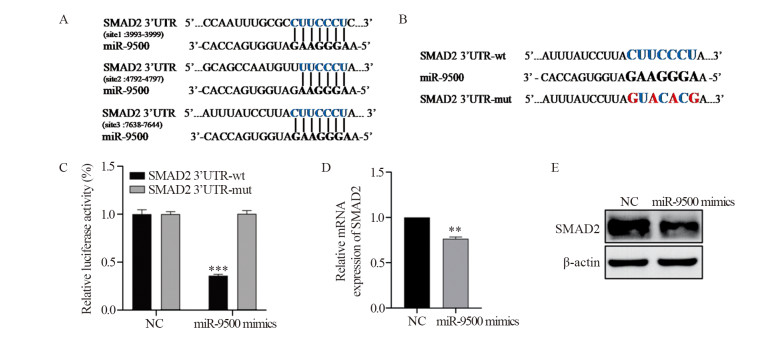
生物信息学分析结果提示:SMAD2可能是miR-9500的关键靶基因之一。TargetScan数据库分析发现,SMAD2 3’UTR有3个miR-9500的结合位点,这两者有着较为可靠的靶向关系,见图 5A。本研究中所验证的结合位点为7638~7644位,其野生型和突变型序列见图 5B。合成含有miR-9500结合位点的SMAD2 3’UTR序列及miR-9500结合位点突变的SMAD2 3’UTR序列,分别构建SMAD2-wt与SMAD2-mut双荧光素酶报告重组质粒,并通过测序比对证实重组质粒载体构建成功(图请扫描本文OSID码)。双荧光素酶报告实验结果显示,miR-9500 mimics转染组与NC组相比,SMAD2-wt共转染组荧光素酶活性显著降低;而SMAD2-mut共转染组荧光素酶活性无明显变化,见图 5C。qRT-PCR与Western blot检测结果显示,过表达miR-9500可显著降低SMAD2的mRNA相对表达水平和蛋白表达水平,见图5D~E。综上,miR-9500可靶向结合SMAD2并下调SMAD2的表达水平。
|
| A: Predicted binding sites of miR-9500 and SMAD2; B: Wild-type and mutant sequences of SMAD2 3'UTR; C: Dual luciferase reporter assay; D-E: The effects of miR-9500 overexpression on the relative mRNA expression level and protein expression level of SMAD2. **: P < 0.01, ***: P < 0.001, compared with NC group. 图 5 miR-9500靶向结合SMAD2并抑制SMAD2表达 Figure 5 miR-9500 targeted SMAD2 and inhibited the expression of SMAD2 |
划痕实验与Transwell实验结果均表明,miR-9500 mimics处理组肺腺癌细胞的迁移能力显著低于NC对照组,见图6A~B。基质胶侵袭实验结果表明,过表达miR-9500可显著降低肺腺癌细胞的侵袭能力,见图 6C。Western blot结果表明,过表达miR-9500可显著降低肺腺癌细胞迁移侵袭标志分子MMP9和MMP2的蛋白表达水平,见图 6D。综上,miR-9500可通过靶向SMAD2进而显著降低肺腺癌细胞的迁移侵袭能力。
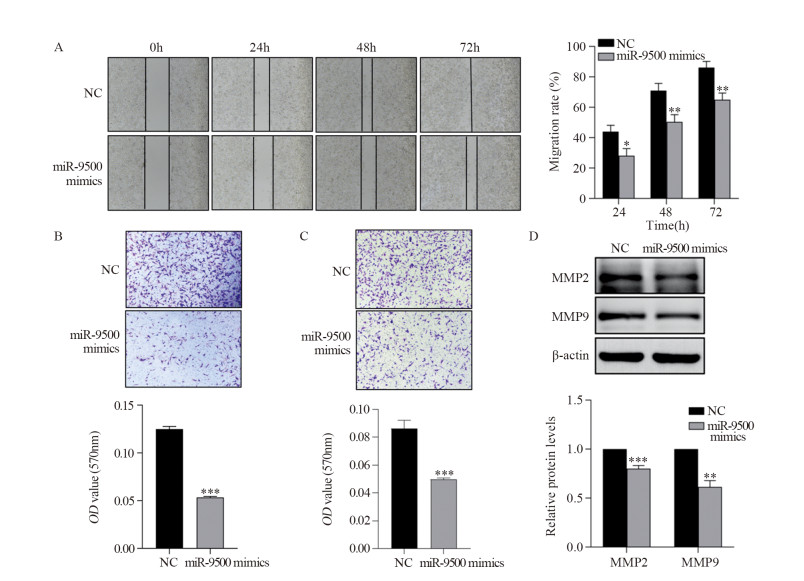
|
| A-B: Effect of miR-9500 overexpression on the migration of lung adenocarcinoma cells was analyzed by Wound healing and Transwell assays; C: Matrigel invasion assay was used to determine the effect of miR-9500 overexpression on the invasion ability of lung adenocarcinoma cells; D: Western blot was used to analyze the effect of miR-9500 overexpression on the protein expression levels of MMP2 and MMP9 in lung adenocarcinoma cells; *: P < 0.05, **: P < 0.01, ***: P < 0.001, compared with NC group. 图 6 过表达miR-9500可抑制肺腺癌细胞迁移侵袭 Figure 6 Overexpression of miR-9500 could inhibit the migration and invasion of lung adenocarcinoma cells |
肺腺癌是肺癌的常见类型,约占所有肺癌病例的40%,是最具侵袭性和致命性的肿瘤之一[9]。发生转移是造成晚期肺腺癌患者治疗失败和死亡的主要原因之一,但目前关于肺腺癌转移的分子机制尚未完全阐明[10-11]。本研究结果表明,miR-9500可通过靶向SMAD2来抑制肺腺癌细胞的迁移和侵袭,提示miR-9500可能作为肿瘤抑制因子在肺腺癌的发生发展中发挥重要调控作用。
研究表明miRNAs在肺腺癌细胞的迁移侵袭调控中扮演着重要角色。如:miR-21-5p在肺腺癌中呈显著高表达,可通过靶向WWC2(WW和C2结构域蛋白2)促进肺腺癌细胞的迁移和侵袭[12]。而miR-191-5p可通过靶向SATB同源盒1(SATB1)来抑制Wnt/β-catenin信号通路激活,从而降低肺腺癌细胞的迁移能力[13]。miR-365可通过靶向ETS1(ETS原癌基因1)显著降低肺腺癌细胞的迁移和侵袭能力[14]。除Yoo等首次发现并报道miR-9500可通过靶向AKT丝/苏氨酸蛋白激酶1(AKT1)来抑制肺腺癌进展外,目前鲜有关于miR-9500在肺腺癌调控中的报道[15]。本研究发现过表达miR-9500可显著抑制肺腺癌细胞A549迁移侵袭,与Yoo等研究结果一致。
由于miRNAs与其靶基因3’UTR区配对的非严格性,使得一个miRNA可与多个靶基因结合,反之,不同miRNA也可与同一个靶基因结合进而发挥调控作用[16]。故本研究中,我们利用3个不同的数据库预测miR-9500的靶基因,取交集得到698个基因作为miR-9500的共同靶基因;并借助STRING和Cytoscape筛选出包括SMAD2在内的前20个基因作为miR-9500的核心靶基因。这些核心靶基因相关信号通路主要富集于癌症通路、ErbB信号通路、黏着斑、TGF-β信号通路等。ErbB家族由四个成员组成,包括EGFR、ErbB2、ErbB3和ErbB4且均为受体酪氨酸激酶。其中EGFR异常激活与包括肺腺癌在内的多种肿瘤发病机制密切相关[17-18]。黏着斑是细胞与周围环境的接触点,与肿瘤细胞迁移侵袭过程密切相关,该通路的异常激活可显著提高肺腺癌细胞的迁移侵袭能力,进而促进肺腺癌的发生发展[19-21]。TGF-β信号通路对细胞的增殖、分化、黏附、衰老和凋亡等多方面具有调控作用,并且在肿瘤迁移侵袭、肿瘤微环境及免疫反应中发挥重要作用[22]。如:非SMC凝聚素I复合亚基G(NCAPG)可通过TGF-β信号通路促进肺腺癌细胞的迁移[23]。miR-206可通过抑制TGF-β信号通路进而抑制肺腺癌的生长转移[24]。研究发现TGF-β/SMAD2信号通路在肿瘤进展早期可抑制肿瘤增殖,但在肿瘤进展后期发挥促进上皮间质转化(EMT)进程和肿瘤转移的作用[25]。这些研究证据提示,miR-9500可能通过ErbB信号通路、黏着斑、TGF-β信号通路等途径参与调控肺腺癌细胞迁移侵袭。
有研究表明MDM2原癌基因(MDM2)可通过激活SMAD2/3信号通路促进肺腺癌细胞迁移侵袭[26]。此外,miR-155-5p可通过靶向SMAD2进而显著降低肺腺癌细胞的迁移侵袭能力[27]。本研究通过生信分析发现排名前10的miR-9500关键靶基因中,仅有VAMP2、SMAD2及RXRA三者的表达水平与肺腺癌患者的总生存期显著相关。且与低表达组相比,VAMP2和RXRA高表达组肺腺癌患者的总生存期均显著延长,而SMAD2高表达组肺腺癌患者的总生存期显著缩短。这些研究证据表明SMAD2可能作为促癌因子参与调控肺腺癌细胞迁移侵袭。考虑到SMAD2与miR-9500之间的靶向评分高于VAMP2和RXRA这两个靶基因,且其3’ UTR有3个miR-9500结合位点,两者间可能存在较为可靠的靶向关系,我们进一步探究了miR-9500是否通过靶向SMAD2来抑制肺腺癌细胞迁移侵袭。结果表明:miR-9500可靶向结合SMAD2进而抑制SMAD2表达;过表达miR-9500可显著降低肺腺癌细胞的迁移侵袭能力。综上,miR-9500可通过靶向SMAD2来抑制肺腺癌细胞迁移侵袭,其可能作为肿瘤抑制因子在肺腺癌发生发展中发挥重要调控作用。
作者贡献:李 芳:选题与实验设计、论文审阅校正 韩采利:生信分析与作图、论文起草与修改 王 丽:细胞实验设计与实施 沈 慧:参与数据分析以及论文校对
| [1] |
Sung H, Ferlay J, Siegel RL, et al. Global Cancer Statistics 2020: GLOBOCAN Estimates of Incidence and Mortality Worldwide for 36 Cancers in 185 Countries[J]. CA Cancer J Clin, 2021, 71(3): 209-249. DOI:10.3322/caac.21660 |
| [2] |
崔国辉, 胡海. 肺癌分子标志物的研究进展及转化应用[J]. 生物医学转化, 2020, 1(1): 79-85. [Cui GH, Hu H. Research progress and translational application of molecular markers of lung cancer[J]. Sheng Wu Yi Xue Zhuan Hua, 2020, 1(1): 79-85.] |
| [3] |
Siegel RL, Miller KD, Fuchs HE, et al. Cancer Statistics, 2021[J]. CA Cancer J Clin, 2021, 71(1): 7-33. DOI:10.3322/caac.21654 |
| [4] |
Barta JA, Powell CA, Wisnivesky JP. Global Epidemiology of Lung Cancer[J]. Ann Glob Health, 2019, 85(1): 8. DOI:10.5334/aogh.2419 |
| [5] |
Wu KL, Tsai YM, Lien CT, et al. The Roles of MicroRNA in Lung Cancer[J]. Int J Mol Sci, 2019, 20(7): 1611. DOI:10.3390/ijms20071611 |
| [6] |
Wu X, Wu G, Zhang H, et al. MiR-196b Promotes the Invasion and Migration of Lung Adenocarcinoma Cells by Targeting AQP4[J]. Technol Cancer Res Treat, 2021, 20: 1533033820985868. |
| [7] |
Wang J, Zhang Y, Ge F. MiR-186 Suppressed Growth, Migration, and Invasion of Lung Adenocarcinoma Cells via Targeting Dicer1[J]. J Oncol, 2021, 2021: 6217469. |
| [8] |
Liu Y, Liang L, Ji L, et al. Potentiated lung adenocarcinoma (LUAD) cell growth, migration and invasion by lncRNA DARSAS1 via miR-188-5p/KLF12 axis[J]. Aging (Albany NY), 2021, 13(19): 23376-23392. |
| [9] |
Denisenko TV, Budkevich IN, Zhivotovsky B. Cell death-based treatment of lung adenocarcinoma[J]. Cell Death Dis, 2018, 9(2): 117. DOI:10.1038/s41419-017-0063-y |
| [10] |
Zhang Y, Wang LF, Gao JH, et al. Clinical significance of epithelial-mesenchymal transition-related molecules in lung adenocarcinoma[J]. Curr Oncol, 2019, 26(2): e121-e127. DOI:10.3747/co.26.4471 |
| [11] |
Wang Y, Zhang J, Zhong LY, et al. Hsa-miR-335 enhances cell migration and invasion in lung adenocarcinoma through targeting Copine-1[J]. MedComm (2020), 2021, 2(4): 810-820. |
| [12] |
Wang G, Zhou Y, Chen W, et al. miR-21-5p promotes lung adenocarcinoma cell proliferation, migration and invasion via targeting WWC2[J]. Cancer Biomark, 2020, 28(4): 549-559. DOI:10.3233/CBM-201489 |
| [13] |
Zhou LY, Zhang FW, Tong J, et al. MiR-191-5p inhibits lung adenocarcinoma by repressing SATB1 to inhibit Wnt pathway[J]. Mol Genet Genomic Med, 2020, 8(1): e1043. |
| [14] |
Tong L, Han WZ, Wang JL, et al. MicroRNA-365 inhibits the progression of lung adenocarcinoma through targeting ETS1 and inactivating AKT/mTOR pathway[J]. Eur Rev Med Pharmacol Sci, 2020, 24(9): 4836-4845. |
| [15] |
Yoo JK, Jung HY, Lee JM, et al. The novel miR-9500 regulates the proliferation and migration of human lung cancer cells by targeting Akt1[J]. Cell Death Differ, 2014, 21(7): 1150-1159. DOI:10.1038/cdd.2014.33 |
| [16] |
Pinweha P, Rattanapornsompong K, Charoensawan V, et al. MicroRNAs and oncogenic transcriptional regulatory networks controlling metabolic reprogramming in cancers[J]. Comput Struct Biotechnol J, 2016, 14: 223-233. DOI:10.1016/j.csbj.2016.05.005 |
| [17] |
Cho J. Mechanistic insights into differential requirement of receptor dimerization for oncogenic activation of mutant EGFRand its clinical perspective[J]. BMB Rep, 2020, 53(3): 133-141. DOI:10.5483/BMBRep.2020.53.3.025 |
| [18] |
Deng M, Liu B, Zhang Z, et al. Loss of G-protein-signaling modulator 2 accelerates proliferation of lung adenocarcinoma via EGFR signaling pathway[J]. Int J Biochem Cell Biol, 2020, 122: 105716. DOI:10.1016/j.biocel.2020.105716 |
| [19] |
Zhu L, Plow EF, Qin J. Initiation of focal adhesion assembly by talin and kindlin: A dynamic view[J]. Protein Sci, 2021, 30(3): 531-542. DOI:10.1002/pro.4014 |
| [20] |
Wu Y, Liu L, Shen X, et al. Plakophilin-2 Promotes Lung Adenocarcinoma Development via Enhancing Focal Adhesion and Epithelial-Mesenchymal Transition[J]. Cancer Manag Res, 2021, 13: 559-570. DOI:10.2147/CMAR.S281663 |
| [21] |
Kosibaty Z, Murata Y, Minami Y, et al. ECT2 promotes lung adenocarcinoma progression through extracellular matrix dynamics and focal adhesion signaling[J]. Cancer Sci, 2021, 112(2): 703-714. DOI:10.1111/cas.14743 |
| [22] |
Zhao H, Wei J, Sun J. Roles of TGF-β signaling pathway in tumor microenvirionment and cancer therapy[J]. Int Immunopharmacol, 2020, 89(Pt B): 107101. |
| [23] |
Wu Y, Lin Y, Pan J, et al. NCAPG promotes the progression of lung adenocarcinoma via the TGF-β signaling pathway[J]. Cancer Cell Int, 2021, 21(1): 443. DOI:10.1186/s12935-021-02138-w |
| [24] |
Watt K, Newsted D, Voorand E, et al. MicroRNA-206 suppresses TGF-β signalling to limit tumor growth and metastasis in lung adenocarcinoma[J]. Cell Signal, 2018, 50: 25-36. DOI:10.1016/j.cellsig.2018.06.008 |
| [25] |
Hao Y, Baker D, Ten Dijke P. TGF-β-Mediated Epithelial-Mesenchymal Transition and Cancer Metastasis[J]. Int J Mol Sci, 2019, 20(11): 2767. DOI:10.3390/ijms20112767 |
| [26] |
Tang Y, Xuan Y, Qiao G, et al. MDM2 promotes epithelial-mesenchymal transition through activation of Smad2/3 signaling pathway in lung adenocarcinoma[J]. Onco Targets Ther, 2019, 12: 2247-2258. DOI:10.2147/OTT.S185076 |
| [27] |
Lin J, Chen Y, Liu L, et al. MicroRNA-155-5p suppresses the migration and invasion of lung adenocarcinoma A549 cells by targeting Smad2[J]. Oncol Lett, 2018, 16(2): 2444-2452. |
 2023, Vol. 50
2023, Vol. 50
