文章信息
- 肝细胞癌中免疫相关基因的预后作用
- Prognostic Role of Immune-related Genes in Hepatocellular Carcinoma
- 肿瘤防治研究, 2022, 49(6): 599-605
- Cancer Research on Prevention and Treatment, 2022, 49(6): 599-605
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2022.21.1240
- 收稿日期: 2021-11-01
- 修回日期: 2022-01-22
肝癌是最常见的消化系统恶性肿瘤之一,居世界癌症死亡原因第三位,每年造成70多万人死亡[1]。肝细胞癌(HCC)占原发性肝癌的90%,已经成为全球公共卫生的一大挑战[1-2]。尽管目前对于HCC有多种治疗手段,但五年生存率不超过19%[3]。早期诊断率低、肿瘤进展迅速和高复发率可能与HCC患者预后不良有关,然而,HCC的进展机制尚不清楚。目前,生物信息学已经成为癌症研究的重要手段之一。因此,寻找预测HCC诊断、进展、预后的可靠生物标志物,对HCC的预防和治疗有重要意义。
研究表明,免疫微环境在HCC的发病机制中具有关键作用[4-5]。作为典型的炎性肿瘤,免疫耐受及逃避机制在HCC的发展中起着重要作用[6]。例如,骨髓来源的抑制性细胞通过增强免疫抑制因子的表达促进肿瘤的发展[7-8],所以需要探究如何找到免疫相关因素。本研究利用TCGA数据库对HCC免疫相关基因进行分析,并且建立HCC的预后预测模型,旨在为HCC的治疗提供新的思路。
1 资料与方法 1.1 原始资料下载与整理从癌症基因组图谱数据库(TCGA,https://portal.gdc.cancer.gov/)中获得371例HCC患者的转录组数据和临床病理信息。总生存时间≤90天的样本被排除在外,共获得329例患者生存数据。另外,收集了6例患者的HCC和癌旁样本用于关键基因的验证。收集的组织立即在液氮中冷冻,并储存在-80℃的冰箱中。本研究均通过患者知情同意,并得到了广西医科大学第一附属医院伦理委员会的批准。
1.2 免疫浸润评估和分组根据Bindea等的结果[9],本研究使用了一组免疫细胞表面标志基因。利用29个免疫数据集和ssGSEA方法结合R软件“GSVA”包将TCGA肝细胞癌转录组数据进行分组。来自TCGA的肝细胞癌样本被“hclust”包分为低免疫细胞浸润组和高免疫细胞浸润组[10]。ESTIMATE算法用于分析免疫评分、基质评分、肿瘤纯度和ESTIMATE评分,并绘制聚类热图以进行有效分组。
1.3 肝细胞癌免疫相关基因的筛选TCGA数据分为高免疫细胞浸润组和低免疫细胞浸润组。根据FDR < 0.05和|log2FC| > 1的标准,使用“limma”包分析差异基因,其中FDR为错误发现率,FC为差异倍数。另外,从immport数据库中下载1 793个免疫相关基因。维恩图从上述两个分析中确定真正的免疫相关基因。
1.4 功能富集分析为了更好地了解差异基因的潜在生物学功能,在R中使用“clusterProfiler”包[11]进行了涵盖生物过程(BP)、分子功能(MF)和细胞成分(CC)的基因本体(GO)功能富集和京都基因百科全书(KEGG)分析。P < 0.05为差异有统计意义,结果用“ggplot2”包可视化。
1.5 预后模型的建立和验证进行单因素Cox回归分析,以确定差异基因的预后价值,P < 0.05为差异有统计学意义。LASSO回归算法过滤预后基因,筛选出4个预后相关免疫基因。基于上述预后基因,进行逐步回归多因素Cox分析来构建预后基因特征。预后基因风险评分公式的构建如下[12]:风险评分=∑i(βi*Ei)。Ei指预后基因的表达;βi指预后基因的回归系数。根据中位风险评分将患者分为高风险组和低风险组。Kaplan-Meier生存分析用于评估预后模型的预测能力。
1.6 独立预测分析和列线图构建进行单因素和多因素Cox比例风险回归分析,以确定预后模型的预测能力是否独立于常规临床特征。P < 0.05为差异有统计学意义。计算危险比和95%置信区间。基于临床病理特征,对整个队列样本进行分层分析。所有独立的预后因素用于建立列线图,以评估HCC患者的1、3和5年生存概率。
1.7 qRT-PCR检测使用RNAisoPlusreagent(日本TaKaRa公司)提取HCC组织和癌旁组织的总RNA,使用PrimeScriptTMRT reagent Kit with gDNA Eraser(日本TaKaRa公司)在37℃ 15 min和85℃ 5 s将其反转录为cDNA。在ABI7500system系统(美国API公司)上,用TBGreen®PremixExTaqTMIIKit(日本TaKaRa公司)进行qRT-PCR实验。引物序列见表 1。
使用R(v.3.6.0)软件进行统计分析。连续变量总结为均数±标准差。使用Wilcox检验比较各组差异。定性变量采用费希尔精确检验或皮尔逊卡方检验。使用对数秩检验计算生存时间差异,P < 0.05为差异有统计学意义。绘制Kaplan-Meier曲线显示存活时间差异。
2 结果 2.1 肝细胞癌组织的免疫分组肝细胞癌样本根据免疫浸润程度分为高免疫细胞浸润组和低免疫细胞浸润组。为验证上述分组方案的真实性,ESTIMATE算法分析结果提示高免疫细胞浸润组的肿瘤纯度低于低免疫细胞浸润组,而ESTIMATE评分、免疫评分和基质评分值高于低免疫细胞浸润组,见图 1。高免疫细胞浸润组的免疫成分多于低免疫细胞浸润组,TIGIT、PD-L1、PD-1、LAG3、TIM-3、CTLA4和HLA家族在高免疫细胞浸润组中表达也较高。CIBERSORT方法分析结果表明,高免疫细胞浸润组中免疫细胞类型较多。
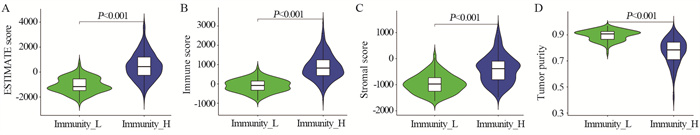
|
| 图 1 肝细胞癌的分组和验证 Figure 1 Grouping and validation of hepatocellular carcinoma |
基于截止值(|log2FC| > 1和FDR < 0.05),在低免疫细胞浸润组和高免疫细胞浸润组之间鉴定了1 123个差异基因,其中包括946个上调的差异基因和177个下调的差异基因。基于来自immport数据库的免疫基因和来自高、低免疫细胞浸润组的差异基因进行维恩分析,发现了337个重叠基因,它们被认为是真正的差异基因。
2.3 差异基因的GO和KEGG分析为了探索差异基因的潜在功能,分析了337个差异基因的GO和KEGG的功能富集。在BP方面,差异基因在免疫反应−激活细胞表面受体信号通路、循环免疫球蛋白介导的体液免疫应答和体液免疫反应等方面均显著富集。对于CC,差异基因在免疫球蛋白复合物、质膜外侧和循环免疫球蛋白复合物等方面富集。对于MF,差异基因在抗原结合、免疫球蛋白受体结合和细胞因子活性等方面富集,见图 2A。此外,KEGG分析表明,大多数差异基因相关途径与免疫反应显著相关,见图 2B。

|
| BP: biological process; CC: cellular component; MF: molecular function. 图 2 差异表达基因的GO(A)和KEGG(B)分析 Figure 2 GO(A) and KEGG(B) analyses of differentially-expressed genes |
将329例患者的差异基因表达谱与生存数据相结合,进行单因素Cox回归分析,获得36个与患者总体生存期(OS)相关的mRNAs。执行LASSO回归算法。通过多因素Cox回归确定由4个mRNAs组成的模型为预测患者OS的最佳预后模型,见图 3A。基于TCGA队列分析,4个mRNAs在肿瘤组织中均表达下调,包括S100A9、HMOX1、IL18RAP和FCER1G。

|
| A: multivariate Cox proportional hazards regression analysis; B: Kaplan-Meier curves of the low and high-risk groups. 图 3 肝细胞癌预后模型的构建及总体生存的评估b Figure 3 Construction of prognostic model and evaluation of overall survival of HCC |
根据中位风险评分,将329例肝癌样本分为高风险组(n=164)和低风险组(n=165)。随着风险评分的升高,高风险组患者死亡人数多于低风险组。生存曲线提示,与低风险组相比,高风险组的总生存时间明显缩短(P=1.547X10-7),见图 3B。1、3和5年OS ROC曲线的AUC值分别为0.752、0.775和0.705,表明该模型具有良好的敏感度和特异性(图请扫描OSID码)。
排除临床数据不完整的患者后,共获得261例患者临床病理特征,其中高风险组124例,低风险组137例,两组患者的生存时间按TNM分期Ⅰ+Ⅱ、组织学分级1+2、组织学分级3+4、年龄 > 60岁、女性、男性、有血管侵犯和无血管侵犯不同亚组进行分析,结果显示高风险组患者的总生存时间比低风险组短,见图 4。
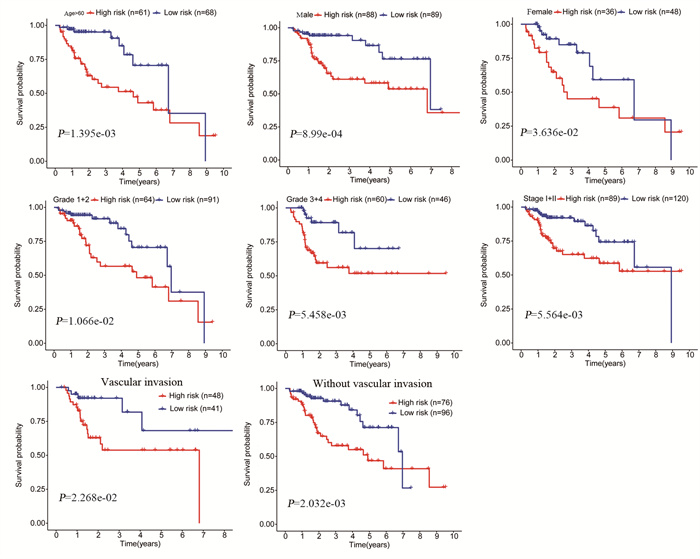
|
| 图 4 不同亚组中低风险和高风险人群的生存时间 Figure 4 Survival time of low- and high-risk patients in different subgroups |
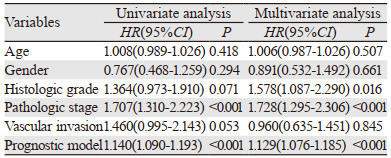
单因素分析显示,病理分期(P < 0.001)和4-mRNAs模型(P < 0.001)与总生存期显著相关。多因素分析显示,4-mRNAs模型(P < 0.001)和病理分期(P < 0.001)仍然是总生存率的独立影响因素,见表 2,证实了模型的可靠性。根据ROC曲线下面积的比较,确定风险评分(AUC=0.784)比其他HCC预后因素能更准确地预测死亡率(相关数据请扫描OSID码)。
|
使用以下3个独立的预后因素来预测列线图:TNM分期、组织学分级和风险评分,见图 5。将不同的独立因子与相应的分值进行匹配,通过相加得到总分值。最后,获得每例患者的1、3和5年生存概率。总分越高,列线图中的预后越差。校准图显示列线图的有效性好,45°线代表了最佳预测情况(图请扫描OSID码)。
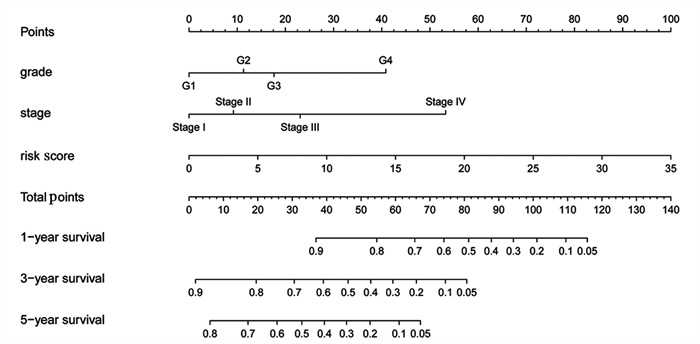
|
| 图 5 列线图分析 Figure 5 Nomogram analysis |
分析6对肝细胞癌和癌旁样本,以验证4个基因的表达水平。结果表明,与正常肝组织相比,S100A9、HMOX1、IL18RAP和FCER1G在肝细胞癌组织中的表达显著下调,且差异均有统计学意义(P < 0.05)。与之前的结果一致,与正常样品相比,肝细胞癌样品中4个基因的表达水平显著下调(图请扫描OSID码)。
3 讨论目前,肝癌的治疗主要包括肝部分切除术、肝移植、射频消融、经动脉化疗栓塞以及药物治疗。然而,这些治疗方法的效果有限,到目前为止,还没有有效的生物标志物来准确预测肝癌患者的生存率。因此,鉴定可靠的生物标志物和肝癌患者预后的预测因素非常重要。
越来越多的研究表明,免疫微环境对肝癌患者的发生、进展、治疗反应和远期预后有很大影响[13-14]。免疫细胞作为机体的监测细胞,可以干扰分子信号,识别肿瘤细胞的异常增殖。这些细胞在癌症的生物学功能中具有重要作用,包括肿瘤的增殖、转移和侵袭[15]。此外,免疫逃逸机制的存在往往会影响肿瘤的预后。HCC肿瘤微环境的多种免疫抑制细胞参与了肿瘤细胞的免疫逃逸[16]。以往研究表明,肿瘤相关巨噬细胞中的M2表型为肿瘤生长产生炎性反应环境,并通过诱导肝癌的肿瘤血管生成促进肿瘤进展和转移,大大降低了患者的生存率[17]。然而,一些癌细胞可以避免被免疫系统检测到。这些癌细胞可以通过抑制免疫反应,如抗原呈递等来逃避免疫系统,从而促进肿瘤侵袭[18]。研究报道,β-连环蛋白的激活促进免疫逃逸和对程序性死亡受体1(PD-1)的抵抗,并且可能代表HCC患者T细胞排斥的新生物标志物[19]。PD-1作为一种免疫检查点分子,通过降低T细胞活性和增强肿瘤细胞的免疫耐受性,导致肿瘤患者预后不良。也有报道称,纳武单抗作为PD-1的免疫抑制剂,可能在晚期肝癌中发挥作用[20-21]。
本研究以肝细胞癌的免疫浸润状态为重点,通过分析肝细胞癌样本之间的差异,筛选了337个基因作为免疫细胞浸润相关的差异基因。进一步探讨了免疫细胞浸润相关的差异基因的功能富集,这可能为探索差异基因调控肝细胞癌发展和预后的机制提供更多细节。
本研究建立的肝癌预后模型由四个免疫相关基因组成,即S100A9、HMOX1、IL18RAP和FCER1G。S100钙结合蛋白A9(S100A9)是一种钙卫蛋白,在多种细胞类型中表达,并且在骨髓细胞中大量存在,因此也被称为骨髓相关蛋白[22-23]。S100A9在多种癌症中过度表达[24-26]。S100A9招募髓源性抑制细胞(MDSCs)通过炎性反应途径促进癌症生长[27]。另外,有研究表明S100A9主要位于HCC肿瘤组织中浸润的巨噬细胞和中性粒细胞中,很少有HCC细胞本身表达S100A9[28]。血红素加氧酶1(HMOX1)是血红素分解代谢的核心酶,与肿瘤生长和转移有关[29-30]。迄今为止,许多研究人员提出HMOX1与肿瘤发生密切相关,如抗细胞凋亡、细胞增殖、侵袭和转移,并且它可以成为潜在的癌症治疗靶点。有研究表明,增加的细胞内HO-1蛋白通过抑制IL-6表达显著抑制了人类HCC细胞的迁移和生长[31]。白介素18受体辅助蛋白(IL18RAP)是白介素1受体家族的成员,由多种细胞类型表达,包括免疫细胞和基质细胞[32]。IL18RAP通过抑制涉及caspase-3的血管生成和凋亡信号转导,有效抑制HCC细胞的生长,充分证明了IL18RAP的抗肿瘤作用[33]。IgE的Fc组分高亲和性1受体(FCER1G)是参与过敏反应的关键分子[34]。FCER1G对慢性炎性反应至关重要,并在死亡激活信号转导、诱导细胞凋亡中发挥重要作用[35]。FCER1G与肾透明细胞癌的进展和预后相关[35]。目前,FCER1G在肝细胞癌中的作用未见报道。本研究使用六对肝细胞癌组织和癌旁组织检测这四个基因的mRNA水平。结果表明,这四种mRNA在肿瘤组织中表达下调。实验结果与TCGA数据库结果基本一致,这也证实了模型的可靠性。
目前,一些由多个基因组成的预后模型可以预测肝癌的预后,如Xu等开发的由8个免疫相关mRNA组成的预后模型[36]。然而,该模型在肝癌免疫微环境的研究中没有得到广泛应用。本研究结果表明,由这四个基因组成的风险模型能够区分高风险人群和低风险人群,并准确预测预后。根据TCGA数据分析,发现风险评分高的HCC患者预后较差。值得注意的是,风险模型是肝癌的独立预后风险因素。基于这一特征,构建了预后列线图,以帮助患者制定短期治疗策略。因此,该预后模型可能参与HCC的发展,并可能成为一种可靠的临床生物标志物。
人类白细胞抗原(HLA)基因的多态性被认为与多种恶性肿瘤的敏感度有关,并参与致癌、肿瘤增殖和免疫逃逸的进程[37-38]。有研究发现,与早期实体癌的周围正常组织相比,HLAI类在癌细胞中高表达,这可能导致基于细胞毒性T淋巴细胞(CTL)的癌症免疫疗法的抗肿瘤效应[39]。有研究结果显示,CD8+CTL通过HLAI类特异性上调肿瘤细胞系中PD-L1的表达[40]。
根据目前的研究,已鉴定的mRNAs可能成为免疫治疗的潜在靶点,并在预测和评估HCC患者的OS方面具有较大潜力。本研究的优势在于挖掘和分析TCGA转录组数据,并建立一种新的免疫相关预后模型。该模型在预测HCC患者的OS方面效果显著,反映了HCC肿瘤微环境的免疫状态。然而,目前研究的缺点是该模型没有得到其他数据库的验证。未来,4个mRNA标记的预后价值需要在其他独立的HCC数据集和更多的临床患者中进一步验证。同时,本研究缺乏蛋白质组学和免疫组织化学的检测。因此,这些免疫相关mRNAs的应用价值需要在后续研究中进一步阐明。尽管存在这些局限性,目前的研究建立了一种新的HCC免疫相关基因特征,它与患者风险和OS密切相关。
综上所述,本研究通过分析肝细胞癌样本和免疫细胞浸润数据之间的差异,构建了预后模型并确定了与免疫相关的肝细胞癌核心基因。鉴定出的基因可能用于靶向治疗和免疫治疗,为临床提供新的治疗思路。
作者贡献:
王珏、金宗睿:设计实验、撰写论文
王维、易麒麟:数据下载、文献搜集
王继龙、朱海、徐邦浩:数据整理、分析数据
郭雅、文张:论文审阅、指导实验
| [1] |
Sung H, Ferlay J, Siegel RL, et al. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2021, 71(3): 209-249. DOI:10.3322/caac.21660 |
| [2] |
Nault JC, Villanueva A. Biomarkers for Hepatobiliary Cancers[J]. Hepatology, 2021, 73(Suppl 1): 115-127. |
| [3] |
Allemani C, Matsuda T, Di Carlo V, et al. Global surveillance of trends in cancer survival 2000-14 (CONCORD-3): analysis of individual records for 37 513 025 patients diagnosed with one of 18 cancers from 322 population-based registries in 71 countries[J]. Lancet, 2018, 391(10125): 1023-1075. DOI:10.1016/S0140-6736(17)33326-3 |
| [4] |
Oura K, Morishita A, Tani J, et al. Tumor Immune Microenvironment and Immunosuppressive Therapy in Hepatocellular Carcinoma: A Review[J]. Int J Mol Sci, 2021, 22(11): 5801. DOI:10.3390/ijms22115801 |
| [5] |
邹添添, 覃伟, 朱迎, 等. 肝癌免疫微环境与免疫治疗: 研究进展与发展趋势[J]. 中国普通外科杂志, 2020, 29(7): 785-797. [Zou TT, Qin W, Zhu Y, et al. Immune microenvironment and immunotherapy in hepatocellular carcinoma: research progress and development directions[J]. Zhongguo Pu Tong Wai Ke Za Zhi, 2020, 29(7): 785-797.] |
| [6] |
Singh A, Beechinor RJ, Huynh JC, et al. Immunotherapy Updates in Advanced Hepatocellular Carcinoma[J]. Cancers (Basel), 2021, 13(9): 2164. DOI:10.3390/cancers13092164 |
| [7] |
Law AMK, Valdes-Mora F, Gallego-Ortega D. Myeloid-Derived Suppressor Cells as a Therapeutic Target for Cancer[J]. Cells, 2020, 9(3): 561. DOI:10.3390/cells9030561 |
| [8] |
Liu M, Zhou J, Liu X, et al. Targeting monocyte-intrinsic enhancer reprogramming improves immunotherapy efficacy in hepatocellular carcinoma[J]. Gut, 2020, 69(2): 365-379. DOI:10.1136/gutjnl-2018-317257 |
| [9] |
Bindea G, Mlecnik B, Tosolini M, et al. Spatiotemporal Dynamics of Intratumoral Immune Cells Reveal the Immune Landscape in Human Cancer[J]. Immunity, 2013, 39(4): 782-795. DOI:10.1016/j.immuni.2013.10.003 |
| [10] |
Gonnord P, Costa M, Abreu A, et al. Multiparametric analysis of CD8+ T cell compartment phenotype in chronic lymphocytic leukemia reveals a signature associated with progression toward therapy[J]. Oncoimmunology, 2019, 8(4): e1570774. DOI:10.1080/2162402X.2019.1570774 |
| [11] |
Yu G, Wang LG, Han Y, et al. clusterProfiler: an R package for comparing biological themes among gene clusters[J]. OMICS, 2012, 16(5): 284-287. DOI:10.1089/omi.2011.0118 |
| [12] |
Liao X, Huang K, Huang R, et al. Genome-scale analysis to identify prognostic markers in patients with early-stage pancreatic ductal adenocarcinoma after pancreaticoduodenectomy[J]. Onco Targets Ther, 2017, 10: 4493-4506. DOI:10.2147/OTT.S142557 |
| [13] |
Giraud J, Chalopin D, Blanc JF, et al. Hepatocellular Carcinoma Immune Landscape and the Potential of Immunotherapies[J]. Front Immunol, 2021, 12: 655697. DOI:10.3389/fimmu.2021.655697 |
| [14] |
Lim CJ, Lee YH, Pan L, et al. Multidimensional analyses reveal distinct immune microenvironment in hepatitis B virus-related hepatocellular carcinoma[J]. Gut, 2019, 68(5): 916-927. DOI:10.1136/gutjnl-2018-316510 |
| [15] |
Bindea G, Mlecnik B, Galon J. Tumor spread or siege immunity: dissemination to distant metastasis or not[J]. OncoImmunology, 2021, 10(1): 1919377. DOI:10.1080/2162402X.2021.1919377 |
| [16] |
Hao X, Sun G, Zhang Y, et al. Targeting Immune Cells in the Tumor Microenvironment of HCC: New Opportunities and Challenges[J]. Front Cell Dev Biol, 2021, 9: 775462. DOI:10.3389/fcell.2021.775462 |
| [17] |
Zhu S, Yi M, Wu Y, et al. Roles of tumor-associated macrophages in tumor progression: implications on therapeutic strategies[J]. Exp Hematol Oncol, 2021, 10(1): 60. DOI:10.1186/s40164-021-00252-z |
| [18] |
Fu Y, Liu S, Zeng S, et al. From bench to bed: the tumor immune microenvironment and current immunotherapeutic strategies for hepatocellular carcinoma[J]. J Exp Clin Cancer Res, 2019, 38(1): 396. DOI:10.1186/s13046-019-1396-4 |
| [19] |
Ruiz De Galarreta M, Bresnahan E, Molina-Sánchez P, et al. β-Catenin Activation Promotes Immune Escape and Resistance to Anti-PD-1 Therapy in Hepatocellular Carcinoma[J]. Cancer Discov, 2019, 9(8): 1124-1141. DOI:10.1158/2159-8290.CD-19-0074 |
| [20] |
El-Khoueiry AB, Sangro B, Yau T, et al. Nivolumab in patients with advanced hepatocellular carcinoma (CheckMate 040): an open-label, non-comparative, phase 1/2 dose escalation and expansion trial[J]. Lancet, 2017, 389(10088): 2492-2502. DOI:10.1016/S0140-6736(17)31046-2 |
| [21] |
吴占波, 任秀宝. 免疫检查点阻断剂在肝癌治疗中的研究进展[J]. 肿瘤防治研究, 2016, 43(9): 811-813. [Wu ZB, Ren XB. Advance of Immune Checkpoint Inhibitors in Therapy of Liver Cancer[J]. Zhong Liu Fang Zhi Yan Jiu, 2016, 43(9): 811-813. DOI:10.3971/j.issn.1000-8578.2016.09.017] |
| [22] |
Fan ZP, Peng ML, Chen YY, et al. S100A9 Activates the Immunosuppressive Switch Through the PI3K/Akt Pathway to Maintain the Immune Suppression Function of Testicular Macrophages[J]. Front Immunol, 2021, 12: 743354. DOI:10.3389/fimmu.2021.743354 |
| [23] |
Shi L, Zhao Y, Fei C, et al. Cellular senescence induced by S100A9 in mesenchymal stromal cells through NLRP3 inflammasome activation[J]. Aging(Albany NY), 2019, 11(21): 9626-9642. |
| [24] |
Jo SH, Heo WH, Son HY, et al. S100A8/A9 mediate the reprograming of normal mammary epithelial cells induced by dynamic cell-cell interactions with adjacent breast cancer cells[J]. Sci Rep, 2021, 11(1): 1337. DOI:10.1038/s41598-020-80625-2 |
| [25] |
Zhao Z, Zhang C, Zhao Q. S100A9 as a novel diagnostic and prognostic biomarker in human gastric cancer[J]. Scand J Gastroenterol, 2020, 55(3): 338-346. DOI:10.1080/00365521.2020.1737883 |
| [26] |
杜雪梅, 昌红, 孙焕英, 等. 钙离子结合蛋白S100A9在乳腺癌中表达及临床意义[J]. 细胞与分子免疫学杂志, 2012, 28(6): 637-639. [Du XM, Chang H, Sun HH, et al. Expression and clinical significance of calcium-binding protein S100A9 in breast cancer[J]. Xi Bao Yu Fen Zi Mian Yi Xue Za Zhi, 2012, 28(6): 637-639.] |
| [27] |
Huang M, Wu R, Chen L, et al. S100A9 Regulates MDSCs-Mediated Immune Suppression via the RAGE and TLR4 Signaling Pathways in Colorectal Carcinoma[J]. Front Immunol, 2019, 10: 2243. DOI:10.3389/fimmu.2019.02243 |
| [28] |
Liao J, Li JZ, Xu J, et al. High S100A9 + cell density predicts a poor prognosis in hepatocellular carcinoma patients after curative resection[J]. Aging(Albany NY), 2021, 13(12): 16367-16380. |
| [29] |
Chiang SK, Chen SE, Chang LC, et al. A Dual Role of Heme Oxygenase-1 in Cancer Cells[J]. Int J Mol Sci, 2018, 20(1): 39. DOI:10.3390/ijms20010039 |
| [30] |
巫亚龙, 唐圣松. 血红素加氧酶-1的抗肿瘤细胞凋亡作用研究进展[J]. 临床与病理杂志, 2016, 36(4): 496-500. [Wu YL, Tang SS. Advances in apoptosis of heme oxygenase-1 anti-tumor cell[J]. Lin Chuang Yu Bing Li Za Zhi, 2016, 36(4): 496-500.] |
| [31] |
Zou C, Zhang H, Li Q, et al. Heme oxygenase-1: a molecular brake on hepatocellular carcinoma cell migration[J]. Carcinogenesis, 2011, 32(12): 1840-1848. DOI:10.1093/carcin/bgr225 |
| [32] |
Mantovani A, Dinarello CA, Molgora M, et al. Interleukin-1 and Related Cytokines in the Regulation of Inflammation and Immunity[J]. Immunity, 2019, 50(4): 778-795. DOI:10.1016/j.immuni.2019.03.012 |
| [33] |
Liu WW, Hu M, Wang YM, et al. Overexpression of interleukin-18 protein reduces viability and induces apoptosis of tongue squamous cell carcinoma cells by activation of glycogen synthase kinase-3 beta signaling[J]. Oncol Rep, 2015, 33(3): 1049-1056. DOI:10.3892/or.2015.3724 |
| [34] |
Fu L, Cheng Z, Dong F, et al. Enhanced expression of FCER1G predicts positive prognosis in multiple myeloma[J]. J Cancer, 2020, 11(5): 1182-1194. DOI:10.7150/jca.37313 |
| [35] |
Chen L, Yuan L, Wang Y, et al. Co-expression network analysis identified FCER1G in association with progression and prognosis in human clear cell renal cell carcinoma[J]. Int J Biol Sci, 2017, 13(11): 1361-1372. DOI:10.7150/ijbs.21657 |
| [36] |
Xu D, Wang Y, Zhou K, et al. Development and Validation of a Novel 8 Immune Gene Prognostic Signature Based on the Immune Expression Profile for Hepatocellular Carcinoma[J]. Onco Targets Ther, 2020, 13: 8125-8140. DOI:10.2147/OTT.S263047 |
| [37] |
Sabbatino F, Liguori L, Polcaro G, et al. Role of Human Leukocyte Antigen System as A Predictive Biomarker for Checkpoint-Based Immunotherapy in Cancer Patients[J]. Int J Mol Sci, 2020, 21(19): 7295. DOI:10.3390/ijms21197295 |
| [38] |
王静, 魏素菊. HLA-Ⅰ类在肺癌免疫治疗中相关研究进展[J]. 中国免疫学杂志, 2021, 37(19): 2428-2432. [Wang J, Wei SJ. Research progress of HLA-Ⅰin lung cancer immunotherapy[J]. Zhongguo Mian Yi Xue Za Zhi, 2021, 37(19): 2428-2432. DOI:10.3969/j.issn.1000-484X.2021.19.023] |
| [39] |
Akazawa Y, Nobuoka D, Takahashi M, et al. Higher human lymphocyte antigen class I expression in early-stage cancer cells leads to high sensitivity for cytotoxic T lymphocytes[J]. Cancer Sci, 2019, 110(6): 1842-1852. DOI:10.1111/cas.14022 |
| [40] |
Huang CY, Wang Y, Luo GY, et al. Relationship Between PD-L1 Expression and CD8+ T-cell Immune Responses in Hepatocellular Carcinoma[J]. J Immunother, 2017, 40(9): 323-333. DOI:10.1097/CJI.0000000000000187 |
 2022, Vol. 49
2022, Vol. 49



