文章信息
- 基于TCGA数据库的消化道肿瘤LncRNA预后风险评分模型
- LncRNA Prognostic Risk Scoring Model for Gastrointestinal Tumors Based on TCGA Database
- 肿瘤防治研究, 2022, 49(6): 606-611
- Cancer Research on Prevention and Treatment, 2022, 49(6): 606-611
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2022.21.1159
- 收稿日期: 2021-10-14
- 修回日期: 2022-03-01
我国是消化系统肿瘤高发国家。2020年全球癌症统计报告显示,结肠癌、胃癌、直肠癌、食管癌均位于世界新发与死亡癌症的前十位,占新发病例总数的18.5%、新增死亡例数的22.4%[1]。得益于免疫治疗、靶向治疗等新技术的不断发展[2-3],消化道肿瘤的发病率、死亡率持续下降,但患者的总生存期(overall survi val, OS)依然较低,预后不良[4]。因此,我们急需行之有效的预后预测模型来指导临床治疗。
肿瘤分子标志物在基因组学的研究不断深入,使得肿瘤生物学研究逐渐步入泛癌研究阶段。泛癌同时对多种不同类型的肿瘤基因组进行分析,能够寻找不同肿瘤的共同特征,寻找多种肿瘤的诊断、治疗及预后广谱靶点[5]。由于消化道解剖结构与生理功能相似,食管癌、胃癌、结肠癌与直肠癌数据合并分析并建立消化道泛癌的预后模型有助于寻找四种肿瘤在发生发展过程中的相同点。
肿瘤的精准治疗要求根据患者特异性指标寻找精准的生物标志物。长非编码RNA是一类特殊的的基因转录本,虽然绝大多数不参与蛋白质的编码[6-7],但与转录及转录后调控密切相关[8],广泛参与了肿瘤的发生发展[9],能够成为肿瘤诊断和预后的潜在标志物[10]。
癌症基因组图谱TCGA数据库共收集了33种癌症的11 000多例患者的相关数据,极大地提高了研究者对肿瘤生物学的理解[11]。本研究收集整理了来自TCGA数据库的消化道肿瘤相关数据,运用Cox回归和Lasso回归分析进行预后相关基因筛选和模型构建,以期能够对临床治疗进行辅助和指导。
1 资料与方法 1.1 资料来源从UCSC-Xena(http://xena.ucsc.edu)数据库下载经过归一化处理后的TCGA数据库中的消化道肿瘤患者(共1 117例,其中食管癌172例、胃癌381例、结肠癌487例、直肠癌167例)的RNA测序(RNA Seq)数据和临床相关数据,整合数据,用于后续分析。
1.2 方法 1.2.1 消化道肿瘤lncRNA表达数据及临床资料收集与处理首先对病例样本进行筛选:(1)排除无完整临床病理资料的病例;(2)排除预后相关信息不全的病例。纳入有完整临床病理及预后资料(包括性别、年龄、病理分期和生存状况)的病例,Ensemble数据库进行基因注释得到纳入研究的lncRNA表达谱。
1.2.2 消化道肿瘤lncRNA差异分析及单因素Cox分析使用配对t检验对94例患者的肿瘤样本及配对正常样本进行统计学分析,计算logFC值并初步筛选差异表达的lncRNA。对结果进行单因素Cox回归分析筛选预后相关的差异lncRNA。
1.2.3 Lasso及多因素Cox回归分析最小绝对值收敛和选择算子(least absolute shrinkage and selection operator, Lasso)算法,可调整自变量的回归系数,首先将对模型贡献小的变量的系数压缩为零,再进行剔除,降低模型过度拟合的程度,使模型中均为相对更加重要的变量。
将1 117例样本随机分成训练集(894例)和验证集(223例)两部分。R语言中的survival包和glmnet包进行Lasso回归,选择最优的λ纳入模型中,得到与预后显著相关的lncRNA,再利用多因素Cox回归分析计算每个lncRNA的回归系数,根据每个lncRNA的表达值及回归系数按照如下公式构建消化道肿瘤预后风险模型:
Risk Score(RS)=∑explncRNAn*αlncRNAn
该公式中,Risk Score代表风险值,exp代表每个lncRNA在样本中的表达值,α代表多因素Cox回归分析中计算得到的每个lncRNA的回归系数。
1.2.4 模型效能评估基于预后风险模型,得出每例患者的评分,将患者进一步分为高、低风险组。同时将模型中的各个基因在不同肿瘤高低风险组中的表达量进行分析。
采用时间依赖的受试者工作特征曲线(receiver operating characteristic curve, ROC)评估模型预测性能,Kaplan-Meier生存曲线和Log rank检验分析患者生存差异,Cox回归综合评价包括模型风险评分在内的各个临床病理特征对预后的影响。
1.2.5 功能富集分析根据每例消化道肿瘤样本的高低风险状态划分,R包“clusterProfiler”对高风险组与低风险组mRNA表达数据进行差异分析后分别对上调基因与下调基因进行基因富集分析。
1.3 统计学方法数据预处理、模型的构建、验证及评价均基于R语言(Version 4.0.3)及相关的R程序包。所有统计学检验均为双侧检验,P < 0.05为差异有统计学意义。
2 结果 2.1 差异表达lncRNA筛选本研究以校正后的P < 0.05且差异表达倍数 > 1(FDR < 0.05且|logFC| > 1)为筛选标准,最终得到了1 426个差异表达的lncRNA,其中1 280个lncRNA表达上调,146个lncRNA表达下调。
2.2 构建模型并评估效能采用单因素Cox回归在全体差异表达的lncRNA中筛选与预后显著相关的lncRNA,以P < 0.001为标准选择出94个lncRNA。为避免模型过度拟合并筛选出与消化道肿瘤患者预后更加相关的lncRNA,对单因素分析得到的lncRNA进行Lasso回归分析,λ取最优值时,得到36个lncRNA(图请扫描本文OSID码),对结果继续进行多因素Cox回归分析,得到最相关的13个lncRNA,见图 1A。计算每个lncRNA的危险比及95%CI,通过将每个lncRNA对应的系数代入公式得到消化道肿瘤预后风险评分模型,具体如下:Riskscore=0.5117*expAC006357.1+0.9657*expAC010998.1+0.5926*expAC011586.2+(-0.4004)*expAC099792.1+(-0.3537)*expAC110772.1+0.4013*expAC12493.1+0.3443*expAL513123.1+(-0.6706)*expAL590483.1+0.8417*expDSCR10+1.0330*expLINC02382+0.7515*expLINC02866+(-0.2830)*expMIR3142HG+0.3331*expRHOXF1P1。

|
| A: forest plot of the prognostic model; B: ROC curve of predicted survival C: survival status distribution of the high-risk group and the low-risk group; D: survival curves of the patients in the high-risk group and the low-risk group. 图 1 模型在训练组中预后预测效能的评估 Figure 1 Evaluation of prognostic prediction effectiveness of the model in training group |
利用该模型对训练集894例样本进行时间依赖ROC分析,结果显示,三年曲线下面积(AUC)=0.746,提示该模型对患者预后具有良好的预测能力,见图 1B。
根据模型,计算训练集患者的风险评分,采用评分中位数(1.279)作为cut-off值将所有样本分为高风险组和低风险组。生存时间分布图显示,随着风险评分的升高,消化道肿瘤患者死亡例数逐渐增多,见图 1C。K-M生存曲线的Log rank检验显示,比较五年总生存率,低分险组显著高于高风险组(P < 0.01),见图 1D。
分析模型中各个基因在食管癌、胃腺癌、结肠癌和直肠癌高低风险组中的表达,结果显示,在绝大多数分组中,高低风险组之间差异显著,见图 2。
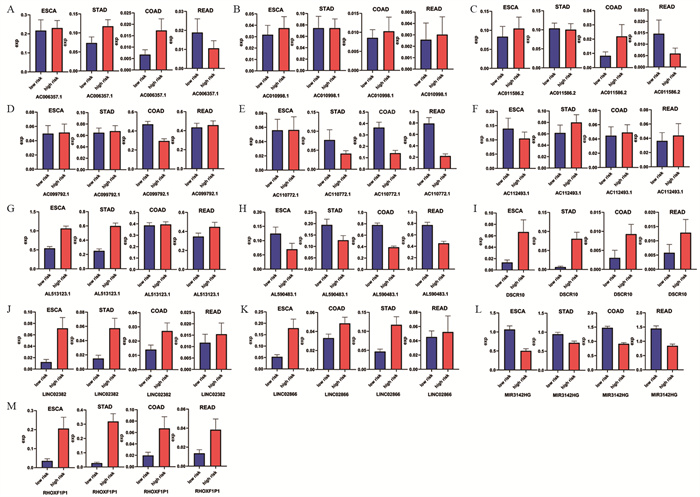
|
| A: AC006357.1; B: AC010998.1; C: AC011586.2; D: AC099792.1; E: AC110772.1; F: AC112493.1; G: AL513123.1; H: AL590483.1; I: DSCR10; J: LINC02382; K: LINC02866; L: MIR3142HG; M: RHOXF1P1. ESCA: esophageal carcinoma; STAD: stomach adenocarcinoma; COAD: colon adenocarcinoma; READ: rectum adenocarcinoma. 图 2 模型中基因的表达量在四种消化道肿瘤高低风险组中差异显著 Figure 2 Genes expression in the model were significantly different between high and low risk groups in four gastrointestinal tumors |
利用223例验证集样本数据进行内部验证分析,结果显示,该模型的三年AUC为0.704,提示该模型在验证集中预测能力良好,具有较好的外推性,见图 3A。生存分析显示,低风险组总生存率依然显著高于高风险组(P < 0.01),见图 3B。与其他临床性状评价模型效果相比,风险评分作为变量时AUC最大,见图 3C。
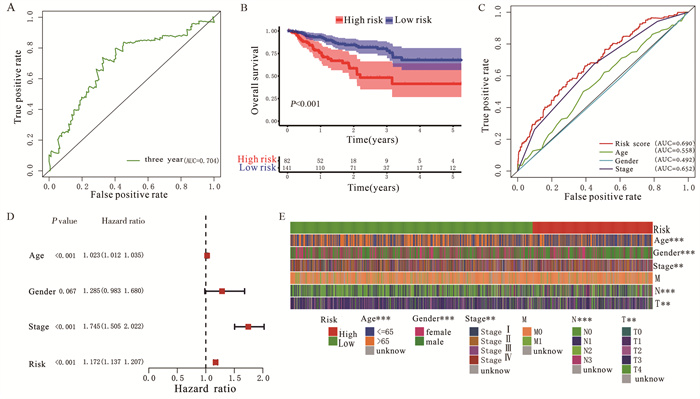
|
| A: ROC curve of predicted survival based on the prognostic risk score model in the internal validation; B: the survival curves of patients in the highrisk group and the low-risk group in the internal validation; C: ROC curve of predicted survival based on clinical risk factors; D: the forest plot of clinical risk factors; E: the heat map of risk score and other clinicopathological characteristics. 图 3 验证组中模型预后预测效能的评估 Figure 3 Evaluation of prognostic prediction effectiveness of the model in the validation group |
由于本研究需要评价该13-lncRNA模型是否能独立于其他临床病理特征进行预后预测,将894例样本数据进行Cox回归分析,提示该预后风险评分模型可以独立于其他临床病理特征进行预后预测,见图 3D。同时,热图提示风险评分与年龄、性别、TNM分期、T分级和N分级显著相关,见图 3E。
为证明模型在不同类型肿瘤中的预测价值,本研究将该预后模型在172例食管癌,381例胃癌,487例结肠癌与167例直肠癌数据中独立进行验证。结果显示,四种肿瘤的高低风险组均有显著的生存差异,见图 4。

|
| 图 4 四种消化道肿瘤中模型预测生存效能的分层分析 Figure 4 Stratified analysis of predictive survival effectiveness of the model within four gastrointestinal tumors |
为进一步确认消化道肿瘤发生发展过程中涉及到的基因功能、信号通路,将高低风险组中具有显著差异的基因进行GO、KEGG功能富集分析。通路主要富集在细胞顶端成分的组成和检测知觉等化学刺激的生物过程中,见图 5。
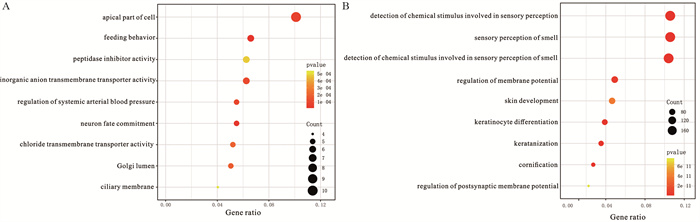
|
| 图 5 上调基因(A)和下调基因(B)功能富集通路气泡图 Figure 5 Bubble chart of function enrichment pathway analysis of up-regulated genes (A) and down-regulated genes (B) |
消化道肿瘤早期症状隐匿,并且筛查在大部分地区未得到普及,患者往往出现症状后到医疗机构就诊,诊断结果很多已处于晚期,手术治疗后容易出现转移复发,严重影响患者的预后[12-14]。本研究通过制定风险评分模型辅助临床评估患者的预后。
既往研究大多采用血清学标志物等对消化道肿瘤的预后进行预测,Zheng等对血清中外源性miR-590-5p作为胃癌诊断和预后生物标志物的有效性进行了研究验证[15],但这些标志物单独应用的预测效能不高,且不同研究没有一致的判断标准。本研究使用lncRNA测序数据构建模型,随着基因测序技术的不断突破为肿瘤研究提供大量数据,曾被认为不发挥生物学功能的lncRNA被证实通过染色体修饰、转录激活和干扰等方式,在转录和转录后及表观遗传水平调控相关靶基因,从而在多种恶性肿瘤的发生发展过程中发挥作用[16]。因此使用lncRNA构建的预后风险评分模型是综合考虑每一患者基因学的疾病预后评价体系。不同部位的肿瘤细胞的形态、增殖和转移等显著不同造成的肿瘤异质性是肿瘤治疗面临的重要难题[17],但是这些肿瘤细胞在分子水平上的相似性超过同部位肿瘤内部的相似性,因此根据患者的基因信息等可以进行准确治疗与预后评估,从而实现精准治疗[18]。
消化道肿瘤泛癌分析通过联合食管癌、胃癌、结肠癌与直肠癌数据分析肿瘤之间的相似与差异,从分子水平上认识肿瘤发展导致不同患者出现不同预后状况的机制,最终指导预后判断。目前已有的预后模型大多为单个肿瘤的研究,Liu等对食管癌预后相关lncRNA进行研究并构建6-lncRNA风险评分模型[19],该模型AUC为0.785,提示lncRNA模型对食管癌患者预后风险具有较好的预测效能。但是不能从整体上对消化道肿瘤的发展过程做出分析,从而无法基于消化道的相似性做出整体预后评价。本文是针对消化道泛癌的分析,弥补了当下科学研究中消化道泛癌预后状态评价方面的空白。
本研究尚具有一定的局限性:(1)高通量测序数据具有一定的背景噪音,即使在分析前进行了批次矫正,仍需要临床试验来证明;(2)通过生物信息学及统计学方法获得13基因模型,分子机制仍需进一步通过基础实验验证。
综上所述,本研究基于TCGA数据库中的消化道肿瘤患者的lncRNA表达和预后数据,使用Lasso和Cox回归,构建了消化道肿瘤lncRNA预后风险评分模型。结果显示:模型对消化道肿瘤患者预后具有较好的预测能力,且模型所得评分为独立预后因素。
作者贡献:
李梦涵:收集数据、统计分析、论文撰写与修改
肖琼、高鹏:审核统计分析结果、论文撰写与修改
付昱、孙晨蕊:审核统计分析结果、论文修改
宋永喜:指导研究设计、论文修改
| [1] |
Sung H, Ferlay J, Siegel RL, et al. Global Cancer Statistics 2020: GLOBOCAN Estimates of Incidence and Mortality Worldwide for 36 Cancers in 185 Countries[J]. CA Cancer J Clin, 2021, 71(3): 209-249. DOI:10.3322/caac.21660 |
| [2] |
Wei SC, Duffy CR, Allison JP. Fundamental Mechanisms of Immune Checkpoint Blockade Therapy[J]. Cancer Discov, 2018, 8(9): 1069-1086. DOI:10.1158/2159-8290.CD-18-0367 |
| [3] |
Sayles LC, Breese MR, Koehne AL, et al. Genome-Informed Targeted Therapy for Osteosarcoma[J]. Cancer Discov, 2019, 9(1): 46-63. DOI:10.1158/2159-8290.CD-17-1152 |
| [4] |
Chen W, Zheng R, Baade PD, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin, 2016, 66(2): 115-132. DOI:10.3322/caac.21338 |
| [5] |
王印祥. 泛肿瘤研究和肿瘤免疫研究——未来抗肿瘤药的发展趋势[J]. 中国药物化学杂志, 2015, 25(2): 149-152. [Wang YX. Pan-cancer research and immunotherapy: New trends in antitumor drug development[J]. Zhongguo Yao Wu Hua Xue Za Zhi, 2015, 25(2): 149-152.] |
| [6] |
Yamamura S, Imai-Sumida M, Tanaka Y, et al. Interaction and cross-talk between non-coding RNAs[J]. Cell Mol Life Sci, 2018, 75(3): 467-484. DOI:10.1007/s00018-017-2626-6 |
| [7] |
Kapranov P, Cheng J, Dike S, et al. RNA Maps Reveal New RNA Classes and a Possible Function for Pervasive Transcription[J]. Science, 2007, 316(5830): 1484-1488. DOI:10.1126/science.1138341 |
| [8] |
Ali T, Grote P. Beyond the RNA-dependent function of LncRNA genes[J]. Elife, 2020, 9: e60583. DOI:10.7554/eLife.60583 |
| [9] |
Ponting CP, Oliver PL, Reik W. Evolution and functions of long noncoding RNAs[J]. Cell, 2009, 136(4): 629-641. DOI:10.1016/j.cell.2009.02.006 |
| [10] |
Gómez-Maldonado L, Tiana M, Roche O, et al. EFNA3 long noncoding RNAs induced by hypoxia promote metastatic dissemination[J]. Oncogene, 2015, 34(20): 2609-2620. DOI:10.1038/onc.2014.200 |
| [11] |
Liu J, Lichtenberg T, Hoadley KA, et al. An Integrated TCGA Pan-Cancer Clinical Data Resource to Drive High-Quality Survival Outcome Analytics[J]. Cell, 2018, 173(2): 400-416. e11. DOI:10.1016/j.cell.2018.02.052 |
| [12] |
Smyth EC, Nilsson M, Grabsch HI, et al. Gastric cancer[J]. Lancet, 2020, 396(10251): 635-648. DOI:10.1016/S0140-6736(20)31288-5 |
| [13] |
Anderegg MCJ, Ruurda JP, Gisbertz SS, et al. Feasibility of extended chemoradiotherapy plus surgery for patients with cT4b esophageal carcinoma[J]. Eur J Surg Oncol, 2020, 46((4 Pt A)): 626-631. |
| [14] |
Weitz J, Koch M, Debus J, et al. Colorectal cancer[J]. Lancet, 2005, 365(9454): 153-165. DOI:10.1016/S0140-6736(05)17706-X |
| [15] |
Zheng GD, Xu ZY, Hu C, et al. Exosomal miR-590-5p in Serum as a Biomarker for the Diagnosis and Prognosis of Gastric Cancer[J]. Front Mol Biosci, 2021, 8: 636566. DOI:10.3389/fmolb.2021.636566 |
| [16] |
Maass PG, Luft FC, Bahring S. Long non-coding RNA in health and disease[J]. J Mol Med (Berl), 2014, 92(4): 337-346. DOI:10.1007/s00109-014-1131-8 |
| [17] |
Marusyk A, Polyak K. Tumor heterogeneity: causes and consequences[J]. Biochim Biophys Acta, 2010, 1805(1): 105-117. |
| [18] |
Collins FS, Varmus H. A new initiative on precision medicine[J]. N Engl J Med, 2015, 372(9): 793-795. DOI:10.1056/NEJMp1500523 |
| [19] |
Liu Y, Wang L, Liu H, et al. The Prognostic Significance of Metabolic Syndrome and a Related Six-lncRNA Signature in Esophageal Squamous Cell Carcinoma[J]. Front Oncol, 2020, 10: 61. DOI:10.3389/fonc.2020.00061 |
 2022, Vol. 49
2022, Vol. 49


