文章信息
- CircRNA hsa_circ_0044569在肺癌诊断中的价值
- CircRNA hsa_circ_0044569 as A Potential Biomarker for Diagnosis of Lung Cancer
- 肿瘤防治研究, 2021, 48(12): 1061-1065
- Cancer Research on Prevention and Treatment, 2021, 48(12): 1061-1065
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2021.21.0488
- 收稿日期: 2021-04-29
- 修回日期: 2021-06-21
2. 570311 海口,海南医学院第二附属医院心血管外科;
3. 750004 银川,宁夏医科大学总医院生物芯片国家工程中心宁夏分中心
2. Department of Cardiovascular Surgery, Second Affiliated Hospital of Hainan Medical College, Haikou 570311, China;
3. Department of Beijing National Biochip Research Center Sub-center in Ningxia, The General Hospital of Ningxia Medical University, Yinchuan 750004, China
肺癌已经是发达国家和发展中国家男性癌症死亡的主要原因[1],中国肿瘤流行病现状显示,中国肺癌男性发病率居首位,女性发病率居第二位,但是中国肺癌在男性及女性的死亡率中均居首位[2],肺癌分为非小细胞肺癌(NSCLC)和小细胞肺癌(SCLC),非小细胞肺癌主要分为:腺癌、鳞癌和大细胞肺癌[3]。非小细胞肺癌较小细胞肺癌发展和转移慢,受到现有的检查方式,体检意识,医疗资源分配等相关因素的影响,大部分患者诊断肺癌时已为晚期,5年生存率不足20%[4],晚期诊断的主要原因是患者症状出现晚,不易早期发现,所以发现新的可以早期诊断肺癌的标志物迫在眉睫。
环状RNA(circular RNA,circRNA)在最初发现时被认为是剪接错误的产物[5],随着科技的发展高通量RNA测序的普及,发现环状RNA在真核细胞中丰富且稳定[6]。它是一类新兴的非编码RNA分子,与传统的线性RNA不同,circRNA的分子结构呈封闭环形,不易被核酸内切酶降解[7],并且具有疾病和组织特异性。越来越多的研究显示,circRNA在肿瘤的发生发展过程中扮演了重要的角色[8-9]。有报道已经表明circRNA参与了很多肿瘤的发生、发展和转移等过程[10-13],结合circRNA的这些特点,其具备可以成为疾病诊断标志物的潜力。
1 资料与方法 1.1 研究对象收集2018年4月—2019年8月宁夏医科大学总医院普胸外科手术的56对肺癌组织和与其配对的癌旁组织标本,其中4对用于芯片筛选。排除标准为:(1)合并其他肿瘤的患者;(2)术前有放化疗病史;(3)临床信息不完善。纳入标准:(1)有明确肺癌的病理诊断;(2)既往没有癌症病史;(3)没有艾滋/艾滋病病毒感染;(4)患者术前均无放疗或化疗史。本研究获宁夏医科大学总医院医学伦理委员会批准,所有患者或其家属均签署知情同意书。
1.2 样本及临床信息采集收集肺癌组织和离肿瘤边缘 > 5 cm处癌旁组织,立即放入液氮内,之后取出整理放于-80℃冰箱长期保存,最终病理诊断由病理科2名主任医师确诊。其中Ⅰ期28例,Ⅱ期7例,Ⅲ期11例,Ⅳ期6例,其中Ⅰ期和Ⅱ期为早期肺癌,Ⅲ期和Ⅳ期为中晚期肺癌。收集所有患者信息,包括:(1)基本信息:患者年龄、性别、电话、登记号、住院号、患者手术时间、吸烟、饮酒等;(2)肺癌诊断和分型;(3)肺癌患者的临床特征:肿瘤大小、是否转移、肿瘤TNM分期等临床指标;(4)血清检验结果:血常规、肿瘤标志物指标(CA125、CA199、CEA)等。
1.3 circRNA芯片分析使用Arraystar Human circRNA Array(V2.0)(北京博奥生物有限公司,中国)和GeneSpring 13.0(Agilent)软件对4对肺癌患者的肺癌和癌旁组织样品进行芯片筛选。对RNA进行消化、扩增及标记后,通过人circRNA表达谱芯片(Human circRNA array version 2.0,北京博奥生物有限公司,中国)检测样品中circRNA表达情况,该芯片覆盖162351条已知circRNA的探针。检测结果进行初步过滤和质控去除不达标部分,剩余数据进行筛选,筛选条件为:差异倍数(fold change, FC)≥5, P < 0.01,荧光信号值≥100。使用Feature Extraction(CapitalBio)软件提取芯片数据,GeneSpring V13.0软件(Agilent)分析差异倍数、荧光信号值和显著性,Cluster3.0软件进行差异聚类分析。
1.4 RNA提取及反转录首先从组织中提取总RNA,应用OMEGA的E.Z.N.A.Total RNA Kit Ⅰ试剂盒按照说明进行组织RNA的提取,提取后使用分光光度计NanoDrop2000(Thermo Fisher Scientific)测定RNA的纯度和浓度,OD260/280的比值约为1.9~2.0,然后通过GoScript反转录(RT)系统(Promega Corporation,Fitchburg,WI,USA)将RNA反转录为cDNA。反转录体系为:1 µl总RNA(约500 ng),1 µl随机引物,10 µl ddH2O,2 µl dNTP,4 µl反转录缓冲液,1 µl RNA酶抑制剂,1 µl反转录酶,总体积20 µl;反应条件为:25℃ 5 min;42℃ 60 min,70℃ 5 min。RNA及cDNA置于-80℃储存备用。
1.5 qRT-PCR检测circRNA hsa_circ_0044569在肺癌及癌旁组织的表达差异使用反转录系统合成cDNA。qRT-PCR使用TB Green qPCR Mastermix(TaKaRa,日本),在LightCycler480实施PCR反应。qRT-PCR反应的总体积为20 μl,其中包括0.8 μl正向/反向引物,10 μl TB Green,6.4 μl二次蒸馏水和2 μl cDNA。循环程序95℃下持续10 s,然后再40个循环中95℃持续15 s,退火温度30 s,β-actin在表达中起内参对照作用,基因的相对表达量使用ΔCT法计算。所有样品进行3次重复实验,见表 1。
使用SPSS23.0,GraphPad Prism7.0进行统计分析。数据表示为平均值±标准差(x±s),从肺癌组和癌旁对照组之间的分布差异计算每个circRNA的倍数变化,并通过独立样本t检验分析显著性,使用SPSS23.0构建接收器工作特征曲线(ROC),当AUC≤0.5时,circRNA没有诊断价值,P < 0.05为差异有统计学意义。
2 结果 2.1 肺癌患者组织中的circRNA表达谱芯片分析结果显示:肺癌及癌旁组织间存在大量差异表达的circRNA,见图 1。通过芯片设定的差异筛选标准和生物信息学分析最终获得19个差异表达的circRNA,其中11个表达上调,8个表达下调的circRNA作为候选分子进行进一步研究,经临床标本验证最终选择在肺癌中稳定显著上调的hsa_circ_0044569作为研究目标,见图 2。
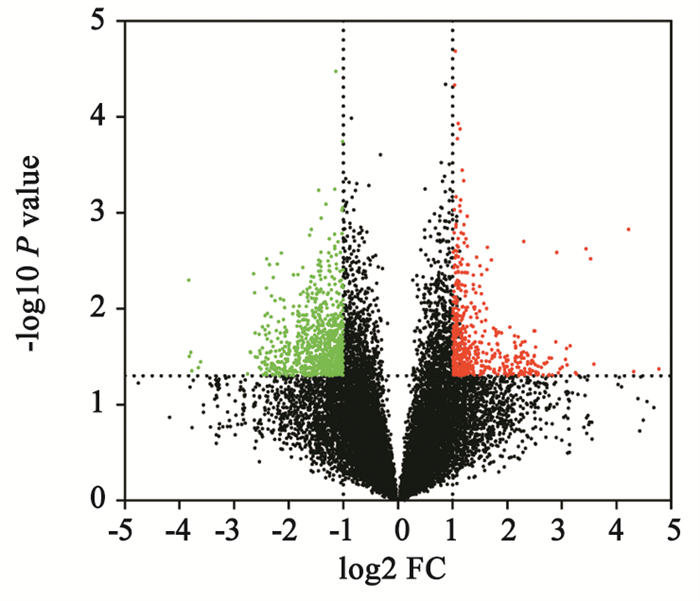
|
| Green: the down-regulated circRNA; Red: the up-regulated circRNA; Black: the circRNA without different expression. 图 1 肺癌与癌旁组织之间差异表达的circRNA火山图 Figure 1 Volcano map of differentially-expressed circRNA between lung cancer and adjacent tissues |
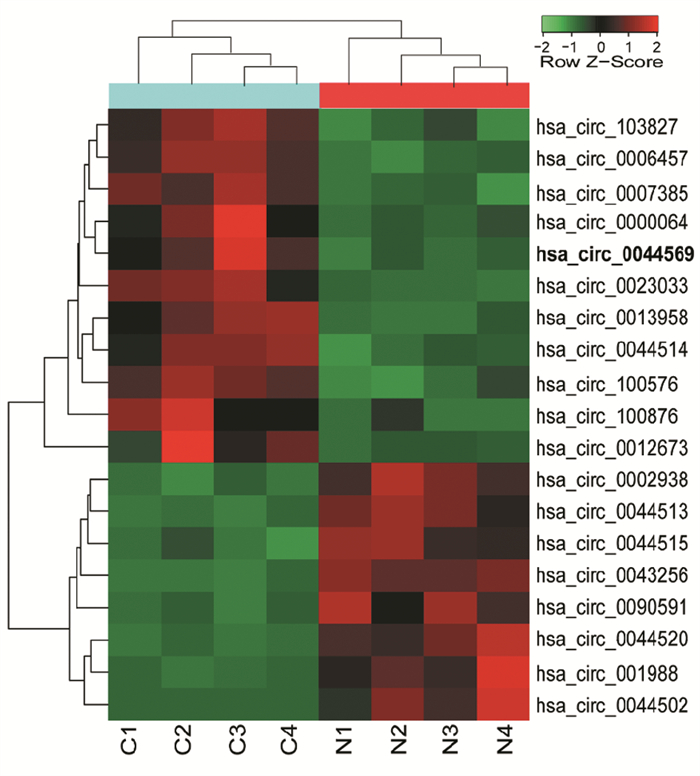
|
| C: cancer tissues; N: adjacent tissues. 图 2 肺癌与癌旁组织之间差异表达的circRNA聚类图 Figure 2 Clustergram of differentially-expressed circRNA between lung cancer and adjacent tissues |
结果显示:hsa_circ_0044569表达与肺癌患者年龄、性别、肿瘤大小、TNM分期、淋巴结转移、CEA、CA12-5、CA19-9、饮酒、吸烟等差异无统计学意义,但与肺癌的病理类型之间差异有统计学意义(P < 0.05),见表 2。
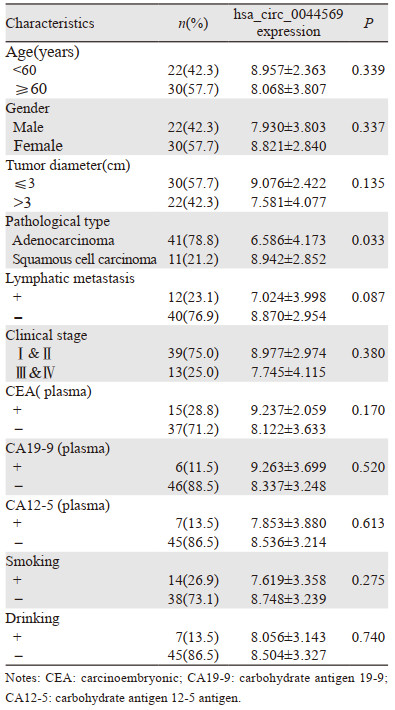
|
对52对肺癌患者癌和对应的癌旁组织中hsa_circ_0044569表达水平进行分析结果显示,hsa_circ_0044569在肺癌组织中的表达水平高于与其对应的癌旁组织,见图 3A~B;肺癌组织46例表现出高表达(88.5%),6例表现为低表达(11.5%),见图 3C。
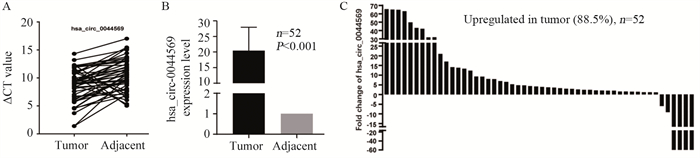
|
| 图 3 qRT-PCR验证在肺癌和癌旁组织中差异表达的hsa_circ_0044569 Figure 3 Differential hsa_circ_0044569 expression between lung cancer tissues and adjacent tissues verified by qRT-PCR |
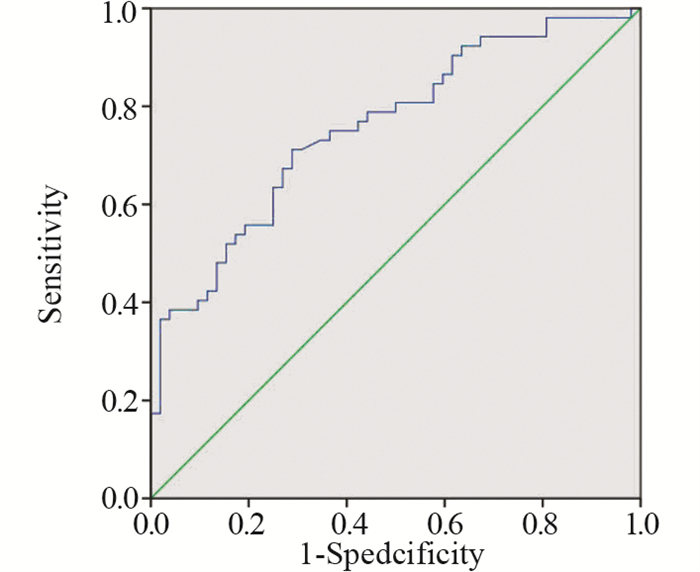
应用ROC曲线评价hsa_circ_0044569对肺癌的诊断价值,获得hsa_circ_0044569在52对肺癌组织中的临界值值为9.62,曲线下面积AUC值为0.758, P < 0.01,敏感度为0.712,特异性为0.712,见图 4。因此,hsa_circ_0044569具备早期肺癌诊断的潜能。
|
| 图 4 肺癌与配对癌旁组织中hsa_circ_0044569表达的ROC曲线 Figure 4 ROC curves of hsa_circ_0044569 expression in lung cancer and adjacent tissues |
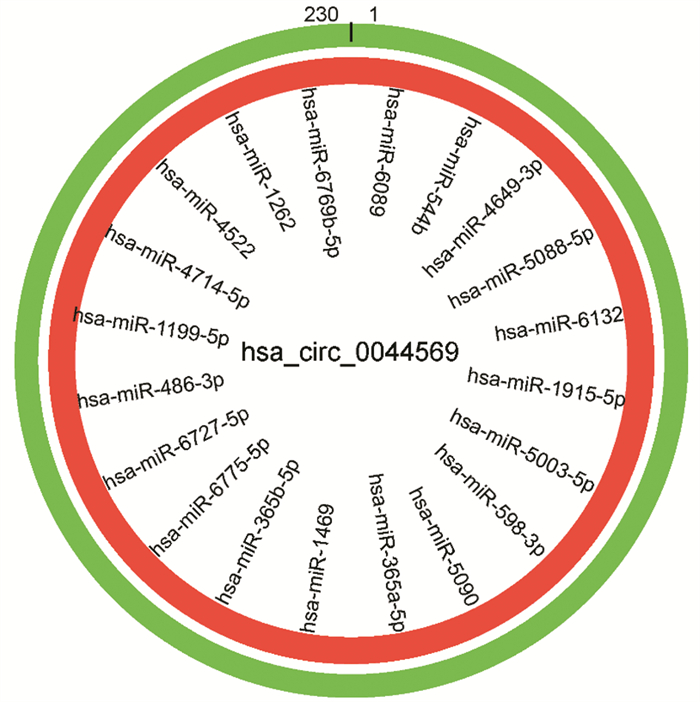
hsa_circ_0044569结合的miRNA预测结果,见图 5。本研究采用miRanda数据库预测与hsa_circ_0044569结合miRNA情况,hsa_circ_0044569共计有138个miRNA结合位点,根据结合位点类型(Site type)、结合位点总评分(Context++ score)及miRNA家族的保守靶向概率(PCT)计算与hsa_circ_0044569结合的靶向miRNA排名靠前的前20个,供进一步机制研究参考。
|
| 图 5 通过miRanda数据库预测到与hsa_circ_0044569结合的miRNA结合位点分布 Figure 5 Distribution of miRNA binding sites that bind to hsa_circ_0044569 predicted by miRanda database |
环状RNA的种类和形式多种多样,可以起源于基因组序列的任何区域[14],根据来源主要分为3类: 内含子序列形成的环状RNA(circular intronic RNA, ciRNA),外显子序列形成的环状RNA(exonic circular RNA, ecircRNA)以及内含子和外显子序列共同形成的环状RNA(exon-intron circular RNA, EIciRNA)外显子环状RNA可以由一个或者多个外显子序列组成,主要存在于细胞质内,而内含子环状RNA主要分布在细胞核内[15]。环状RNA广泛存在于哺乳动物细胞中,与线性RNA相比,它们有着较高的表达水平。circRNA的两个最重要的特性是它们高度保守且非常稳定。与其他非编码RNA相比,这些优势为circRNA提供了成为诊断癌症疾病的理想生物标志物的潜力[16]。随着RNA测序和其他检测技术的应用,我们注意到,环状RNA在许多肿瘤中都存在差异表达的现象,如膀胱癌[17]、结肠癌[18]、肝细胞癌[19]等肿瘤,由于环状RNA比线性RNA更稳定,并且环状RNA由细胞囊泡储存并通过胞吐方式释放进入血液,致使环状RNA在临床工作中易于获取。以上特点为环状RNA在肿瘤诊断方面提供了临床优势。
本研究发现hsa_circ_0044569在鉴别肺腺癌及肺鳞癌之间差异有统计学意义,但是与患者的年龄,性别、肿瘤大小、TNM分期、淋巴结转移、CEA、CA12-5、CA19-9、饮酒、吸烟等之间差异无统计学意义。目前为止,很多实验都验证了circRNA与肺癌患者临床特点之间的关系。Cheng等[20]的研究表明,肺鳞癌中circTP63被上调,并且其上调与肺鳞癌患者中更大的肿瘤大小和更高的TNM分期相关。升高的circTP63在体外和体内均可促进肿瘤细胞增殖。Wang等[21]的研究显示,环状RNA circ_0020123通过海绵miR-590-5p调控THBS2促进NSCLC细胞增殖和迁移,抑制细胞凋亡。Liu等[22]的研究显示,基因微阵列检测结果表明hsa_circ_0046264是最显著的上调基因,qRT-PCR检测进一步证明hsa_circ_0046264在肿瘤患者组织中表达显着上调。临床分析表明,hsa_circ_0046264的表达水平与患者的年龄、肿瘤大小、肿瘤分期(TNM)和淋巴结转移显著相关(P < 0.01)。
早期诊断是有效治疗肺癌的重要前提,胸部CT可早期发现肺部病变,然而,有些病变通过CT很难区分良恶性;支气管镜检查和经皮肺穿刺可以获取病理组织,但侵入性检查通常有不良反应甚至会危及到患者生命;痰液脱落细胞学检查和血清中与肺癌相关蛋白质标志物检测的敏感度和特异性有限,需要新的标志物来辅助肺癌的临床诊断。基于circRNA的特点其不易被核酸外切酶降解,故它可以作为肺癌早期诊断的有效生物标志物。
本研究结果显示hsa_circ_0044569在肺癌中表达显著上调,且稳定性较好,ROC曲线分析其具备成为肺癌早期诊断标志物的潜能。hsa_circ_0044569对于诊断肺癌具有重要意义,且对于肺腺癌和肺鳞癌的鉴别有一定的作用。因本实验纳入的样本数量有限,存在选样偏倚的可能性,后期需要更多的临床实验数据进一步的研究其可行性。
作者贡献:
侯岩君:标本收集、实验操作及论文撰写
田进海:实验操作、数据统计及论文修改
王立斌、何进喜:实验设计及指导
| [1] |
Sung H, Ferlay J, Siegel RL, et al. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2021, 71(3): 209-249. DOI:10.3322/caac.21660 |
| [2] |
郑荣寿, 孙可欣, 张思维, 等. 2015年中国恶性肿瘤流行情况分析[J]. 中华肿瘤杂志, 2019, 41(1): 19-28. [Zheng RS, Sun KX, Zhang SW, et al. Report of cancer epidemiology in China, 2015[J]. Zhonghua Zhong Liu Za Zhi, 2019, 41(1): 19-28.] |
| [3] |
Travis WD, Brambilla E, Burke AP, et al. WHO classification of tumours of the lung, pleura, thymus and heart[M]. 2015: 9-96.
|
| [4] |
Zeng H, Chen W, Zheng R, et al. Changing cancer survival in China during 2003-15: a pooled analysis of 17 population-based cancer registries[J]. Lancet Glob Health, 2018, 6(5): e555-e567. DOI:10.1016/S2214-109X(18)30127-X |
| [5] |
Sanger HL, Klotz G, Riesner D, et al. Viroids are single-stranded covalently closed circular RNA molecules existing as highly base-paired rod-like structures[J]. Proc Natl Acad Sci U S A, 1976, 73(11): 3852-3856. DOI:10.1073/pnas.73.11.3852 |
| [6] |
余昊, 王刚, 孙备. 环状RNA和癌症[J]. 中国肿瘤生物治疗杂志, 2018, 25(9): 940-944. [Yu H, Wang G, Sun B. circ RNA and Cancer[J]. Zhongguo Zhong Liu Sheng Wu Zhi Liao Za Zhi, 2018, 25(9): 940-944.] |
| [7] |
Zhang M, Xin Y. Circular RNAs: a new frontier for cancer diagnosis and therapy[J]. J Hematol Oncol, 2018, 11(1): 21. DOI:10.1186/s13045-018-0569-5 |
| [8] |
Su M, Xiao Y, Ma J, et al. Circular RNAs in Cancer: emerging functions in hallmarks, stemness, resistance and roles as potential biomarkers[J]. Mol Cancer, 2019, 18(1): 90. DOI:10.1186/s12943-019-1002-6 |
| [9] |
宗曾艳, 孔凡虹, 王萌萌, 等. 环状RNA在恶性肿瘤发生发展和诊疗中的研究进展[J]. 国际检验医学杂志, 2020, 41(1): 98-103. [Zong ZY, Kong FH, Wang MM, et al. Research progress of circular RNA in carcinogenesisi, development, diagnosis and treatment of malignant tumors[J]. Guo Ji Jian Yan Yi Xue Za Zhi, 2020, 41(1): 98-103.] |
| [10] |
Qian P, Linbo L, Xiaomei Z, et al. Circ_0002770, acting as a competitive endogenous RNA, promotes proliferation and invasion by targeting miR-331-3p in melanoma[J]. Cell Death Dis, 2020, 11(4): 264. DOI:10.1038/s41419-020-2444-x |
| [11] |
Xia T, Pan Z, Zhang J. CircSMC3 regulates gastric cancer tumorigenesis by targeting miR-4720-3p/TJP1 axis[J]. Cancer Med, 2020, 9(12): 4299-4309. DOI:10.1002/cam4.3057 |
| [12] |
Xu Y, Yu J, Huang Z, et al. Circular RNA hsa_circ_0000326 acts as a miR-338-3p sponge to facilitate lung adenocarcinoma progression[J]. J Exp Clin Cancer Res, 2020, 39(1): 57. DOI:10.1186/s13046-020-01556-4 |
| [13] |
Xu F, Ni M, Li J, et al. Circ0004390 promotes cell proliferation through sponging miR-198 in ovarian cancer[J]. Biochem Biophys Res Commun, 2020, 526(1): 14-20. DOI:10.1016/j.bbrc.2020.03.024 |
| [14] |
Greene J, Baird AM, Brady L, et al. Circular RNAs: Biogenesis, Function and Role in Human Diseases[J]. Front Mol Biosci, 2017, 4: 38. DOI:10.3389/fmolb.2017.00038 |
| [15] |
Jiang F, Shen X. Current prevalence status of gastric cancer and recent studies on the roles of circular RNAs and methods used to investigate circular RNAs[J]. Cell Mol Biol Lett, 2019, 24: 53. DOI:10.1186/s11658-019-0178-5 |
| [16] |
Zhao ZJ, Shen J. Circular RNA participates in the carcinogenesis and the malignant behavior of cancer[J]. RNA Biol, 2017, 14(5): 514-521. DOI:10.1080/15476286.2015.1122162 |
| [17] |
Liu Q, Zhou Q, Zhong P. circ_0067934 increases bladder cancer cell proliferation, migration and invasion through suppressing miR-1304 expression and increasing Myc expression levels[J]. Exp Ther Med, 2020, 19(6): 3751-3759. |
| [18] |
夏林, 余国政, 唐晶. 环状RNA在结直肠癌中诊断价值的Meta分析[J]. 肿瘤防治研究, 2021, 48(9): 864-870. [Xia L, Yu GZ, Tang J. Diagnostic Value of Circular RNAs for Colorectal Cancer: A Meta-analysis[J]. Zhong Liu Fang Zhi Yan Jiu, 2021, 48(9): 864-870.] |
| [19] |
Zhi Q, Wan D, Ren R, et al. Circular RNA profiling identifies circ102049 as a key regulator of colorectal liver metastasis[J]. Mol Oncol, 2021, 15(2): 623-641. DOI:10.1002/1878-0261.12840 |
| [20] |
Cheng Z, Yu C, Cui S, et al. circTP63 functions as a ceRNA to promote lung squamous cell carcinoma progression by upregulating FOXM1[J]. Nat Commun, 2019, 10(1): 3200. |
| [21] |
Wang L, Zhao L, Wang Y. Circular RNA circ_0020123 promotes non-small cell lung cancer progression by sponging miR-590-5p to regulate THBS2[J]. Cancer Cell Int, 2020, 20: 387. |
| [22] |
Liu ZH, Yang SZ, Chen XT, et al. Correlations of hsa_circ_0046264 expression with onset, pathological stage and chemotherapy resistance of lung cancer[J]. Eur Rev Med Pharmacol Sci, 2020, 24(18): 9511-9521. |
 2021, Vol. 48
2021, Vol. 48



