文章信息
- 环状RNA在结直肠癌中诊断价值的Meta分析
- Diagnostic Value of Circular RNAs for Colorectal Cancer: A Meta-analysis
- 肿瘤防治研究, 2021, 48(9): 864-870
- Cancer Research on Prevention and Treatment, 2021, 48(9): 864-870
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2021.21.0069
- 收稿日期: 2021-01-19
- 修回日期: 2021-07-08
2. 435700 黄石,鄂东医疗集团黄石市中心医院(湖北理工学院附属医院)普外科;
3. 412007 株洲,中南大学湘雅医学院附属株洲医院肿瘤科
2. Department of General Surgery, Huangshi Central Hospital of Edong Healthcare Group (Affiliated Hospital of Hubei Polytechnic University), Huangshi 435700, China;
3. Department of Oncology, Zhuzhou Hospital Affiliated to Xiangya School of Medicine of Central South University, Zhuzhou 412007, China
结直肠癌(colorectal cancer, CRC)是全球范围内具有高发病率和高死亡率的恶性肿瘤。最新的中国癌症报告显示,CRC的发病率和死亡率分别占据第四位和第三位[1]。CRC的早期症状不明显,很多CRC患者明确诊断时已是中晚期,临床预后很差[2]。大便隐血试验检测CRC的敏感度很低,极易造成误诊和漏诊[3]。内镜组织活检结合组织病理学是诊断CRC的金标准,然而因检查有创侵入性而不易被广大患者接受[4]。寻找新型标志物是CRC早期诊断和预后判断的重要手段。环形RNA(circular RNA, circRNA)属于非编码RNA的一种,其源于基因组的外显子和(或)内含子,但无5’末端的帽子和3’末端poly(A)尾巴结构,通过共价键形成闭合环状结构[5]。相比线性RNA,circRNA结构更为稳定,因其对核酸酶等消化不敏感[6]。circRNA在CRC的发生、发展中扮演重要角色,并在CRC的临床诊断中具有潜在的应用价值[7-23]。鉴于单项单中心研究存在样本小、结果偏倚大等诸多问题,本研究拟通过定量的Meta分析,系统性评价circRNA对CRC临床诊断的综合效能,为circRNA后期能否作为新型分子标志物用于CRC诊断提供循证医学依据。
1 资料与方法 1.1 纳入、排除标准全文依照系统评价和荟萃分析优先报告的条目(PRISMA)规范(2009版)进行报告[24]。纳入条件:(1)内容关于circRNA在CRC中的诊断价值评价,研究对象为国内外CRC患者,研究样本例数≥20例; (2)研究中可直接或间接获得数据用于构建2×2四格表。排除标准:(1)与circRNA诊断CRC无关的文献; (2)用于统计分析的数据提供不足且联系原作者失败; (3)质量评分为低质量的研究; (4)文章类型为基础研究、综述、会议摘要等。
1.2 文献检索检索数据包括PubMed、EMBASE、Wiley Online Library、Web of Science、CNKI、万方、维普以中文或英文发表的文献,日期截至2021年3月1日。检索词为:“circular RNA、circRNA、环状RNA、结直肠癌、colorectal cancer、colorectal carcinoma、colorectal neoplasms、诊断、敏感度、特异性、曲线下面积、ROC曲线、diagnosis、sensitivity、specificity、AUC、ROC curve”,检索时通过“and”或“or”进行组合。
1.3 数据提取和质量评价安排两作者同时筛选数据信息,包括论文作者、发文时间、人群来源、样本数量、对照样本类型/来源、circRNA名称、检测方法、参照基因、cut-off值设置、灵敏度、特异度及AUC值等,对于不能直接获得的数据通过联系作者或软件间接提取。参照“诊断性试验质量评价量表 2(QUADAS-2)”工具对文献偏倚和质量进行评分[25],包括病例选择、待评价实验、金标准、病例流程和进展情况等方面。项目评为风险“低”时记1分,风险评为“未知”或“高”时均记0分。总评分≥4分认为文献研究偏倚较小、质量较高[26]。
1.4 统计学方法诊断统计量的合并基于Stata12.0。异质性分析采用Spearman相关系数评估阈值效应,Cochran’-Q、I2检验评估非阈值效应,P < 0.01或I2 > 50%时认为异质性显著,选用随机效应模型合并统计量,否则选用固定效应模型。设置亚组分析、敏感度检验和Meta回归分析异质性生成原因。进行Deek’s定量漏斗图检验,评估研究间发表偏倚,P < 0.1提示出现发表偏倚。
2 结果 2.1 文献筛选结果图 1所示,在线数据库检索共获得513篇相关论文(剔除重复)。认真阅读文献标题和摘要后,排除435篇无关文献,最后全文阅读剩余78篇文献,进一步排除61篇,最终有17项研究纳入统计分析[7-23]。
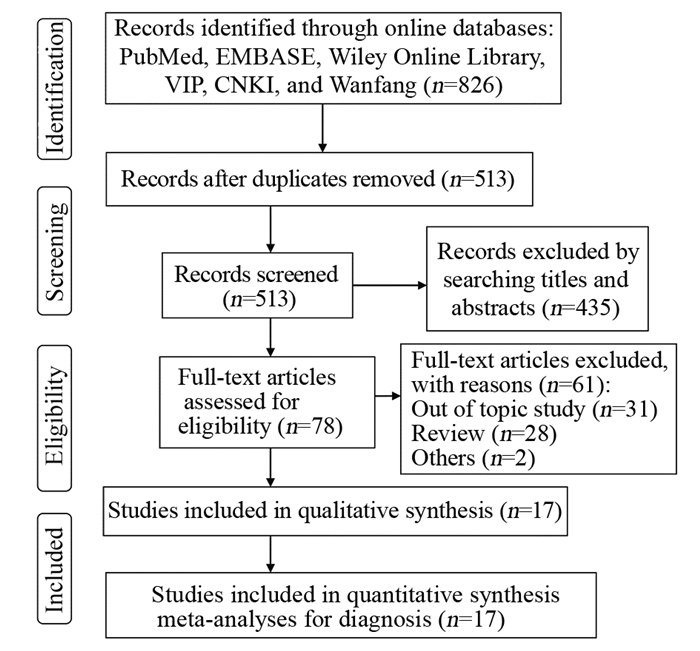
|
| 图 1 相关研究的纳入和排除过程 Figure 1 Inclusion and exclusion process of relevant studies |
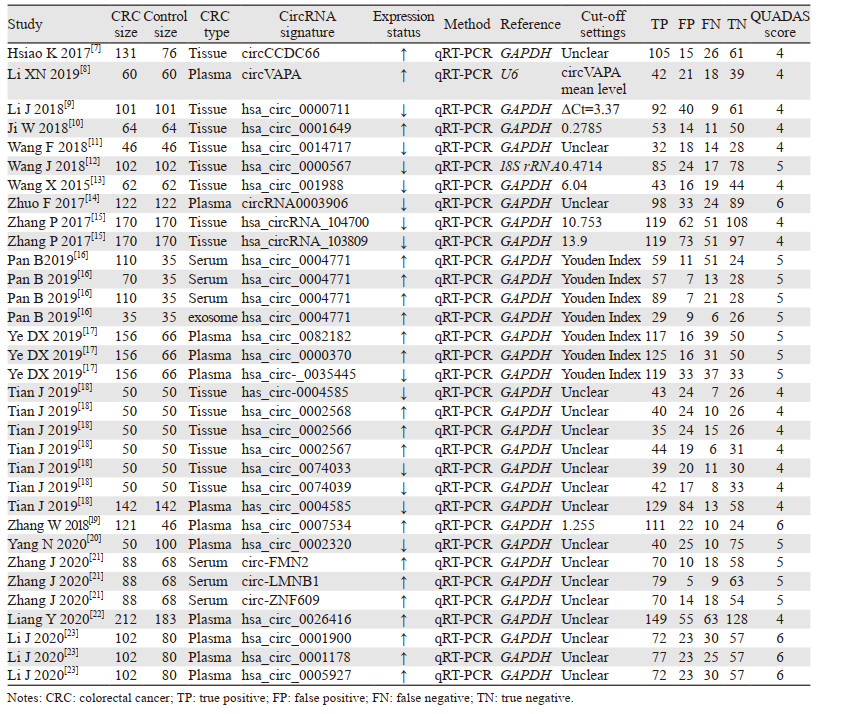
获得的17项研究共含1873例CRC患者和1573例匹配非癌对照。所有CRC患者均经病理组织细胞学确诊,检测样本类型包括组织、血浆、血清和外泌体,均于治疗前手术前获得。纳入研究人群包括亚洲人和欧洲人。纳入的circRNA有29种,分别为circCCDC66、hsa_circ_0001649、hsa_circ_0000711、circVAPA、hsa_circ_001988、hsa_circ_0000567、hsa_circ_0014717、hsa_circRNA_104700、hsa_circRNA_103809、circRNA0003906、hsa_circ_0004771、hsa_circ_0082182、hsa_circ_0000370、hsa_circ_0035445、has_circ_0004585、hsa_circ_0002568、hsa_circ_0002566、hsa_circ_0002567、hsa_circ_0074033、hsa_circ_0074039、hsa_circ_0007534、hsa_circ_0002320、circ-FMN2、circ-LMNB1、circ-ZNF609、hsa_circ_0026416、hsa_circ_0001900、hsa_circ_0001178和hsa_circ_0005927,其中19种circRNA在CRC组织中表达上调,10种表达下调。circRNA的表达检测均通过RT-qPCR,内参基因包括GAPDH,U6和18S rRNA,见表 1。
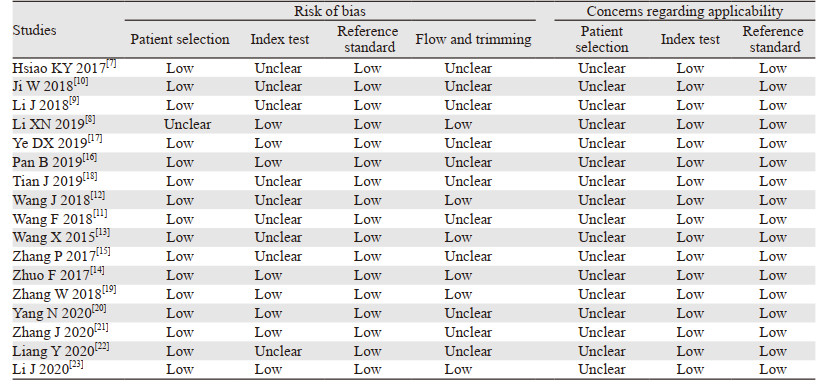
按照QUADAS Ⅱ的7项条目严格评估纳入文献的质量[17]。表 2所示所有纳入研究的累计得分均高于4分,提示纳入的文献整体研究质量较高。
异质性分析结果显示,Spearman相关系数的P=0.646,提示研究间不存在阈值效应引起的异质性,同时Cochran’-Q检验P=0.000,I2=71.9%,提示研究间存在中等程度异质性,主要来自非阈值效应,故选取随机效应模型进行诊断指标的合并。
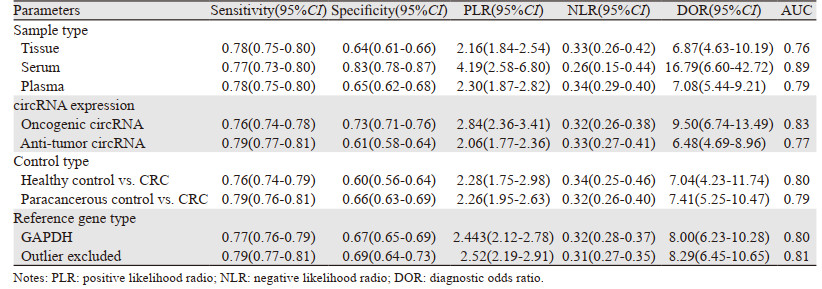
2.4 诊断Meta分析CRC患者中异常表达的circRNA合并的AUC=0.81,见图 2,对应的敏感度、特异性、阳性似然比(PLR)、阴性似然比(NLR)和诊断优势比(DOR)分别为0.79(95%CI: 0.76~0.81)、0.69(95%CI: 0.64~0.73)、2.52(95%CI: 2.20~2.89)、0.31(95%CI: 0.26~0.36)和8.02(95%CI: 6.21~10.36),提示异常表达的circRNA表达谱在CRC诊断中具有较高的价值。
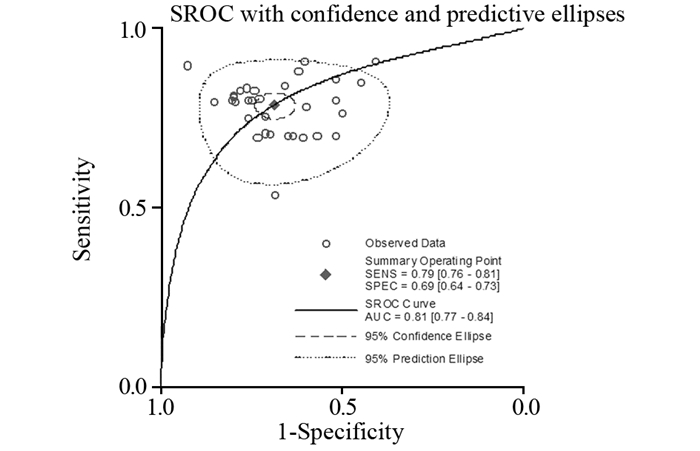
|
| 图 2 circRNA异常表达谱诊断CRC的SROC曲线 Figure 2 SROC curve of abnormal circRNA expression profile in diagnosing CRC |
亚组分析结果显示,血清来源的circRNA分析检测(AUC=0.89, DOR=16.79)诊断CRC效能高于血浆(AUC=0.79, DOR=7.08)和组织(AUC=0.76, DOR=6.87),提示血清可作为circRNA分析检测的最好基质。据circRNA的表达情况分析显示,致癌性的circRNA表达谱诊断CRC效能高于抑癌性的circRNA(AUC: 0.83 vs. 0.77; DOR: 9.50 vs. 6.48)。基于参考基因GAPDH的circRNA分析与整体诊断效能相当。基于GAPDH为内参的circRNA诊断CRC效能与整体效应相当(AUC: 0.80 vs. 0.81, DOR: 8.29 vs. 8.00),见表 3。
敏感度分析显示,纳入研究[16]的一项数据偏离上限,提示为离群数据,见图 3,故将该数据重新剔除后进行Meta分析:对应合并的敏感度、特异性、PLR、NLR、DOR和AUC分别为0.79(95%CI: 0.77~0.81)、0.69(95%CI: 0.64~0.73)、2.52(95%CI: 2.19~2.91)、0.31(95%CI: 0.27~0.35)、8.29(95%CI: 6.45~10.65)和0.81,对应I2由71.9%降至71.2%,说明纳入的离群数据是对合并整体效应的结果及异质性的影响均不大。
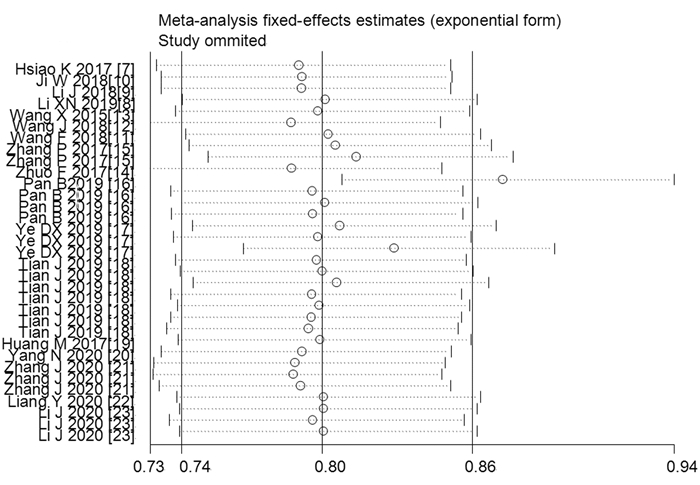
|
| 图 3 敏感度分析检测研究的同质性 Figure 3 Sensitivity analysis of homogeneity among studies |
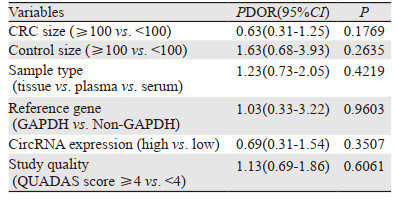
Meta回归分析基于病例数、对照组例数、样本类型、circRNA表达状态,文献质量评分等变量进一步分析异质性来源。结果显示,上述变量均不是引起研究异质性产生的因素(均P > 0.05),见表 4。
研究间的发表偏倚经Deek’s定量漏斗图检测诊断性,结果显示,该检验得到P=0.310,提示研究间不存在发表偏倚,见图 4。
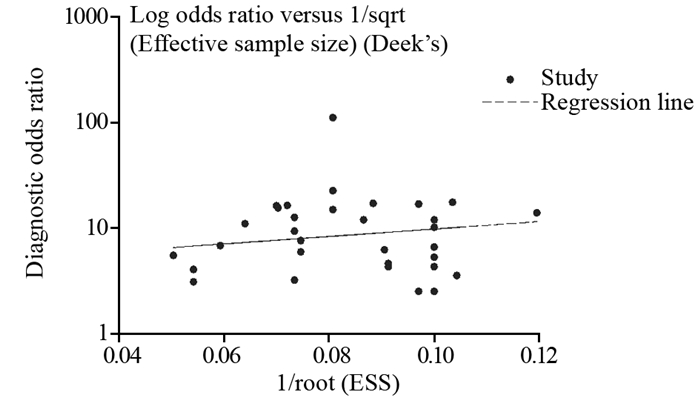
|
| 图 4 Deek’s定量漏斗图显示研究的发表偏倚 Figure 4 Publication bias assessed by Deek's quantitative funnel plot |
2015年中国癌症统计显示,CRC的发病率和死亡率分别占据第四位和第三位,且发病率呈逐年上升趋势[1]。不断开发CRC早期诊断的生物标志物迫在眉睫。近年研究发现,环形RNA(circRNA)的异常表达与CRC的临床分期、复发及转移等临床病理特征密切相关[7-23],提示可作为CRC诊断的重要指标。本研究通过定量Meta分析系统性评价了异常表达的circRNA分子对CRC的综合诊断效能。
目前研究已报道,异常的circRNA分子表达谱在肝癌[27]、肺癌[28]中均具有很高的诊断应用价值。Huang等[27]的Meta分析结果显示,异常的circRNA分子表达谱用于区分肝细胞癌和非癌对照的合并AUC为0.81,对应合并敏感度为0.82,合并特异性为0.72。本研究合并效应量分析显示,异常表达的circRNA用于诊断CRC和非癌对照的合并敏感度为0.79,特异性为0.69,AUC为0.81,说明异常circRNA的表达异常可作为CRC较好的诊断分子指标。另一方面,诊断的优势比(DOR)可用于反映诊断试验的结果与疾病的关联程度,当DOR > 1时,其值越大说明该诊断试验的判别准确度越高[29]。本研究中circRNA合并DOR为8.02,说明整体的判别准确度很高。另外,我们发现阳性似然比(PLR)为2.52,说明CRC患者中circRNA检测阳性结果的概率约为非癌对照检测阳性概率的2.3倍; 阴性似然比(NLR)为0.31提示,在阴性结果中,circRNA检测存在约31%的假阴性率。本研究进一步基于circRNA的功能(表达形式)及参考基因类型等进行亚组分析,发现表达上调的circRNA对CRC的综合诊断效能优于下调的circRNA,血浆来源的circRNA检测诊断CRC价值高于组织来源,但基于GAPDH为内参的circRNA检测诊断CRC效能与整体效应相当。尽管如此,因亚组分析的纳入样本量少,故以上发现有待于进一步证实。
异质性的产生是Meta分析过程中不可避免的一个问题[30]。异质性的产生主要源自阈值和非阈值效应:阈值效应即诊断实验阈值选择的不同而引起,而患者病情的严重程度、疾病不同病理组织学类型、不同的试剂来源、检测温度、操作人员不同等为非阈值因素[31]。由于诊断试验的独立性,加上非阈值因素诸多差异,异质性便由此产生[26]。本研究合并的统计量存在较大的异质性,考虑可能与纳入研究的不同cut-off值设定、纳入研究人群的不同状态、circRNA的不同分子类型及表达水平等相关。因此,本研究通过敏感度分析和单因素回归分析深入探讨异质性来源。敏感度分析提示研究间存在离群数据,故将该数据重新剔除后再次进行Meta分析,显示I2由71.9%降至71.2%,说明纳入的离群数据对整体合并结果及异质性影响不大。尽管如此,回归分析提示样本例数、样本类型、circRNA表达状态,文献质量评分等均不是引起研究间异质性的因素。
本研究存在不足之处如下:(1)可能存在一定的人群偏倚:纳入的研究主要来自亚洲人群(中国和中国台湾); (2)circRNA表达谱涵盖不全面; (3)纳入的部分研究数据为通过计算间接提取,存在一定误差; (4)获得的样本类型大部分为组织,缺乏与血清、血浆样本的对比。因此,本研究结论仍需后期更大样本的临床研究加以证实。
综上,本研究系统性评价了异常表达的circRNA在CRC诊断中的临床应用价值,认为合并的circRNA异常表达谱可作为CRC诊断的较好辅助指标应用于临床。
作者贡献
夏林:数据分析、提取及撰写文章
余国政:数据提取
唐晶:课题思路设计、指导文章写作
| [1] |
Chen W, Zheng R, Baade PD, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin, 2016, 66(2): 115-132. DOI:10.3322/caac.21338 |
| [2] |
Miller KD, Nogueira L, Mariotto AB, et al. Cancer treatment and survivorship statistics, 2019[J]. CA Cancer J Clin, 2019, 69(5): 363-385. DOI:10.3322/caac.21565 |
| [3] |
钟选芳, 肖丹, 许岸高, 等. 粪便DNA与粪隐血试验机会性筛查大肠肿瘤的比较研究[J]. 临床消化病杂志, 2016, 28(2): 106-109. [Zhong XF, Xiao D, Xu AG, et al. Fecal DNA versus fecal occult blood test in opportunity screening for colorectal neoplasm[J]. Lin Chuang Xiao Hua Bing Za Zhi, 2016, 28(2): 106-109. DOI:10.3870/1cxh.j.issn.1005-541X.2016.02.13] |
| [4] |
苏便欣, 张学彦. 结肠镜检查肠息肉漏诊及其相关因素研究进展[J]. 胃肠病学, 2016, 21(2): 115-117. [Su BX, Zhang XY. Advances in Studies on Missed Diagnosis of Intestinal Polyps and Related Factors in Colonoscopic Examination[J]. Wei Chang Bing Xue, 2016, 21(2): 115-117. DOI:10.3969/j.issn.1008-7125.2016.02.013] |
| [5] |
Connelly CM, Moon MH, Schneekloth JS Jr. The Emerging Role of RNA as a Therapeutic Target for Small Molecules[J]. Cell Chem Biol, 2016, 23(9): 1077-1090. DOI:10.1016/j.chembiol.2016.05.021 |
| [6] |
Li Y, Zheng Q, Bao C, et al. Circular RNA is enriched and stable in exosomes: a promising biomarker for cancer diagnosis[J]. Cell Res, 2015, 25(8): 981-984. DOI:10.1038/cr.2015.82 |
| [7] |
Hsiao KY, Lin YC, Gupta SK, et al. Noncoding Effects of Circular RNA CCDC66 Promote Colon Cancer Growth and Metastasis[J]. Cancer Res, 2017, 77(9): 2339-2350. DOI:10.1158/0008-5472.CAN-16-1883 |
| [8] |
Li XN, Wang ZJ, Ye CX, et al. Circular RNA circVAPA is up-regulated and exerts oncogenic properties by sponging miR-101 in colorectal cancer[J]. Biomed Pharmacother, 2019, 112: 108611. DOI:10.1016/j.biopha.2019.108611 |
| [9] |
Li J, Ni S, Zhou C, et al. The expression profile and clinical application potential of hsa_circ_0000711 in colorectal cancer[J]. Cancer Manag Res, 2018, 10: 2777-2784. DOI:10.2147/CMAR.S172388 |
| [10] |
Ji W, Qiu C, Wang M, et al. Hsa_circ_0001649: A circular RNA and potential novel biomarker for colorectal cancer[J]. Biochem Biophys Res Commun, 2018, 497(1): 122-126. DOI:10.1016/j.bbrc.2018.02.036 |
| [11] |
Wang F, Wang J, Cao X, et al. Hsa_circ_0014717 is downregulated in colorectal cancer and inhibits tumor growth by promoting p16 expression[J]. Biomed Pharmacother, 2018, 98: 775-782. DOI:10.1016/j.biopha.2018.01.015 |
| [12] |
Wang J, Li X, Lu L, et al. Circular RNA hsa_circ_0000567 can be used as a promising diagnostic biomarker for human colorectal cancer[J]. J Clin Lab Anal, 2018, 32(5): e22379. DOI:10.1002/jcla.22379 |
| [13] |
Wang X, Zhang Y, Huang L, et al. Decreased expression of hsa_circ_001988 in colorectal cancer and its clinical significances[J]. Int J Clin Exp Pathol, 2015, 8(12): 16020-16025. |
| [14] |
Zhuo F, Lin H, Chen Z, et al. The expression profile and clinical significance of circRNA0003906 in colorectal cancer[J]. Onco Targets Ther, 2017, 10: 5187-5193. DOI:10.2147/OTT.S147378 |
| [15] |
Zhang P, Zuo Z, Shang W, et al. Identification of differentially expressed circular RNAs in human colorectal cancer[J]. Tumour Biol, 2017, 39(3): 1010428317694546. |
| [16] |
Pan B, Qin J, Liu X, et al. Identification of Serum Exosomal hsa-circ_0004771 as a Novel Diagnostic Biomarker of Colorectal Cancer[J]. Front Genet, 2019, 10: 1096. DOI:10.3389/fgene.2019.01096 |
| [17] |
Ye DX, Wang SS, Huang Y, et al. A 3-circular RNA signature as a noninvasive biomarker for diagnosis of colorectal cancer[J]. Cancer Cell Int, 2019, 19: 276. DOI:10.1186/s12935-019-0995-7 |
| [18] |
Tian J, Xi X, Wang J, et al. CircRNA hsa_circ_0004585 as a potential biomarker for colorectal cancer[J]. Cancer Manag Res, 2019, 11: 5413-5423. DOI:10.2147/CMAR.S199436 |
| [19] |
Zhang W, Yang S, Liu Y, et al. Hsa_circ_0007534 as a blood-based marker for the diagnosis of colorectal cancer and its prognostic value[J]. Int J Clin Exp Pathol, 2018, 11(3): 1399-1406. |
| [20] |
Yang N, Xu B, Kong P, et al. Hsa_circ_0002320: a novel clinical biomarker for colorectal cancer prognosis[J]. Medicine (Baltimore), 2020, 99(28): e21224. DOI:10.1097/MD.0000000000021224 |
| [21] |
Zhang J, Cai A, Zhao Y. Three CircRNAs Function as Potential Biomarkers for Colorectal Cancer[J]. Clin Lab, 2020, 66(12): 2405-2412. |
| [22] |
Liang Y, Shi J, He Q, et al. Hsa_circ_0026416 promotes proliferation and migration in colorectal cancer via miR-346/NFIB axis[J]. Cancer Cell Int, 2020, 20: 494. DOI:10.1186/s12935-020-01593-1 |
| [23] |
Li J, Song Y, Wang J, et al. Plasma circular RNA panel acts as a novel diagnostic biomarker for colorectal cancer detection[J]. Am J Transl Res, 2020, 12(11): 7395-7403. |
| [24] |
David Moher, Alessandro Liberati, Jennifer Tetzlaff, 等. 系统综述和荟萃分析优先报告的条目: PRISMA声明[J]. 中西医结合学报, 2009, 7(9): 889-896. [David Moher, Alessandro Liberati, Jennifer Tetzlaff, et al. Preferred reporting items for systematic reviews and meta-analyses: the PRISMA statement (Chinese edition)[J]. Zhong Xi Yi Jie He Xue Bao, 2009, 7(9): 889-896.] |
| [25] |
Whiting PF, Rutjes AW, Westwood ME, et al. QUADAS-2:a revised tool for the quality assessment of diagnostic accuracy studies[J]. Ann Intern Med, 2011, 155(8): 529-536. DOI:10.7326/0003-4819-155-8-201110180-00009 |
| [26] |
罗晓莉, 肖燕萍, 林颖烽, 等. 长链非编码RNA-UCA1对膀胱癌诊断价值的Meta分析[J]. 中国医药导报, 2019, 16(11): 77-80. [Luo XL, Xiao YP, Lin YF, et al. Meta-analysis of the value of long-chain non-coding RNA-UCA1 in the diagnosis of bladder cancer[J]. Zhongguo Yi Yao Dao Bao, 2019, 16(11): 77-80.] |
| [27] |
Huang X, Zhang W, Shao Z. Prognostic and diagnostic significance of circRNAs expression in hepatocellular carcinoma patients: A meta-analysis[J]. Cancer Med, 2019, 8(3): 1148-1156. DOI:10.1002/cam4.1939 |
| [28] |
Huang X, Zhang W, Shao Z. Prognostic and diagnostic significance of circRNAs expression in lung cancer[J]. J Cell Physiol, 2019, 234(10): 18459-18465. DOI:10.1002/jcp.28481 |
| [29] |
童峰, 陈坤, 何寒青. 优势比在诊断试验评价中的应用[J]. 中华流行病学杂志, 2005, 26(10): 813-814. [Tong F, Chen K, He HQ. Application of odds ratio in the evaluation of a diagnostic test[J]. Zhonghua Liu Xing Bing Xue Za Zhi, 2005, 26(10): 813-814. DOI:10.3760/j.issn:0254-6450.2005.10.020] |
| [30] |
王丹, 翟俊霞, 牟振云, 等. Meta分析中的异质性及其处理方法[J]. 中国循证医学杂志, 2009, 9(10): 1115-1118. [Wang D, Zhai JX, Mou ZY, et al. Discussing on the Research of Heterogeneity in Meta-analysis[J]. Zhongguo Xun Zheng Yi Xue Za Zhi, 2009, 9(10): 1115-1118.] |
| [31] |
李冬秀, 陈敬林, 黄湘, 等. 血清AFU对原发性肝癌诊断价值的meta分析[J]. 国际检验医学杂志, 2015, 1: 1-4. [Li DX, Chen JL, Huang X, et al. Diagnostic value of serum AFU for primary hepatic carcinoma: a meta-analysis[J]. Guo Ji Jian Yan Yi Xue Za Zhi, 2015, 1: 1-4.] |
 2021, Vol. 48
2021, Vol. 48