文章信息
- 内源性HMGB1调节低氧乏养胰腺癌细胞脂肪酸代谢及其机制
- Endogenous HMGB1 Modulates Fatty Acid Metabolism of Hypoxic and Nutrient-poor Pancreatic Cancer Cells and Related Mechanism
- 肿瘤防治研究, 2021, 48(7): 667-673
- Cancer Research on Prevention and Treatment, 2021, 48(7): 667-673
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2021.21.0038
- 收稿日期: 2021-01-13
- 修回日期: 2021-04-03
2. 212013 镇江,江苏大学医学院
2. School of Medicine, Jiangsu University, Zhenjiang 212013, China
胰腺癌恶性程度极高,具有早期诊断困难,放化疗不敏感,致死率高等特点[1]。由于丰富的纤维基质成分和不成熟的血管结构,低氧乏养是胰腺癌微环境最显著特点之一[2-3], 癌细胞在相对缺氧和营养缺乏的环境中恶性生物学行为增加,然而其确切机制仍不十分明确。
高迁移率族蛋白B1(high-mobility group box 1, HMGB1)损伤相关模式分子,具有细胞内和细胞外两种存在模式[4-5]。细胞内HMGB1,又称内源性HMGB1,是一种高丰度的细胞核非组蛋白DNA结合蛋白[6],核内的HMGB1参与稳定核小体、DNA复制、转录翻译等进程[7]。在血供和(或)氧供不足、营养缺乏、炎性反应、氧化应激等因素下,内源性HMGB1从肿瘤细胞细胞核转位至细胞质[8-10],进而发生激活肿瘤细胞自噬、抑制凋亡等行为[11]。近年来,内源性HMGB1在肿瘤代谢重编程中的作用得到越来越多的关注,但是其在胰腺癌脂质代谢重编程和线粒体功能调控中的作用却鲜见报道。本研究通过研究胰腺癌Patu8988细胞内源性HMGB1在脂质代谢重编程和线粒体功能中的作用,进一步明确其在胰腺癌增殖、侵袭以及迁移等恶性生物学行为中的作用。
1 材料与方法 1.1 实验材料人胰腺癌细胞株Patu8988、AsPC1、BXPC3和Panc1细胞和人胚胎肾细胞HEK293T购自中国科学院上海细胞研究所;胰蛋白酶、澳洲胎牛血清购自美国Gibco公司;高糖型DMEM、磷酸盐溶液PBS购自美国Hyclone公司;兔抗人单克隆抗体β-微管蛋白(β-tubulin)、兔抗人多克隆抗体高迁移率族蛋白B1(HMGB1)购于美国Abcam公司;兔抗人单克隆抗体脂肪酸合酶(FASN)、三磷酸腺苷柠檬酸裂解酶(ACLY)、磷酸化三磷酸腺苷柠檬酸裂解酶(p-ACLY)、线粒体融合蛋白1(MFN1)、线粒体融合蛋白2(MFN2)、线粒体分裂蛋白(FIS1)、动力相关蛋白(DRP1)、磷酸化动力相关蛋白1(p-DRP1(Ser637))以及鼠抗单克隆抗体酰基辅酶A合成酶短链家族成员2(ACSS2)均购自美国Cell Signaling Technology公司;HRP标记的羊抗兔二抗和羊抗鼠二抗购于美国Santa Cruz Biotechnology公司;MitoTracker®Red CMXRos线粒体红色荧光探针购于上海前尘生物科技有限公司;DAPI溶液购于北京Solarbio公司。
1.2 实验方法 1.2.1 细胞培养人胰腺癌Patu8988细胞培养于DMEM高糖培养基(含10%胎牛血清、100 u/L青霉素、100 mg/L链霉素),在37℃恒温、5%CO2及饱和湿度的培养箱中培养(10%FBS)。乏养培养条件(2%FBS)为DMEM高糖培养基(含2%胎牛血清、100 u/L青霉素、100 mg/L链霉素),在37℃恒温、5%CO2及饱和湿度的培养箱中培养。低氧培养条件(Hypoxia)为将含10%胎牛血清的细胞培养皿置于密封的低氧培养盒中,向低氧培养盒里充入混合气(1%O2、5%CO2、94%N2),持续充入10 min,低氧混合气购自南大恒通气体厂,然后将低氧培养盒转移到37℃培养箱中培养。低氧乏养培养条件(2%FBS+Hypoxia)为DMEM高糖培养基(含2%胎牛血清、100 u/L青霉素、100 mg/L链霉素),将其置于密封的低氧培养盒中,向低氧培养盒里充入混合气,持续充入10 min,随后将低氧培养盒放入37℃恒温培养箱中。
1.2.2 病毒包装与感染在六孔板中接种适量HEK293T细胞,待第二天细胞40%~60%融合时,进行病毒质粒转染。取1.5 ml灭菌EP管,加入1 μg包装混合质粒和1 μg表达质粒(空载质粒、HMGB1-shRNA1、HMGB1-shRNA2)以及100 μl的无血清培养基(高糖DMEM)。轻柔混匀,室温孵育5 min。取1.5 ml灭菌EP管,5 μl PEI脂质体(质粒: PEI脂质体=1:2.5)溶于100 μl无血清培养基中。轻柔混匀,室温孵育5 min。将DNA溶液和脂质体溶液轻柔混匀,室温孵育25 min。在六孔板中每孔加入2 ml含血清的生长培养基,再加入DNA-脂质体复合物。转染后48~72 h收获含病毒的上清液。3 000 r/min离心20 min,去除沉淀。六孔板中接种待感染细胞Patu8988,用含有10%FBS的DMEM培养基培养;待细胞铺板,第二天更换新培养基,加入聚凝胺和上述病毒液进行感染,混匀;第三天重复感染一次。72 h后,荧光显微镜下观察GFP的表达情况。收取细胞系蛋白及RNA,蛋白质免疫印迹法和实时荧光定量PCR鉴定细胞系中HMGB1的表达情况,验证敲减效率。
1.2.3 蛋白质免疫印迹法检测相关蛋白表达用无菌PBS清洗细胞三次后,加入蛋白裂解液,提取总蛋白,100℃加热5 min后12 000 g离心5 min。蛋白经10%十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(SDS-PAGE)分离,每个泳道20 μl的蛋白样品上样,然后将蛋白转印至PVDF膜上,5%脱脂牛奶室温封闭1 h;一抗[β-tubulin、HMGB1、FASN、p-ACLY、ACLY、MFN1、MFN2、FIS1、DRP1、p-DRP1(S637)、ACSS2(1:1 000)] 4℃孵育过夜;TBST洗膜,每次10 min,重复3次;相应二抗[羊抗兔二抗(1:10 000),羊抗鼠二抗(1:10 000)]室温孵育1 h;TBST洗膜3次,每次10 min;ECL发光试剂显影,在凝胶成像系统上拍照并分析。
1.2.4 实时荧光定量PCR检测相关指标mRNA水平的表达用TRIzol法提取总RNA, 根据反转录试剂盒的操作方法获得单链cDNA,以cDNA为模板进行实时荧光定量PCR检测,SYBR Green染料法操作,以β-actin为靶基因的内参,采用10 μl反应体系(2×SYBR Premix ExTaqTM 5 μl,含上游引物0.2 μl、下游引物0.2 μl、cDNA溶液0.5 μl、DEPC水4.1 μl),95℃预变性180 s,95℃ 10 s,56℃ 30 s,40个循环。
1.2.5 激光共聚焦显微镜观察线粒体形态变化在24孔板内放置盖玻片,加入适量培养基进行爬片培养。细胞贴壁后进行低氧乏养处理,24 h后吸除培养液,加入37℃预热的MitoTracker®Red CMXRos染色工作液,与细胞共孵育15~45 min。染色结束后,去除染色工作液,在24孔板中用PBS清洗玻片一次;换用新鲜配制且预热的4%多聚甲醛室温固定20 min后PBST浸洗玻片3次;滴加DAPI避光孵育10~15 min,PBST浸洗4次; 吸干爬片上的液体,封片液封片,然后在激光共聚焦显微镜下观察细胞内线粒体形态,并采集相应图像。
1.2.6 细胞增殖能力检测接种对照组(Ctrl-HMGB1)及干扰组(HMGB1-shRNA1、HMGB1-shRNA2)Patu8988细胞于96孔板内,每孔1×103个,细胞贴壁后低氧乏养培养,每24 h加入10 μl CCK-8检测试剂,避光孵育2 h,检测450 nm处吸光度值。
1.2.7 细胞侵袭和转移能力检测(1)Transwell小室迁移实验将Transwell小室放入24孔板孔洞中,小室内为上室,24孔板孔洞为下室,将对照组(Ctrl-HMGB1)和干扰组(HMGB1-shRNA1、HMGB1-shRNA2)Patu8988细胞用胰酶消化,离心后1 ml无血清培养基重悬,计数,吸取100 μl细胞悬液(约含5×104个细胞)滴加入小室上方,低氧乏养培养。小室外侧加入500 μl含2%FBS的新鲜培养基,将其置于密封的低氧培养盒中,向低氧培养盒里充入混合气,持续充入10 min,随后将低氧培养盒放入37℃恒温培养箱中。当下室底部出现穿过Transwell上小孔的癌细胞时,对下室侧癌细胞进行结晶紫染色。在显微镜下随机选取视野进行拍摄;此实验重复三次。(2)Transwell小室侵袭实验将BD胶放置于冰上融化。每孔吸取100 μl无血清培养基,待BD胶完全融化后吸取4 μl至EP管中,与无血清培养基混合快速吹匀后加入到小室内,放置于细胞恒温培养箱。待BD胶完全凝固后,弃去无血清培养基,加入细胞悬液。实验步骤同Transwell迁移实验。
1.2.8 细胞迁移能力检测将对照组(Ctrl-shRNA)和干扰组(HMGB1-shRNA1、HMGB1-shRNA2)Patu8988细胞接种至24孔板,每孔接种细胞约2×105个,放置于细胞恒温培养箱中过夜。第二天细胞贴壁且融合度达到90%~100%,用10 μl小枪头在孔底划两条平行直线。用PBS清洗划痕后的孔,清洗2~3次后,低氧乏养处理,选取合适位置进行显微镜下拍照记为0 h,24 h后同一位置拍照。实验重复3次。
1.2.9 生物信息学分析利用GEPIA数据库(http://gepia.cancer-pku.cn/)中的临床患者相关数据,分析HMGB1在胰腺癌患者和非胰腺癌患者中的表达水平。将胰腺癌患者分为HMGB1高表达组和低表达组,分析两组的生存期。
1.3 统计学方法所有实验均独立重复三次,采用SPSS17.0统计学软件对数据进行分析。计量资料用均数±标准差表示, 两组间均数比较采用两独立样本t检验,多组间均数比较采用单因素方差分析(one-way ANOVA),组间多重比较采用LSD-t检验,以P < 0.05为差异有统计学意义。
2 结果 2.1 HMGB1表达水平与胰腺癌患者生存率的关系经GEPIA数据库分析临床胰腺癌标本发现:与正常胰腺组织相比,胰腺癌组织HMGB1 mRNA表达水平明显增高(P < 0.01),见图 1A。TCGA转录组数据提示:HMGB1高表达组胰腺癌患者总体生存率明显低于低表达组(P=0.00097),见图 1B。
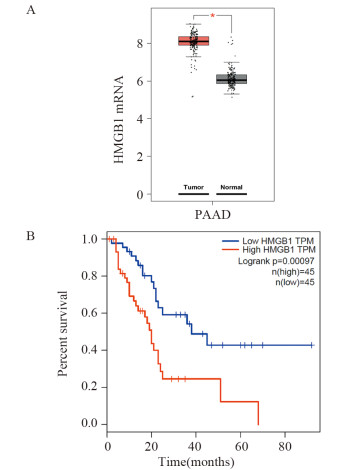
|
| A: GEPIA database analysis of HMGB1 mRNA expression levels in clinical pancreatic cancer specimens; B: TCGA transcriptome data analysis of the correlation between the expression level of HMGB1 and overall survival of pancreatic cancer patients; *: P < 0.01. 图 1 GEPIA数据库及TCGA转录组数据分析HMGB1在胰腺癌中的表达及其临床意义 Figure 1 GEPIA database and TCGA transcriptome data analyses of HMGB1 expression in pancreatic cancer tissues and its clinical significance |
蛋白质免疫印迹结果表明在胰腺癌细胞AsPC1、BXPC3、Patu8988和Panc1中,Patu8988的HMGB1基础表达量最高,见图 2A,因此选择Patu8988细胞株进行稳定干扰HMGB1的细胞株构建。荧光显微镜结果显示Patu8988细胞绿色荧光蛋白(green fluorescent protein, GFP)表达呈阳性,提示质粒转染成功,见图 2B。蛋白免疫印迹法及实时荧光PCR法分别检测显示:干扰组(HMGB1-shRNA1和HMGB1-shRNA2)HMGB1的蛋白及mRNA表达量明显下降,表明Patu8988 HMGB1干扰细胞系构建成功,见图 2C~E。
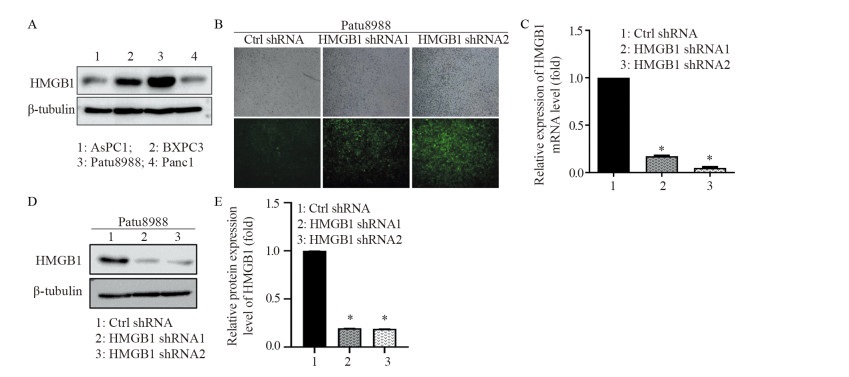
|
| A: the protein expression levels of HMGB1 in human pancreatic cancer cell lines AsPC1, BXPC3, Patu8988 and Panc1; B: the expression of GFP detected by fluorescence microscope; C: the expression of HMGB1 mRNA level detected by qRT-PCR; D: the expression of HMGB1 protein level detected by Western blot; E: histogram; *: P < 0.05, compared with Ctrl shRNA. 图 2 HMGB1敲减细胞株构建及效率验证 Figure 2 Construction and efficiency verification of HMGB1-knockdown cell line |
与对照组(Ctrl-shRNA)相比,Transwell实验结果显示:细胞的侵袭迁移能力下降,见图 3A、3B;CCK-8法检测Patu8988细胞的增殖能力,敲减低氧乏养Patu8988细胞的HMGB1后,细胞增殖能力明显下降,见图 3C。因此,低氧乏养的肿瘤微环境中,内源性HMGB1维持胰腺癌Patu8988细胞的增殖、侵袭及迁移运动能力。
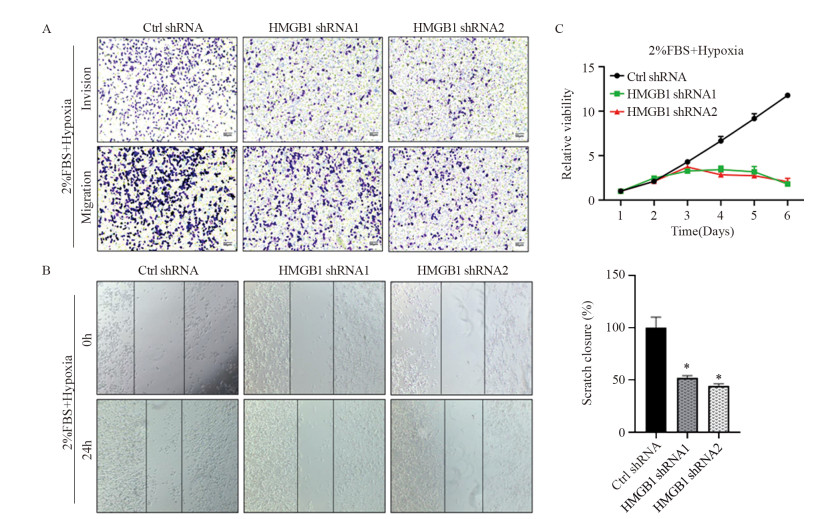
|
| *: P < 0.05, compared with Ctrl shRNA. 图 3 HMGB1敲减对Patu8988细胞增殖(A)、侵袭(B)及迁移运动(C)能力的影响 Figure 3 Effect of HMGB1 knockdown on proliferation(A), invasion(B) and migration(C) of Patu8988 cells |
将Patu8988细胞在不同培养条件下培养24 h后,蛋白质免疫印迹结果显示Patu8988细胞ACLY及p-ACLY蛋白表达水平明显升高,见图 4A,ACLY作用的柠檬酸途径来源的线粒体乙酰辅酶A增加,FA合成相关指标脂肪酸合酶(FASN)升高,表明低氧乏养Patu8988细胞的FA合成增加。
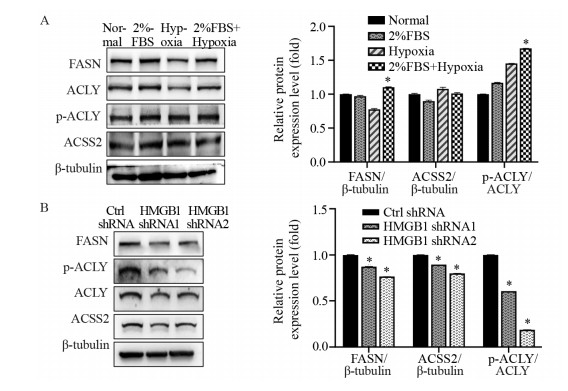
|
| *: P < 0.05, compared with normal culture condition. 图 4 蛋白质免疫印迹法检测HMGB1敲减对Patu8988细胞脂肪酸从头合成相关蛋白表达量的影响 Figure 4 Effect of HMGB1 knockdown on expression of de novo fatty acid synthesis related proteins in Patu8988 cells detected by Western blot |
蛋白质免疫印迹法结果显示:与对照组(Ctrl-HMGB1)相比,干扰HMGB1组的低氧乏养Patu8988细胞ACLY及p-ACLY蛋白表达水平明显降低,见图 4B,表明内源性HMGB1上调低氧乏养Patu8988细胞ACLY蛋白表达,提高其磷酸化水平,增强ACLY的活性,促进肿瘤的FA合成,从而维持胰腺癌细胞的恶性增殖、侵袭及迁移运动能力。
2.4 内源性HMGB1促进低氧乏养Patu8988细胞线粒体融合激光共聚焦显微镜观察到干扰HMGB1的低氧乏养Patu8988细胞与未干扰组相比,线粒体网状结构破坏,融合减少,分裂增多,见图 5A。蛋白质免疫印迹结果表明干扰组的Patu8988细胞线粒体外膜融合蛋白1(MFN1)和线粒体外膜融合蛋白2(MFN2)表达量下降,线粒体分裂蛋白FIS1表达量明显升高,p-DRP1(Ser637)降低,见图 5B。上述结果表明内源性HMGB1参与调节低氧乏养Patu8988细胞线粒体的动态变化,促进线粒体的融合,抑制线粒体的分裂。
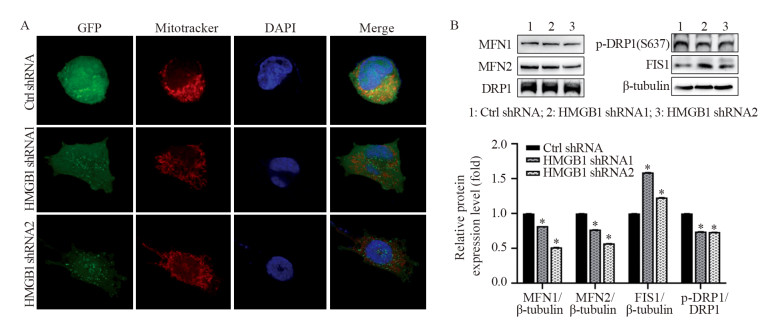
|
| *: P < 0.05, compared with Ctrl shRNA. 图 5 激光共聚焦显微镜和蛋白质免疫印迹法检测HMGB1敲减对Patu8988细胞线粒体形态(A)及线粒体分裂和融合相关蛋白表达量(B)的影响 Figure 5 Effect of HMGB1 knockdown on mitochondrial morphology(A) and expression of mitochondrial division and fusion related proteins(B) in Patu8988 cells detected by laser confocal microscope and Western blot |
HMGB1在多种肿瘤中过度表达[12-13],对肿瘤细胞的增殖、迁移、浸润和肿瘤新生血管形成等有着重要的影响。HMGB1在细胞核内合成,通过自分泌和旁分泌方式分泌到胞质和细胞外基质中[14]。根据其定位和修饰的不同,HMGB1的功能也不同。胞质和细胞外基质中的HMGB1可结合多种受体,比如Toll样受体(TLR)和糖基化终产物受体(RAGE)等[15],激活多种信号通路,参与细胞发育、分化、免疫、转移、新陈代谢、自噬和死亡,乙酰化和磷酸化有助于HMGB1在胞质中的积累,阻止其重新定位于细胞核中;细胞核内HMGB1调节多种蛋白的转录活性,例如,HMGB1直接与P53(抑癌基因)相互作用,增强其DNA结合[16-17]。近年来对HMGB1的研究指出,内源性HMGB1在细胞能量代谢中发挥重要作用[18]。本研究结果显示敲减HMGB1基因后,低氧乏养Patu8988细胞ACLY和p-ACLY均显著下调,FASN蛋白表达水平降低,Patu8988细胞的增殖、侵袭和迁移运动能力明显下降,表明内源性HMGB1通过调节ACLY的表达和活性,促进胰腺癌细胞FA合成,维持其恶性生物学行为。这对研究内源性HMGB1在胰腺癌脂质代谢中的作用及其机制具有重要意义。
代谢重编程是肿瘤的重要特征,肿瘤细胞通过代谢重编程为其在严酷的微环境中(如缺氧、酸中毒、营养不良)提供了生存和增殖的选择优势[19-20]。大量研究表明:脂质代谢在肿瘤中发生高度的重编程[21-23]。除Warburg效应外,在多种肿瘤细胞中发现脂质合成增加,以维持肿瘤细胞在生长过程中的膜合成以及能量代谢等过程[24]。与正常组织相比,细胞恶性行为增加的重要特征之一就是FA从头合成的增加[25]。FA代谢模式的调整会影响细胞生长、增殖、分化和运动性能[26]。FA合成是一种合成代谢过程,可将营养素衍生的碳转化为脂肪酸。这些碳主要由柠檬酸盐提供,通常由葡萄糖衍生的丙酮酸盐进入线粒体三羧酸(TCA)循环产生,在ACLY的作用下裂解为草酰乙酸和乙酰辅酶A,后者是FA合成的主要底物,是糖代谢和脂肪酸代谢的枢纽[27]。本研究结果证明了在低氧乏养的微环境下,胰腺癌Patu8988细胞ACLY表达及活性明显升高,FASN表达增高,FA合成增加。值得注意的是,癌细胞在产生用于FA合成的柠檬酸盐过程中表现出代谢可塑性。在缺氧或线粒体功能异常的癌细胞中,TCA循环受到抑制,线粒体来源的柠檬酸盐减少[28],因此线粒体功能在FA合成中至关重要。正常情况下,线粒体融合与分裂处于平衡状态,其数目与形态保持稳定。线粒体融合与分裂平衡失调会导致细胞功能障碍。融合增多,线粒体呈长管网状结构,能量代谢更加丰富,可对细胞起到一定的保护作用;分裂增多,线粒体变得短小、碎片化,呈点状结构,导致线粒体损伤[29]。有证据表明HMGB1可以促进肝癌细胞线粒体的生物合成,并且维持线粒体的形态,干扰HMGB1之后,线粒体发生功能障碍[30]。但也有文章表明HMGB1可以通过诱导线粒体分裂促进成纤维样滑膜细胞迁移[31]。本实验进一步研究HMGB1对低氧乏养胰腺癌细胞线粒体分裂与融合的影响,结果显示,敲减HMGB1后,线粒体分裂增加,功能障碍,FA合成减少。但实验中并未对线粒体形态变化导致的线粒体内其他代谢的改变进行深入的研究,因此线粒体代谢的变化与脂质代谢的关系有待进一步研究。
本研究证明HMGB1可以通过维持低氧乏养胰腺癌Patu8988细胞线粒体的功能,上调ACLY表达和活性,促进胰腺癌的FA合成,维持肿瘤细胞的恶性生物学行为,促进肿瘤的发生发展。本研究可能为针对胰腺癌微环境的靶向治疗提供新的思路和依据。
作者贡献:
高洁:实验操作及论文撰写
吴琪炜、宋廉、石卉、王鸣、龚爱华:实验操作及数据分析
朱海涛、王冬青:实验设计、论文指导及审阅
| [1] |
Quaresma M, Coleman MP, Rachet B. 40-year trends in an index of survival for all cancers combined and survival adjusted for age and sex for each cancer in England and Wales, 1971-2011: a population-based study[J]. Lancet, 2015, 385(9974): 1206-1218. DOI:10.1016/S0140-6736(14)61396-9 |
| [2] |
Haqq J, Howells LM, Garcea G, et al. Pancreatic stellate cells and pancreas cancer: current perspectives and future strategies[J]. Eur J Cancer, 2014, 50(15): 2570-2582. DOI:10.1016/j.ejca.2014.06.021 |
| [3] |
Huang Y, Don-Wauchope AC. The clinical utility of kidney injury molecule 1 in the prediction, diagnosis and prognosis of acute kidney injury: a systematic review[J]. Inflamm Allergy Drug Targets, 2011, 10(4): 260-271. DOI:10.2174/187152811796117735 |
| [4] |
Lu B, Antoine DJ, Kwan K, et al. JAK/STAT1 signaling promotes HMGB1 hyperacetylation and nuclear translocation[J]. Proc Natl Acad Sci U S A, 2014, 111(8): 3068-3073. DOI:10.1073/pnas.1316925111 |
| [5] |
Napolitano A, Antoine DJ, Pellegrini L, et al. HMGB1 and Its Hyperacetylated Isoform are Sensitive and Specific Serum Biomarkers to Detect Asbestos Exposure and to Identify Mesothelioma Patients[J]. Clin Cancer Res, 2016, 22(12): 3087-3096. DOI:10.1158/1078-0432.CCR-15-1130 |
| [6] |
Murugesapillai D, McCauley MJ, Maher LJ 3rd, et al. Single-molecule studies of high-mobility group B architectural DNA bending proteins[J]. Biophys Rev, 2016, 9(1): 17-40. |
| [7] |
Zheng H, Chen JN, Yu X, et al. HMGB1 Enhances Drug Resistance and Promotes In Vivo Tumor Growth of Lung Cancer Cells[J]. DNA Cell Biol, 2016, 35(10): 622-627. DOI:10.1089/dna.2016.3360 |
| [8] |
Yang H, Antoine DJ, Andersson U, et al. The many faces of HMGB1: molecular structure-functional activity in inflammation, apoptosis, and chemotaxis[J]. J Leukoc Biol, 2013, 93(6): 865-873. DOI:10.1189/jlb.1212662 |
| [9] |
Kawanishi S, Ohnishi S, Ma N, et al. Crosstalk between DNA Damage and Inflammation in the Multiple Steps of Carcinogenesis[J]. Int J Mol Sci, 2017, 18(8): 1808. DOI:10.3390/ijms18081808 |
| [10] |
Pistoia V, Pezzolo A. Involvement of HMGB1 in Resistance to Tumor Vessel-Targeted, Monoclonal Antibody-Based Immunotherapy[J]. J Immunol Res, 2016, 2016: 3142365. |
| [11] |
Min HJ, Suh KD, Lee YH, et al. Cytoplasmic HMGB1 and HMGB1-Beclin1 complex are increased in radioresistant oral squamous cell carcinoma[J]. Br J Oral Maxillofac Surg, 2019, 57(3): 219-225. DOI:10.1016/j.bjoms.2019.01.011 |
| [12] |
Chung HW, Lee SG, Kim H, et al. Serum high mobility group box-1 (HMGB1) is closely associated with the clinical and pathologic features of gastric cancer[J]. J Transl Med, 2009, 7: 38. DOI:10.1186/1479-5876-7-38 |
| [13] |
王华, 蔡红兵, 李树炜, 等. HMGB1对宫颈癌干性基因OCT4、Sox2和Nanog表达的影响[J]. 肿瘤防治研究, 2018, 45(10): 786-791. [Wang H, Cai HB, Li SW, et al. Effect of High-mobility Group Box-1 on Expression of Stem Cell Markers OCT4, Sox2 and Nanog in Cervical Carcinoma[J]. Zhong Liu Fang Zhi Yan Jiu, 2018, 45(10): 786-791. DOI:10.3971/j.issn.1000-8578.2018.17.1374] |
| [14] |
Fujii K, Luo Y, Sasahira T, et al. Co-treatment with deoxycholic acid and azoxymethane accelerates secretion of HMGB1 in IEC6 intestinal epithelial cells[J]. Cell Prolif, 2009, 42(5): 701-709. DOI:10.1111/j.1365-2184.2009.00624.x |
| [15] |
Hreggvidsdottir HS, Lundberg AM, Aveberger AC, et al. High mobility group box protein 1 (HMGB1)-partner molecule complexes enhance cytokine production by signaling through the partner molecule receptor[J]. Mol Med, 2012, 18(1): 224-230. |
| [16] |
Tang D, Kang R, Zeh HJ 3rd, et al. High-mobility group box 1 and cancer[J]. Biochim Biophys Acta, 2010, 1799(1-2): 131-140. DOI:10.1016/j.bbagrm.2009.11.014 |
| [17] |
Banerjee S, Kundu TK. The acidic C-terminal domain and A-box of HMGB-1 regulates p53-mediated transcription[J]. Nucleic Acids Res, 2003, 31(12): 3236-3247. DOI:10.1093/nar/gkg412 |
| [18] |
Gdynia G, Sauer SW, Kopitz J, et al. The HMGB1 protein induces a metabolic type of tumour cell death by blocking aerobic respiration[J]. Nat Commun, 2016, 7: 10764. DOI:10.1038/ncomms10764 |
| [19] |
Pavlova NN, Thompson CB. The Emerging Hallmarks of Cancer Metabolism[J]. Cell Metab, 2016, 23(1): 27-47. DOI:10.1016/j.cmet.2015.12.006 |
| [20] |
Finley LW, Zhang J, Ye J, et al. SnapShot: cancer metabolism pathways[J]. Cell Metab, 2013, 17(3): 466-466. e2. DOI:10.1016/j.cmet.2013.02.016 |
| [21] |
Abramson HN. The lipogenesis pathway as a cancer target[J]. J Med Chem, 2011, 54(16): 5615-5638. DOI:10.1021/jm2005805 |
| [22] |
Santos CR, Schulze A. Lipid metabolism in cancer[J]. FEBS J, 2012, 279(15): 2610-2623. DOI:10.1111/j.1742-4658.2012.08644.x |
| [23] |
Schulze A, Harris AL. How cancer metabolism is tuned for proliferation and vulnerable to disruption[J]. Nature, 2012, 491(7424): 364-373. DOI:10.1038/nature11706 |
| [24] |
Luo X, Cheng C, Tan Z, et al. Emerging roles of lipid metabolism in cancer metastasis[J]. Mol Cancer, 2017, 16(1): 76. DOI:10.1186/s12943-017-0646-3 |
| [25] |
Menendez JA, Lupu R. Fatty acid synthase and the lipogenic phenotype in cancer pathogenesis[J]. Nat Rev Cancer, 2007, 7(10): 763-777. DOI:10.1038/nrc2222 |
| [26] |
Hatziapostolou M, Polytarchou C, Iliopoulos D. miRNAs link metabolic reprogramming to oncogenesis[J]. Trends Endocrinol Metab, 2013, 24(7): 361-373. DOI:10.1016/j.tem.2013.03.002 |
| [27] |
Zaidi N, Swinnen JV, Smans K. ATP-citrate lyase: a key player in cancer metabolism[J]. Cancer Res, 2012, 72(15): 3709-3714. DOI:10.1158/0008-5472.CAN-11-4112 |
| [28] |
Chen M, Huang J. The expanded role of fatty acid metabolism in cancer: new aspects and targets[J]. Precis Clin Med, 2019, 2(3): 183-191. DOI:10.1093/pcmedi/pbz017 |
| [29] |
Chan DC. Mitochondrial fusion and fission in mammals[J]. Annu Rev Cell Dev Biol, 2006, 22: 79-99. DOI:10.1146/annurev.cellbio.22.010305.104638 |
| [30] |
Tohme S, Yazdani HO, Liu Y, et al. Hypoxia mediates mitochondrial biogenesis in hepatocellular carcinoma to promote tumor growth through HMGB1 and TLR9 interaction[J]. Hepatology, 2017, 66(1): 182-197. DOI:10.1002/hep.29184 |
| [31] |
吴玉娟, 曹镐禄, 何华宾, 等. 高迁移率族蛋白1通过诱导线粒体分裂促进成纤维样滑膜细胞迁移[J]. 赣南医学院学报, 2017, 37(6): 851-855. [Wu YJ, Cao GL, He HB, et al. High-mobility Group Protein B1 promotes the Migration of Fibroblast-like Synovial Cells by Inducing Mitochondrial Fission[J]. Gannan Yi Xue Yuan Xue Bao, 2017, 37(6): 851-855. DOI:10.3969/j.issn.1001-5779.2017.06.004] |
 2021, Vol. 48
2021, Vol. 48


