文章信息
- 2012-2019年中国未知HAdV病毒株分型及基因特征分析
- Classification and Gene Features of Unverified HAdV in China, 2012-2019
- 肿瘤防治研究, 2021, 48(6): 581-585
- Cancer Research on Prevention and Treatment, 2021, 48(6): 581-585
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2021.20.1416
- 收稿日期: 2020-12-07
- 修回日期: 2021-04-09
人腺病毒(human adenovirus, HAdV)最初由挪威Rowe等于1953年在健康成年人中的扁桃腺样组织中进行分离培养出来,1954年希勒曼对患有急性呼吸道感染的患者进行研究,从咽喉分泌物中分离到相同的病毒,1956年,国际病毒委员会(ICTV)将这种病毒命名为人腺病毒[1]。
人腺病毒基因组长约36 kb[2],含九类基因,按照复制时间分为早期转录基因和晚期转录基因[3-4],其中关于早期表达与腺病毒有关的基因为E1~E3、E7基因,晚期表达与腺病毒有关的基因有5种,为L1~L5基因[4]。
癌症是造成人类死亡的重要原因之一,对大多数肿瘤类型来说,目前的医疗技术仍无法治愈。溶瘤病毒疗法一直被认为是一种有前途的抗癌基因治疗策略[5]。人腺病毒是研究广泛的溶瘤病毒之一,能特异性感染并裂解肿瘤细胞,溶瘤腺病毒疗法在治疗癌症方面已经取得了一定进展[6]。HAdV-C5型人腺病毒是使用最广泛的溶瘤腺病毒之一,目前有临床试验发现HAdV-C5型人腺病毒的中和性免疫阻碍了对人前列腺癌的治疗效果[7],B型溶瘤腺病毒能够明显地裂解骨肉瘤细胞[8]。因此,针对不同癌症选用合适类型的人腺病毒用于溶瘤腺病毒疗法非常重要。
根据人腺病毒基因组的DNA序列特点、六邻体蛋白特点、纤突特点以及免疫原性可将人腺病毒分为HAdV-A-G共7个亚属[3],102个基因型[9]。截至目前,仍有少部分HAdV基因组未能明确分型。为此,本研究从GenBank数据库中共收集到11株2012-2019年来自中国未明确分型的HAdV完整基因组序列,并收集国内外已明确分型的HAdV基因组序列,通过生物信息学方法对这些未能明确分型的HAdV进行基因结构、基因同源性及系统进化分析,从基因层面上对这些未知HAdV序列进行分型。
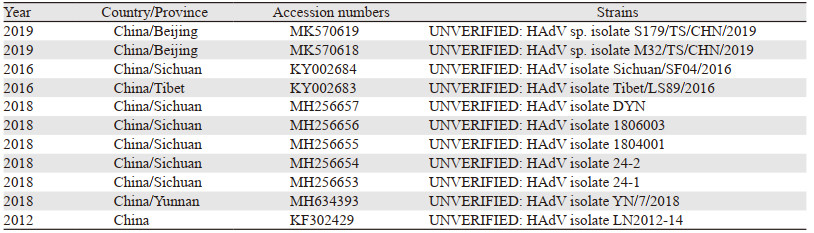
1 材料与方法 1.1 病毒株来源与基本信息本研究所需的基因序列全部来自GenBank数据库(https://www.ncbi.nlm.nih.gov/)。其中11株尚未明确分型的完整HAdV基因组序列均来自中国,另外67株已明确基因型的HAdV病毒株,分别来自HAdV-A到HAdV-G不同亚属共67个基因型,以及7株国际上公认的HAdV参照基因组序列,将这74株基因组序列作为系统进化分析的参照序列。本研究11株未知亚属病毒株序列的基本信息,包括病毒株收集时间和地区、基因登录号(Accession Numbers)、病毒株的名称,见表 1。
使用DNA Star软件中的EditSeq工具将收集到的所有HAdV基因组序列以FASTA格式保存以便于后续分析[10]。将11株未明确分型的HAdV基因组序列、67株已明确基因型的参照HAdV基因组序列以及7株国际公认的HAdV参照序列利用MEGA7.0软件中的邻接法构建HAdV病毒株的系统进化树,分析未明确分型的HAdV序列与已知HAdV序列之间的进化关系,选择Kimura 2-parameter模型,自展次数选择1 000次[11]。
1.2.2 基因同源性分析利用DNA Star软件中的MegAlign工具将未明确分型HAdV序列与已知序列进行比对,分析基因序列之间的同源性,算法选择Clustal W[10]。
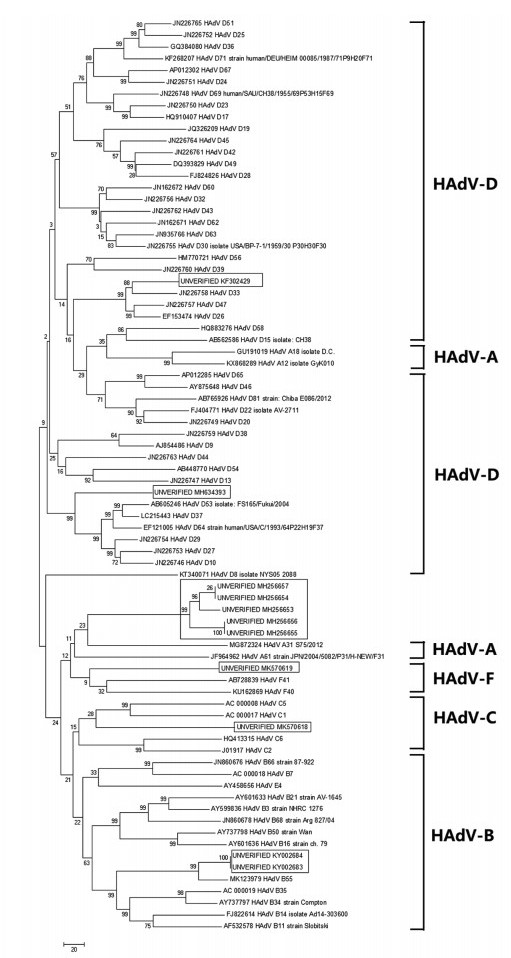
2 结果 2.1 HAdV基因组的系统进化分析系统进化树结果显示:同亚属的HAdV之间进化关系十分明显,可以用来推测未明确分型病毒株的进化关系,见图 1。
|
| 图 1 基于HAdV基因组数据库构建系统进化树 Figure 1 Phylogenetic tree of whole HAdV genome |
MH634393和KF302429两株HAdV病毒株与D型HAdV病毒株序列进化关系较近;KY002683和KY002684两株HAdV病毒株与B型HAdV病毒株序列进化关系较近;MK570618的HAdV病毒株与C型HAdV病毒株序列进化关系较近;MK570619的HAdV病毒株与F型HAdV病毒株序列进化关系较近。剩下5株HAdV病毒株(MH256653、MH256654、MH256655、MH256656、MH256657)相互之间的进化关系比较接近,但是与其他参照病毒株进化关系不是非常明显,对这5株HAdV病毒株的分型还有待进一步验证。
2.2 基因组序列同源性分析根据系统进化分析结果,预测出6株未明确分型HAdV基因组序列的可能分型,将这6株HAdV序列分别与预测分型的参照序列进行多序列比对分析,观察基因组序列之间的同源性。
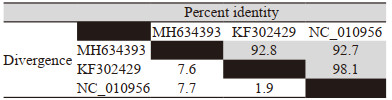
2.2.1将MH634393和KF302429基因组序列与HAdV-D参照基因组序列以及进化关系最近的基因组序列进行多序列比对,结果发现:MH634393与D型HAdV参照基因组序列同源性为92.7%,KF302429与D型HAdV参照基因组序列同源性为98.1%,见表 2,与之前系统进化分析结果相符合,可以推断出这两株未明确类型的HAdV病毒株为D型HAdV。
|
将MK570618基因组序列与HAdV-C参照基因组序列以及进化关系最近的基因组序列进行多序列比对,结果发现:MK570618与3株C型HAdV序列的同源性都比较低,基因组序列上存在较大差异,见表 3,与系统进化分析结果不符,推测MK570618不是C型HAdV。

|
将KY002683和KY002684基因组序列与HAdV-B参照基因组序列以及进化关系最近的基因组序列进行多序列比对,结果发现:KY002683和KY002684之间的同源性是100%,与B1型HAdV基因组序列(NC_011203)的同源性为84.8%,与B2型HAdV基因组序列(NC_011202)的同源性为97.8%,见表 4,故推测这两株HAdV应该属于B2型HAdV,此结果也与系统进化树结果相符合。

|
将MK570619基因组序列与HAdV-F参照基因组序列以及进化关系最近的基因组序列进行多序列比对,结果发现:MK570619和3株HAdV-F的基因组序列之间的同源性较低,只有64.1%~64.4%,见表 5。说明MK570619与F型HAdV的基因组序列存在较大差异,推测可能是由于在做系统进化分析时结果存在一定误差,排除是F型HAdV的可能性。这株HAdV无法从基因层面上对其分型,可能需要通过对其蛋白质的分析来推断出MK570619的分型。

|
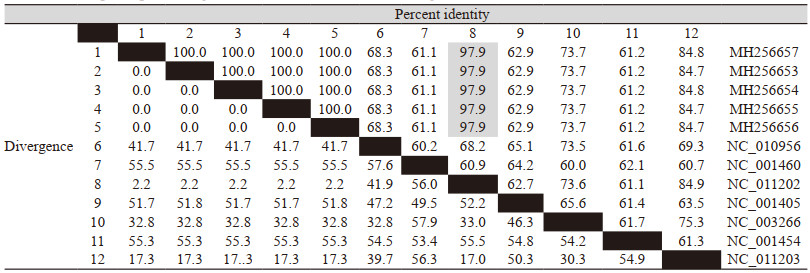
通过系统进化分析预测出了6株未明确分型的HAdV所属亚属,但仍有5株进化关系不明显,无法从系统进化分析中推测出具体分型。将这5株HAdV与7株HAdV参照序列(HAdV-A、HAdV-B1、HAdV-B2、HAdV-C、HAdV-D、HAdV-E、HAdV-F)进行多序列比对,观察其同源性特点发现:这5株HAdV相互之间的序列同源性为100%,应属于同一分型,与B2型HAdV基因组序列的同源性达到了97.9%,推测这5株HAdV序列应该属于B型HAdV,见表 6。
人腺病毒是常见的传染病原体,会引起呼吸道、胃肠道等疾病[12]。在过去几十年里对于人腺病毒的研究不断深入,对人腺病毒的治疗也有了相应的临床方案。同时,人腺病毒也是很常用的基因载体,在临床应用方面经常用于肿瘤基因治疗[13]。
溶瘤腺病毒疗法被认为是有希望的新型抗癌疗法,通过对腺病毒载体的改造,利用病毒特异性靶向杀死肿瘤细胞[14]。HAdV-C5型人腺病毒作为溶瘤腺病毒已广泛用于癌症的临床治疗中,有研究评估了15种不同人腺病毒对前列腺、肾脏肿瘤和骨肉瘤的溶瘤活性。在对20例临床患者骨肉瘤病理切片观察中发现B型溶瘤腺病毒对骨肉瘤细胞具有杀伤作用,同时抑制了骨肉瘤细胞的自噬过程,从而增强了细胞对化疗的敏感度[8]。因此,B型HAdV作为溶瘤腺病毒在骨肉瘤治疗方面具有较高的临床应用潜力。在人前列腺肿瘤的测试中,HAdV-C5型、HAdV-B11和HAdV-B35型人腺病毒在静脉注射后造成了较严重的肝毒性,HAdV-C6型毒性相对较小,同时显著延缓了肿瘤生长[15],说明HAdV-C6型人腺病毒比HAdV-C5型、HAdV-B11和HAdV-B35型人腺病毒更适合于人前列腺肿瘤的溶瘤病毒治疗。因此并不是所有类型的人腺病毒都可以用于溶瘤病毒治疗[9],找出合适类型的人腺病毒进行抗癌治疗是十分必要的。
本研究将11株未明确分型的完整HAdV基因组序列进行分型,并找出适合做溶瘤腺病毒的病毒株。通过系统进化分析结果显示有6株未知的HAdV序列与参照HAdV序列有着很近的进化关系,可以推测出这6株可能的分型。剩余5株在系统进化树上彼此之间的进化关系很接近,但是与参照序列无明显进化关系,无法通过系统进化分析来判别其类型。
对之前已推测出可能类型的HAdV序列与各自参照序列进行多序列比对结果发现:KY002683和KY002684基因组序列与HAdV-B参照基因组序列之间的同源性为97.8%,综合系统进化分析结果判断出这两株HAdV应该属于B型HAdV;MH634393和KF302429基因组序列与HAdV-D参照基因组序列之间的同源性分别为92.7%和98.1%,推测这两株HAdV应该属于D型HAdV;剩余两株为MK570618和MK570619的HAdV与系统进化分析得出的可能类型的参照序列之间同源性较低,与进化分析结果不一致,无法从基因层面上判断出这两株病毒的类型,可能还需要进一步对这两株病毒株的蛋白质序列进行相关分析才能判断出其分型。
剩余5株HAdV基因组序列未能从系统进化分析结果上推断出病毒类型,可能是由于HAdV之间本身的差异比较小,导致系统进化关系比较接近,于是将这5株HAdV基因组序列与HAdV-A-HAdV-F参照序列进行多序列比对分析,发现这5株HAdV的序列与B型HAdV基因组序列的同源性达到了97.9%,推测这5株应该属于B型HAdV,这5株B型HAdV病毒株与之前分型的两株B型HAdV病毒株可能适合作为溶瘤腺病毒进行骨肉瘤治疗的相关研究。
本文对2012-2019年在中国发现的11株未分型HAdV病毒株的基因组序列进行了初步研究,对其中9株病毒株进行了分型,并找出7株可能适合作为溶瘤腺病毒的B型HAdV病毒株,为之后对未知HAdV病毒株的分型以及溶瘤腺病毒类型的选择提供了一定思路。
作者贡献:
马骁:数据收集分析、论文设计和撰写
刘万红:论文选题及修改
| [1] |
Mennechet FJD, Paris O, Ouoba AR, et al. A review of 65 years of human adenovirus seroprevalence[J]. Expert Rev Vaccines, 2019, 18(6): 597-613. DOI:10.1080/14760584.2019.1588113 |
| [2] |
Lynch 3rd JP, Kajon AE. Adenovirus: Epidemiology, Global Spread of Novel Serotypes, and Advances in Treatment and Prevention[J]. Semin Respir Crit Care Med, 2016, 37(4): 586-602. DOI:10.1055/s-0036-1584923 |
| [3] |
Chartier C, Degryse E, Gantzer M, et al. Efficient generation of recombinant adenovirus vectors by homologous recombination in Escherichia coli[J]. J Virol, 1996, 70(7): 4805-4810. DOI:10.1128/jvi.70.7.4805-4810.1996 |
| [4] |
Xing L, Tikoo SK. Promoter activity of left inverted terminal repeat and downstream sequences of porcine adenovirus type 3[J]. Virus Res, 2005, 109(1): 51-58. DOI:10.1016/j.virusres.2004.10.006 |
| [5] |
Kelly E, Russell SJ. History of oncolytic viruses: genesis to genetic engineering[J]. Mol Ther, 2007, 15(4): 651-659. DOI:10.1038/sj.mt.6300108 |
| [6] |
Twumasi-Boateng K, Pettigrew JL, Kwok YYE, et al. Oncolytic viruses as engineering platforms for combination immunotherapy[J]. Nat Rev Cancer, 2018, 18(7): 419-432. DOI:10.1038/s41568-018-0009-4 |
| [7] |
Vogels R, Zuijdeest D, van Rijnsoever R, et al. Replication-deficient human adenovirus type 35 vectors for gene transfer and vaccination: efficient human cell infection and bypass of preexisting adenovirus immunity[J]. J Virol, 2003, 77(15): 8263-8271. DOI:10.1128/JVI.77.15.8263-8271.2003 |
| [8] |
Bishop MW, Janeway KA, Gorlick R. Future directions in the treatment of osteosarcoma[J]. Curr Opin Pediatr, 2016, 28(1): 26-33. DOI:10.1097/MOP.0000000000000298 |
| [9] |
Baker AT, Aguirre-Hernández C, Halldén G, et al. Designer Oncolytic Adenovirus: Coming of Age[J]. Cancers (Basel), 2018, 10(6): 201. DOI:10.3390/cancers10060201 |
| [10] |
Burland TG. DNASTAR's Lasergene sequence analysis software[J]. Methods Mol Biol, 2000, 132: 71-91. |
| [11] |
Kumar S, Stecher G, Tamura K. MEGA7: Molecular Evolutionary Genetics Analysis Version 7.0 for Bigger Datasets[J]. Mol Biol Evol, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [12] |
Scott MK, Chommanard C, Lu X, et al. Human Adenovirus Associated with Severe Respiratory Infection, Oregon, USA, 2013-2014[J]. Emerg Infect Dis, 2016, 22(6): 1044-1051. DOI:10.3201/eid2206.151898 |
| [13] |
Arnberg N. Adenovirus receptors: implications for targeting of viral vectors[J]. Trends Pharmacol Sci, 2012, 33(8): 442-448. DOI:10.1016/j.tips.2012.04.005 |
| [14] |
de Graaf JF, de Vor L, Fouchier RAM, et al. Armed oncolytic viruses: A kick-start for anti-tumor immunity[J]. Cytokine Growth Factor Rev, 2018, 41: 28-39. DOI:10.1016/j.cytogfr.2018.03.006 |
| [15] |
Nguyen TV, Crosby CM, Heller GJ, et al. Oncolytic adenovirus Ad657 for systemic virotherapy against prostate cancer[J]. Oncolytic Virother, 2018, 7: 43-51. DOI:10.2147/OV.S155946 |
 2021, Vol. 48
2021, Vol. 48