文章信息
- 各基因亚型乳腺癌间基因组拷贝数变异的比较
- Comparison of Genomic Copy Number Variations Among Breast Cancer Subtypes
- 肿瘤防治研究, 2021, 48(4): 341-346
- Cancer Research on Prevention and Treatment, 2021, 48(4): 341-346
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2021.20.0921
- 收稿日期: 2020-08-03
- 修回日期: 2020-11-17
2. 529000 江门,江门市妇幼保健院药学部;
3. 529030 江门,江门市中心医院中心实验室;
4. 521021 潮州,广东省人乳头状瘤病毒(HPV)相关疾病分子诊断工程技术研究开发中心
2. Department of Pharmacy, Jiangmen Maternity and Child Health Care Hospital, Jiangmen 529000, China;
3. Clinical Experimental Center, Jiangmen Central Hospital, Jiangmen 529030, China;
4. Research and Development Center for Molecular Diagnosis Engineering Technology of Human Papillomavirus Related Diseases of Guangdong Province, Chaozhou 521021, China
乳腺癌是最常见的女性恶性肿瘤,近四十年间,全球乳腺癌发病率保持上升趋势,严重威胁着女性健康[1-2]。乳腺癌是生物学高度异质的恶性肿瘤,临床最为常见且恶性程度较高的是浸润性乳腺癌,根据组织形态进一步分为浸润性导管癌(invasive ductal carcinoma, IDC)和浸润性小叶癌(invasive lobular carcinoma, ILC)等,并以IDC为临床的主要病理类型[3]。然而,同样的病理类型在临床生物学特征及治疗反应性上常常截然不同,这是因为特征基因的突变与异常表达往往更能反映乳腺癌的治疗反应及患者预后[4],随着高通量基因检测技术的成熟应用,乳腺癌基因特征与临床特征的关系逐渐清晰,由此建立病理基因分型逐步被临床所接受并广泛应用。
乳腺癌基因分型的方法有多种,主要通过基因的表达谱算法和免疫组织化学判读等,虽然不同的分型方法在具体样品的判断中存在差异,但群体的分类重复性上基本相似[5]。临床上公认的四个基因亚型为管腔A型(Luminal A, LumA)、管腔B型(Luminal B, LumB)、人表皮生长因子受体2过表达型(HER2 overexpression, Her2)和基底细胞样型(Basal-like, BasL),其中BasL型基本等同于免疫组织化学分型中的三阴乳腺癌(triple negative breast carcinoma, TNBC)[6]。现已知Her2型乳腺癌的特征是人表皮生长因子受体2(human epidermal growth factor receptor 2, HER2)的过表达及下游通路的持续激活,而其编码基因ERBB2 (erb-B2 receptor tyrosine kinase 2)的扩增是HER2蛋白过表达的主要原因[4],迄今未见系统性报道其余三个基因亚型的基因组拷贝数变异(copy number variations, CNV)特征,因此,本课题组通过全基因组CNV分析,在癌症基因组图谱数据库(The Cancer Genome Atlas, TCGA, https://gdc-portal.nci.nih.gov/)中挖掘出与基因分型相关的CNV并加以验证。
1 资料与方法 1.1 TCGA乳腺癌的AIMS分型及全基因组CNV分析下载TCGA中乳腺癌的表达谱测序数据、基因组拷贝信息及对应样品的临床信息,数据库的更新下载日期为2020年1月31日。参考本课题组已发表文献[5],在R语言(版本3.2.2)程序平台上,使用AIMS程序包(版本1.2.0)对每个样品的表达谱进行基因分型,纳入LumA、LumB、Her2和BasL四个基因亚型及临床信息详细的1 006例乳腺癌病例。参考本课题组已发表文献[7],利用GISTIC 2.0软件分析肿瘤组织的全基因组拷贝信息,运行所得的关键数据有:(1)每个探针标记位点的扩增或缺失情况,即G评分(G-score)。G评分的高低能综合反映该位点的扩增或缺失的深度和频率;(2)每个样品中单个基因的拷贝数变异情况,包括深度缺失(Deep deletion)、浅度缺失(Shallow deletion)、二倍体(Diploid)、第三拷贝获得(Gain)和显著扩增(Amplification)五个状况;其中在分析缺失中,将深度缺失及浅度缺失均纳入为缺失,而在扩增分析中,仅将显著扩增定义为扩增,并在R4.0.2软件中对各基因在不同基因亚型的拷贝数分布进行卡方检验(Chi-square test函数),并采用Benjamin-Hochberg方法(p.adjust函数)进行P值校正,得到Q值。
1.2 患者一般信息收集整理2017年1月—2019年12月在江门市中心医院就诊的浸润性乳腺癌324例,均为原发灶的手术或活检检查后的剩余标本,取样前均未行任何新辅助治疗,保存在该院临床生物资源库中。TCGA与江门市中心医院乳腺癌患者的一般信息见表 1,两组样品在性别、年龄、病理类型、临床分期和基因分型的分布差异均无统计学意义(均P > 0.05)。本研究为回顾性研究,已申请并获批免患者知情同意,且整个项目经江门市中心医院伦理委员会审查并批准(2020-51号)。
|
参考本课题组已发表文献[7],调取石蜡组织切片,利用GeneRead DNA FFPE Kit按操作说明书提取石蜡切片的DNA;利用TaqMan Copy Number Assay的荧光定量PCR引物Hs00450668_cn检测ERBB2基因拷贝数,引物Hs03853983_cn检测TFDP1基因拷贝数,引物Hs06966674_cn检测MIR148B基因拷贝数,引物Hs01978234_cn检测CCND1基因拷贝数,引物Hs00738157_cn检测MDM2基因拷贝数,引物Hs06266734_cn检测MIR139基因拷贝数,其中内参引物为TaqMan Copy Number Reference Assay RNase P,二倍体参考样品为健康人血浆白膜层样品,扩增试剂盒为TaqMan Fast Advanced Master Mix,按说明书在CFX96荧光定量PCR仪中检测。规定样品中目标基因的相对拷贝数检测值小于0.75为缺失(Deletion),大于1.9为显著扩增(Amplification)。
1.4 统计学方法计数资料用频数表示,用R4.0.2、Excel 2016及GraphPad Prism7.0软件处理数据,各组间频数数据统计使用卡方检验(Chi-squared test)或Fisher's精确检验(Fisher's exact test),检验水准α=0.05,组间频数的两两比较使用rcompanion程序包的pairwiseNominalIndependence函数,并采用函数内默认的Benjamin-Hochberg方法进行P值校正,得到Q值。
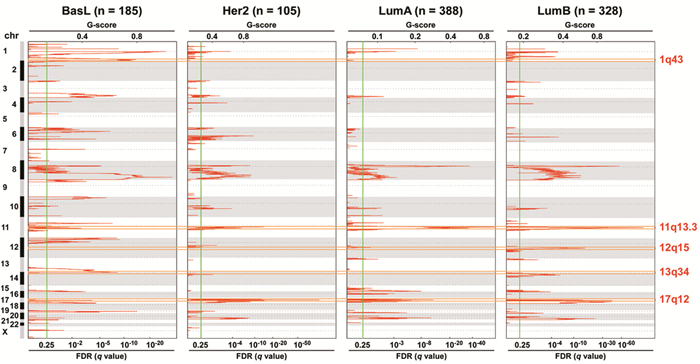
2 结果 2.1 TCGA中各亚型的扩增区段分析除BasL型外,另外3种亚型乳腺癌的扩增峰较为聚集,且热点扩增峰的G评分较高,可达0.8以上。以软件默认的FDR < 0.25为显著性扩增的阈值,可发现BasL型的特征峰有1q43和13q34,LumB型的特征峰为12q15,Her2型的特征峰为17q12;而11q13.3在Her2型、LumA型及LumB型中扩增,在BasL型中无扩增,见图 1。
|
| BasL: basl-like type; Her2: HER2 overexpression type; LumA: luminal A type; LumB: luminal B type; Chr: chromosome; FDR: false discovery rates. 图 1 TCGA中各亚型的扩增区段分析 Figure 1 Amplified chromosome regions of each subtype from TCGA |
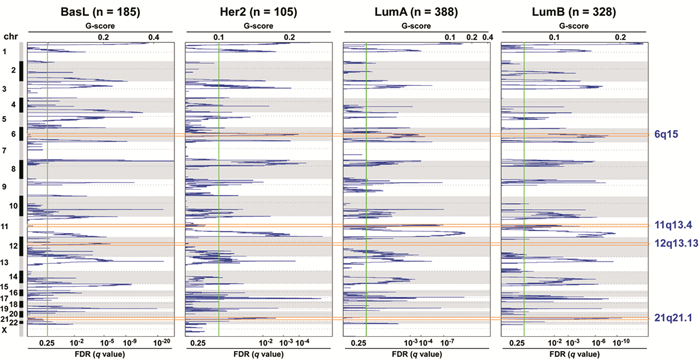
相对于全基因组的扩增情况,各亚型缺失区段缺少明显的规律,且G评分也普遍较低,在0.2左右。6q15在Her2型、LumA型及LumB型中缺失,11q13.4特异性在LumB型中缺失,12q13.13特异性在BasL型中缺失,21q21.1在Her2型和LumB型中缺失,见图 2。
|
| 图 2 TCGA中各亚型的缺失区段分析 Figure 2 Deleted chromosome regions of each subtype from TCGA |
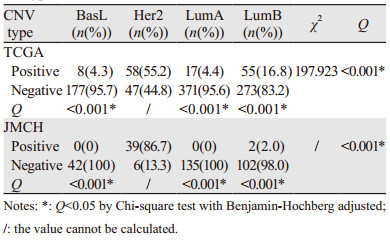
利用IGV软件并结合相关文献报道(见讨论部分),确定各区段的扩增或缺失关键基因:1q43为丝氨酸/苏氨酸蛋白激酶Akt-3(AKT serine/threonine kinase 3, Akt3)、11q13.3为细胞周期蛋白D1(Cyclin D1, CCND1)、12q15为原癌基因MDM2(MDM2 proto-oncogene, MDM2)、13q34为转录因子Dp-1(transcription factor Dp-1, TFDP1)、17q12为Erb-B2受体酪氨酸激酶2(Erb-B2 receptor tyrosine kinase 2, ERBB2)、6q15为丝裂原活化蛋白激酶7(mitogen-activated protein kinase kinase kinase 7, MAP3K7)、11q13.4为微小RNA-139(microRNA 139, MIR139)、12q13.13为微小RNA-148B(microRNA 148B, MIR148B)及21q21.1为微小RNA Let-7c(microRNA Let-7c, MIRLET7C)。上述热点基因的CNV在四种亚型的分布差异均有统计学意义(均P < 0.05),其中,在BasL型中,TFDP1(13q34)的扩增和MIR148B(12q13.13)的缺失占比高;在Her2型中,ERBB2(17q12)的扩增是最显著的特征;LumB型中,CCND1(11q13.3)和MDM2(12q15)的扩增及MIR139(11q13.4)的缺失占比高;而未见LumA型的特异性CNV热点基因,见表 2。
将ERBB2的扩增定义为阳性事件,比较发现,在TCGA中Her2型阳性事件发生率为55.2%,BasL型和LumA型的阳性率均低于或等于4.4%,而LumB型较高,可达16.8%;在江门市中心医院样品中,Her2型阳性事件发生率为86.7%,BasL型和LumA型的阳性率均为0,而LumB型为2.0%,组间分布差异均有统计学意义(均P < 0.05),见表 3。
将TFDP1的扩增或MIR148B的缺失定义为阳性事件,分析发现57.8%的BasL型发生了TFDP1的扩增或MIR148B的缺失,而其他亚型的阳性事件发生率均低于或等于15.2%,分布差异均有统计学意义(均P < 0.05)。江门市中心医院样品中71.4%的BasL型发生了TFDP1的扩增或MIR148B的缺失,而其他亚型的阳性事件发生率均低于或等于19.5%,组间分布均差异有统计学意义(均P < 0.05),见表 4。
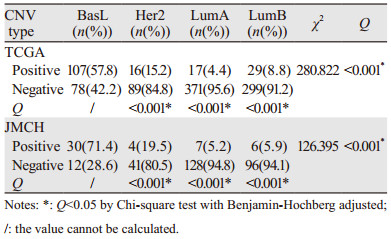
|
将CCND1或MDM2的扩增或MIR139的缺失定义为阳性事件,分析发现,47.6%的LumB型发生了CCND1或MDM2的扩增或MIR139的缺失,而其他亚型的阳性事件发生率均低于35%,分布差异均有统计学意义(P < 0.05)。江门市中心医院样品中61.8%的LumB型发生了CCND1或MDM2的扩增或MIR139的缺失,而其他亚型乳腺癌的阳性事件发生率均低于或等于20.0%,组间分布差异有统计学意义(P < 0.05),见表 5。
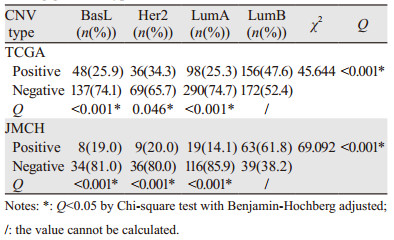
|
本研究首先利用AIMS分型软件,将TCGA数据库中的乳腺癌患者按表达谱进行基因分型,再利用GISTIC2.0软件分别对BasL型、Her2型、LumA型和LumB型患者的肿瘤组织进行全基因组CNV分析,发现:(1)相对于其他三种亚型,BasL型的扩增峰较为杂乱,表明BasL型乳腺癌的发生过程中维持基因组稳定及参与修复的功能受损,这与BasL型有高频率的肿瘤相关蛋白53(tumor protein 53, TP53)和乳腺癌易感基因1(breast cancer 1, BRCA1)突变相一致[4];(2)相对于全基因组的扩增情况,各亚型缺失区段缺少明显的规律,且具体缺失区段的程度和频率均较低,表明在各亚型中抑癌基因的功能失活只有小部分与缺失相关,这与同行的研究[8]结论相似;(3)BasL型中有高比例的13q34扩增和12q13.13缺失,Her2型有高比例的17q12扩增,LumB型的特征为高比例的12q15扩增和11q13.4缺失,而LumA型则无特异性扩增或缺失的染色体区段,这部分发现值得进一步讨论。
位于17q12区段的关键癌基因ERBB2的扩增是Her2型乳腺癌的重要分子特征,江门市中心医院样品结果显示,ERBB2(17q12)在Her2型乳腺癌中显著扩增,虽然与其他亚型间的分布比例有显著性差异,但无法做到100%的区分,其原因主要是:(1)相对于临床常用的免疫组织化学及荧光原位杂交法[9],本研究采用的qPCR法和TCGA的高通量分析均无法完全排除肿瘤组织中的原位癌、坏死及间质成分;(2)各种方法中,对于临界值的争议依然存在,无法完全做到客观分析。不过,相比于TCGA,江门市中心医院样品的验证过程中,尽可能采用显微切割的方法,剔除了大部分非目标区域组织,其临床一致性较高,可以作为TCGA分析结果的验证支持数据。
随后,用TFDP1(13q34)的扩增或MIR148B (12q13.13)的缺失定义为BasL型乳腺癌的特征分子事件,通过对TCGA的分析及江门市中心医院样品的验证,可知该事件在BasL型的阳性率比其他各亚型高3.6~7.3倍,且差异具统计学意义,表明BasL型乳腺癌中有高比例的TFDP1/MIR148B拷贝数变异。已有研究报道,BasL型乳腺癌或TNBC存在较高比例的13q34区段扩增[10],且该区段的扩增能导致TFDP1蛋白水平的上调,而TFDP1为细胞周期启动的关键转录因子家族E2Fs的共激活因子,体外实验证明,沉默TFDP1能有效抑制生长因子驱动的乳腺癌细胞增殖[11],这表明TFDP1很可能参与了BasL型乳腺癌的恶性生物学行为,是潜在的特异性靶点。同样,值得我们注意的是,有报道指出miR-148b具有抑制多种恶性肿瘤的转移能力,过表达miR-148b能抑制乳腺癌细胞浸润破坏血管内皮层,抑制乳腺癌等多种恶性肿瘤的体内转移[12-13],这提示MIR148B的缺失可能与BasL型乳腺癌部分患者的高转移临床表型相关,值得进一步挖掘其临床转化价值。
在四个分子分型中,LumA型和LumB型的区分标准还存在一定的争议,我们利用全基因组CNV分型也未能发现LumA型中较典型的扩增或缺失峰,只观察到CCND1(11q13.3)和MDM2 (12q15)的扩增及MIR139(11q13.4)的缺失在LumB型中的比例更高,其中,TCGA的LumA型和LumB型发生CCND1/MDM2/MIR139拷贝数变异情况的比例分别是25%和48%,江门市中心医院样品的比例分别是14%和62%,差异均有统计学意义。虽然未见MIR139在乳腺癌组织中缺失的报道,但已有大量文献报道,CCND1和MDM2在LumB型乳腺癌中的显著扩增,其中CCND1的扩增是细胞周期素依赖性激酶4/6(cyclin-dependent kinase 4/6, CDK4/6)抑制剂帕博西尼(Palbociclib)的伴随诊断用药指标[14],而MDM2的扩增能通过抑制p53蛋白的功能促进乳腺癌化疗抵抗,是潜在的治疗靶点[15]。虽然本研究挖掘到了较新颖的乳腺癌分子分型标志物,但具体的临床价值还需要进一步加以验证。
作者贡献:
王智辉:收集整理分析数据、撰写论文
钟媚共:生物信息学分析、撰写论文
陈秋旋、孟子杰:组织切片和核酸提取
伍婉婷、郑焱:PCR操作及数据分析
张鑫:制定研究方案、审核论文
| [1] |
Siegel RL, Miller KD, Jemal A. Cancer statistics, 2019[J]. CA Cancer J Clin, 2019, 69(1): 7-34. DOI:10.3322/caac.21551 |
| [2] |
陈万青, 郑荣寿, 张思维, 等. 2013年中国恶性肿瘤发病和死亡分析[J]. 中国肿瘤, 2017, 26(1): 1-7. [Chen WQ, Zheng RS, Zhang SW, et al. Report of Cancer Incidene and Mortality in China 2013[J]. Zhongguo Zhong Liu, 2017, 26(1): 1-7.] |
| [3] |
刘青, 张英, 周馨, 等. 2009—2018年北京地区单中心乳腺癌临床流行病学及病理特征回顾性分析[J]. 肿瘤, 2020, 40(6): 431-439. [Liu Q, Zhang Y, Zhou X, et al. A single center, retrospective analysis on clinical epidemiology and pathologic characteristics of breast cancer in Beijing from 2009 to 2018[J]. Zhong Liu, 2020, 40(6): 431-439.] |
| [4] |
赵翠翠, 刘红. 年轻乳腺癌的分子生物学发病机制[J]. 肿瘤防治研究, 2020, 47(3): 213-217. [Zhao CC, Liu H. Molecular Biological Pathogenesis of Young Breast Cancer[J]. Zhong Liu Fang Zhi Yan Jiu, 2020, 47(3): 213-217. DOI:10.3971/j.issn.1000-8578.2020.19.1130] |
| [5] |
陈美慧, 钟媚共, 蒋旭, 等. 乳腺癌基因分型与患者临床病理及预后的关系[J]. 肿瘤防治研究, 2018, 45(10): 752-757. [Chen MH, Zhong MG, Jiang X, et al. Correlation of Genotypes with Clinicopathological and Prognostic Characteristies of Breast Cancer Patients[J]. Zhong Liu Fang Zhi Yan Jiu, 2018, 45(10): 752-757. DOI:10.3971/j.issn.1000-8578.2018.17.1658] |
| [6] |
Herrera Juarez M, Tolosa Ortega P, Sanchez de Torre A, et al. Biology of the Triple-Negative Breast Cancer: Immunohistochemical, RNA, and DNA Features[J]. Breast Care (Basel), 2020, 15(3): 208-216. DOI:10.1159/000508758 |
| [7] |
Chen J, Liu A, Lin Z, et al. Downregulation of the Circadian Rhythm Regulator HLF Promotes Multiple-Organ Distant Metastases in Non-Small Cell Lung Cancer through PPAR/NF-kappaB Signaling[J]. Cancer Lett, 2020, 482: 56-71. DOI:10.1016/j.canlet.2020.04.007 |
| [8] |
Zhang G, Wang Y, Chen B, et al. Characterization of frequently mutated cancer genes in Chinese breast tumors: a comparison of Chinese and TCGA cohorts[J]. Ann Transl Med, 2019, 7(8): 179. DOI:10.21037/atm.2019.04.23 |
| [9] |
乳腺癌HER2检测指南编写组. 乳腺癌HER2检测指南(2019版)[J]. 中华病理学杂志, 2019, 48(3): 169-175. [Breast Cancer Expert Panel. Guideline for HER2 detection in breast cancer, the 2019 version[J]. Zhonghua Bing Li Xue Za Zhi, 2019, 48(3): 169-175. DOI:10.3760/cma.j.issn.0529-5807.2019.03.001] |
| [10] |
Saucedo-Cuevas LP, Ruppen I, Ximenez-Embun P, et al. CUL4A contributes to the biology of basal-like breast tumors through modulation of cell growth and antitumor immune response[J]. Oncotarget, 2014, 5(8): 2330-2343. DOI:10.18632/oncotarget.1915 |
| [11] |
Reimer D, Sadr S, Wiedemair A, et al. Heterogeneous cross-talk of E2F family members is crucially involved in growth modulatory effects of interferon-gamma and EGF[J]. Cancer Biol Ther, 2006, 5(7): 771-776. DOI:10.4161/cbt.5.7.2750 |
| [12] |
Orso F, Quirico L, Virga F, et al. miR-214 and miR-148b Targeting Inhibits Dissemination of Melanoma and Breast Cancer[J]. Cancer Res, 2016, 76(17): 5151-5162. DOI:10.1158/0008-5472.CAN-15-1322 |
| [13] |
Jiang Z, Zhang J, Chen F, et al. MiR-148b suppressed non-small cell lung cancer progression via inhibiting ALCAM through the NF-kappaB signaling pathway[J]. Thorac Cancer, 2020, 11(2): 415-425. DOI:10.1111/1759-7714.13285 |
| [14] |
Li Z, Cui J, Yu Q, et al. Evaluation of CCND1 amplification and CyclinD1 expression: diffuse and strong staining of CyclinD1 could have same predictive roles as CCND1 amplification in ER positive breast cancers[J]. Am J Transl Res, 2016, 8(1): 142-153. |
| [15] |
Colaluca IN, Basile A, Freiburger L, et al. A Numb-Mdm2 fuzzy complex reveals an isoform-specific involvement of Numb in breast cancer[J]. J Cell Biol, 2018, 217(2): 745-762. DOI:10.1083/jcb.201709092 |
 2021, Vol. 48
2021, Vol. 48