文章信息
- 非小细胞肺癌患者外周血和肺癌组织中ZIC1启动子甲基化检测的临床意义
- Clinical Significance of Detection of ZIC1 Promoter Methylation in Peripheral Blood and Cancer Tissues of Non-small Cell Lung Cancer Patients
- 肿瘤防治研究, 2021, 48(4): 354-357
- Cancer Research on Prevention and Treatment, 2021, 48(4): 354-357
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2021.20.0911
- 收稿日期: 2020-07-29
- 修回日期: 2021-02-03
2. 430033 武汉,湖北省第三人民医院病理科
2. Department of Pathology, The Third People's Hospital of Hubei Province, Wuhan 430033, China
肺癌是全世界最常见的死亡原因之一,非小细胞肺癌(non-small cell lung cancer, NSCLC)是肺癌的主要亚型之一,约占80%~85%,具有发病率高、死亡率高、预后差等特点[1]。基因甲基化在癌症中普遍存在[2],基因的甲基化异常能有效预测癌症的发生,并可作为预后评估的标志物[3-5]。自Aruga等[6]发现了新基因ZIC(zinc finger of thecerebellum),ZIC家族的其他基因逐一被发现,并命名为ZIC2~ZIC5,ZIC1基因定位于人类染色体3q24区域,为重要的锌指结构转录因子。笪晨星等[7]研究表明,ZIC1基因启动子区甲基化和肝癌相关,认为ZIC1基因启动子区甲基化导致ZIC1基因不表达,因此促进肝癌的发生和发展。最近研究[8]发现,构建甲基化敏感位点对非小细胞肺癌早期检测具有重大意义,其中包括ZIC1,可作为早期检测的生物标志物。为了进一步探讨ZIC1甲基化与非小细胞肺癌的关系,本文用甲基化特异性聚合酶链反应(methylation-specific polymerase chain reaction, MSP)的方法比较NSCLC患者和健康人群外周血中的ZIC1基因甲基化状态,并比较NSCLC患者肺癌和癌旁组织中的ZIC1甲基化状态,以期为ZIC1基因甲基化作为非小细胞肺癌临床诊断标志物提供一定的数据资料。
1 资料和方法 1.1 临床对象和样本收集选取本院2016年3月—2018年7月的95例NSCLC患者,其中男58例,女37例,平均年龄为(59.5±4.5)岁,有吸烟史61例,KPS评分为(90.1±2.8)分。手术过程中取肺癌组织和离癌组织3 cm以上的癌旁组织,迅速置于液氮中速冻。纳入标准:(1)通过症状体征和肺部增强CT以及病理组织学检查确诊为NSCLC;(2)无肺部其他感染性疾病,无合并其他肿瘤;(3)功能状态KPS评分 > 70分;(4)原发肿瘤发生于单侧肺,未进行过手术、放疗、化疗等治疗手段。另外选择健康人群为对照组,95例正常外周血来自于本院体检人群,男性58例,女性37例;平均年龄为(54.8±3.4)岁,有吸烟史55例,KPS评分为(90.7±2.6)分。NSCLC组和对照组在性别、年龄、吸烟史、KPS评分上差异均无统计学意义(P > 0.05)。本研究经伦理委员会批准且所有参与实验的患者和健康人群均签署知情同意书。
1.2 检测方法 1.2.1 DNA提取取200 mg组织,冰上研磨成悬液,离心后取上清液。取两组患者的血清标本200 μl和上述组织上清液,按照EasyPure基因组DNA试剂盒(购于北京全式金生物技术有限公司,编号:EE101-02)说明和步骤,分别提取NSCLC患者的肺癌组织、癌旁组织、外周血和健康者外周血中的DNA,并采用紫外分光光度计检测提取的DNA含量和纯度。
1.2.2 亚硫酸氢钠处理取纯化的DNA样品20 μl,以DNA重亚硫酸盐转化试剂盒(购于德国Qiagen公司)对组织和血清样本DNA进行亚硫酸盐修饰。将DNA置于95℃水浴中加热20 min,再冰浴20 min,然后置于42℃浓度为3 mol/L的NaOH溶液中温浴20 min。加入NaHSO3处理液,50℃恒温16 h。添加1 ml的37℃树脂,将混匀后的液体真空抽滤,以80%的异丙醇洗涤,析出DNA,以NaOH、冰乙醇、醋酸铰沉淀后,以80%的乙醇洗涤,低温保存备用。
1.2.3 MSP实验引物由上海生工生物工程设计并合成。ZIC1甲基化引物正义:5'-TAAAGATAGGTTAATCGAGGGTC-3';反义:5'-GAACGCGATCTTTATTACCGAC-3'。非甲基化引物正义:5'-GTAAAGATAGGTTAATTGAGGGTT-3';反义:5'-AAAAACAAACACAATCTTTATTACCAAC-3'。反应体系25 μl:其中Bisulfite修饰后基因组DNA模板4 μl,10×缓冲液2.5 μl,上游和下游引物各1 μl,dNTP各Mix 2 μl、MgCl2 3 μl、TaqTMDNA 0.5 μl,去离子水11 μl。试剂盒由KAPA Biosystems公司提供,甲基化的反应条件为:95℃预变性10 min,95℃变性60 s,62℃退火,72℃延伸40 s,35个循环,最后一个循环72℃延伸8 min。非甲基化的反应条件为:95℃预变性10 min,95℃变性60 s,58℃退火,72℃延伸40 s,35个循环,最后一个循环72℃延伸8 min。MSP产物经2%琼脂糖凝胶电泳,GelRed染色,用紫外检测仪Tanon 3500凝胶图像分析系统观察检测结果。根据结果计算其敏感度和特异性:敏感度=真阳性例数/(真阳性例数+假阴性例数)×100%;特异性=真阴性例数/(真阴性例数+假阳性例数)×100%。
1.3 统计学方法采用SPSS20.0进行统计学分析。NSCLC患者ZIC1基因甲基化和临床病理因素的相关性采用卡方检验,NSCLC组和对照组ZIC1甲基化检出率采用t检验进行分析。P < 0.05为差异具有统计学意义。
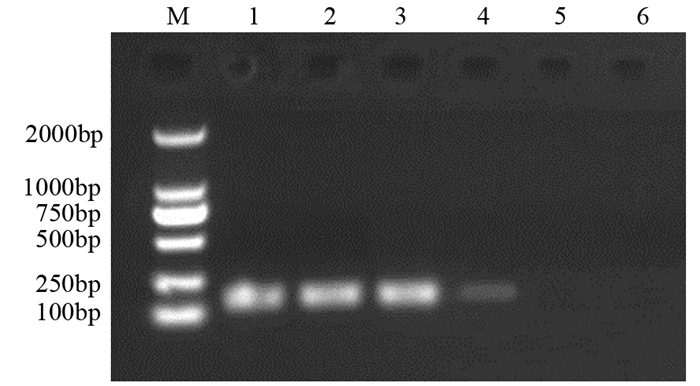
2 结果 2.1 ZIC1基因在NSCLC组患者和健康人群外周血中的甲基化状况95例NSCLC组患者的外周血中,67例(70.52%)检出ZIC1甲基化,而95例健康人群的外周血中,仅33例(34.73%)检测出ZIC1甲基化,差异有统计学意义(χ2=24.404, P=0.000);而在肿瘤组织和癌旁组织中ZIC1甲基化的检出率分别为83.15%和3.15%,差异亦有统计学意义(χ2=123.921, P=0.000)。综上,ZIC1甲基化检测外周血和肿瘤组织的敏感度分别为70.52%和83.15%(P=0.000),特异性分别为65.27%和96.85%(P=0.000)。ZIC1甲基化的电泳图见图 1。
|
| M: the marker of DL2000; 1, 2, 3: the amplification results of ZIC1 gene target fragment in NSCLC group; 4, 5, 6: the amplification results of ZIC1 gene target fragment in control group. 图 1 ZIC1基因目的片段PCR扩增产物电泳图 Figure 1 Electrophoretogram of PCR amplified products of ZIC1 gene target fragment |
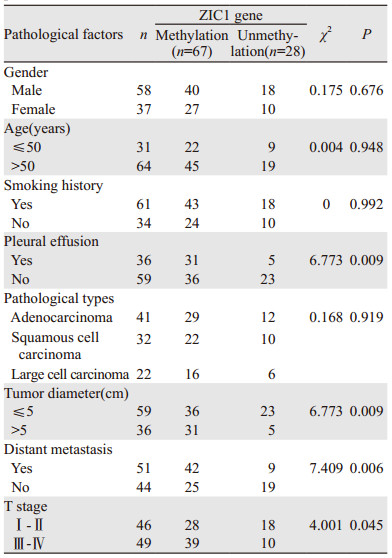
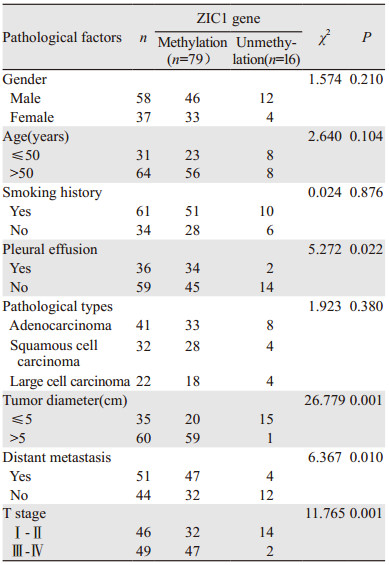
NSCLC组患者外周血及肿瘤组织ZIC1基因甲基化与患者性别、病理类型、年龄、吸烟史无显著相关性(均P > 0.05),而与肿瘤大小、远处转移情况、T分期、有无胸腔积液具有相关性(均P < 0.05),见表 1~2。
|
|
随着基因研究的不断深入,表观遗传在肿瘤发生过程中的重要作用日益凸显,DNA的甲基化是肿瘤发生机制中的表观遗传形式之一[9]。CpG岛是基因中明显甲基化的区域,主要位于基因的启动子和外显子区域,常见于哺乳动物细胞中[10]。5'甲基化的mCpG结构在结合甲基结合蛋白后,能够阻止启动子和转录因子的结合过程,导致基因低表达甚至不表达,异常的甲基化可能导致基因表达异常或癌症的发生[11]。在非小细胞肺癌中发现ZIC1正是CpG甲基化敏感位点之一[8]。
ZIC1参与调节细胞的生长与凋亡,是一种潜在的预后生物标志物[12-13]。研究发现,ZIC1在胃癌被表观遗传沉默,ZIC1启动子甲基化被作为胃癌早期诊断和检测的新分子标记[14]。Lin等[15]的研究也发现ZIC1的甲基化率在胃癌发生过程中显著增加,联合检测ZIC1和HOXD10、RUNX3启动子甲基化可能是早期检测胃癌的一种新策略。ZIC1在乳腺恶性肿瘤患者中发挥抑癌作用,可抑制癌细胞增殖、诱导凋亡、抑制肿瘤生长[16]。在本研究中,健康人群的外周血ZIC1基因甲基化检出率显著低于NSCLC组,肿瘤组织ZIC1基因甲基化检出率显著高于癌旁组织,且ZIC1基因甲基化和肿瘤大小、远处转移情况、T分期、胸腔积液等具有显著相关性。以上检测结果提示ZIC1甲基化率在NSCLC发生发展过程中升高,ZIC1甲基化在肿瘤组织中诊断NSCLC的敏感度和特异度均高于外周血,且具有预后评估意义。
和传统的肿瘤标志物检测比较,基因甲基化的预后具有更高的敏感度和特异性。近年来,以DNA甲基化修饰为代表的表观遗传学在肿瘤临床中的应用价值越来越凸显,在肺癌早期诊断、治疗、复发以及预后等方面均有体现[17]。结合本研究结果,在NSCLC患者外周血及肿瘤组织中,ZIC1基因甲基化与肿瘤的发生发展、转移、分期等相关,且ZIC1基因甲基化检测在NSCLC组织中的诊断敏感度和特异性高于外周血。后续我们将通过基础实验来验证ZIC1表达对NSCLC发生发展的影响,以期能进一步了解ZIC1在NSCLC中的可能作用机制。
作者贡献:
徐瑶:执行实验,撰写文章
刘世国:主导研究思路
张畅、张军霞、彭黎:整理分析数据
张黎:提供病理阅片资料
姜姗:病理技术操作
| [1] |
Zeng FY, Yu N, Han YY, et al. The long non-coding RNA MIAT/miR-139-5p/MMP2 axis regulates cell migration and invasion in non-small-cell lung cancer[J]. J Biosci, 2020, 45: 51. DOI:10.1007/s12038-020-0019-8 |
| [2] |
Nanavaty V, Abrash EW, Hong C, et al. DNA Methylation Regulates Alternative Polyadenylation via CTCF and the Cohesin Complex[J]. Mol Cell, 2020, 78(4): 752-764.e6. DOI:10.1016/j.molcel.2020.03.024 |
| [3] |
Shou F, Xu F, Li G, et al. RASSF1A promoter methylation is associated with increased risk of thyroid cancer: a meta-analysis[J]. Onco Targets Ther, 2017, 10: 247-257. DOI:10.2147/OTT.S124417 |
| [4] |
Hao X, Luo H, Krawczyk M, et al. DNA methylation markers for diagnosis and prognosis of common cancers[J]. Proc Natl Acad Sci U S A, 2017, 114(28): 7414-7419. DOI:10.1073/pnas.1703577114 |
| [5] |
Nian J, Sun X, Ming SY, et al. Diagnostic accuracy of methylated SEPT9 for blood-based colorectal cancer detection: A systematic review and Meta-analysis[J]. Clin Transl Gastroenterol, 2017, 8(1): e216. DOI:10.1038/ctg.2016.66 |
| [6] |
Aruga J, Yokota N, Hashimoto M, et al. A novel zinc finger protein, zic, is involved in neurogenesis, especially in the cell lineage of cerebellar granule cells[J]. J Neurochem, 1994, 63(5): 1880-1890. |
| [7] |
笪晨星, 顾勇, 段炜. 肝细胞癌ZIC1基因启动子区甲基化与临床病理特征的关系[J]. 中国肿瘤生物治疗杂志, 2016, 23(3): 416-418. [Da CX, Gu Y, Duan W. Relationship between promoter methylation of the ZIC1 gene and clinical pathological characteristics of the patients with hepatocellular carcinoma[J]. Zhongguo Zhong Liu Sheng Wu Zhi Liao Za Zhi, 2016, 23(3): 416-418.] |
| [8] |
周嘉梁, 卫海民, 吴佳, 等. 全基因组测序筛选非小细胞肺癌敏感甲基化位点的肺癌预警体系的构建[J]. 中华实验外科杂志, 2020, 37(1): 155-157. [Zhou JL, Wei HM, Wu J, et al. Construction of warning system for lung cancer by whole genome sequencing screening for sensitive methylation sites of non-small-cell lung carcinoma[J]. Zhonghua Shi Yan Wai Ke Za Zhi, 2020, 37(1): 155-157. DOI:10.3760/cma.j.issn.1001-9030.2020.01.045] |
| [9] |
薛伍君, 陈清江, 张旭东. DNA甲基化相关肿瘤标志物临床研究进展[J]. 实用肿瘤杂志, 2020, 35(2): 187-192. [Xue WJ, Chen QJ, Zhang XD. Clinical research progress of DNA methylation related tumor markers[J]. Shi Yong Zhong Liu Za Zhi, 2020, 35(2): 187-192.] |
| [10] |
Yang YJ, Dong HL, Qiang XW, et al. Cytosine methylation enhances DNA condensation revealed by equilibrium measurements using magnetic tweezers[J]. J Am Chem Soc, 2020, 142(20): 9203-9209. DOI:10.1021/jacs.9b11957 |
| [11] |
Iida T, Iwanami A, Sanosaka T, et al. Whole-genome DNA methylation analyses revealed epigenetic instability in tumorigenic human iPS cell-derived neural stem/progenitor cells[J]. Stem Cells, 2017, 35(5): 1316-1327. DOI:10.1002/stem.2581 |
| [12] |
Ge QW, Hu YY, He JM, et al. Zic1 suppresses gastric cancer metastasis by regulating Wnt/β-catenin signaling and epithelial‐mesenchymal transition[J]. FASEB J, 2020, 34(2): 2161-2172. DOI:10.1096/fj.201901372RR |
| [13] |
Gu X, Guo XK, Chen BH, et al. Prognostic and clinicopathological value of ZIC1 in patients with cervical squamous cell carcinoma[J]. Oncol Lett, 2019, 18(6): 6621-6627. |
| [14] |
Chen X, Lin Z, Xue M, et al. Zic1 promoter hypermethylation in plasma DNA is a potential biomarker for gastric cancer and intraepithelial neoplasia[J]. PLoS One, 2015, 10(7): e0133906. DOI:10.1371/journal.pone.0133906 |
| [15] |
Lin ZH, Lou MZ, Chen XQ, et al. Combined detection of plasma ZIC1, HOXD10 and RUNX3 methylation is a promising strategy for early detection of gastric cancer and precancerous lesions[J]. J Cancer, 2017, 8(6): 1038-1044. DOI:10.7150/jca.18169 |
| [16] |
Han W, Cao F, Gao XJ, et al. ZIC1 acts a tumor suppressor in breast cancer by targeting survivin[J]. Int J Oncol, 2018, 53(3): 937-948. |
| [17] |
李国术, 谢栓栓, 王昌惠. DNA甲基化修饰在肺癌研究中的进展[J]. 国际呼吸杂志, 2018, 38(22): 1734-1738. [Li GS, Xie SS, Wang CH. Research advances of DNA methylation in lung cancer[J]. Guo Ji Hu Xi Za Zhi, 2018, 38(22): 1734-1738. DOI:10.3760/cma.j.issn.1673-436X.2018.22.012] |
 2021, Vol. 48
2021, Vol. 48


