文章信息
- 食管鳞癌中LncRNA DLEU1的表达及对食管鳞癌细胞增殖和迁移的影响
- Expression of LncRNA DLEU1 in Esophageal Squamous Cell Carcinoma and Its Effect on Proliferation and Migration of ESCC Cells
- 肿瘤防治研究, 2021, 48(3): 255-260
- Cancer Research on Prevention and Treatment, 2021, 48(3): 255-260
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2021.20.0749
- 收稿日期: 2020-06-30
- 修回日期: 2020-10-19
2. 050011 石家庄,河北省肿瘤研究所
2. Hebei Cancer Institute, Shijiazhuang 050011, China
随着基因组学研究的进展,发现90%以上的人类基因组为非编码RNA(non-coding RNA, ncRNAs),其中大部分以长链非编码RNA(long non-coding RNA, LncRNA)的形式存在,长度超过200个核苷酸[1]。LncRNA可在表观遗传水平、转录水平和转录后水平直接或者间接参与靶基因的调控,广泛参与细胞内的重要生理调节作用[2]。但是,LncRNA表达失调也可通过不同的信号通路调节多种恶性肿瘤的表型,从而参与肿瘤细胞的增殖、分化、迁移和侵袭等恶性生物学进程[3-5]。近年来,越来越多的研究表明LncRNA可成为恶性肿瘤诊断、治疗和评价预后的潜在生物标志物[6-8]。
食管癌是消化系统常见的恶性肿瘤之一,我国食管癌的病理类型主要是鳞状细胞癌(esophageal squamous cell carcinoma, ESCC)[9]。由于ESCC患者早期症状不典型,大多数患者就诊时已为中晚期,且具有进展快和复发转移率高等恶性特点[10],严重影响ESCC的治疗效果。因此,迫切需要寻找新的预测标志物和治疗靶点以提高整体疗效。LncRNA淋巴细胞白血病缺失基因1(deleted in lymphocytic leukemia 1, DLEU1)位于染色体13q14.3上,最早发现其在慢性淋巴细胞白血病等造血系统肿瘤中表达失调并发挥重要调控作用[11],后续多项研究表明DLEU1在口腔癌、宫颈癌和肾癌等实体肿瘤中表达失调,且其异常表达与肿瘤的发生发展密切相关[12-14]。但是DLEU1在ESCC中的表达及相关功能研究目前尚无报道。本研究拟分析DLEU1在ESCC患者组织中的表达和临床意义,并探讨DLEU1对ESCC细胞增殖和迁移的恶性生物学行为,为深入研究其作用机制奠定理论基础。
1 资料与方法 1.1 临床资料收集2016年8月—2017年2月在河北医科大学第四医院胸外科行根治性肿瘤切除术的58例ESCC患者的癌及癌旁组织(距离癌灶边缘5 cm以外的癌旁组织)标本,标本取材后迅速置于RNA保存液中,以待提取RNA。所有患者术前3月内未行放化疗和免疫治疗等抗肿瘤治疗,并均于术后经病理学确诊为ESCC。58例患者中男44例,女14例,年龄50~76岁,中位年龄63岁。组织标本按照国际抗癌联盟(UICC)第八版标准进行TNM分期:Ⅰ期6例、Ⅱ期18例、Ⅲ期34例;肿瘤 < 5 cm者37例,≥5 cm者21例;有淋巴结转移者42例,无淋巴结转移者16例。所有研究对象均签署知情同意书,研究方案经河北医科大学第四医院伦理委员会批准。
1.2 细胞株和主要试剂人ESCC细胞株TE1、Yes-2、Kyse150、Kyse170和Eca9706来自河北医科大学第四医院东院科研中心。胎牛血清、DMEM培养基及LipofectamineTM2000购自美国Invitrogen公司。0.25%胰蛋白酶和DMSO购自上海索莱宝公司。DLEU1引物由广州瑞博生物科技有限公司合成,GAPDH引物由上海生工生物工程有限公司合成。RNA保存液购自北京庄盟国际生物基因科技有限公司。Tri Quick总RNA提取试剂购自北京索莱宝科技有限公司。qPCR Mix购自武汉爱博泰克生物科技有限公司。反转录试剂购自美国Thermo Fisher Scientific公司。siRNA转染试剂购自上海吉玛公司。CCK-8试剂购自大连美仑生物技术有限公司。
1.3 RT-qPCR检测ESCC组织和细胞中DLEU1的表达水平从RNA保存液中将ESCC及其相应癌旁组织取出,在研钵中将其研磨成粉末后,加入1 ml TRIzol试剂。另外按照TRIzol试剂说明书提取ESCC及其相应癌旁组织和ESCC细胞系TE1、Yes-2、Kyse150、Kyse170和Eca9706中的总RNA,测定RNA纯度及浓度。然后按照反转录试剂盒合成cDNA,反应条件为:40℃ 60 min,25℃ 5 min,75℃ 5 min。以1 μl cDNA配置qPCR体系,反应管中加入5 μl SYBR Green荧光染料、0.4 μl上下游引物和3.6 μl RNase-free水,混匀后以cDNA为模板、GAPDH为内参,对底物进行扩增,扩增条件为:95℃ 3 min;95℃ 5 s、60℃ 32 s,共40个循环。引物序列:DLEU1: F: 5’-GCGGAGGTGAAGTGAACTTAGA-3’, R: 5’-CTCCTAAGCAGGACCCGTATT-3’; GAPDH: F: 5’-TCAGAGTGGCGAAGGCAGGAG-3’, R: 5’-GCAACTCAAGTAAGTCCGCACCT-3’。实验结果采用2-ΔΔCt法计算目的基因的相对表达量,实验重复3次。
1.4 分析DLEU1表达水平与ESCC患者临床病理特征的关系收集58例ESCC患者的临床病理资料,分析ESCC患者中DLEU1表达量与临床病理特征的相关性。根据ESCC组织中DLEU1的相对表达倍数分为高表达组和低表达组。本研究将所有ESCC患者DLEU1的相对表达倍数临界阈值定义为1,其中 > 1为DLEU1高表达组(n=37),≤1为DLEU1低表达组(n=21)。分析DLEU1表达与ESCC患者临床病理特征的关系。
1.5 多因素Cox回归分析DLEU1表达与ESCC患者预后的关系随访时间定义为术后病理确诊时间至患者出现转移或随访截止时间,随访时间为36月,随访方式主要为电话随访和门诊随访两种方式,随访内容包括一般情况、临床症状及影像学检查。如随访期间患者出现转移,随访时间终止;如患者随访期间失访,则以末次有效随访时间为止。计算各例患者的生存时间(月),根据分组情况用乘积极限法(Kaplan-Meier)进行单因素生存分析并行Log rank检验。并釆用多因素Cox回归方法分析ESCC患者的独立预后因素。
1.6 细胞转染取对数生长期的Eca9706细胞,以1×105个/孔密度接种于6孔板中,待细胞融合度达到70%~80%时,按LipofectamineTM2000转染试剂的说明书,将LipofectamineTM2000及5 μl siRNA-DLEU1或5 μl siRNA-NC转染入Eca9706细胞系,4~6 h后更换含FBS新鲜培养基,继续培养进行后续功能试验。si-DLEU1和si-NC的序列分别是5’-GCAGUCUGUUCUGAACAUA-3’和5’-GUCUGCAGCUGAGAGUAT- 3’。
1.7 CCK-8法检测ESCC细胞增殖能力取转染后的各组Eca9706细胞,以1.5×103个/孔的密度接种于96孔板,每组设4个平行复孔,培养0、24、48、72、96 h后分别每孔加入10 μl CCK-8试剂,常规孵育1 h,用酶标仪检测450 nm波长的光密度(OD)值,计算细胞增殖能力,实验重复3次。
1.8 划痕实验检测ESCC细胞的迁移能力6孔板接种约5×105个细胞,待细胞长满后,用200 μl枪头在细胞中划2条垂直于培养板底的直线,PBS冲洗3次,加入2 ml无血清DMEM培养基继续培养,分别于0和24 h在显微镜下观察细胞的迁移距离,实验重复3次。
1.9 统计学方法采用SPSS20.0软件进行统计分析,正态分布的计量资料以(x±s)表示,组间比较采用t检验;计数资料以率表示,采用χ2检验比较各组表达情况与临床病理特征的关系;生存分析比较采用Kaplan-Meier检验;多变量Cox回归模型评估DLEU1及其他各变量对预后的作用。GraphPad Prism8对实验数据进行相关图片的绘制。以P < 0.05为差异有统计学意义。
2 结果 2.1 DLEU1在ESCC组织中的表达水平RT-qPCR结果显示,ESCC组织中DLEU1的表达水平(1.97±2.87)显著高于配对癌旁组织(1±1.87),差异有统计学意义(t=2.907, P < 0.01),见图 1。表明DLEU1在ESCC组织中高表达。
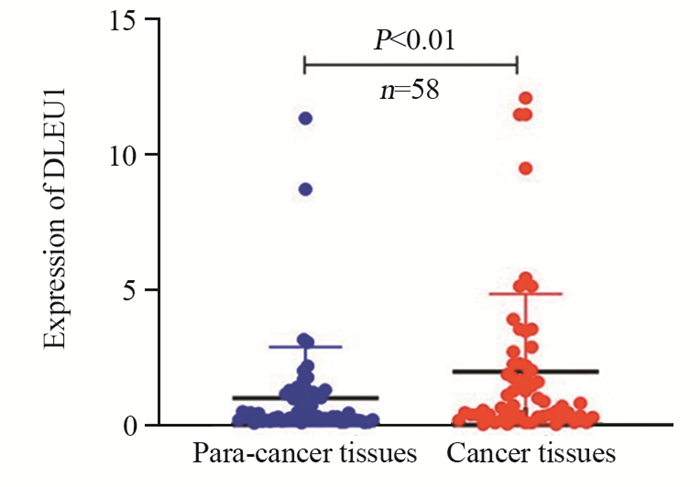
|
| 图 1 RT-qPCR法检测ESCC组织中DLEU1的表达水平 Figure 1 Expression levels of DLEU1 in ESCC tissues detected by RT-qPCR |
ESCC组织中DLEU1高表达组37例(63.79%),DLEU1低表达组21例(36.21%)。DLEU1高表达组中DLEU1的表达水平是相应癌旁组织的(4.59±3.16)倍(P < 0.0001,DLEU1低表达组中DLEU1的表达水平是相应癌旁组织的(0.73±0.32)倍(P=0.000397)。进一步分析DLEU1基因表达水平对ESCC患者临床病理特征的影响,结果显示,ESCC组织中DLEU1表达水平与ESCC患者的肿瘤大小(P=0.0089)、TNM分期(P=0.0437)和淋巴结转移(P=0.0015)具有相关性(均P < 0.05),而与患者性别(P=0.4949)和年龄(P=0.9845)无相关性(均P > 0.05),见表 1。
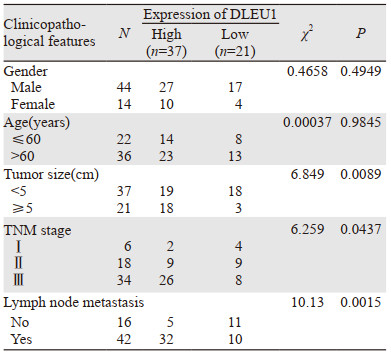
|
本研究共随访病例58例,无失访病例,随访率100%。Kaplan-Meier生存曲线结果显示,与DLEU1基因低表达组相比,DLEU1基因高表达组ESCC患者3年生存率显著降低,两组间差异有统计学意义(HR=2.808, P < 0.01),见图 2。以上结果表明ESCC组织中高表达DLEU1基因提示患者预后不良。
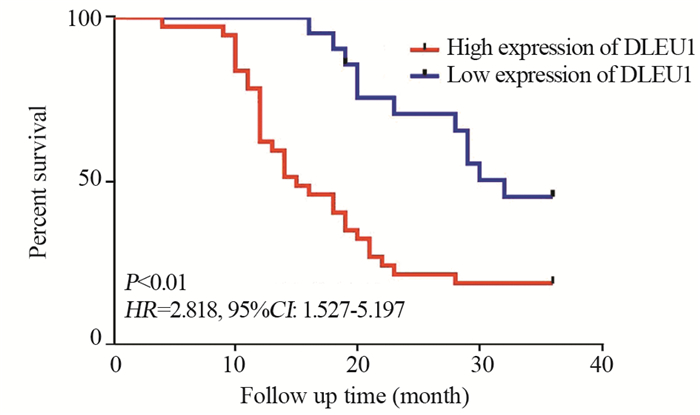
|
| 图 2 DLEU1表达与ESCC患者预后的关系 Figure 2 Relation between DLEU1 expression and prognosis of ESCC patients |
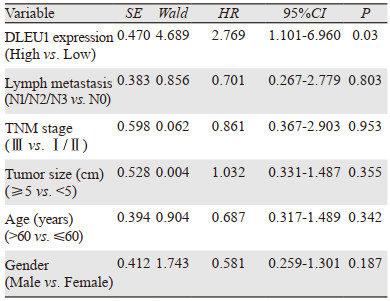
进一步进行多因素分析,结果表明,DLEU1高表达是ESCC患者较短生存期的独立危险因素(HR=2.769, 95%CI: 1.101~6.96, P=0.03),见图 3、表 2。说明高表达DLEU1是影响ESCC患者预后的独立危险因素。
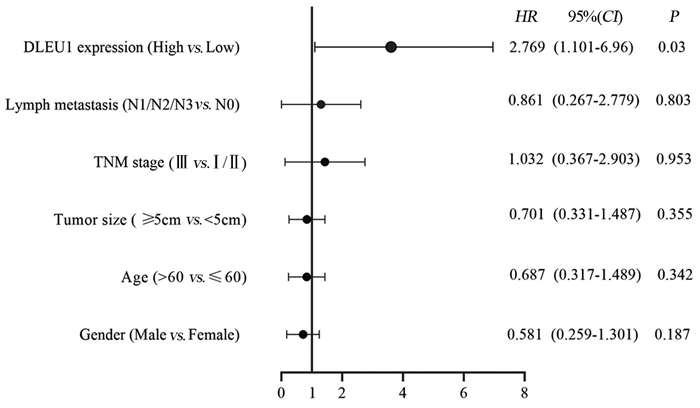
|
| 图 3 ESCC患者Cox风险回归模型多因素分析 Figure 3 Multivariate Cox risk regression analysis of ESCC patients |
|
RT-qPCR检测结果显示,与TE1细胞相比,食管鳞癌Eca9706、Yes-2、Kyse170和Kyse150细胞中DLEU1表达水平明显升高(t=27.14, 21.89, 22.97, 32.06,均P < 0.01),见图 4A,因此我们选择Eca9706细胞进行后续研究。进一步RT-qPCR检测结果显示,与si-NC组相比,si-DLEU1组中DLEU1表达水平明显降低(t=6.682, P < 0.01),见图 4B,证实成功构建低表达DLEU1的食管癌细胞系。

|
| **: compared with TE1 cell, all P < 0.01. 图 4 RT-qPCR检测ESCC细胞系中(A)和Eca9706细胞转染si-DLEU1、si-NC后(B)DLEU1的表达水平 Figure 4 Expression levels of DLEU1 in ESCC cell line(A) and Eca9706 cells transfected with si-DLEU1 or si-NC(B) detected by RT-qPCR |
CCK-8法检测结果显示,siRNA-DLEU1组在48、72、96 h细胞增殖能力明显低于si-NC组,分别为(t48=5.2, t72=5.716, t96=5.346,均P < 0.01),见图 5A。划痕愈合实验表明,siRNA-DLEU1组细胞的迁移能力明显小于siRNA-NC组(t=7.333, P < 0.01),见图 5B。综上所述,敲低DLEU1可抑制食管癌Eca9706细胞的增殖和迁移能力。

|
| 图 5 DLEU1促进Eca9706细胞的增殖(A)和迁移(B)能力 Figure 5 DLEU1 promoted proliferation(A) and migration(B) of Eca9706 cells |
近年来,越来越多的研究表明LncRNA的异常表达与ESCC的增殖、侵袭、迁移等恶性生物学行为密切相关,并参与ESCC的发生发展[15-16]。DLEU1是一种定位于人染色体13q14.2-q14.3区域,高度保守的LncRNA。早期研究[11]发现B细胞慢性淋巴细胞白血病的患者中存在DLEU1失调,其过表达可以提高患者治愈率及生存率,推测DLEU1作为肿瘤抑制基因参与B细胞慢性淋巴细胞白血病的发生发展。随后多项研究[17-19]表明,DLEU1在多种实体肿瘤中表达上调,可作为一种癌基因调控肿瘤的恶性生物学进程。有研究发现[20],LncRNA DLEU1在胰腺癌组织中高表达,敲低DLEU1可明显抑制胰腺癌细胞的增殖、迁移和侵袭能力,LncRNA DLEU1高表达为预后不良指标。在胃癌中,DLEU1高表达状态和肿瘤大小、病理分期和淋巴结转移有关,其作用机制可能通过上调KLF2表达参与胃癌的恶性生物学行为[21]。以上研究结果表明DLEU1的表达失调调控肿瘤的发生发展。然而,关于DLEU1在ESCC中的研究缺乏相关报道。本研究检测了ESCC组织和癌旁组织中DLEU1的表达水平,分析其与ESCC患者的临床病理特征以及预后的相关性,并初步探索了DLEU1在食管癌细胞系中的功能影响,旨在为ESCC的早期诊断和判断预后提供新的生物标志物。
本研究结果发现,与相应的癌旁组织表达相比,ESCC组织中DLEU1的相对表达水平显著升高。表明DLEU1在ESCC组织中呈高表达状态,提示我们增高的DLEU1可能参与了食管鳞癌肿瘤生物学特性的形成和肿瘤进展过程。进一步分析了ESCC组织中DLEU1表达与ESCC患者临床病理特征的相关性。结果显示DLEU1表达水平与年龄和性别无关,与肿瘤组织大小、TNM分期和淋巴结转移密切相关,即肿瘤组织越大、TNM分期越晚和有淋巴结转移,DLEU1表达水平越高。综上所述,DLEU1可能通过上调其表达参与ESCC肿瘤组织的浸润生长、转移及侵袭过程。
本研究Kaplan-Meier生存分析结果发现,DLEU1高表达患者具有较短的生存期,提示DLEU1高表达预示ESCC患者预后不良。后续采用多因素Cox回归分析证实了DLEU1高表达是ESCC患者预后的独立影响因素。另外,本研究成功构建DLEU1低表达的食管癌细胞系,初步探讨DLEU1在食管癌细胞系中的恶性生物学行为。通过CCK-8和划痕愈合实验发现沉默DLEU1可明显抑制ESCC细胞的增殖和迁移能力。因此,DLEU1可作为癌基因参与ESCC的恶性生物学行为。
综上所述,DLEU1在ESCC组织中高表达,其高表达水平与肿瘤组织大小、TNM分期、淋巴结转移及预后不良密切相关,且是ESCC患者不良预后的独立影响因素。另外,DLEU1可促进ESCC细胞的增殖和迁移能力。以上结果提示DLEU1可以作为ESCC潜在的分子标志物及药物靶点,为深入探讨DLEU1影响食管癌浸润转移的分子机制奠定理论基础。
作者贡献
段玉青:实验设计、实施及开展;数据分析;论文撰写及修改
王郁:细胞培养
王雪晓:临床标本收集
刘丽华:课题设计、数据审核及指导论文撰写
| [1] |
Liu J, Jung C, Xu J, et al. Genome-wide analysis uncovers regulation of long intergenic noncoding rnas in arabidopsis[J]. Plant Cell, 2012, 24(11): 4333-4345. DOI:10.1105/tpc.112.102855 |
| [2] |
Lin C, Yang L. Long noncoding RNA in cancer: Wiring signaling circuitry[J]. Trends Cell Bio, 2018, 28(4): 287-301. DOI:10.1016/j.tcb.2017.11.008 |
| [3] |
Su R, Ma J, Zheng J, et al. Pabpc1-induced stabilization of bdnf-as inhibits malignant progression of glioblastoma cells through stau1-mediated decay[J]. Cell Death Dis, 2020, 11(2): 81. DOI:10.1038/s41419-020-2267-9 |
| [4] |
Wen X, Liu X, Mao YP, et al. Long non-coding rna dancr stabilizes HIF-1α and promotes metastasis by interacting with nf90/nf45 complex in nasopharyngeal carcinoma[J]. Theranostics, 2018, 8(20): 5676-5689. DOI:10.7150/thno.28538 |
| [5] |
Tang T, Shan G, Zeng F. Knockdown of dgcr5 enhances the radiosensitivity of human laryngeal carcinoma cells via inducing mir-195[J]. J Cell Physiol, 2019, 234(8): 12918-12925. DOI:10.1002/jcp.27958 |
| [6] |
Qiu BQ, Lin XH, Ye XD, et al. Long non-coding RNA PSMA3-AS1 promotes malignant phenotypes of esophageal cancer by modulating the miR-101/EZH2 axis as a ceRNA[J]. Aging(Albany NY), 2020, 12(2): 1843-1856. |
| [7] |
Jia Y, Duan Y, Liu T, et al. Lncrna TTN-AS1 promotes migration, invasion, and epithelial mesenchymal transition of lung adenocarcinoma via sponging miR-142-5p to regulate CDK5[J]. Cell Death Dis, 2019, 10(8): 573. DOI:10.1038/s41419-019-1811-y |
| [8] |
Wang Y, Fang Z, Hong M, et al. Long-noncoding RNAs(lncRNAs) in drug metabolism and disposition, implications in cancer chemo-resistance[J]. Acta Pharm Sin B, 2020, 10(1): 105-112. DOI:10.1016/j.apsb.2019.09.011 |
| [9] |
Ohashi S, Miyamoto S, Kikuchi O, et al. Recent advances from basic and clinical studies of esophageal squamous cell carcinoma[J]. Gastroenterology, 2015, 149(7): 1700-1715. DOI:10.1053/j.gastro.2015.08.054 |
| [10] |
Yang L, Song X, Zhu J, et al. Tumor suppressor microrna-34a inhibits cell migration and invasion by targeting MMP-2/MMP-9/FNDC3B in esophageal squamous cell carcinoma[J]. Int J Oncol, 2017, 51(1): 378-388. DOI:10.3892/ijo.2017.4015 |
| [11] |
Garding A, Bhattacharya N, Claus R, et al. Epigenetic upregulation of lncRNAs at 13q14.3 in leukemia is linked to the in Cis downregulation of a gene cluster that targets NF-κB[J]. PLoS Genet, 2013, 9(4): e1003373. DOI:10.1371/journal.pgen.1003373 |
| [12] |
Nishiyama K, Maruyama R, Niinuma T, et al. Screening for long noncoding RNAs associated with oral squamous cell carcinoma reveals the potentially oncogenic actions of DLEU1[J]. Cell Death Dis, 2018, 9(8): 826. DOI:10.1038/s41419-018-0893-2 |
| [13] |
Liu C, Tian X, Zhang J, et al. Long non-coding RNA DLEU1 promotes proliferation and invasion by interacting with miR-381 and enhancing HOXA13 expression in cervical cancer[J]. Front Genet, 2018, 9: 629. DOI:10.3389/fgene.2018.00629 |
| [14] |
He GZ, Yu SY, Zhou QP, et al. Lncrna DLEU1 accelerates the malignant progression of clear cell renal cell carcinoma via regulating miRNA-194-5p[J]. Eur Rev Med Pharmacol Sci, 2019, 23(24): 10691-10698. |
| [15] |
Sheng J, Zhao Q, Zhao J, et al. SRSF1 modulates PTPMT1 alternative splicing to regulate lung cancer cell radioresistance[J]. EBioMedicine, 2018, 38: 113-126. DOI:10.1016/j.ebiom.2018.11.007 |
| [16] |
Hong W, Ying H, Lin F, et al. LncRNA LINC00460 silencing represses EMT in colon cancer through downregulation of ANXA2 via upregulating miR-433-3p[J]. Mol Ther Nucleic Acids, 2020, 19: 1209-1218. DOI:10.1016/j.omtn.2019.12.006 |
| [17] |
Chen X, Zhang C, Wang X. Long noncoding RNA DLEU1 aggravates osteosarcoma carcinogenesis via regulating the miR-671-5p/DDX5 axis[J]. Artif Cells Nanomed Biotechnol, 2019, 47(1): 3322-3328. DOI:10.1080/21691401.2019.1648285 |
| [18] |
Feng L, He M, Rao M, et al. Long noncoding RNA DLEU1 aggravates glioma progression via the miR-421/MEF2D axis[J]. Onco Targets Ther, 2019, 12: 5405-5414. DOI:10.2147/OTT.S207542 |
| [19] |
Zhang W, Liu S, Liu K, et al. Long non-coding RNA deleted in lymphocytic leukaemia 1 promotes hepatocellular carcinoma progression by sponging miR-133a to regulate IGF-1R expression[J]. J Cell Mol Med, 2019, 23(8): 5154-5164. DOI:10.1111/jcmm.14384 |
| [20] |
Gao S, Cai Y, Zhang H, et al. Long noncoding RNA DLEU1 aggravates pancreatic ductal adenocarcinoma carcinogenesis via the miR-381/CXCR4 axis[J]. J Cell Physiol, 2019, 234(5): 6746-6757. DOI:10.1002/jcp.27421 |
| [21] |
Li X, Li Z, Liu Z, et al. Long non-coding RNA DLEU1 predicts poor prognosis of gastric cancer and contributes to cell proliferation by epigenetically suppressing KLF2[J]. Cancer Gene Ther, 2018, 25(3-4): 58-67. DOI:10.1038/s41417-017-0007-9 |
 2021, Vol. 48
2021, Vol. 48


