文章信息
- TP53家族基因TP73与肿瘤发生的研究进展
- Progress of TP53 Family Gene TP73 and Tumorigenesis
- 肿瘤防治研究, 2021, 48(2): 186-190
- Cancer Research on Prevention and Treatment, 2021, 48(2): 186-190
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2021.20.0622
- 收稿日期: 2020-06-05
- 修回日期: 2020-10-03
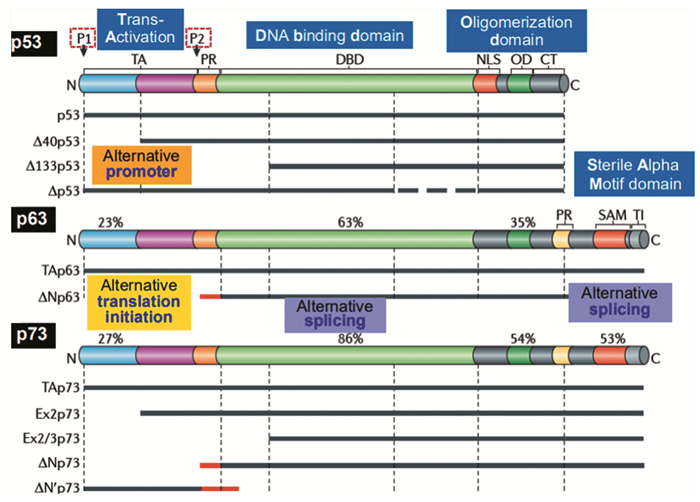
TP73基因,是TP53家族同源基因,基因序列与TP53高度相似,见图 1[1]。因其功能与著名的“基因卫士”TP53类似,所以将TP73、TP53和家族另一重要成员TP63合称为“基因卫士”家族[2]。但与p53基因在恶性肿瘤中广泛发生突变不同,p73很少发生基因突变。据统计p73在初发肿瘤中突变率仅为0.6%,因此一般认为p73作为抑癌因子,在p53突变型肿瘤中起着代偿性的抑癌作用[3]。然而并非完全如此,机体内存在着基因全长型的TAp73亚型(抑癌因子)和N端截短型的DNp73亚型(癌促因子),两种功能对立的亚型在机体内保持一定比例,从而维持机体“稳态”。而在肿瘤发生过程中往往可以观察到两者比例失衡。肿瘤发生与多种关键分子表达失调和细胞信号通路异常相关,一旦重要分子和调节通路发生致癌性异常将最终导致肿瘤发生。本文就p73与肿瘤发生的最新重要研究成果作一综述。
人类TP73基因位于1号染色体的远端短臂(1p36.32),含16个外显子,属于TP53同源家族基因[4]。与TP53基因序列类似,TP73在5’端含有两个选择性启动子P1和P2以及多个可变剪接位点。从P1起始转录的p73蛋白N端含有反式激活结构域(trans-activation domain, TA domain),从P2起始转录将不含TA结构域。同时,TP53和TP73序列中间均含有DNA结合域(DNA binding domain, DBD)。另外,在TP73的3’端含有不育α基序域(sterile alpha motif domain, SAM),在肿瘤细胞基因组中该区域通常缺失[5]。TP73基因,可以转录出35个mRNA转录本,理论上能翻译产生29种p73蛋白质亚型。目前已经发现了14种p73蛋白质亚型。
从功能上可将p73蛋白分为TAp73和DNp73两种亚型。其中,TAp73亚型是从P1启动子起始转录,包含转录激活结构域的转录本,而DNp73主要包括从P2启动子起始转录产生的ΔΝp73转录本和源于P1启动子转录后经选择性剪接作用而导致转录激活结构域缺失的ΔEx2TP73(缺失exon2)、ΔEx2/3TP73(缺乏exons2和3)以及Δ’ΝTp73(缺失exons1和2)转录本。另外,3’端出现的另一种剪接作用将产生至少7种不同的mRNA异形体(α, β, γ, ζ, δ, ε和η)[6]。然而,这些异形体能否被翻译成蛋白质仍有争议。一般认为,TAp73与抑癌因子p53功能类似,能够共同启动p53下游通路,在p53突变型肿瘤细胞中起代偿性的抑癌作用。然而,DNp73与TAp73功能相反,对肿瘤发生发展起着正性调控作用。
2 TP73/p73与肿瘤发生 2.1 TP73调节自噬TP73能够调节自噬调制器DRAM1的表达。实验表明,原代细胞及肿瘤细胞系经mTOR信号抑制剂雷帕霉素的处理后,TP73表达水平提高了,这表明TP73和mTOR信号通路之间存在着联系[7]。最近,体内实验也证实了TP73在自噬过程中的作用[8]。当TP73敲除小鼠受到营养缺乏的打击时,肝脏中有大量的脂质颗粒堆积,而自噬的水平也降低了[8]。因此,合理的推测是甘油三酯水解转化为脂肪酸的过程受阻进而导致自噬缺失。另外,在TP73敲除小鼠的肝脏、结肠和心脏等多个器官中Atg5(自噬关键基因)的表达受抑。进一步研究发现TAp73α和TAp73β结合并激活了Atg5启动子。这说明Atg5是TAp73的直接转录靶基因。TAp73-Atg5轴进一步证实了TAp73在自噬调控中所扮演的角色,并有助于解释TP73敲除小鼠所表现出的上述表型[9]。
2.2 TP73调节血管生成血管生成与肿瘤的发生发展息息相关。已经证实,p73蛋白可以调节血管生成,其中DNp73可诱导血管生成基因VEGF-α表达促进血管生成,而TAp73则具有双重功能[10]。一方面,TAp73可通过MDM2负性调控HIF-1α(低氧诱导因子)的表达来间接抑制血管生成。在TAp73沉默的小鼠模型中观察到血管生成增加,而过表达TAp73则表现出相反的结果,这些实验结果均支持TAp73的抗血管生成作用[11]。然而,另一方面,在低氧环境中(一种生理性血管生成刺激因素),TAp73通过HIF-1α阻遏E3泛素化连接酶SIAH1的表达而维持自身稳定,进而直接激活包含VEGF-α在内的许多血管生成靶基因[12]。这种机制与DNp73促血管生成的机制类似。该机制显示TAp73和DNp73可以直接结合到血管生成靶基因的调节位点,而并不需要依赖HIF-α结合到HREs(缺氧应答元件)。实验表明,敲除TAp73或者DNp73后血管生成靶基因的活性减弱,而相反过表达TAp73或DNp73则可诱导这些靶基因表达。另外,观察到p73缺乏后小鼠视网膜的血管发育受到了干扰。这一系列实验结果均支持TAp73的促血管生成理论。目前认为,TAp73所表现出的双重功能依赖于刺激的强度和特定的生理环境。这种功能的选择性也受到其他因素的影响,比如p53的状态[13],但其具体机制尚未明确。
2.3 药物敏感度、转移和细胞凋亡等最近,有报道称p73是人类化学敏感度的一个重要决定因素。携带特定多态性的p53突变蛋白质可以通过抑制TAp73来诱导耐药性产生[14],并且特定磷酸化、甲基化和乙酰化事件对p73诱导细胞凋亡和药物化学敏感度具有关键作用,例如:TAp73的表达和P1启动子甲基化可降低肌肉浸润性膀胱癌(MIBC)化疗敏感度和顺铂治疗后的生存率[15]。另外有文献表明DNp73在驱动肿瘤细胞转移中起重要作用,这一功能的实现主要是通过切断IGF1R-AKT/STAT3信号通路与其抑制因子EPLIN之间的联系实现的,实验发现DNp73可以通过触发上皮间质转换(EMT)、细胞迁移和瘤细胞入侵来促进皮肤癌转移,而且在高度转移性肿瘤中敲降DNp73水平能够消除这些转移表型[16]。另外有研究人员发现,NAV3(Navigator-3,一种微管结合蛋白)是p73的一个新的转录靶点。它通过DNA损伤以依赖p73的方式上调,并在p73介导的抑制癌细胞侵袭、迁移和转移中发挥重要作用。即DNA损伤诱导p73,上调内源性NAV3 mRNA和蛋白水平,进而抑制E-钙黏素的表达而抑制肿瘤细胞转移和侵袭[17]。对于p53突变后p73能否发挥其潜在的抑癌作用,最近一项针对p53缺失的人肝癌细胞(Hep3B)的研究发现,使用姜黄素后,可以通过一种TAp73/DNp73依赖的方式诱导细胞凋亡[18]。
2.4 TP73调节细胞代谢TP53和TP63在代谢方面均有重要发现,那么自然而然我们会猜想TP73是否也在细胞代谢方面发挥作用,尤其是肿瘤细胞代谢。研究报道,TAp73可通过干预线粒体代谢来调节细胞衰老[19]。结果显示TAp73敲除小鼠表现出更明显的衰老表征,包括生存期下降、体重减轻、体脂减少、角膜退化和脊柱后凸增强。敲除TAp73使得细胞内ATP水平、氧消耗量和线粒体复合物Ⅳ活性降低,这表明线粒体代谢有缺陷。其中线粒体复合体Ⅳ亚基之一的细胞色素C氧化酶亚基4(COX4i1)是线粒体复合物Ⅳ装配和功能完整所必需的[19]。在TAp73缺陷的小鼠中发现COX4i1表达减少,而恢复其在TAp73敲除细胞中的表达后发现线粒体耗氧量增加。同时,在TAp73敲除鼠中观察到氧化应激的增加,可能会导致原癌基因的突变累积,使基因组不稳定性增加。这与在小鼠中观察到自发肿瘤增加的结果相符合[20]。
肿瘤细胞的一个共同特征是代谢重编程。快速增殖的肿瘤细胞需要通过增加营养物质吸收和重组代谢通量来支持生物合成,以适应它们的新陈代谢[21]。最近发现,TAp73直接调控着磷酸戊糖途径(PPP)的限速酶——葡萄糖-6-磷酸脱氢酶(G6PD)的表达[22]。然而TP53和TP63却不能与其启动子结合并驱动G6PD的转录,这表明存在某些与TAp73有特异作用的辅因子参与了G6PD的调控。TAp73对G6PD表达的正性调控增加了PPP的通量,进而促进了NADPH和核糖核酸的产生,为合成代谢提供了能量和原料。总的来说,这些发现颠覆了传统所认为的TA亚型起着抑癌作用,而表现出潜在的致癌作用。
肿瘤细胞依赖于特定的代谢产物来维持细胞存活和增殖。丝氨酸途径为细胞提供了氨基酸、谷胱甘肽(GSH)、脂质和核苷酸[21]。有研究报道,TP53与肿瘤细胞应对丝氨酸饥饿的能力有关,即缺乏TP53的细胞不能对丝氨酸饥饿作出反应[23]。有趣的是,近来Amelio等发现,在人类肺腺癌中,丝氨酸的生物合成途径和TP73的表达之间也存在着相关性。TAp73可激活丝氨酸的生物合成途径,导致细胞内的丝氨酸和甘氨酸的水平增加,而这与谷氨酸、三羧酸循环中间体和GSH的积累有关。TAp73直接调节谷氨酰胺酶-2(GLS2)的表达,促进谷氨酸向谷氨酰胺转化,进而驱动丝氨酸的生物合成。同时,抑制TAp73表达后,发现完全抑制了肿瘤细胞依赖于丝氨酸和甘氨酸的增殖途径[24]。这表明,TAp73在代谢应激下促进肿瘤细胞的增殖,也与传统所认为的抑癌作用相反。
2.5 TP73与miRNAs2009年,Sampath等发现了TP73和miRs之间存在交互作用的第一个证据[25]。该团队发现组蛋白去乙酰化酶抑制剂(HDACis)治疗慢性淋巴细胞白血病(CLL)本质上是一种依赖TP73的细胞凋亡。HDACis促进miR-106b的表达,使其靶向E3泛素连接酶Itch而失活。Itch水平的下降,使促凋亡因子TAp73稳定积累,随后转录激活PUMA(p53上调凋亡调节因子)和导致线粒体功能障碍。另外,在CLL和胰腺癌中均发现miR-106b(上调)与Itch(下调)呈负相关[25-26]。然而,在使用各种刺激物诱导细胞凋亡过程中发现,CLL细胞中的Itch也会被细胞凋亡蛋白酶切除。由于凋亡蛋白酶-3、凋亡蛋白酶-6和凋亡蛋白酶-7中Asp240残基活性而引起Itch的切除,并且使用凋亡蛋白酶抑制剂能够完全解除HDACi治疗后的Itch下调。这表明Itch下调是细胞凋亡的结果,而不是原因。因此,我们可以合理推测出在HDACis治疗CLL过程中miR-106b与TAp73存在直接交互作用。
miR-647在广泛肿瘤中都发挥作用。最近研究发现,miR-647在胃癌中高表达[27],并且miR-647的表达水平与胃癌细胞系MGC-803的增殖和迁移能力呈正相关,而与凋亡呈负相关。进一步研究发现miR-647通过靶向抑制肿瘤抑制蛋白p73而促进肿瘤发生发展。
TP73和miRs的交互作用还体现在卵巢癌中。研究发现,作为miR调节因子的TP53家族蛋白在卵巢癌细胞中过度表达[28]。TP63和TP73的表达与17种miRs的表达密切相关。在这些miRs中,miR-200家族的表达与TP73的表达呈正相关。TP53突变与miR-200家族的表达之间并没有相关性,这表明TP53并不是卵巢癌细胞中这些miRs的主要调控因子。通过荧光素酶报告系统和染色质免疫沉淀实验表明TP73可以直接与miR-200启动子结合,为TP73与miR-200的共表达提供了直接证据,但这一相关性是否与预后有关,目前尚无定论。此外,虽然最近发现了TP63 -miRs轴在肿瘤转移方面具有重要作用,但TP73在这方面所扮演的角色还有待阐明。
3 小结TP73作为“肿瘤卫士家族”—TP53家族的重要成员之一,基因序列与TP53高度相似。由于选择性启动子和可变剪接作用使得TP73可产生多种蛋白亚型,根据结构和功能的差异可分为TAp73和DNp73两大类。与p53常在肿瘤中发生突变不同,p73常表现为TAp73/DNp73表达失衡。两类分子功能既对立又统一,在肿瘤发生发展的许多方面均发挥着非常重要的作用。文章虽然对p73蛋白与肿瘤发生的关系进行了一定阐述,但新的科学问题也愈发凸显,例如p73蛋白如何影响p53蛋白家族中的其他成员、TAp73/DNp73的正常调控机制及表达失衡的原因为何、p73与肿瘤免疫逃逸是否相关等。这些科学问题仍需不断深入挖掘p73的潜在分子机制,从而推动肿瘤精准治疗的发展,最终造福患者。
作者贡献:
许杰:论文构思及撰写
郝牧:课题资金支持和论文修改
| [1] |
Levine AJ, Tomasini R, McKeon FD, et al. The p53 family: guardians of maternal reproduction[J]. Nat Rev Mol Cell Biol, 2011, 12(4): 259-265. DOI:10.1038/nrm3086 |
| [2] |
Napoli M, Flores ER. The p53 family reaches the final frontier: the variegated regulation of the dark matter of the genome by the p53 family in cancer[J]. RNA Biol, 2020, 17(11): 1636-1647. DOI:10.1080/15476286.2019.1710054 |
| [3] |
Hall C, Muller PAJ. The Diverse Functions of Mutant 53, Its Family Members and Isoforms in Cancer[J]. Int J Mol Sci, 2019, 20(24): 6188. DOI:10.3390/ijms20246188 |
| [4] |
Biscotti MA, Barucca M, Carducci F, et al. The p53 gene family in vertebrates: Evolutionary considerations[J]. J Exp Zool B Mol Dev Evol, 2019, 332(6): 171-178. DOI:10.1002/jez.b.22856 |
| [5] |
Vikhreva P, Melino G, Amelio I. p73 Alternative Splicing: Exploring a Biological Role for the C-Terminal Isoforms[J]. J Mol Biol, 2018, 430(13): 1829-1838. DOI:10.1016/j.jmb.2018.04.034 |
| [6] |
Zhang YX, Pan WY, Chen J. p53 and its isoforms in DNA double-stranded break repair[J]. J Zhejiang Univ Sci B, 2019, 20(6): 457-466. DOI:10.1631/jzus.B1900167 |
| [7] |
Wang H, Liu Y, Wang D, et al. The Upstream Pathway of mTOR-Mediated Autophagy in Liver Diseases[J]. Cells, 2019, 8(12): 1597. DOI:10.3390/cells8121597 |
| [8] |
Huang F, Wang BR, Wang YG. Role of autophagy in tumorigenesis, metastasis, targeted therapy and drug resistance of hepatocellular carcinoma[J]. World J Gastroenterol, 2018, 24(41): 4643-4651. DOI:10.3748/wjg.v24.i41.4643 |
| [9] |
Datta S, Chakraborty S, Panja C, et al. Reactive nitrogen species control apoptosis and autophagy in K562 cells: implication of TAp73α induction in controlling autophagy[J]. Free Radic Res, 2018, 52(4): 491-506. DOI:10.1080/10715762.2018.1449210 |
| [10] |
Dulloo I, Hooi PB, Sabapathy K. Hypoxia-induced DNp73 stabilization regulates Vegf-A expression and tumor angiogenesis similar to TAp73[J]. Cell Cycle, 2015, 14(22): 3533-3539. DOI:10.1080/15384101.2015.1078038 |
| [11] |
Stantic M, Wolfsberger J, Sakil H, et al. ΔNp73 enhances HIF-1α protein stability through repression of the ECV complex[J]. Oncogene, 2018, 37(27): 3729-3739. DOI:10.1038/s41388-018-0195-2 |
| [12] |
Marini A, Rotblat B, Sbarrato T, et al. TAp73 contributes to the oxidative stress response by regulating protein synthesis[J]. Proc Natl Acad Sci U S A, 2018, 115(24): 6219-6224. DOI:10.1073/pnas.1718531115 |
| [13] |
Nemajerova A, Moll UM. Tissue-specific roles of p73 in development and homeostasis[J]. J Cell Sci, 2019, 132(19): jcs2333378. |
| [14] |
Gunaratne PH, Pan Y, Rao AK, et al. Activating p53 family member TAp63: A novel therapeutic strategy for targeting p53-altered tumors[J]. Cancer, 2019, 125(14): 2409-2422. DOI:10.1002/cncr.32053 |
| [15] |
Bunch B, Krishnan N, Greenspan RD, et al. TAp73 expression and P1 promoter methylation, a potential marker for chemoresponsiveness to cisplatin therapy and survival in muscle-invasive bladder cancer (MIBC)[J]. Cell Cycle, 2019, 18(17): 2055-2066. DOI:10.1080/15384101.2019.1638693 |
| [16] |
Steder M, Alla V, Meier C, et al. DNp73 exerts function in metastasis initiation by disconnecting the inhibitory role of EPLIN on IGF1R-AKT/STAT3 signaling[J]. Cancer Cell, 2013, 24(4): 512-527. DOI:10.1016/j.ccr.2013.08.023 |
| [17] |
Uboveja A, Satija Y K, Siraj F, et al. p73-NAV3 axis plays a critical role in suppression of colon cancer metastasis[J]. Oncogenesis, 2020, 9(2): 12. DOI:10.1038/s41389-020-0193-4 |
| [18] |
Lee Y, Kwon YH. Regulation of apoptosis and autophagy by luteolin in human hepatocellular cancer Hep3B cells[J]. Biochem Biophys Res Commun, 2019, 517(4): 617-622. DOI:10.1016/j.bbrc.2019.07.073 |
| [19] |
Lopriore P, Capitanio N, Panatta E, et al. TAp73 regulates ATP7A: possible implications for ageing-related diseases[J]. Aging (Albany NY), 2018, 10(12): 3745-3760. |
| [20] |
Nemajerova A, Amelio I, Gebel J, et al. Non-oncogenic roles of TAp73: from multiciliogenesis to metabolism[J]. Cell Death Differ, 2018, 25(1): 144-153. DOI:10.1038/cdd.2017.178 |
| [21] |
Campbell SL, Wellen KE. Metabolic Signaling to the Nucleus in Cancer[J]. Mol Cell, 2018, 71(3): 398-408. DOI:10.1016/j.molcel.2018.07.015 |
| [22] |
Itahana Y, Itahana K. Emerging Roles of p53 Family Members in Glucose Metabolism[J]. Int J Mol Sci, 2018, 19(3): 776. DOI:10.3390/ijms19030776 |
| [23] |
Humpton TJ, Hock AK, Maddocks O, et al. p53-mediated adaptation to serine starvation is retained by a common tumour-derived mutant[J]. Cancer Metab, 2018, 6: 18. DOI:10.1186/s40170-018-0191-6 |
| [24] |
Amelio I, Markert EK, Rufini A, et al. p73 regulates serine biosynthesis in cancer[J]. Oncogene, 2014, 33(42): 5039-5046. DOI:10.1038/onc.2013.456 |
| [25] |
Sampath D, Calin GA, Puduvalli VK, et al. Specific activation of microRNA106b enables the p73 apoptotic response in chronic lymphocytic leukemia by targeting the ubiquitin ligase Itch for degradation[J]. Blood, 2009, 113(16): 3744-3753. DOI:10.1182/blood-2008-09-178707 |
| [26] |
Zhang X, Ren D, Wu X, et al. miR-1266 Contributes to Pancreatic Cancer Progression and Chemoresistance by the STAT3 and NF-κB Signaling Pathways[J]. Mol Ther Nucleic Acids, 2018, 11: 142-158. DOI:10.1016/j.omtn.2018.01.004 |
| [27] |
Zhang X, Zhang M, Wang G, et al. Tumor promoter role of miR647 in gastric cancer via repression of TP73[J]. Mol Med Rep, 2018, 18(4): 3744-3750. |
| [28] |
Knouf EC, Garg K, Arroyo JD, et al. An integrative genomic approach identifies p73 and p63 as activators of miR-200 microRNA family transcription[J]. Nucleic Acids Res, 2012, 40(2): 499-510. DOI:10.1093/nar/gkr731 |
 2021, Vol. 48
2021, Vol. 48