文章信息
- 环状RNA Hsa_circ_0026352在乳腺癌中的表达及临床意义
- Expression of Circular RNA Hsa_circ_0026352 in Breast Cancer and Its Clinical Significance
- 肿瘤防治研究, 2021, 48(1): 43-48
- Cancer Research on Prevention and Treatment, 2021, 48(1): 43-48
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2021.20.0310
- 收稿日期: 2020-04-07
- 修回日期: 2020-08-17
2. 750004 银川,宁夏医科大学临床医学院;
3. 750011 银川,银川市疾病预防控制中心微生物检验科
2. College of Clinical Medicine, Ningxia Medical University, Yinchuan 750004, China;
3. Department of Microbiological Examination, Yinchuan City Center for Disease Control and Prevention, Yinchuan 750011, China
乳腺癌是一种常见的女性高危恶性肿瘤,严重危害女性健康[1]。研究表明,年龄、雌激素水平异常、遗传和表观遗传因素都可导致乳腺癌发生风险增加[2]。尽管乳腺癌的诊疗技术不断发展,但乳腺癌的复发和转移仍频繁发生[3]。早期诊断能够提前预警,减少肿瘤的恶化。因此,探索更高效、敏感的分子标志物,对乳腺癌的临床防治具有重要意义。
环状RNA(circular RNA, circRNA)是一种具有连续闭合环状结构的非编码RNA,由外显子、内含子或两者共同的反向剪切而来[4]。circRNA由于其环状结构,不易被核酸外切酶降解,在生物体内比线性RNA更加稳定[5]。大多数circRNA在不同物种间表达具有组织特异性,在肿瘤疾病中大量差异表达[6-7]。稳定性、组织特异性和疾病特异性使circRNA具有成为检测分子标志物的潜力。然而,大多数circRNA的潜在功能尚不清楚。
因此,本研究通过一系列筛选过滤确定Hsa-circ-0026352(Hsa_circ_0026352)为候选的分子标志物,并验证其在乳腺癌患者外周血及健康人群外周血中的差异表达,探讨Hsa_circ_0026352的表达与乳腺癌患者临床特征的相关性,评价Hsa_circ_0026352作为乳腺癌诊断标志物的价值。为乳腺癌的早期防治提供新的思路和见解。
1 资料与方法 1.1 临床资料收集2017年1月—2018年10月宁夏医科大学总医院肿瘤外科确诊为乳腺癌患者的癌组织及癌旁组织标本共18对,乳腺癌患者外周血标本100例。乳腺癌患者纳入标准:(1)女性;(2)年满18周岁;(3)无既往病史;(4)无艾滋病毒感染;(5)乳房切除术后;(6)无放疗、化疗史。收集2018年10月宁夏医科大学总医院体检中心体检健康人群外周血标本50例。健康人群的纳入标准:(1)女性;(2)无恶性肿瘤病史;(3)无乳房功能障碍;(4)无其他任何临床全身性急性或慢性炎性反应性疾病;(5)无自身免疫疾病;(6)无高血压。所有标本的收集对象在调查取样前签署知情同意书。本研究已获得宁夏医科大学总医院伦理委员会批准。
1.2 标本及患者信息收集组织样本:接受手术乳房切除的乳腺癌患者,收取乳腺癌组织标本及癌旁正常组织标本(正常组织为肿瘤边缘>5 cm处)。外周血样本:乳腺癌患者,健康人群空腹过夜后,用乙二胺四乙酸(EDTA)抗凝采血管从肘部正中静脉采血5 ml。组织样本及血液样本在使用前后均储存于-80℃。
收集乳腺癌患者的一般临床资料和肿瘤病理学特征信息,包括:年龄、肿瘤大小、肿瘤是否转移、肿瘤TNM分期(依据美国肿瘤联合委员会恶性肿瘤分期系统进行分期)[8]、肿瘤组织学分级、糖蛋白抗原153(carbohydrate antigen, CA153)测定结果、癌胚抗原(carcinoembryonic antigen, CEA)测定结果。
1.3 circRNA芯片检测及分析以人类circRNA V2芯片(北京博奥生物科技股份有限公司,中国)对3对乳腺癌和癌旁组织的circRNA芯片检测分析。下机数据通过GeneSpring V13.0软件进行初步归一化及荧光信号值分析。该芯片共包含162 351个人类circRNA的标记探针。以癌旁组织检测结果为对照,筛选乳腺癌组织中产生差异的circRNA。筛选条件为:初始荧光信号值>100,差异倍数(Fold change, FC)≥2,显著性P < 0.05。采用Cluster 3.0,Graphpad Prism 8.0.1软件绘制环状RNA表达聚类图,采用ggPlot2软件分析散点图及火山图。使用circPrimer 1.2软件绘制环状RNA结构图。
1.4 qRT-PCR验证使用TRIzol(Invitrogen, 美国)法提取组织样本中的RNA,RNA大量提取试剂盒(百泰克,中国)提取血液样本中的RNA。取0.3 μg总RNA电泳验证RNA质量(1.3 g琼脂糖/100 ml,1×TAE缓冲液,120 V电压,25 min)。使用PrimeScript® RT reagent Kit(Thermo,美国)将RNA反转录获得cDNA,反转录体系为:1 µl总RNA(约500 ng),1 µl random primer,10 µl ddH2O,2 µl dNTP,4 µl反转录buffer,1 µl RNA酶抑制剂,1 µl反转录酶,总体积20 µl;反转录条件为:25℃ 5 min;42℃ 60 min,70℃ 5 min。RNA及cDNA置于-80℃储存备用。取各组cDNA使用TB Green premix Ex TaqTM Ⅱ(TAKARA,日本)在LightCycler480Ⅱ定量平台(Roche,美国)以β-actin为内参进行qRT-PCR验证实验。反应体系为:1 µl模板cDNA,0.5 µl+0.5 µl上下游引物,8 µl ddH2O,10 µl SYBR MIX,总体积20 µl;PCR条件为:94℃ 30 s预变性反应;94℃ 5 s,60℃ 30 s,72℃ 20 s,PCR反应40个循环;65℃ 5 s分析融解曲线。检测得到的数据通过2-ΔΔCt方法计算基因的相对表达量。所有引物序列见表 1。
使用SPSS23.0软件(IBM,美国)和GraphPad Prism version 8.0软件(GraphPad,美国)对数据结果进行统计分析。计量资料采用平均值±标准差(x±s)表示。两组数据间比较采用t检验,多组数据间比较采用单因素ANOVA分析,采用卡方检验分析分类变量的显著性,采用曲线回归分析及Pearson检验分析相关性,采用ROC曲线评价环状RNA的诊断价值,以曲线下面积(AUC)≥0.6表示具有诊断价值。检验水准P < 0.05为差异有统计学意义。
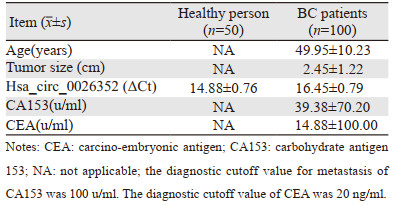
2 结果 2.1 Hsa_circ_0026352的筛选及验证在初始筛选条件下,共发现1 972个差异表达的circRNA,其中在乳腺癌组织中表达上调的环状RNA546个,下调的环状RNA1 426个。热图、火山图和散点图均显示了乳腺癌组织与癌旁组织间差异表达的circRNA,见图 1A。扩大筛选条件为差异倍数≥8,P < 0.01,荧光值≥1000,来源基因位于肿瘤相关的信号通路,最终确定circ-0026352满足筛选要求。通过qRT-PCR在18对乳腺癌及癌旁组织中验证发现,Hsa-circ-0026352在乳腺癌组织中表达显著低于癌旁组织(FC=9.45, P < 0.01),见图 1B。ROC曲线分析显示,Hsa-circ-0026352在乳腺癌及癌旁组织检测中的AUC>0.6(AUC= 0.881, P < 0.01),见图 1C。相关结果提示Hsa_circ_ 0026352具有乳腺组织特异性,具备成为乳腺癌检测标志物的潜力。
|
| PC: para-carcinoma tissue; A: heatmap of differential expression circRNAs profiles in breast cancer tissues and para-carcinoma tissues. Red: upregulated circRNAs expression. Green: downregulated circRNAs expression. Black: no significant difference; B: qRT-PCR for the Hsa_circ_0026352 expression between breast carcinoma tissue and para-carcinoma tissue; C: the ROC analysis results of Hsa_circ_0026352 in breast cancer tissue diagnostic examination. 图 1 Hsa_circ_0026352的芯片筛选及其在乳腺癌组织中表达的验证 Figure 1 Screening of Hsa_circ_0026352 through circular RNAs microarray and verification of its expression in breast cancer tissues |
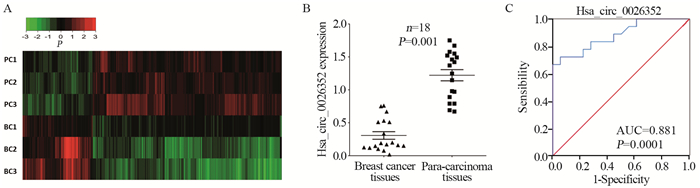
通过circ Primer1.2软件分析,Hsa_circ_0026352是由NR4A1基因第3、4、5外显子反向剪切而成,见图 2 A。为确保qRT-PCR验证有效性,在circRNA环状结构设计两条引物,Hsa_circ_0026352-junction为该环状RNA结合位点特异性引物,Hsa_circ_0026352-linear为该环状RNA臂上引物(可同时识别NR4A1 mRNA),使用核酸外切酶RNase R处理血液中提取的circRNA,结果显示,线性RNA会被核酸酶消化,而Hsa_circ_0026352无法被核酸酶消化,见图 2B。qRT-PCR验证结果显示,相对于健康人群外周血,Hsa_circ_0026352乳腺癌患者外周血中相对表达显著下调,见图 2C。ROC曲线分析显示,Hsa_circ_0026352在乳腺癌患者外周血检测中的AUC=0.826(P < 0.01),敏感度为68%,特异性为90%,见图 2D。相关结果提示Hsa_circ_0026352具有血液样品特异性,具备成为乳腺癌外周血诊断分子标志物的潜力。
|
| PB: peripheral blood; BC: breast cancer; A: structure chart of Hsa_circ_0026352; B: verification of circRNAs in peripheral blood with exonuclease(RNase R) treatment; C:qRT-PCR for the Hsa_circ_0026352 expression between peripheral blood of breast cancer patients and healthy person; D: the ROC analysis results of Hsa_circ_0026352 in peripheral blood of breast cancer patients. 图 2 Hsa_circ_0026352的结构特征及其在乳腺癌患者外周血中表达的验证 Figure 2 Structure characteristics of Hsa_circ_0026352 and its expression verification in peripheral blood of breast cancer patients |
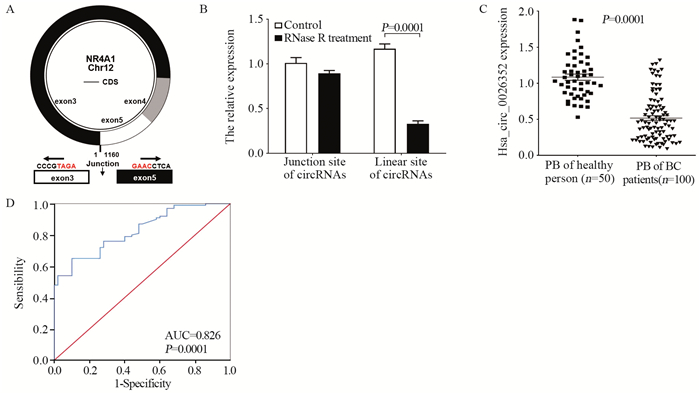
Pearson相关性分析结果显示,Hsa_circ_0026352的ΔCt值与外周血CA153水平呈显著正相关(n=100,r2=0.287, P=0.004);曲线回归分析结果显示,Hsa_circ_0026352相对表达与外周血CA153水平呈显著负相关(P < 0.05),而与年龄、肿瘤大小、CEA值的变化无相关性(P>0.05),见图 3、表 2。提示Hsa_circ_0026352可与CA153检测协同使用。
|
| A-D: curve regression analyses of the correlation between expression levels of hsa_circ_0026352 and the continuous measurement data of breast cancer patients including age, tumor size, CEA level and CA153 level. 图 3 Hsa_circ_0026352在外周血中的表达及其与乳腺癌患者临床特征的相关性 Figure 3 Expression levels of hsa_circ_0026352 in peripheral blood and its correlation with clinicopathological characteristics of breast cancer patients |
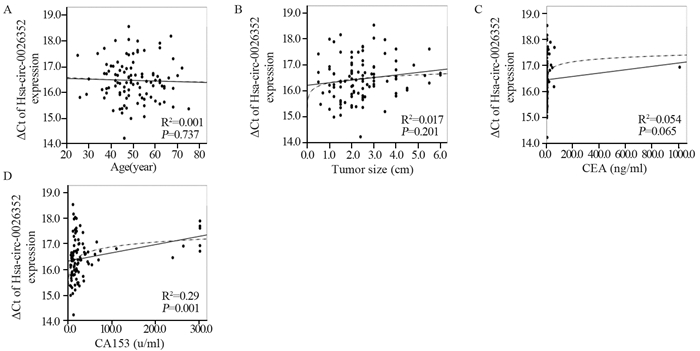
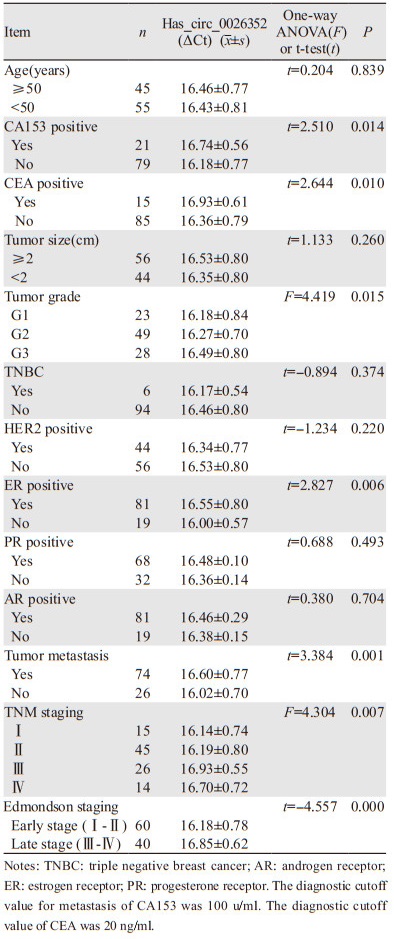
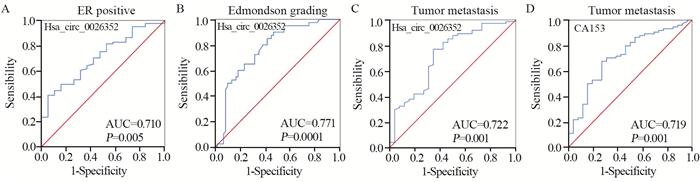
单因素ANOVA分析及ROC曲线结果显示,Hsa_circ_0026352的相对表达在CA153阳性、CEA阳性、ER阳性、肿瘤是否转移、TNM分期、Edmondson分级中分布差异有统计学意义(均P < 0.05),见表 3。ROC曲线分析结果显示,Hsa_circ_0026352在乳腺癌患者中鉴别出ER阳性的AUC为0.710(P < 0.01),鉴别乳腺癌Edmondsen分级的AUC为0.771(P < 0.01);鉴别乳腺肿瘤转移的AUC为0.722(P < 0.01)。与之相比,本组资料中CA153鉴别乳腺肿瘤转移的AUC为0.719(P < 0.01),见图 4。结果表明,Hsa_circ_0026352诊断乳腺癌转移的能力与CA153相当,Hsa_circ_0026352具备成为乳腺癌转移、ER阳性及临床分期辅助外周血分子标志物的潜力。
|
|
| A: Hsa_circ_0026352 and ER positive; B: Hsa_circ_0026352 and Edmondson grading of breast cancer; C: Hsa_circ_0026352 and breast cancer metastasis; D: CA153 and breast cancer metastasis. 图 4 Hsa_circ_0026352用于乳腺癌患者外周血分子标志物的ROC分析 Figure 4 ROC analysis of Hsa_circ_0026352 in peripheral blood of breast cancer patients |
乳腺癌的高发病率和高复发率严重威胁着人类的健康,早期诊断和分型有助于乳腺癌患者的治疗。circRNA是一种非编码RNA,其通过外显子或内含子首尾相连形成一个闭合环状的结构,没有3'和5'端[9]。这种结构的特点赋予circRNA不易被降解的特性,无论在RNA酶作用、煮沸、反复冻融或长期保存等多种影响下,其表达具备稳定性[10]。此外,circRNA在大多数癌组织及血浆中的表达具有组织特异性和疾病特异性[11-12]。这些特性使circRNA有可能成为许多疾病,特别是癌症早期诊断和预后的生物标志物。
本研究使用circRNA芯片筛选乳腺癌与癌旁组织中差异表达的circRNA,在差异倍数≥8,P < 0.01,荧光值≥1 000,来源基因位于肿瘤相关的信号通路,最终确定Hsa_circ_0026352满足筛选要求。其来源于核受体激素基因NR4A1(nuclear receptor subfamily 4 group A member 1)的外显子,有报道证实抑制NR4A1的表达能够促进乳腺癌细胞增殖和侵袭,并抑制乳腺癌细胞凋亡[13-14]。Hsa_circ_0026352结构序列上具有大量miRNA吸附位点及少量蛋白编码阅读框,其在乳腺癌患者组织及外周血中表达显著下调(FC≈9),基于NR4A1在乳腺癌细胞增殖及凋亡过程中的重要作用,我们推测Hsa_circ_0026352可能通过miRNA吸附功能或编码蛋白功能介入NR4A1基因对细胞增殖及凋亡的调控过程。ROC分析显示,Hsa_circ_0026352在乳腺癌患者组织检测中的AUC为0.881(P < 0.01),在外周血检测中的AUC为0.826(P < 0.01)。这证实Hsa_circ_0026352具备作为乳腺癌外周血诊断标志物的潜力。circRNA在肿瘤诊断标志物的应用潜力已在多篇报道中得到证实。Gao等发现Hsa_circ_0006528在乳腺癌中表达上调,且其异常表达与乳腺癌的化疗耐药密切相关,可能作为化疗耐药预测标志物[15]。Yin等研究发现Hsa_circ_0001785能够作为乳腺癌的诊断标志物[16]。而本研究首次报道了Hsa_circ_0026352与乳腺癌的关系。
确定Hsa_circ_0026352在乳腺癌诊断中的价值后,我们进一步探讨了乳腺癌患者临床特征中的分布和相关性,结果显示,作为检测指标,Hsa_circ_0026352在外周血中的相对表达与CA153水平呈显著负相关(P < 0.05),并且当患者以CA153是否阳性分组后,Hsa_circ_0026352的表达差异显著(P < 0.05)。CA153是乳腺癌最重要的特异性标志物之一,是乳腺癌诊断、转移、监测术后复发的最佳指标[17]。但是,只有30%~60%的乳腺癌患者可见CA153明显升高,其在转移性乳腺癌最高达80%[18]。因此,Hsa_circ_0026352可能作为CA153的协同监测指标,可能有效提高CA153的诊断准确性。此外,我们发现Hsa_circ_0026352的相对表达在CA153阳性、ER阳性、肿瘤是否转移、Edmondson分级中分布差异显著(P < 0.05)。ROC曲线分析,Hsa_circ_0026352可能作为ER阳性、乳腺肿瘤转移、乳腺癌分期监测的重要循环系统标志物。在本组数据中,其鉴别乳腺癌转移的效果与CA153相当。
综上所述,本研究显示Hsa_circ_0026352在乳腺癌外周血中特异性表达下调,其具备成为乳腺癌诊断、乳腺癌转移、ER阳性监测标志物的潜力。而其在乳腺癌发生、转移中的作用机制还需进一步研究。
作者贡献
张旭:资金项目支持、设计实施实验、撰写及修改论文
马芳:整理资料、数据信息录入和分组
纳玮、李晓菡、黄琦、于晶晶:参与实验
王嘉:数据收集
王立斌:资金项目支持、统筹实验、撰写及修改论文、技术支持
| [1] |
Waks AG, Winer EP. Breast Cancer Treatment: A Review[J]. JAMA, 2019, 321(3): 288-300. DOI:10.1001/jama.2018.19323 |
| [2] |
Coughlin SS. Social determinants of breast cancer risk, stage, and survival[J]. Breast Cancer Res Treat, 2019, 177(3): 537-548. DOI:10.1007/s10549-019-05340-7 |
| [3] |
Emens LA. Breast cancer immunotherapy: facts and hopes[J]. Clin Cancer Res, 2018, 24(3): 511-520. DOI:10.1158/1078-0432.CCR-16-3001 |
| [4] |
Soslau G. Circular RNA (circRNA) was an important bridge in the switch from the RNA world to the DNA world[J]. J Theor Biol, 2018, 447: 32-40. DOI:10.1016/j.jtbi.2018.03.021 |
| [5] |
Zhang M, Xin Y. Circular RNAs: a new frontier for cancer diagnosis and therapy[J]. J Hematol Oncol, 2018, 11(1): 21. DOI:10.1186/s13045-018-0569-5 |
| [6] |
Su M, Xiao Y, Ma J, et al. Circular RNAs in Cancer: emerging functions in hallmarks, stemness, resistance and roles as potential biomarkers[J]. Mol Cancer, 2019, 18(1): 90. DOI:10.1186/s12943-019-1002-6 |
| [7] |
宗曾艳, 孔凡虹, 王萌萌, 等. 环状RNA在恶性肿瘤发生发展和诊疗中的研究进展[J]. 国际检验医学杂志, 2020, 41(1): 98-103. [Zong ZY, Kong FH, Wang MM, et al. Research progress of circular RNA in carcinogenesisi, development, diagnosis and treatment of malignant tumors[J]. Guo Ji Jian Yan Yi Xue Za Zhi, 2020, 41(1): 98-103. DOI:10.3969/j.issn.1673-4130.2020.01.032] |
| [8] |
Cserni G, Chmielik E, Cserni B, et al. The new TNM-based staging of breast cancer[J]. Virchows Arch, 2018, 472(5): 697-703. DOI:10.1007/s00428-018-2301-9 |
| [9] |
Li Z, Ruan Y, Zhang H, et al. Tumor-suppressive circular RNAs: Mechanisms underlying their suppression of tumor occurrence and use as therapeutic targets[J]. Cancer Sci, 2019, 110(12): 3630-3638. DOI:10.1111/cas.14211 |
| [10] |
Zhang HD, Jiang LH, Sun DW, et al. CircRNA: a novel type of biomarker for cancer[J]. Breast cancer, 2018, 25(1): 1-7. DOI:10.1007/s12282-017-0793-9 |
| [11] |
Ojha R, Nandani R, Chatterjee N, et al. Emerging Role of Circular RNAs as Potential Biomarkers for the Diagnosis of Human Diseases[J]. Adv Exp Med Biol, 2018, 1087: 141-157. DOI:10.1007/978-981-13-1426-1_12 |
| [12] |
Lei B, Tian Z, Fan W, et al. Circular RNA: a novel biomarker and therapeutic target for human cancers[J]. Int J Med Sci, 2019, 16(2): 292-301. DOI:10.7150/ijms.28047 |
| [13] |
Wu L, Chen L. Characteristics of Nur77 and its ligands as potential anticancer compounds[J]. Mol Med Rep, 2018, 18(6): 4793-4801. DOI:10.3892/mmr.2018.9515 |
| [14] |
Hedrick E, Li X, Cheng Y, et al. Potent inhibition of breast cancer by bis-indole-derived nuclear receptor 4A1 (NR4A1) antagonists[J]. Breast Cancer Res Treat, 2019, 177(1): 29-40. DOI:10.1007/s10549-019-05279-9 |
| [15] |
Gao D, Zhang X, Liu B, et al. Screening circular RNA related to chemotherapeutic resistance in breast cancer[J]. Epigenomics, 2017, 9(9): 1175-1188. DOI:10.2217/epi-2017-0055 |
| [16] |
Yin WB, Yan MG, Fang X, et al. Circulating circular RNA hsa_circ_0001785 acts as a diagnostic biomarker for breast cancer detection[J]. Clin Chim Acta, 2018, 487: 363-368. DOI:10.1016/j.cca.2017.10.011 |
| [17] |
Li X, Dai D, Chen B, et al. Clinicopathological and Prognostic Significance of Cancer Antigen 15-3 and Carcinoembryonic Antigen in Breast Cancer: A Meta-Analysis including 12, 993 Patients[J]. Dis Markers, 2018, 2018: 9863092. DOI:10.1155/2018/9863092 |
| [18] |
赖玉玲, 黄丽芳, 许晓峰. 血清多项指标联合检测对乳腺癌的鉴别诊断和预后评估作用[J]. 临床血液学杂志(输血与检验), 2019, 32(6): 922-926. [Lai YL, Huang LF, Xu XF. Role of serum multi-index combined detection in differential diagnosis and prognosis evaluation of breast cancer[J]. Lin Chuang Xue Ye Xue Za Zhi (Shu Xue Yu Jian Yan), 2019, 32(6): 922-926.] |
 2021, Vol. 48
2021, Vol. 48