文章信息
- 脑胶质瘤中CXC趋化因子表达的预后意义及免疫细胞浸润分析
- Prognostic Significance and Immune Cell Infiltration Analysis of CXC Chemokines in Glioblastoma
- 肿瘤防治研究, 2021, 48(4): 347-353
- Cancer Research on Prevention and Treatment, 2021, 48(4): 347-353
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2020.21.0435
- 收稿日期: 2020-04-29
- 修回日期: 2020-08-10
2. 010010 呼和浩特,内蒙古大学生命科学学院
2. College of Life Sciences, Inner Mongolia University, Hohhot 010010, China
多形性胶质母细胞瘤(glioblastoma, GBM)是发病率和死亡率最高的恶性脑肿瘤,5年生存率仅4.7%,患者预后不足15月[1]。因此,寻找有效的生物标志物和治疗靶点对胶质母细胞瘤患者的诊断和治疗十分重要。CXC趋化因子家族共包含16个成员,参与诸多生物学过程,如血管生成[2]、肿瘤生长[3]以及免疫响应[4]等。CXC趋化因子能够通过免疫途径影响肿瘤发生和发展[2, 5],也可以作为多种肿瘤的预后标志物[6-7]。
近年来,有文献报道CXC趋化因子在GBM中能够发挥重要作用[8-9],但其在GBM治疗靶点和预后标志物方面的研究仍然有待深入。生物信息学分析技术的不断发展及相关数据库的建立为分析CXC趋化因子提供了工具和平台。本研究对CXC趋化因子在GBM中的表达进行全面的生物信息学分析,利用TCGA数据库中GBM的表达谱数据和临床表型数据,结合大型公共数据库评估CXC趋化因子作为GBM治疗靶标和预后生物标志物的潜力,旨在为GMB患者早期诊断及治疗方案提供一定的帮助。
1 资料与方法 1.1 查看CXC趋化因子在GBM的表达情况利用ONCOMINE[10]查看CXC趋化因子家族中16个成员在多种肿瘤中的表达情况。利用GEPIA(http://gepia.cancer-pku.cn)数据库[11]分别对16个CXC趋化因子家族成员在肿瘤组织样本(n=163)和正常组织样本(n=207)中的表达情况进行差异分析,使用ANOVA方差分析对差异表达结果进行统计检验,P < 0.05为差异具有统计学意义。
1.2 分析CXC趋化因子对GBM患者预后的影响利用GEPIA计算差异表达CXCL趋化因子的患者总生存期(overall survival, OS)和无病生存期(disease-free survival, DFS)。使用Log rank检验对生存率曲线进行统计分析,以P < 0.05为差异有统计学意义。
1.3 计算CXC趋化因子的表达相关性使用R软件corrplot程序包分析CXC趋化因子在GBM中表达的相关性,使用Pearson相关系数说明和验证分析结果。
1.4 构建CXC趋化因子及其邻近蛋白的PPI网络在String数据库[12]导入差异表达的CXC趋化因子构建PPI网络,根据连接分数得到28个邻近结点(combine score > 0.4)。使用Cytoscape软件对网络进行分析,根据度(连接蛋白的数量)得出网络中的核心基因。
1.5 利用DAVID工具进行GO功能富集分析和KEGG信号通路富集分析使用DAVID(http://david.ncifcrf.gov)在线分析工具对PPI网络中的所有结点进行GO功能富集分析[13],分别得到CXC趋化因子及其邻近基因在生物学过程(biological process, BP)和途径、细胞组分(cellular component, CC)和定位及分子功能(molecular function, MF)的富集情况。使用DAVID分析工具进行KEGG通路富集分析,富集条件以P < 0.05为阈值。
1.6 预测CXC趋化因子的关键转录调控因子和激酶靶标通过TRRUST(https://www.grnpedia.org/trrust/)预测CXC趋化因子的关键转录调控因子[14]。使用LinkedOmics(http://www.linkedomics.org/)预测差异表达趋化因子的激酶靶标。
1.7 分析CXC趋化因子在GBM的免疫浸润水平使用TIMER(https://cistrome.shinyapps.io/timer/)数据库查询CXC趋化因子的表达与肿瘤纯度和免疫细胞浸润程度的关系[15]。在TIMER中,使用Pearson相关性分析计算肿瘤纯度与基因表达之间的相关性,使用Spearman相关性分析计算6种免疫细胞浸润程度与基因表达的关系,采用偏相关分析方法排除肿瘤纯度对免疫细胞浸润分析结果的影响。
2 结果 2.1 CXC趋化因子在GBM中的异常表达ONCOMINE数据库分析结果表明,CXCL8/9/ 10/11在肿瘤中的表达明显上调,见图 1A。GEPIA数据库分析结果表明,CXCL2/3/8/9/10/11/16在GBM中的表达上调,且差异有统计学意义(P < 0.05),见图 1B。此外,CXCL6/13/17在GBM和正常组织中表达差异并不显著(P > 0.05),CXCL14在GBM中则出现表达下调的趋势,CXCL1/4/5/7/12在GBM中的表达高于正常组织,但差异均无统计学意义(P > 0.05)。以上表明,CXC趋化因子在GBM中普遍高表达。
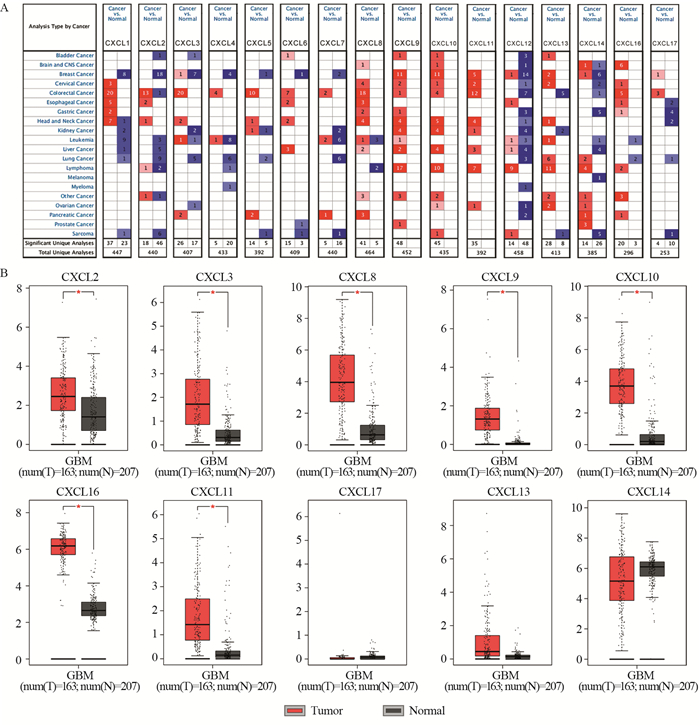
|
| *: P < 0.05. 图 1 CXC趋化因子在泛癌及脑胶质瘤中的表达 Figure 1 Expression of CXC chemokines in pan-cancer and glioblastoma (GBM) |
OS分析曲线结果表明,CXCL3(P=0.021)、CXCL8(P=0.049)与GBM患者的不良预后存在显著相关性。DFS分析曲线显示,CXCL1(P=0.012)、CXCL5(P=0.0078)、CXCL6(P=0.015)和CXCL14(P=0.031)的高表达会导致GBM患者无病生存期缩短,见图 2。
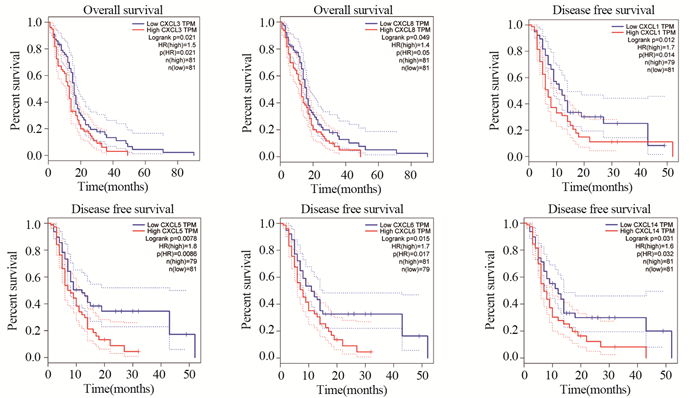
|
| 图 2 脑胶质瘤患者差异表达趋化因子的总生存期和无疾病生存期分析 Figure 2 Overall survival curves and disease-free survival curves of GBM patients with differentially-expressed CXC chemokines |
相关性分析结果表明,CXCL2、CXCL3和CXCL8的表达存在较高的相关性(r2, 3=0.86, r2, 8=0.62, r3, 8=0.76);CXCL9、CXCL10和CXCL11也存在较高的相关性(r9, 10=0.63, r9, 11=0.64, r10, 11= 0.76),构成了另一个表达模块。在预测的与CXC趋化因子相关的28个邻近基因中,一些基因如IL4、IL6、IL10、CCL2、CCL4、STAT3等,均与免疫调节有关,见图 3。
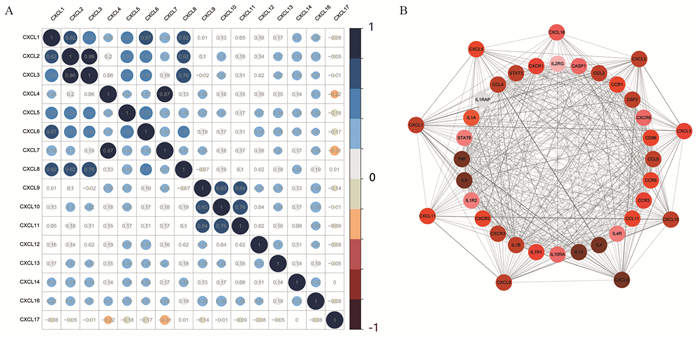
|
| A: r > 0, positively correlated; r < 0, negatively correlated; r=1, completely correlated; B: the color of circles indicated the number of binding nodes (n) 图 3 CXC趋化因子的表达相关性分析(A)及其28个邻近基因的PPI网络(B) Figure 3 Correlation analysis of CXC chemokines expression(A) and PPI network of 28 neighbor genes(B) |
GO功能富集分析显示,BP分析中,基因主要富集在趋化因子介导信号通路、免疫应答、细胞趋化作用和炎性反应响应等与免疫有关的功能上,见图 4A。细胞外区域、胞外基质、细胞质膜及细胞膜外表面则是CC中富集最高的成分,见图 4B。而在分子功能中,CXC趋化因子及其邻近基因主要富集于趋化因子受体结合、细胞因子活性、趋化因子活性和CXCR趋化因子受体结合等趋化因子调节功能,见图 4C。KEGG通路分析显示,基因集主要富集在细胞因子受体相互作用、趋化因子信号通路、NOD-like受体信号通路和TNF信号通路等免疫相关的信号通路上,见图 4D。
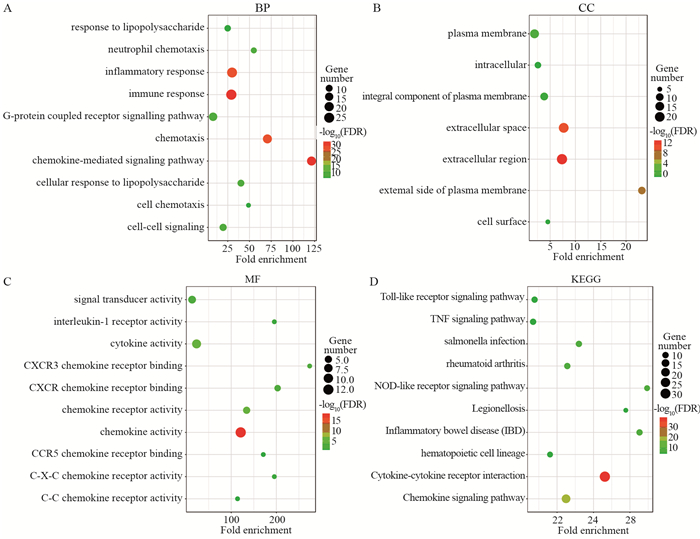
|
| 图 4 PPI网络中基因的GO分析和KEGG通路富集分析 Figure 4 GO analysis and KEGG pathway enrichment analysis of genes in PPI network |
从TRRUST数据库的分析结果中,我们得到两个参与调控CXC趋化因子的转录因子,分别是RELA和NFKB1,重要的是,RELA和NFKB1共同参与了CXCL2、CXCL8和CXCL10的调控。LinkedOmics数据库对CXC趋化因子激酶靶点的预测结果表明,淋巴细胞特异性激酶(lymphocyte-specific kinase)是CXCL3/9/10/11的激酶靶点;此外,Rad3相关蛋白(Rad3-related protein, ATR)被证明为CXCL2的激酶靶点;有丝分裂检验点激酶1(CHEK1)和LCK/YES相关蛋白酪氨酸激酶(Lck/Yes related novel protein tyrosine kinase, LYN)则分别是CXCL8和CXCL16的激酶靶点,见表 1。
TIMER数据库分析结果表明,GBM中CXC趋化因子与肿瘤微环境中6种免疫细胞的浸润水平呈一定相关性。其中,CXCL2表达与中性粒细胞、树突状细胞的浸润呈正相关,与CD8+T细胞浸润呈负相关性,见图 5A。CXCL3表达与中性粒细胞、树突状细胞的浸润呈正相关,与B细胞和巨噬细胞呈负相关;CXCL8(IL8)表达与中性粒细胞、树突状细胞的浸润呈正相关性,与B细胞、CD8+T细胞和巨噬细胞呈负相关,见图 5B;CXCL9的表达与B细胞和树突状细胞的浸润呈正相关,与CD8+T细胞浸润、CD4+T细胞的表达呈负相关;CXCL10的表达与B细胞、中性粒细胞和树突状细胞的浸润呈正相关,与CD8+T细胞浸润、CD4+T细胞的表达呈负相关,见图 5C;与CXCL10类似,CXCL11与B细胞和树突状细胞的浸润呈正相关,与CD8+T细胞浸润、CD4+T细胞的表达呈负相关;而CXCL16的表达仅与CD8+T细胞的浸润呈负相关,与B细胞、CD4+T细胞、巨噬细胞和树突状细胞的浸润均呈正相关。
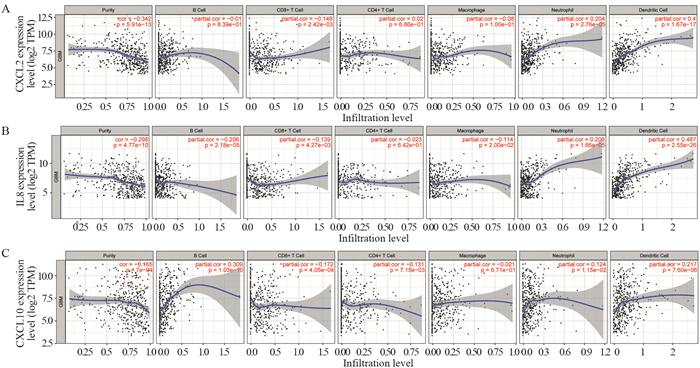
|
| 图 5 差异表达CXC趋化因子与免疫细胞浸润的相关性分析 Figure 5 Correlation between differently-expressed CXC chemokines and immune cell infiltration |
CXC趋化因子在免疫细胞的成熟、分化和迁移过程中发挥重要作用[4]。在肿瘤中,CXC趋化因子被证明对肿瘤微环境中免疫细胞浸润水平和免疫应答产生影响,从而调节肿瘤的发生和发展[16-17]。在GBM中,胶质瘤细胞与基质细胞的通讯会对各类细胞中趋化因子的表达产生影响,从而为肿瘤的发生、侵袭和转移提供特定的微环境[18-19]。CXC趋化因子对肿瘤微环境和肿瘤免疫具有十分重要的意义,其在GBM的靶点研究、预后及免疫治疗等方面具有广阔的研究前景[19]。然而,在GBM中CXC趋化因子的预后价值和作用机制尚不明确。本研究探讨了CXC趋化因子在GBM中的表达情况,结果表明GBM中有7个基因与正常组织有差异表达(CXCL2/3/8/9/10/11/16的表达显著上调)。预后分析结果显示CXCL3和CXCL8的异常表达与GBM患者不良预后显著相关。此外,Bruyère等研究结果还发现CXCL2/3/8在GBM组织中表达上调会影响使用替莫唑胺化疗的疗效[8]。这些结果表明,CXCL3和CXCL8的异常表达对GBM的发展、生存预后和治疗都有影响,但具体机制尚不明确。
由于多种趋化因子在GBM中的表达具有显著差异,本研究进一步分析了它们在GBM中的功能。结果表明CXC趋化因子之间存在或高或低的相关性,并形成了相关性网络,表明这些细胞因子在GBM的发生可能以组成网络的方式发挥作用。然后,我们构建由差异表达的CXC趋化因子及其邻近基因组成的PPI网络,得到了与其相互作用最为密切的28个邻近基因,并对PPI网络内的所有基因进行了GO富集分析和KEGG通路富集分析,以探求CXC趋化因子及其邻近基因主要参与的生物学功能和信号通路。富集分析结果发现,这些PPI调控网络中基因主要富集于趋化因子信号通路、细胞因子间受体相互作用以及TNF信号通路。有研究表明,趋化因子信号通路在多种癌症的增殖、血管生成、转移和免疫逃避中发挥重要作用[20-21],而TNF信号通路的激活也被证明对GBM的发展具有抑制作用[22]。由此得出,差异表达的CXC趋化因子对GBM具有潜在的调控作用。
为了探究CXC趋化因子在GBM中作为潜在的药物治疗靶点的作用,本研究还分析了CXC趋化因子的转录因子,发现RELA和NFKB1可能是调控CXC趋化因子的关键转录因子。RELA蛋白的磷酸化能够影响诸多疾病的进展,尤其是可以通过调节NF-κB信号通路影响炎性疾病和癌症[23-24]。另一项研究结果发现,在胰腺癌中RELA能够影响由癌基因诱导的细胞衰老,从而抑制肿瘤细胞增殖[25]。NFKB1能够通过减少NF-κB信号通路的异常激活抑制多种肿瘤的发生和发展[26]。此外,Plaisier等通过分析后预测NFKB1能够协同ETV6抑制GBM的增殖[27]。
此外,我们进一步预测了CXC趋化因子的激酶靶点,作为CXCL3/9/10/11的激酶靶点,LCK是调节T细胞发育和T细胞应答的关键因子之一。LCK也在多种肿瘤中表达,如乳腺癌、结直肠癌、胆管癌和肺癌,并且可能是这些肿瘤的诊断标志物和治疗靶点[28]。在GBM中,LCK被证实与GBM细胞顺铂耐药性的增强有关,不仅如此,抑制LCK的表达对GBM细胞的增殖能力和自我更新能力也会产生影响[29]。ATR激酶与细胞对基因组不稳定的响应和修复有密切联系,在肿瘤中ATR的激活能够增强药物化疗后细胞内的DNA损伤修复。目前,几种ATR抑制剂,如ETP46464、AZ20、VE822,在癌症小鼠模型体内的功效均得到了验证,临床上也针对ATR抑制剂的治疗敏感度开发了几种生物标志物,用于诊断合适的患者群体并采取对应治疗手段[30]。免疫细胞浸润已被证明可以影响肿瘤的发生和复发,并对肿瘤的免疫治疗和临床效果具有重要的作用[31-32]。CD4+T细胞能够识别癌抗原,而被激活的M1巨噬细胞可以抑制肿瘤生长[33]。本研究发现差异表达CXC趋化因子与六种免疫细胞(B细胞、CD8+T细胞、CD4+T细胞、巨噬细胞、中性粒细胞和树突细胞)的浸润程度具有相关性,表明CXC趋化因子的表达差异对GBM的免疫微环境具有影响,并调控肿瘤的免疫状态,为肿瘤发生和癌症治疗的免疫机制提供新的见解和思路。但是转录组水平的分析并不能完全解释免疫组成和免疫状态对肿瘤细胞的影响,仅能体现细胞的整体水平,因此还需要更多的实验去验证此结果。
综上所述,我们通过分析CXC趋化因子对GBM患者的预后意义,发现CXCL3/8能够作为GBM患者潜在的预后标志物。此外,根据免疫细胞浸润相关性分析结果,提示CXC趋化因子在GBM免疫治疗方面也具有研究价值。转录因子和激酶靶点的分析预测则为进一步研究CXC趋化因子在GBM中的作用机制提供了参考,期望以上分析结果能够为GBM治疗提供新的免疫治疗靶点及预后标志物,帮助临床医生为GBM患者选择合适的治疗方案。
作者贡献:
刘芳远:数据收集及统计分析,论文撰写
苏秀兰:实验思路设计,论文修改
| [1] |
Laperriere N, Weller M, Stupp R, et al. Optimal management of elderly patients with glioblastoma[J]. Cancer Treat Rev, 2013, 39(4): 350-357. DOI:10.1016/j.ctrv.2012.05.008 |
| [2] |
Bikfalvi A, Billottet C. The CC and CXC chemokines: major regulators of tumor progression and the tumor microenvironment[J]. Am J Physiol Cell Physiol, 2020, 318(3): C542-C554. DOI:10.1152/ajpcell.00378.2019 |
| [3] |
Balkwill F. Cancer and the chemokine network[J]. Nat Rev Cancer, 2004, 4(7): 540-550. DOI:10.1038/nrc1388 |
| [4] |
Amedei A, Prisco D, D'Elios MM. The use of cytokines and chemokines in the cancer immunotherapy[J]. Recent Pat Anticancer Drug Discov, 2013, 8(2): 126-142. DOI:10.2174/1574892811308020002 |
| [5] |
Lee NH, Nikfarjam M, He H. Functions of the CXC ligand family in the pancreatic tumor microenvironment[J]. Pancreatology, 2018, 18(7): 705-716. DOI:10.1016/j.pan.2018.07.011 |
| [6] |
Cabrero-de Las Heras S, Martínez-Balibrea E. CXC family of chemokines as prognostic or predictive biomarkers and possible drug targets in colorectal cancer[J]. World J Gastroenterol, 2018, 24(42): 4738-4749. DOI:10.3748/wjg.v24.i42.4738 |
| [7] |
Abdelmageed M, Ali H, Olsson L, et al. The Chemokine CXCL16 Is a New Biomarker for Lymph Node Analysis of Colon Cancer Outcome[J]. Int J Mol Sci, 2019, 20(22): 5793. DOI:10.3390/ijms20225793 |
| [8] |
Bruyère C, Mijatovic T, Lonez C, et al. Temozolomide-induced modification of the CXC chemokine network in experimental gliomas[J]. Int J Oncol, 2011, 38(5): 1453-1464. |
| [9] |
Fazi B, Proserpio C, Galardi S, et al. The Expression of the Chemokine CXCL14 Correlates with Several Aggressive Aspects of Glioblastoma and Promotes Key Properties of Glioblastoma Cells[J]. Int J Mol Sci, 2019, 20(10): 2496. DOI:10.3390/ijms20102496 |
| [10] |
Rhodes DR, Yu J, Shanker K, et al. ONCOMINE: a cancer microarray database and integrated data-mining platform[J]. Neoplasia, 2004, 6(1): 1-6. DOI:10.1016/S1476-5586(04)80047-2 |
| [11] |
Tang ZF, Li CW, Kang BX, et al. GEPIA: a web server for cancer and normal gene expression profiling and interactive analyses[J]. Nucleic Acids Res, 2017, 45(W1): W98-W102. DOI:10.1093/nar/gkx247 |
| [12] |
Szklarczyk D, Gable AL, Lyon D, et al. STRING v11: protein-protein association networks with increased coverage, supporting functional discovery in genome-wide experimental datasets[J]. Nucleic Acids Res, 2019, 47(D1): D607-D613. DOI:10.1093/nar/gky1131 |
| [13] |
Huang da W, Sherman BT, Lempicki RA. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J]. Nat Protoc, 2009, 4(1): 44-57. DOI:10.1038/nprot.2008.211 |
| [14] |
Han H, Cho J W, Lee S, et al. TRRUST v2: an expanded reference database of human and mouse transcriptional regulatory interactions[J]. Nucleic Acids Res, 2018, 46(D1): D380-D386. DOI:10.1093/nar/gkx1013 |
| [15] |
Li T, Fan J, Wang B, et al. TIMER: A Web Server for Comprehensive Analysis of Tumor-Infiltrating Immune Cells[J]. Cancer Res, 2017, 77(21): e108-e110. DOI:10.1158/0008-5472.CAN-17-0307 |
| [16] |
Neagu M, Constantin C, Longo C. Chemokines in the melanoma metastasis biomarkers portrait[J]. J Immunoassay Immunochem, 2015, 36(6): 559-566. DOI:10.1080/15321819.2015.1035593 |
| [17] |
Liang K, Liu Y, Eer D, et al. High CXC Chemokine Ligand 16 (CXCL16) Expression Promotes Proliferation and Metastasis of Lung Cancer via Regulating the NF-kappaB Pathway[J]. Med Sci Monit, 2018, 24: 405-411. DOI:10.12659/MSM.906230 |
| [18] |
Broekman ML, Maas SLN, Abels ER, et al. Multidimensional communication in the microenvirons of glioblastoma[J]. Nat Rev Neurol, 2018, 14(8): 482-495. DOI:10.1038/s41582-018-0025-8 |
| [19] |
Strepkos D, Markouli M, Klonou A, et al. Insights in the immunobiology of glioblastoma[J]. J Mol Med(Berl), 2020, 98(1): 1-10. DOI:10.1007%2Fs00109-019-01835-4 |
| [20] |
Roy I, Getschman AE, Volkman BF, et al. Exploiting agonist biased signaling of chemokines to target cancer[J]. Mol Carcinog, 2017, 56(3): 804-813. DOI:10.1002/mc.22571 |
| [21] |
Zhuang HJ, Cao G, Kou CH, et al. CCL2/CCR2 axis induces hepatocellular carcinoma invasion and epithelial-mesenchymal transition in vitro through activation of the Hedgehog pathway[J]. Oncol Rep, 2018, 39(1): 21-30. |
| [22] |
Guo G, Gong K, Puliyappadamba VT, et al. Efficacy of EGFR plus TNF inhibition in a preclinical model of temozolomide-resistant glioblastoma[J]. Neuro Oncol, 2019, 21(12): 1529-1539. DOI:10.1093/neuonc/noz127 |
| [23] |
Lu X, Yarbrough WG. Negative regulation of RelA phosphorylation: emerging players and their roles in cancer[J]. Cytokine Growth Factor Rev, 2015, 26(1): 7-13. DOI:10.1016/j.cytogfr.2014.09.003 |
| [24] |
Kabacaoglu D, Ruess DA, Ai J, et al. NF-kappaB/Rel Transcription Factors in Pancreatic Cancer: Focusing on RelA, c-Rel, and RelB[J]. Cancers(Basel), 2019, 11(7): 937. |
| [25] |
Lesina M, Wörmann SM, Morton J, et al. RelA regulates CXCL1/CXCR2-dependent oncogene-induced senescence in murine Kras-driven pancreatic carcinogenesis[J]. J Clin Invest, 2016, 126(8): 2919-2932. DOI:10.1172/JCI86477 |
| [26] |
Cartwright T, Perkins ND, Wilson CL. NFKB1: a suppressor of inflammation, ageing and cancer[J]. FEBS J, 2016, 283(10): 1812-1822. DOI:10.1111/febs.13627 |
| [27] |
Plaisier CL, O'Brien S, Bernard B, et al. Causal Mechanistic Regulatory Network for Glioblastoma Deciphered Using Systems Genetics Network Analysis[J]. Cell Syst, 2016, 3(2): 172-186. DOI:10.1016/j.cels.2016.06.006 |
| [28] |
Bommhardt U, Schraven B, Simeoni L. Beyond TCR Signaling: Emerging Functions of Lck in Cancer and Immunotherapy[J]. Int J Mol Sci, 2019, 20(14): 3500. DOI:10.3390/ijms20143500 |
| [29] |
Kim RK, Yoon CH, Hyun KH, et al. Role of lymphocyte-specific protein tyrosine kinase (lck) in the expansion of glioma-initiating cells by fractionated radiation[J]. Biochem Biophys Res Commun, 2010, 402(4): 631-636. DOI:10.1016/j.bbrc.2010.10.072 |
| [30] |
Lecona E, Fernandez-Capetillo O. Targeting ATR in cancer[J]. Nat Rev Cancer, 2018, 18(9): 586-595. DOI:10.1038/s41568-018-0034-3 |
| [31] |
Liu X, Wu S, Yang Y, et al. The prognostic landscape of tumor-infiltrating immune cell and immunomodulators in lung cancer[J]. Biomed Pharmacother, 2017, 95: 55-61. DOI:10.1016/j.biopha.2017.08.003 |
| [32] |
Galli F, Aguilera JV, Palermo B, et al. Relevance of immune cell and tumor microenvironment imaging in the new era of immunotherapy[J]. J Exp Clin Cancer Res, 2020, 39(1): 89. DOI:10.1186/s13046-020-01586-y |
| [33] |
Lin P, Guo Y N, Shi L, et al. Development of a prognostic index based on an immunogenomic landscape analysis of papillary thyroid cancer[J]. Aging (Albany NY), 2019, 11(2): 480-500. |
 2021, Vol. 48
2021, Vol. 48



