文章信息
- 基于TCGA数据库筛选和鉴定肺腺癌免疫治疗临床获益关键基因
- Screening and Identification of Key Genes for Clinical Benefit of Immunotherapy on Lung Adenocarcinoma Based on TCGA Database
- 肿瘤防治研究, 2020, 47(11): 839-845
- Cancer Research on Prevention and Treatment, 2020, 47(11): 839-845
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2020.20.0115
- 收稿日期: 2020-02-19
- 修回日期: 2020-09-10
2. 100853 北京,解放军总医院国宾一科;
3. 100853 北京,解放军总医院生物治疗科
2. Department of Special Ward, Chinese PLA General Hospital, Beijing 100853, China;
3. Department of Biotherapy, Chinese PLA General Hospital, Beijing 100853, China
肺癌作为目前国际、国内最常见的肿瘤之一,已经成为影响公共健康的重要社会及医学热点问题;其中肺腺癌是肺癌最常见的病理类型。最近,随着免疫治疗的应用,尤其是程序性死亡受体1(programmed cell death protein 1, PD-1)单抗的应用,给晚期肺腺癌带来了新的突破及希望。然而,在免疫治疗疗效预测方面中,目前广泛认为PD-L1表达水平可以作为肺腺癌帕博利珠单抗的疗效预测指标,但是在临床实践中单纯依靠PD-L1表达水平来预测能否从免疫治疗中获益存在一定的局限性。而新型的疗效预测标志物,比如肿瘤突变负荷等,在预测免疫治疗的疗效方面还存在一定的争议,提示寻找新的免疫治疗疗效预测标志物是目前精准免疫治疗迫切需要解决的问题。
近年来随着基因测序技术的发展,全基因组测序日愈成熟,那么对于免疫治疗持续临床获益的患者与未能获益的患者之间的基因突变谱特征方面,是否存在差异及其这些差异基因突变谱能否成为预测疗效的新的生物标志物等问题,值得进一步研究。本研究正是基于此出发点,探索帕博利珠单抗在肺腺癌免疫治疗中持久临床获益关键基因突变谱的筛选及相关信号通路分析。
1 资料与方法 1.1 肺腺癌基因突变数据集筛选在TCGA数据库检索肺腺癌与免疫治疗相关数据集,共筛选到三个数据集,包括Lung Adenocarcinoma(MSKCC, Science 2015)、Non-small Cell Lung Cancer(MSK, Cancer Cell 2018)和Non-small Cell Lung Cancer (MSKCC, J Clin Oncol 2018),按照如下纳入及排除标准进行筛选。
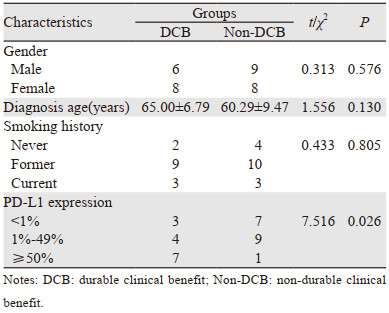
纳入标准:(1)晚期肺腺癌患者;(2)免疫治疗均为帕博利珠单抗单药治疗;(3)评估标准采用ir-RECIST(immune-related response evaluation criteria in solid tumors, ir-RECIST)标准;(4)临床指标定义:应用免疫治疗无进展生存期(progression-free survival, PFS)超过6个月为持久临床获益组(durable clinical benefit, DCB),反之定义为非持久临床获益组(non-durable clinical benefit, Non-DCB);(5)全外显子测序。剔除标准:PD-1联合CTLA-4单抗数据集(MSK, Cancer Cell 2018),其他的PD-1单抗及PD-L1单抗治疗患者(MSKCC, J Clin Oncol 2018);肺鳞癌等其他病理类型肺癌患者;经筛选选定Lung Adenocarcinoma(MSKCC, Science 2015)为研究对象,该数据集共35例患者,剔除1例未做基因突变测序分析患者及随访未超过6个月无法评估的3例,共31例患者纳入研究,其中Non-DCB组17例,DCB组14例。同时分析两组患者基线特征,并进一步分析两组之间在PD-L1表达、突变负荷及其PFS等方面的不同,为后续进一步筛选基因突变谱的差异奠定临床理论基础。
1.2 差异基因的筛选下载数据集肺腺癌患者临床特征资料,同时下载两组患者全基因组或全外显子组基因突变情况,将所有样本中基因突变频率在10%以上的基因突变谱情况纳入研究分析,根据疗效判断标准,探索DCB组和Non-DCB组基因突变特征的差异,并利用韦恩图在线分析软件(http://bioinformatics.psb.ugent.be/webtools/Venn/),对两组进行差异基因突变谱分析,筛选并比较两组患者之间在基因突变特征方面的不同。
1.3 基因功能注释与通路富集分析利用DAVID(https://david.ncifcrf.gov/)生物信息学分析软件,筛选以人为分析对象的研究,对DCB组患者基因突变差异基因,进行基因本体论(gene ontology, GO)及京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes, KEGG)富集通路分析。
1.4 蛋白互作网络分析及关键基因的筛选先采用String在线软件(https://string-db.org/cgi/input.pl),分析DCB组中差异基因的蛋白互作网络,剔除孤立节点,下载蛋白互作网络结果;再将互作网络结果导入Cytoscape软件进一步分析,该软件是一款图形化显示网络并进行分析和编辑的软件。本研究采用Cytoscape 3.7.2版本,对于DCB组中的关键基因网络进行分析;并应用cytoHubba插件,采用Closeness算法,筛选能够预测肺腺癌免疫治疗通路中的关键核心基因。
1.5 关键核心基因与预后分析GEPIA是一个肿瘤数据挖掘网站,数据来源主要基于TGCA和GTEx数据库。本研究通过GEPIA在线生存分析工具(http://gepia.cancer-pku.cn/detail.php),分析核心基因表达水平与肺腺癌预后的关系,并绘制生存曲线。
1.6 统计学方法应用SPSS 21.0软件进行统计分析。计量资料以(x±s)表示,两组间比较采用t检验;计数资料以例数和百分比表示,两组间比较采用χ2检验。所有统计检验均为双侧,P < 0.05为差异有统计学意义。
2 结果 2.1 患者基线特征分析患者基线特征分析显示,患者在年龄、性别、吸烟病史等方面差异无统计学意义,见表 1。DCB组具有更高的PD-L1表达及基因突变负荷(96.47±24.87 vs. 236.50±46.60, t=2.784),且差异有统计学意义(P=0.009)。
|
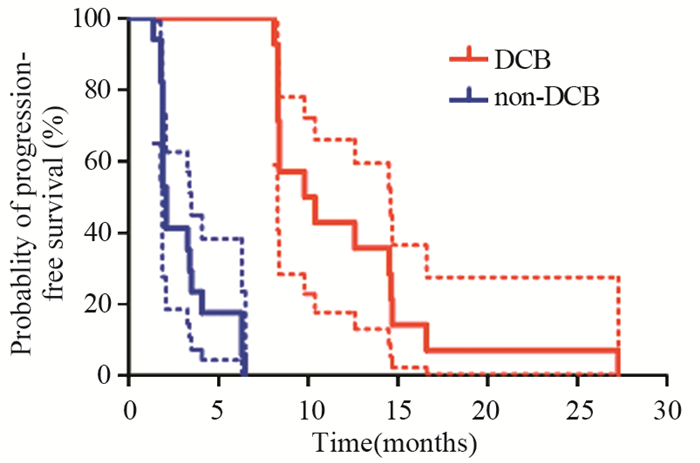
结果显示,DCB组的PFS明显优于non-DCB组,且两组之间差异有统计学意义(χ2=33.00, P < 0.0001),见图 1。
|
| 图 1 DCB组与Non-DCB组之间PFS的比较 Figure 1 Comparison of PFS between DCB group and Non-DCB group |
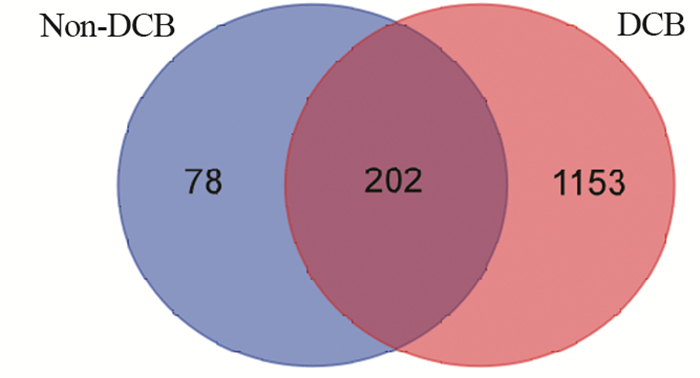
帕博利珠单抗在肺腺癌治疗中,基因突变谱在DCB组和Non-DCB组之间存在差异,对所有样本中基因突变频率20%以上的基因突变谱进一步分析,发现DCB组中更常见基因突变谱包括:FLNC、CRB2、KRAS、MUC4、TNRC18、CSMD3等基因。运用韦恩图展示两组之间差异基因突变谱情况,见图 2。
|
| Blue: genes mutation profile in the Non-DCB group; Pink: genes mutation profile in the DCB group; Brown: common mutant genes. 图 2 DCB组与Non-DCB组基因突变差异谱 Figure 2 Differential genes mutation signature between DCB group and Non-DCB group |
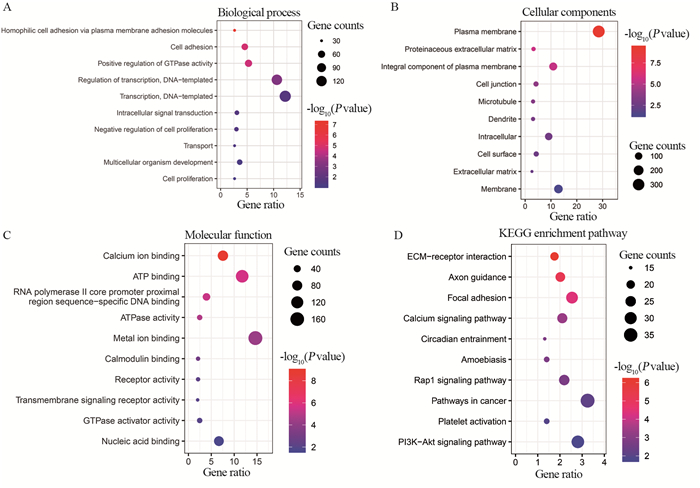
Gene Ontology可分为分子功能(molecular function)、生物学过程(biological process)和细胞组分(cellular component)三个部分。在分子功能方面,包括核酸绑定、GTPase激活活动、跨膜信号受体活性、受体的活动、钙调蛋白结合、RNA聚合酶Ⅱ核心启动子近端区序列特异性DNA结合、腺苷三磷酸酶活性等方面;在生物学过程方面,包括细胞增殖、多细胞生物的发展、细胞增殖的负调控、细胞内信号转导、转录的调节,DNA模板化、GTPase活性的调节、细胞黏附、嗜同性细胞通过质膜黏附分子黏附等;在细胞组分分析中,包括细胞膜的组成部分、细胞外基质、细胞表面、树突、微管、蛋白质的细胞外基质等方面,见图 3。
|
| 图 3 DCB组GO功能及KEGG富集分析 Figure 3 GO function and KEGG pathway enrichment in DCB group |
通过KEGG通路富集分析提示,这些候选基因主要参与了ECM受体交互作用,Ca2+通道、Rap1、PI3K-Akt信号通路等,提示这些信号通路可能为肺腺癌免疫治疗中的关键通路。同时通过KEGG Chord作图分析各个差异基因在各个通路中的分布情况,见图 3。
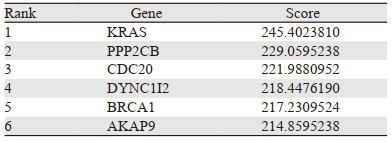
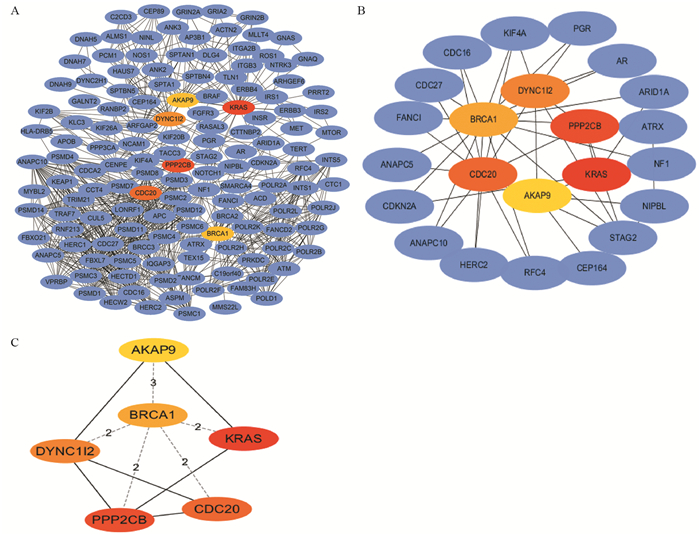
2.5 蛋白互作网络构建及关键基因分析通过String在线分析软件,DCB组中的1 153个差异基因进行蛋白互作网络分析,剔除孤立节点,并下载蛋白互作结果文件;再将蛋白互作文件导入Cytoscape 3.7.2软件,采用cytoHubba插件,进一步分析互作网络中的潜在关键基因。结果显示,基于Closeness算法,其中节点度最高的6个基因为:KRAS(Degree=245.402381)、PPP2CB(Degree=229.0595238)、CDC20(Degree=221.9880952)、DYNCLI2(Degree= 218.447619)、BRCA1(Degree=217.2309524)、AKAP9(Degree=214.8595238),见图 4、表 2。
|
| A: PPI networks of genes in DCB group; B: PPI networks of hub genes in DCB group; C: The top 6 hub genes were screened using the Closeness algorithm. 图 4 DCB组中蛋白-蛋白互作网络分析 Figure 4 PPI networks of hub genes in DCB group |
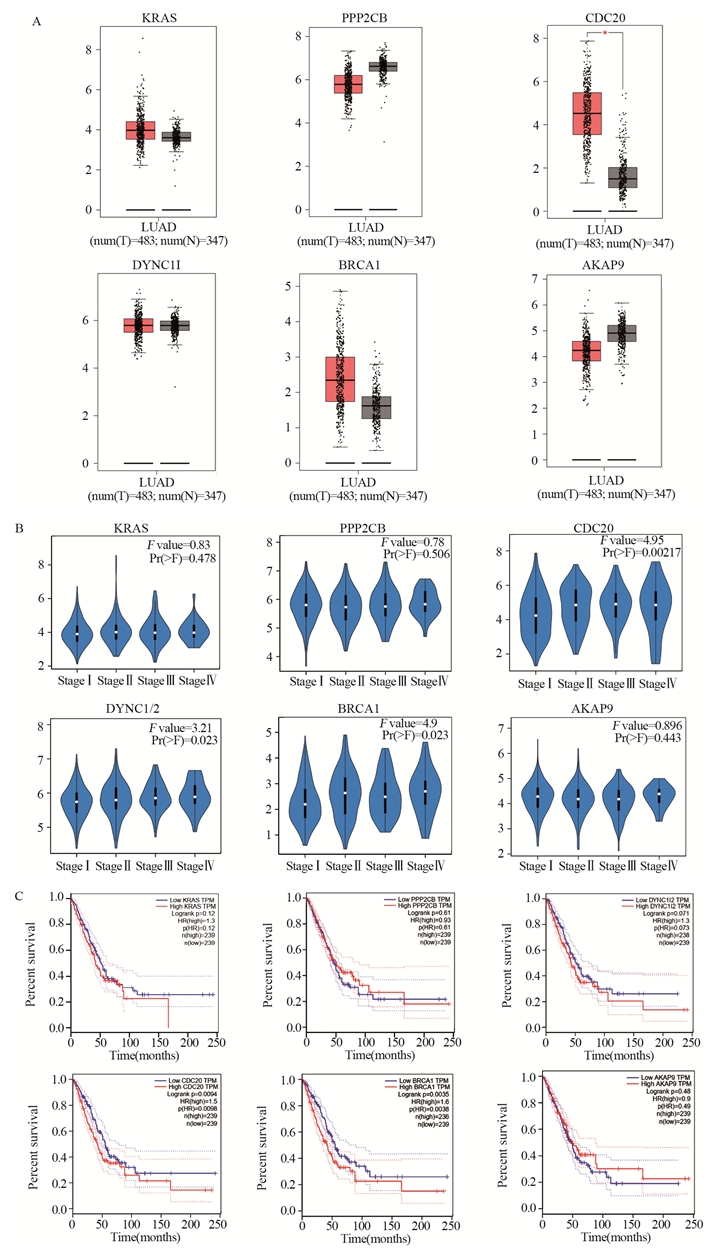
利用GEPIA在线分析软件,首先分析了这6个核心基因在肺腺癌及其癌旁正常组织的表达水平,结果显示,CDC20在肺腺癌中表达高于其癌旁正常组织,且差异具有统计学意义(P < 0.05),而其他关键核心基因未见表达差异;其次,分析这6个核心基因与临床分期关系,结果显示,CDC20、DYNC1I2、BRCA1等表达与临床分期呈正相关,且差异有统计学意义(P < 0.05)。最后,进一步分析6个关键核心基因与预后的关系,结果显示,CDC20、BRCA1等高表达组的中位总生存时间明显短于低表达组,且差异具有统计学意义。KRAS、PPP2CB、DYNC1I2、AKAP9等高表达组与低表达组之间中位总生存之间差异无统计学意义(P > 0.05),见图 5。
|
| A: hub gene expression in lung adenocarcinoma; B: the relation between hub genes and pathological staging of lung adenocarcinoma. C: survival analysis of patients with lung adenocarcinoma in relation to the expression of hub genes. 图 5 肺腺癌中6个关键核心基因与临床分期及预后的关系 Figure 5 Relation between hub genes and pathological staging, prognosis of lung adenocarcinoma |
肿瘤的免疫治疗,主要是通过激活人体自身抗肿瘤的免疫功能来杀死肿瘤细胞,具有疗效持久、不良反应小的特点[1],被诸多医学科学家寄予厚望。目前临床应用较多的是PD-1、PD-L1、CTLA-4单抗等药物[2],在肺癌的研究中,PD-1单抗已被批准用于晚期肺癌一线、二线等治疗[3-4]。本研究主要涉及帕博利珠单抗在肺腺癌中的疗效预测相关生物标志物分析。目前除了PD-L1可以比较明确地作为帕博利珠单抗免疫治疗疗效预测生物标志物之外[5],其他的疗效预测标志物,如肿瘤突变负荷、新抗原、人类白细胞抗原等新型生物标志物[6-8]能否有效指导免疫治疗尚存在争议,所以寻找新的疗效相关生物标志物是当前急需解决的临床难题之一。本研究正是基于此出发点,探索相对于帕博利珠单抗治疗疗效不佳的Non-DCB组而言,能否通过生物信息学分析DCB组的基因突变谱情况,进而更好的指导帕博利珠单抗治疗。
本研究结果显示,在帕博利珠单抗用于肺腺癌的治疗中,基因突变谱在DCB组和Non-DCB组之间存在差异,DCB组中肺腺癌患者具有更多的基因突变数量,更多的突变负荷及更长的PFS等。既往研究显示,具有更多突变负荷的肺癌患者,其免疫治疗的疗效会相对更好[8],与我们的研究结果一致。为了进一步寻找更具普遍性的基因突变特征,对样本中突变频率20%以上的基因突变谱进一步分析,发现DCB组中更常见基因突变谱包括:FLNC、CRB2、KRAS、MUC4、TNRC18、CSMD3等基因突变,提示临床工作中,结合全基因组分析,不同的分子突变特征有助于进一步指导并提高肺腺癌免疫治疗的疗效。
为进一步分析肺腺癌患者应用帕博利珠单抗治疗获益的可能机制,本研究应用在GO及KEGG富集通路分析,结果分析显示,获益人群中的突变谱基因,主要参与了跨膜信号受体活性信号转导、细胞增殖的调控、激酶的活性调节、Ca2+通道信号通路、Rap1通路、PI3K-Akt通路等生物学行为及通路;提示这些生物学功能及信号通路可能在肺腺癌免疫治疗通路中起到关键作用,但其确切的机制有待进一步基础实验验证。
通过Cytoscape软件分析,构建蛋白互作网络图形,筛选了DCB组中6个关键核心基因。进一步对这些核心基因与肺腺癌的预后的关系研究提示,CDC20和BRCA1等的高表达是肺腺癌患者预后不良的标志物。
已有多项研究提示CDC20的高表达与多种肿瘤的不良预后有关,包括前列腺肿瘤、肉瘤,乳腺癌等,其机制可能是参与了肿瘤的转移及侵袭等生物学过程[9-12],Zhang等在吸烟相关肺癌研究中发现CDC20的高表达与肺癌的预后相关[13],Yi等亦发现CDC20在肺癌中表达明显上调,其主要的突变形式是基因扩增,更重要的是,该基因的过表达与肺癌患者更短的生存期有关[14-15]。这些研究均与我们当前的研究结果一致。Budczies等研究发现,CDC20是癌症免疫反应中的潜在药物靶点[16],对这些靶点深入研究,有望提高免疫治疗的精准化。也在一定程度上提示,CDC20在肺癌免疫治疗的相关通路中起到了重要的作用。
本研究发现,BRCA1基因既往研究主要集中在胚系突变研究中,结果显示,伴有BRCA1基因突变的女性患者更容易发生胰腺肿瘤、乳腺癌、卵巢癌等[17-21],且对于伴有BRCA1突变患者,应用奥拉帕利靶向治疗,具有很好的靶向药物指导作用。而在本研究中,结果提示,对于肺腺癌患者,BRCA1高表达患者,具有更差的预后;Wang等研究提示[18],其可能的机制是通过TGF-β/PI3K/AKT/mTOR等信号通路参与了肿瘤的增殖及侵袭等生物学过程。但是对于kras基因突变问题,既往研究中提示为肺癌的预后不良生物标志物[22],与本研究结果不一致,其可能的原因一是研究样本的选择及样本量的不同,二是预后分析软件及算法等统计学方面的差异。
另外,本研究一个局限性在于样本量偏少,其主要原因在于,检索TCGA数据库,共检索肺腺癌免疫疗效预测的三个数据集,包括MSKCC, Science 2015, MSK, Cancer Cell 2018和MSKCC, J Clin Oncol 2018,但是MSK, Cancer Cell 2018数据集包含了鳞癌,免疫治疗药物是PD-1单抗联合CTLA-4单抗;另外,MSKCC, J Clin Oncol 2018数据集也包括了鳞癌及其他类型,治疗中除了含有帕博利珠单抗外,还包括纳武单抗及PD-L1单抗等。为了排除不同的PD-1单抗及免疫联合之间的效应差别,本研究只纳入统一MSKCC, Science 2015,统一的帕博利珠单抗及肺腺癌患者。所以某种程度上,是为了研究的严谨性,本研究排除了病理类型、治疗药物、免疫单药还是联合治疗等的差异,同时选择全外显子测序,由于受到研究设计的影响,导致纳入研究的肺腺癌样本量偏少。
总之,本研究基于TCGA数据库中数据挖掘分析提示,在免疫治疗不同临床疗效的患者人群中,在基因突变谱及特征方面存在差异,进一步研究临床获益人群的基因突变谱特征分析,为肺腺癌新的分子分型提供了一定的理论支撑。另外,在通路富集分析及关键基因筛选方面,初步筛选了参与重要免疫调节通路的潜在关键基因,有助于进一步加深对免疫治疗的理解,从而更好地对肺腺癌患者进行精准化、个体化治疗。
作者贡献
齐晓光:设计实验、分析数据、撰写论文
祁春艳、秦博宇:数据分析整理
胡毅、韩为东:指导实验,审阅论文
| [1] |
Suresh K, Naidoo J, Lin CT, et al. Immune Checkpoint Immunotherapy for Non-Small Cell Lung Cancer: Benefits and Pulmonary Toxicities[J]. Chest, 2018, 154(6): 1416-1423. DOI:10.1016/j.chest.2018.08.1048 |
| [2] |
Haanen JB, Robert C. Immune Checkpoint Inhibitors[J]. Prog Tumor Res, 2015, 42: 55-66. |
| [3] |
Khan M, Lin J, Liao G, et al. Comparative analysis of immune checkpoint inhibitors and chemotherapy in the treatment of advanced non-small cell lung cancer: A meta-analysis of randomized controlled trials[J]. Medicine (Baltimore), 2018, 97(33): e11936. DOI:10.1097/MD.0000000000011936 |
| [4] |
Janakiram M, Pareek V, Cheng H, et al. Immune checkpoint blockade in human cancer therapy: lung cancer and hematologic malignancies[J]. Immunotherapy, 2016, 8(7): 809-819. DOI:10.2217/imt-2016-0001 |
| [5] |
Lantuejoul S, Damotte D, Hofman V, et al. Programmed death ligand 1 immunohistochemistry in non-small cell lung carcinoma[J]. J Thorac Dis, 2019, 11(Suppl 1): S89-S101. |
| [6] |
Jang HJ, Lee HS, Ramos D, et al. Transcriptome-based molecular subtyping of non-small cell lung cancer may predict response to immune checkpoint inhibitors[J]. J Thorac Cardiovasc Surg, 2019, 159(4): 1589-1610. |
| [7] |
Rizvi H, Sanchez-Vega F, La K, et al. Molecular Determinants of Response to Anti-Programmed Cell Death (PD)-1 and Anti-Programmed Death-Ligand 1 (PD-L1) Blockade in Patients With Non-Small-Cell Lung Cancer Profiled With Targeted Next-Generation Sequencing[J]. J Clin Oncol, 2018, 36(7): 633-641. DOI:10.1200/JCO.2017.75.3384 |
| [8] |
Gandara DR, Paul SM, Kowanetz M, et al. Blood-based tumor mutational burden as a predictor of clinical benefit in non-small-cell lung cancer patients treated with atezolizumab[J]. Nat Med, 2018, 24(9): 1441-1448. DOI:10.1038/s41591-018-0134-3 |
| [9] |
Wang S, Chen B, Zhu Z, et al. CDC20 overexpression leads to poor prognosis in solid tumors: A system review and meta-analysis[J]. Medicine (Baltimore), 2018, 97(52): e13832. DOI:10.1097/MD.0000000000013832 |
| [10] |
Alfarsi LH, Ansari RE, Craze ML, et al. CDC20 expression in oestrogen receptor positive breast cancer predicts poor prognosis and lack of response to endocrine therapy[J]. Breast Cancer Res Treat, 2019, 178(3): 535-544. DOI:10.1007/s10549-019-05420-8 |
| [11] |
Zhang Q, Huang H, Liu A, et al. Cell division cycle 20 (CDC20) drives prostate cancer progression via stabilization of β-catenin in cancer stem-like cells[J]. EBio Medicine, 2019, 42: 397-407. |
| [12] |
Cheng S, Castillo V, Sliva D, et al. CDC20 associated with cancer metastasis and novel mushroom-derived CDC20 inhibitors with antimetastatic activity[J]. Int J Oncol, 2019, 54(6): 2250-2256. |
| [13] |
Zhang MY, Liu XX, Li H, et al. Elevated mRNA Levels of AURKA, CDC20 and TPX2 are associated with poor prognosis of smoking related lung adenocarcinoma using bioinformatics analysis[J]. Int J Med Sci, 2018, 15(14): 1676-1685. DOI:10.7150/ijms.28728 |
| [14] |
Yi J, Wei X, Li X, et al. A Genome-Wide Comprehensive Analysis of Alterations in Driver Genes in Non-Small-Cell Lung Cancer[J]. Anticancer Drugs, 2018, 29(1): 10-18. DOI:10.1097/CAD.0000000000000571 |
| [15] |
Zhang Y, Wang H, Wang J, et al. Global Analysis of Chromosome 1 Genes Among Patients With Lung Adenocarcinoma, Squamous Carcinoma, Large-Cell Carcinoma, Small-Cell Carcinoma, or Non-Cancer[J]. Cancer Metastasis Rev, 2015, 34(2): 249-264. DOI:10.1007/s10555-015-9558-0 |
| [16] |
Jan Budczies, Carsten Denkert, Balázs Győrffy, et al. Chromosome 9p Copy Number Gains Involving PD-L1 Are Associated With a Specific Proliferation and Immune-Modulating Gene Expression Program Active Across Major Cancer Types[J]. BMC Med Genomics, 2017, 10(1): 74. DOI:10.1186/s12920-017-0308-8 |
| [17] |
Lee A, Moon BI, Kim TH. BRCA1/BRCA2 Pathogenic Variant Breast Cancer: Treatment and Prevention Strategies[J]. Ann Lab Med, 2020, 40(2): 114-121. DOI:10.3343/alm.2020.40.2.114 |
| [18] |
Wang B, Cao C, Liu X, et al. BRCA1-associated protein inhibits glioma cell proliferation and migration and glioma stem cell self-renewal via the TGF-β/PI3K/AKT/mTOR signalling pathway[J]. Cell Oncol (Dordr), 2020, 43(2): 223-235. |
| [19] |
Li AG, Murphy EC, Culhane AC, et al. BRCA1-IRIS promotes human tumor progression through PTEN blockade and HIF-1α activation[J]. Proc Natl Acad Sci U S A, 2018, 115(41): E9600-E9609. DOI:10.1073/pnas.1807112115 |
| [20] |
Sun X, Cui F, Yin H, et al. Association between EGFR mutation and expression of BRCA1 and RAP80 in non-small cell lung cancer[J]. Oncol Lett, 2018, 16(2): 2201-2206. |
| [21] |
Leng XF, Chen MW, Xian L, et al. Combined analysis of mRNA expression of ERCC1, BAG-1, BRCA1, RRM1 and TUBB3 to predict prognosis in patients with non-small cell lung cancer who received adjuvant chemotherapy[J]. J Exp Clin Cancer Res, 2012, 31(1): 25. DOI:10.1186/1756-9966-31-25 |
| [22] |
Wood K, Hensing T, Malik R, et al. Prognostic and Predictive Value in KRAS in Non-Small-Cell Lung Cancer: A Review[J]. JAMA Oncol, 2016, 2(6): 805-812. DOI:10.1001/jamaoncol.2016.0405 |
 2020, Vol. 47
2020, Vol. 47