文章信息
- 靶向沉默髓鞘转录因子MyT1对脑胶质瘤细胞恶性生物学行为的影响
- Effect of Targeted Knockdown of MyT1 Gene on Malignant Biological Behavior of Glioblastoma Cells
- 肿瘤防治研究, 2020, 47(10): 740-745
- Cancer Research on Prevention and Treatment, 2020, 47(10): 740-745
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2020.20.0088
- 收稿日期: 2020-02-07
- 修回日期: 2020-07-28
2. 300060 天津,天津医科大学肿瘤医院放射科,国家肿瘤临床医学研究中心,天津市“肿瘤防治”重点实验室,天津市恶性肿瘤临床医学研究中心
2. Department of Radiology, Tianjin Medical University Cancer Institute and Hospital, National Clinical Research Center for Cancer, Key Laboratory of Cancer Prevention and Therapy, Tianjin's Clinical Research Center for Cancer, Tianjin 300060, China
脑胶质瘤是最常见的原发性中枢神经系统肿瘤之一,约占原发性中枢神经系统肿瘤的30%~40%,具有复发率高、治愈率低等特点[1-2]。近年来越来越多的研究发现,SOX2[3]、ASCL1[4]、Notch1[5]等神经系统发育相关转录因子可能在脑胶质瘤的发生发展中具有重要作用,逐渐引起了人们的广泛关注。有研究显示髓鞘转录因子MyT1在脑胶质瘤中表达异常,但其生物学功能研究尚少[6-7]。本课题通过shRNA慢病毒技术在人脑胶质瘤U-118MG和U-87MG细胞中靶向沉默MyT1基因,观察它对细胞迁移、侵袭和黏附能力的影响及相关分子机制,为深入研究MyT1基因的生物学功能及人脑胶质瘤发生发展的分子机制提供实验依据。
1 材料与方法 1.1 材料人胚肾细胞HEK293T、人脑胶质瘤U-118MG和U-87MG细胞株为天津大学精仪学院神经工程与康复实验室保存,均使用DMEM+10%FBS在37℃、5%CO2条件下培养。FuGene转染试剂、TRIzol试剂、RevertAidTM第一链cDNA合成试剂盒和SYBR® green PCR试剂盒等购自美国LifeTechnology公司,Transwell小室购自中国Corning公司,兔抗人MyT1与鼠抗人β-actin等抗体购自美国Abcam公司。引物合成和DNA测序由中国金唯智公司完成。荧光定量PCR仪型号为ABI 7900HT Fast real-time PCR System。
1.2 方法 1.2.1 pLVX-iMyT1质粒的构建及鉴定利用在线Ambion shRNA软件设计一组靶向MyT1基因的shRNA序列,合成对应的正义和反义DNA模板,引物序列见表 1。37℃退火1 h,连接到经BglⅡ和HindⅢ酶切的慢病毒表达载体pLVX上,转化到E.coli DH5α感受态细胞获得重组质粒,将该质粒命名为pLVX-iMyT1。阳性克隆送交华大基因公司进行DNA测序验证。
分别在3种慢病毒包装质粒VSVG(3 μg)、pMDL(5 μg)和REV(2.5 μg)中加入shMyT1、shRNA空载体质粒12 μg制备成复合物,加入FuGene试剂转染HEK293T细胞,收集转染48 h后的病毒上清液。将病毒液过滤后分别感染到U-118MG和U-87MG细胞以产生稳定的细胞系,荧光显微镜下观察不同细胞系中GFP标签的表达情况以表征感染效率。
1.2.3 qPCR和Western blot法检测细胞中mRNA与蛋白的表达在100 mm培养皿中将shMyT1组和对照组细胞培养至密度达到80%,分别使用TRIzol法提取其总RNA,使用反转录试剂盒将其反转录为cDNA。按照qPCR试剂说明配制20 μl体系PCR反应混合液进行扩增,检测各基因mRNA的表达。qPCR反应条件:95℃预变性10 min、95℃变性30 s、55℃退火30 s,72℃延伸30 s,重复30个循环,72℃延伸7 min,4℃保存。
同样在上述细胞中加入600 μl Lysis Buffer冰上裂解后提取细胞总蛋白,将总蛋白进行SDS-PAGE分离、转移到PVDF膜上,5%脱脂牛奶室温封闭2 h,一抗β-actin(1:10 000)、MyT1(1:5 000),二抗羊抗兔和羊抗鼠(1:10 000)孵育,依次进行底物发光和曝光等操作。重复实验3次,采用BandScan 5.0软件分析结果。
1.2.4 划痕愈合实验检测细胞的迁移能力将U-118MG和U-87MG细胞制成5×108/L细胞悬液铺于60 mm培养皿中长至80%~90%密度,分别设shMyT1组和对照组,用灭菌枪头在孔板中央笔直划痕,PBS洗去脱落细胞后加入不含胎牛血清的RPMI 1640培养基置于孵育箱培养,分别在0、60 h拍照,计算迁移率。划痕迁移率=(原始划痕宽度-迁移后划痕宽度)/原始划痕宽度×100%。
1.2.5 Transwell实验检测细胞的侵袭能力将U-118MG和U-87MG细胞制成5×108/L细胞悬液,分别设shMyT1组和对照组,将200 μl细胞悬液滴加至预先铺好Matrigel基质胶的Transwell小室中,6孔板下室预先加入500 μl DMEM+10%FBS培养基,细胞培养箱内培养48 h,用棉签擦去Transwell小室内Matrigel胶后,4%多聚甲醛固定15 min,0.1%结晶紫染色,再经PBS洗涤3次,在显微镜下观察上室下表面上、中、下、左、右的5个视野,计数侵袭细胞数。
1.2.6 细胞黏附实验检测细胞的黏附能力将U-118MG和U-87MG细胞制成5×108/L细胞悬液,分别设shMyT1组和对照组,将细胞悬液滴加至Fibrenection隔夜包被的60 mm培养皿中,培养箱内培养1 h,吸去上清培养基及未贴壁细胞后,轻轻用PBS洗3次,使用4%多聚甲醛固定15 min,0.1%结晶紫染色,再经PBS洗涤3次,在显微镜下拍照,并通过计数统计黏附细胞数。
1.3 统计学方法实验数据采用SPSS17.0软件分析,实验重复3次。实验检测结果均符合正态分布,计量资料以均值±标准差(x±s)表示,组间比较用独立样本t检验,采用两因素两水平方差分析进行析因分析。P < 0.05为差异有统计学意义。
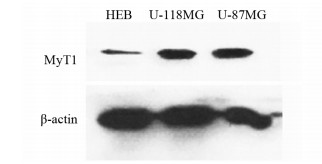
2 结果 2.1 脑胶质瘤细胞中MyT1的表达水平Western blot检测了MyT1在不同细胞系中的表达情况,与人正常脑胶质细胞HEB相比,MyT1在脑胶质瘤U-118MG和U-87MG细胞中显著高表达,见图 1。
|
| 图 1 Western blot检测人正常脑胶质细胞和脑胶质瘤细胞中MyT1的表达水平 Figure 1 MyT1 expression level in normal human brain glia and glioma cells detected by Western blot |
我们在将构建成功的pLVX-iMyT1质粒及空载体质粒在HEK293T细胞中包装成shRNA慢病毒,感染了U-118MG和U-87MG细胞,通过qPCR和Western blot从mRNA和蛋白水平分别检测细胞中MyT1基因的沉默效果。
qPCR结果显示,与对照组相比,靶向沉默MyT1后U-118MG和U-87MG细胞中MyT1 mRNA水平分别下降了约85%和90%,见图 2A。Western blot结果显示,与对照组相比,U-118MG和U-87MG细胞中MyT1蛋白水平也显著降低,见图 2B。这些结果说明,不论在mRNA水平还是蛋白水平,U-118MG和U-87MG细胞中MyT1的表达都发生了显著下调。

|
| 图 2 qPCR(A)和Western blot(B)法检测脑胶质瘤细胞中靶向沉默MyT1基因的效果 Figure 2 Targeted silencing of MyT1 gene in glioma cells detected by qPCR(A) and Western blot(B) |
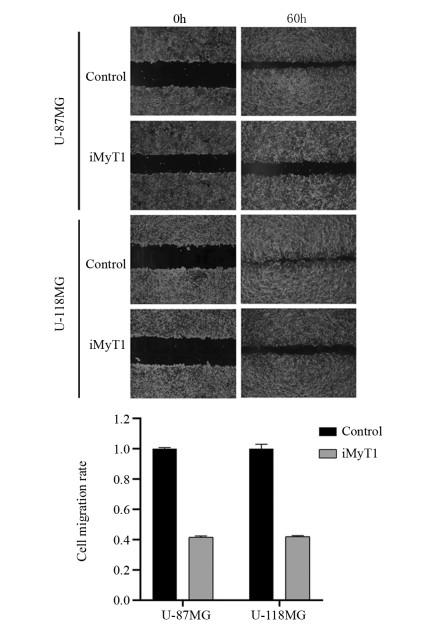
细胞划痕实验结果表明,靶向沉默MyT1后,U-118MG和U-87MG细胞的迁移距离显著短于对照组(P=0.010),60 h时只有对照组的41.5%和42.0%(P=0.012),见图 3。结果表明,敲低MyT1基因后脑胶质瘤U-118MG和U-87MG细胞的迁移能力显著下降。
|
| 图 3 细胞划痕实验检测靶向沉默MyT1对脑胶质瘤细胞迁移能力的影响 Figure 3 Effect of targeted silencing of MyT1 on migration of glioma cells detected by wound-healing assay |
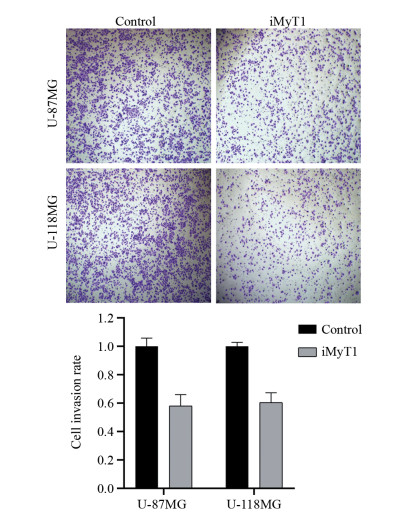
Transwell实验结果表明,靶向沉默MyT1后,U-118MG和U-87MG细胞侵袭穿透基底膜的细胞数显著少于对照组(P=0.015),分别只有对照组的58.0%和60.5%(P=0.022),见图 4。结果表明,敲低MyT1基因后脑胶质瘤U-118MG和U-87MG细胞的侵袭能力显著下降。
|
| 图 4 Transwell实验检测靶向沉默MyT1对脑胶质瘤细胞侵袭能力的影响 Figure 4 Effect of targeted silencing of MyT1 on invasion of glioma cells detected by Transwell assay |
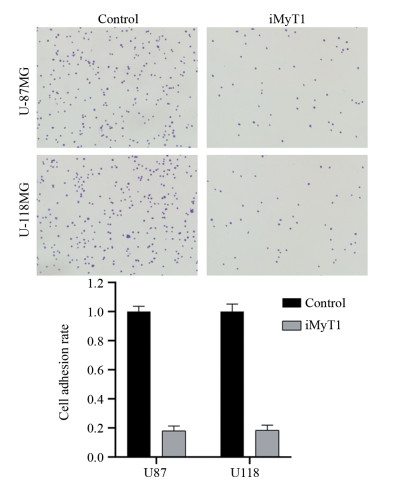
细胞黏附实验结果表明,靶向沉默MyT1后,U-118MG和U-87MG细胞黏附在Fibronection包被培养皿上的细胞数显著少于对照组(P=0.021),分别只有对照组的18.0%和18.3%(P=0.026),见图 5。结果表明,敲低MyT1基因后脑胶质瘤U-118MG和U-87MG细胞的黏附能力显著下降。
|
| 图 5 细胞黏附实验检测靶向沉默MyT1对脑胶质瘤细胞黏附能力的影响 Figure 5 Effect of targeted silencing of MyT1 on adhesion of glioma cells detected by cell adhesion assay |
qPCR结果显示,与对照组相比,靶向沉默MyT1后,U-118MG和U-87MG细胞中E-cadherin、CLDN1、CLDN2、ITGA5等相关基因的表达水平显著下降,而MMP9、CXCR4、c-FOS等肿瘤转移相关基因的表达水平显著上升,见图 6。结果说明,靶向沉默MyT1基因后脑胶质瘤细胞恶性生物学行为的改变可能与这些基因改变有关。
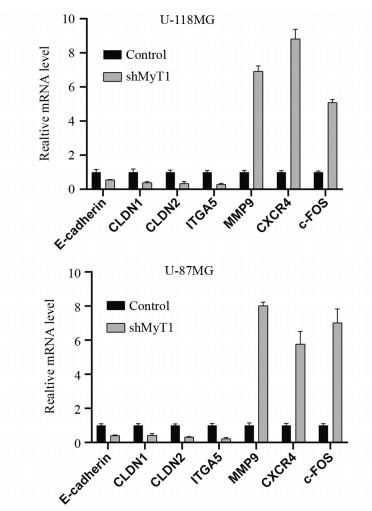
|
| 图 6 靶向沉默MyT1对细胞黏附和肿瘤转移相关基因表达水平的影响 Figure 6 Effects of targeted silencing of MyT1 on expression of cell adhesion- and tumor metastasis-related genes |
正常生理状态下,髓鞘转录因子MyT1主要表达于发育中的中枢神经系统细胞中[8-9],介导少突胶细胞的增殖分化和神经细胞髓鞘的形成[10-12]。Myt1蛋白的DNA结合结构域具有特殊的CCHHC(即Cys-X4-Cys-X4-His-X7-His-X5-Cys)结构,能够识别以“AAGTT”为核心的靶序列,特异性结合在多种神经发育相关基因的启动子上[13],募集HDAC蛋白Sin3B[14]或者和LSD1组成复合物[15],通过改变相关基因的组蛋白修饰特征等方式调控其表达,促进细胞的神经元性分化和增殖[16]。这些结果说明MyT1是神经细胞发育过程中的重要调控基因。1997年Hessler研究小组[6]首次报道了MyT1基因在人高级别脑胶质瘤中存在显著的表达上调现象,随后Sebolt-Leopold小组[17]报道敲低MyT1基因能够导致肿瘤细胞周期抑制并诱导凋亡的产生,最近Wotton小组[7]报道MyT1能够通过影响YAP1基因的表达影响脑胶质瘤细胞的增殖,由此可见,MyT1基因在脑胶质瘤中可能发挥着重要的作用,影响着脑胶质瘤的发生发展和患者的预后。
为深入研究MyT1基因在脑胶质瘤发生发展中的生物学功能及其对肿瘤细胞恶性生物学行为的影响,我们构建了特异性靶向MyT1基因的shRNA质粒,在HEK293中包装得到shRNA慢病毒,分别感染脑胶质瘤U-118MG和U-87MG细胞,得到稳定感染的细胞株。通过qPCR和Western blot实验发现该shRNA慢病毒在mRNA和蛋白水平均很好的实现了对MyT1基因的沉默效应,通过BrdU实验、细胞划痕实验、Transwell实验、细胞黏附实验检测发现脑胶质瘤U-118MG和U-87MG细胞中敲低MyT1基因后细胞的迁移、侵袭、黏附能力明显低于对照组。进一步检测发现,敲低MyT1后细胞中的E-cadherin、CLDN1、CLDN2、ITGA5等细胞黏附相关基因的表达水平显著下降,而MMP9、CXCR4、c-FOS等肿瘤转移相关基因的表达水平显著上升。这些结果说明,敲低MyT1基因可以从多个方面显著抑制脑胶质瘤U-118MG和U-87MG细胞的恶性生物学行为,从而影响脑胶质瘤的发生发展过程。未来,本课题组将以此为基础,深入研究MyT1的生物学功能及表观遗传调控机制,期望明确MyT1是否可成为脑胶质瘤诊疗的分子标志。
作者贡献
丁红军:实验设计,收集、整理数据,统计分析及论文撰写与修改
张艳龙、孟慧鹏、王克强、孙倩、李燕菊:实验技术支持,收集数据,论文修改
何峰:指导实验设计、审核数据及统计分析结果、指导论文撰写与修改
| [1] |
Tomaszewski W, Sanchez-Perez L, Gajewski TF, et al. Brain Tumor Microenvironment and Host State: Implications for Immunotherapy[J]. Clin Cancer Res, 2019, 25(14): 4202-4210. DOI:10.1158/1078-0432.CCR-18-1627 |
| [2] |
Raucher D. Tumor targeting peptides: novel therapeutic strategies in glioblastoma[J]. Curr Opin Pharmacol, 2019, 47: 14-19. DOI:10.1016/j.coph.2019.01.006 |
| [3] |
Peñaranda-Fajardo NM, Meijer C, Liang Y, et al. ER stress and UPR activation in glioblastoma: identification of a noncanonical PERK mechanism regulating GBM stem cells through SOX2 modulation[J]. Cell Death Dis, 2019, 10(10): 690. DOI:10.1038/s41419-019-1934-1 |
| [4] |
Hakes AE, Brand AH. Tailless/TLX reverts intermediate neural progenitors to stem cells driving tumourigenesis via repression of asense/ASCL1[J]. Elife, 2020, 9: e53377. DOI:10.7554/eLife.53377 |
| [5] |
Hai L, Zhang C, Li T, et al. Notch1 is a prognostic factor that is distinctly activated in the classical and proneural subtype of glioblastoma and that promotes glioma cell survival via the NF-κB(p65) pathway[J]. Cell Death Dis, 2018, 9(2): 158. DOI:10.1038/s41419-017-0119-z |
| [6] |
Armstrong RC, Migneault A, Shegog ML, et al. High-grade human brain tumors exhibit increased expression of myelin transcription factor 1 (MYT1), a zinc finger DNA-binding protein[J]. J Neuropathol Exp Neurol, 1997, 56(7): 772-781. DOI:10.1097/00005072-199756070-00003 |
| [7] |
Melhuish TA, Kowalczyk I, Manukyan A, et al. Myt1 and Myt1l transcription factors limit proliferation in GBM cells by repressing YAP1 expression[J]. Biochim Biophys Acta Gene Regul Mech, 2018, 1861(11): 983-995. DOI:10.1016/j.bbagrm.2018.10.005 |
| [8] |
Kim JG, Armstrong RC, v Agoston D, et al. Myelin transcription factor 1 (Myt1) of the oligodendrocyte lineage, along with a closely related CCHC zinc finger, is expressed in developing neurons in the mammalian central nervous system[J]. J Neurosci Res, 1997, 50(2): 272-290. |
| [9] |
Wrathall JR, Li W, Hudson LD. Myelin gene expression after experimental contusive spinal cord injury[J]. J Neurosci, 1998, 18(21): 8780-8793. DOI:10.1523/JNEUROSCI.18-21-08780.1998 |
| [10] |
Pandya P, Pasvolsky R, Babichev Y, et al. PICOT binding to the polycomb group protein, EED, alters H3K27 methylation at the MYT1 PRC2 target gene[J]. Biochem Biophys Res Commun, 2019, 509(2): 469-475. DOI:10.1016/j.bbrc.2018.12.153 |
| [11] |
Vasconcelos FF, Sessa A, Laranjeira C, et al. MyT1 Counteracts the Neural Progenitor Program to Promote Vertebrate Neurogenesis[J]. Cell Rep, 2016, 17(2): 469-483. DOI:10.1016/j.celrep.2016.09.024 |
| [12] |
Nielsen JA, Berndt JA, Hudson LD, et al. Myelin transcription factor 1 (Myt1) modulates the proliferation and differentiation of oligodendrocyte lineage cells[J]. Mol Cell Neurosci, 2004, 25(1): 111-123. DOI:10.1016/j.mcn.2003.10.001 |
| [13] |
Manukyan A, Kowalczyk I, Melhuish TA, et al. Analysis of transcriptional activity by the Myt1 and Myt1l transcription factors[J]. J Cell Biochem, 2018, 119(6): 4644-4655. DOI:10.1002/jcb.26636 |
| [14] |
Romm E, Nielsen JA, Kim JG, et al. Myt1 family recruits histone deacetylase to regulate neural transcription[J]. J Neurochem, 2005, 93(6): 1444-1453. DOI:10.1111/j.1471-4159.2005.03131.x |
| [15] |
Yokoyama A, Igarashi K, Sato T, et al. Identification of myelin transcription factor 1 (MyT1) as a subunit of the neural cell type-specific lysine-specific demethylase 1 (LSD1) complex[J]. J Biol Chem, 2014, 289(26): 18152-18162. DOI:10.1074/jbc.M114.566448 |
| [16] |
Kameyama T, Matsushita F, Kadokawa Y, et al. Myt/NZF family transcription factors regulate neuronal differentiation of P19 cells[J]. Neurosci Lett, 2011, 497(2): 74-79. DOI:10.1016/j.neulet.2011.04.033 |
| [17] |
Wang Y, Decker SJ, Sebolt-Leopold J. Knockdown of Chk1, Wee1 and Myt1 by RNA interference abrogates G2 checkpoint and induces apoptosis[J]. Cancer Biol Ther, 2004, 3(3): 305-313. DOI:10.4161/cbt.3.3.697 |
 2020, Vol. 47
2020, Vol. 47



