文章信息
- HNF4A充当肿瘤抑制基因抑制结直肠肿瘤进展的实验
- Experiment of HNF4A as a Tumor Suppressor Gene Inhibiting Progression of Colorectal Cancer
- 肿瘤防治研究, 2020, 47(11): 817-822
- Cancer Research on Prevention and Treatment, 2020, 47(11): 817-822
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2020.20.0069
- 收稿日期: 2020-02-04
- 修回日期: 2020-03-05
全球癌症调查报告指出每年约有180万新发结直肠癌病例和86万结直肠肿瘤特异性死亡病例,分别占总发病例数和总死亡例数的10%和9%[1]。肿瘤进展是肿瘤特异性死亡的主要原因之一,多种机制参与了肿瘤进展,包括增长信号的自给自足、对抑制生长信号不敏感、规避凋亡、无限的自我复制能力、持续的血管生成及组织浸润和转移及炎性反应[2]。肝细胞核因子4α(hepatocyte nuclear factor 4α, HNF4A)又称NR2A1是配体依赖性转录因子的核受体超家族成员之一,其在肝脏、肾、胰腺、胃肠道中表达。Pearson等研究发现HNF4A突变新生儿与未突变的新生儿相比,突变者与新生儿出生体重及新生儿低血糖密切相关[3],Chen等研究表明HNF4A调节脂肪酸的氧化并且是小鼠肠道干细胞更新所必须的[4]。HNF4A是主要的肝特异性基因表达调节因子,并且在多种肿瘤中具有抑癌特性,HNF4A参与了肝癌的发生与发展,HCV感染后miR-122通过STAT3介导HNF4A的转录抑制促进了肝炎向肝纤维化及肝癌的转变[5],HNF4A与WNT/β-catenin之间的双向负反馈促进了肝癌细胞的上皮间质转化(epithelial-mesenchymal transition, EMT)[6],也有研究表明HNF4A参与了小儿神经母细胞瘤、白血病、食管癌等恶性肿瘤的进展[7-9]。直到最近HNF4A在肠道中的作用才开始被研究,HNF4A不但维持了正常肠道上皮的完整性也是病理条件下失调的关键转录因子[8],但是HNF4A在结直肠癌中的作用仍存在争议,有研究表明HNF4A缺乏的小鼠与对照小鼠比较肠道息肉与肿瘤的发生率显著减少,并且HNF4A保护了肿瘤细胞免受活性氧的损害[10],相反Saandi等研究指出CDX2通过抑制HNF4A功能,促进了结肠恶性肿瘤的进展[11]。但HNF4A如何调节结直肠肿瘤进展的机制尚未被解释,本研究基于既往报道HND4A在结直肠肿瘤中的争议及不足拟探究HNF4A在结直肠肿瘤进展中的具体机制。
1 材料及方法 1.1 细胞及培养结直肠肿瘤细胞系SW480、HT-29、SW620及LoVo购自中国科学院典型培养物保藏委员会细胞库,其中SW480与SW620培养于L-15培养基、HT-29培养于MoCoy’s 5a培养基、LoVo培养于F-12K培养基,所有培养基加入10%胎牛血清及1%的青霉素链霉素双抗、所有细胞培养于37℃、含5%CO2的温箱中。
1.2 试剂L-15、McCoy’s 5a、F-12K培养基、胎牛血清、0.25%的胰蛋白酶、双抗(×100)购自武汉博士德生物有限公司;HNF4A、CD133、CD44、EpCAM、MMP2、MMP9、GADPH一抗及辣根过氧化物酶二抗购自美国Cell Signaling Technology公司;TRIzol试剂盒购自美国Invitrogen公司,Superscript反转录试剂盒及SYBR GreenⅠqPCR试剂盒购自北京Transgen Biotech公司。细胞培养皿、移液枪头、六孔板等耗材购自武汉启动子生物有限公司。
1.3 慢病毒转染HNF4A质粒、shHNF4A(短发夹RNA)或空载体由本实验室既往构建并保存[12];通过HNF4A高表达、敲降质粒或空载体与慢病毒包装试剂(磷酸钙转染试剂盒)共转染HEK 293T细胞,转染72 h后收集细胞上清液,并通过0.45 μm过滤器过滤,将收集的病毒感染预先铺板的细胞,72 h后加入嘌呤霉素筛选,挑选能稳定增殖的单克隆进行Western blot和qPCR检测。
1.4 实时定量PCR(qPCR)将培养细胞的六孔板使用PBS清洗两次,按照TRIzol(美国Invitrogen公司)试剂提取RNA操作步骤从细胞中分离总RNA。使用Superscript反转录酶试剂盒制备cDNA,使用SYBR Green ⅠqPCR试剂盒在ABI 7300系统上进行实时定量反转录PCR(qRT-PCR)。将基因表达数据标准化为GAPDH表达水平。HNF4A引物:F: 5’-CACGGGCAAACACTACGGT-3’,R: 5’-TTGACCTTCGAGTGCTGATCC-3’;GAPDH引物:F: 5’-GGAGCGAGATCCCTCCAAAAT-3’,R: 5’-GGCTGTTGTCATACTTCTCATGG-3’。
1.5 克隆形成实验药物筛选成功转染的结直肠肿瘤细胞系,取约1×103个细胞接种于软琼脂或平板中,于37℃、5%CO2培养箱中培养14 d后取出,弃上清液、PBS清洗2遍,纯甲醇固定15 min,结晶紫染色拍照。
1.6 CCK-8实验将生长状态良好的转染成功的细胞系经胰酶消化后取约1×104个细胞接种于96孔板中,分别于0、24、48、72和96 h后加入CCK-8反应底物,经酶标仪测定492 nm处的吸光度值。
1.7 迁移及侵袭实验使用Transwell聚碳酸酯膜和Boyden室测定法测定HNF4A对实验肿瘤细胞系的迁移和侵袭能力的影响。将200 μl无血清培养基中的1×105个细胞加入到小室的上部。下腔室填充有10%FBS的相应培养基,并在37℃、5%CO2下孵育48 h,在室温下使用4%多聚甲醛将Transwell膜固定30 min,PBS洗涤3次,Giemsa染色30 min,用棉签从插入物中除去未迁移的细胞。使用显微镜计数迁移细胞并定量。对于侵袭测定,在接种细胞之前,用Matrigel包被聚碳酸酯膜,之后与Transwell迁移实验步骤相同。
1.8 Western blot法检测蛋白表达收集转染后的细胞经蛋白酶抑制剂的NP-40冰上裂解30 min,收集蛋白裂解液,采用BCA法检测蛋白浓度,样品于100℃水浴15 min,在SDS-PAGE凝胶上分离大约15 μg蛋白质并转移至PVDF膜,5%脱脂牛奶封闭2 h,之后根据蛋白Marker剪膜,根据检测蛋白分子量,分别孵育一抗(1:1 000),4℃过夜,以TBST缓冲液漂洗3次后,在室温下孵育辣根过氧化物酶标记二抗(1:5 000)2 h,再次TBST洗膜后曝光显影。
1.9 统计学方法本研究均设计了对照实验,每组实验最少重复3次,所有数据均表示为平均值±标准差(x±s)。组间和组间比较分别用t检验。P < 0.05为差异有统计学意义。
2 结果 2.1 LoVo、SW620高表达HNF4A及HT-29、SW480敲降HNF4A的稳定细胞系的构建通过Western blot检测了结直肠癌细胞系中HNF4A基础表达情况,HNF4A在LoVo及SW620中表达较低,在HT-29及SW480中表达较高,见图 1A。进一步构建了HNF4A高表达的LoVo及SW620细胞系,Western blot检测结果显示构建的目的细胞系中HNF4A表达明显升高,见图 1B,同样的结果在qPCR中也得到了证实,见图 1D;在HT-29及SW480中转染HNF4A敲降的慢病毒后Western blot和qPCR结果显示HNF4A表达明显降低,见图 1C、1E。

|
| HNF4A: hepatocyte nuclear factor 4α; **: P < 0.001. 图 1 HNF4A高表达的LoVo、SW620细胞系及HNF4A敲降的HT-29、SW480细胞系的构建 Figure 1 Construction of LoVo, SW620 cell lines with high expression of HNF4A and HT-29, SW480 cell lines with knockdown of HNF4A |
克隆形成实验结果显示,平板中转染HNF4A的细胞相对转染空载体的细胞相比克隆形成数目明显减少,差异有统计学意义(PLoVo=0.002, Psw620=0.007);见图 2A~B;同样在软琼脂克隆形成实验中得到了相同的结论(PLoVo=0.009; Psw620=0.001),见图 2C~D。CCK-8实验结果显示,HNF4A组与对照组相比细胞增殖能力降低,见图 2E~F。我们进一步通过Western blot验证了结直肠肿瘤细胞中干细胞标志物的表达,结果显示,HNF4A过表达组与空白转染组相比干细胞标志物CD133、CD44及EpCAM表达降低,见图 2G。
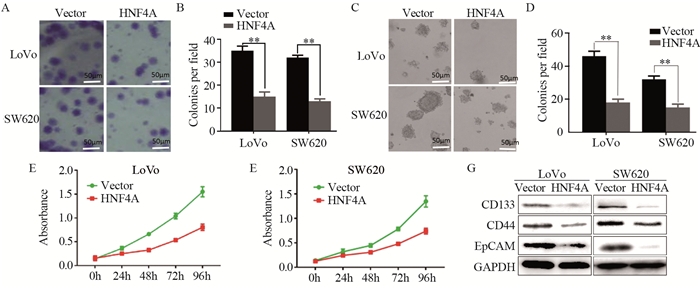
|
| **: P < 0.001; A, C: ×100. 图 2 HNF4A抑制结直肠肿瘤细胞的克隆、增殖能力及降低干细胞标志物的表达 Figure 2 HNF4A inhibited cloning and proliferation of colorectal cancer cells and decreased expression of stem cell markers |
将慢病毒转染的HT-29和SW480细胞行软琼脂及平板克隆实验、CCK-8实验和Western blot法检测干细胞标志物。结果显示,平板及软琼脂中转染shHNF4A的细胞相对转染sh-control的细胞相比克隆形成数目明显增多(PHT-29 < 0.001; Psw480 < 0.001)见图 3A~B与(PHT-29=0.015; sw480 < 0.001)图 3C~D;CCK-8结果显示敲降HNF4A的细胞系增殖能力升高,见图 3E~F;Western blot结果显示,HNF4A敲降组干细胞标志物CD133、CD44及EpCAM表达升高,见图 3G。

|
| **: P < 0.001;A, C: ×100. 图 3 敲降HNF4A促进结直肠肿瘤细胞的克隆、增殖能力及增强干细胞标志物的表达 Figure 3 Knockdown of HNF4A promoted cloning and proliferation of colorectal cancer cells and enhanced expression of stem cell markers |
对过表达HNF4A的LoVo及SW620细胞与转染空载体的细胞行Transwell迁移与侵袭实验检测细胞的迁移侵袭能力,结果显示:与对照相比,高表达HNF4A的LoVo细胞迁移能力降低(PLoVo=0.001, Psw620=0.032),见图 4A~B;在侵袭实验中观察到了同样的结论(PLoVo=0.004, Psw620=0.024),见图 4C~D。
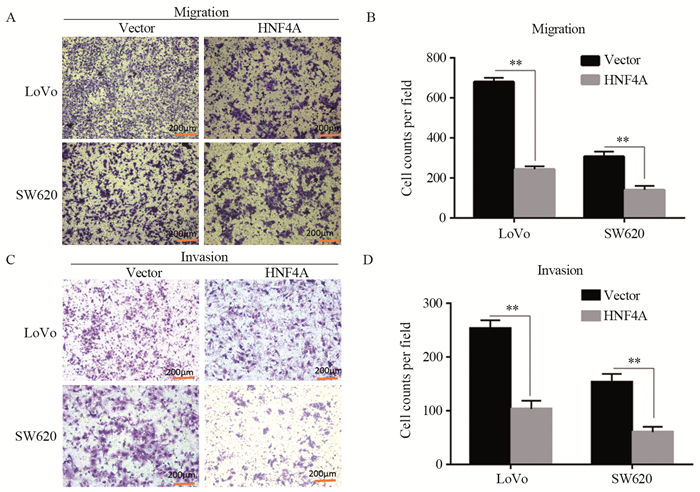
|
| **: P < 0.001; A, C: ×50. 图 4 HNF4A抑制结直肠癌细胞的迁移侵袭能力 Figure 4 HNF4A inhibited migration and invasion of colorectal cancer cells |
对敲降HNF4A的HT-29及SW480细胞与对照细胞进行Transwell迁移与侵袭实验检测细胞的迁移侵袭能力的改变,结果显示:与对照相比,敲降HNF4A的HT-29细胞迁移能力升高(PHT-29=0.008, Psw480=0.007),见图 5A~B;在侵袭实验中观察到了同样的结论(PHT-29=0.004, Psw480=0.020),见图 5C~D。
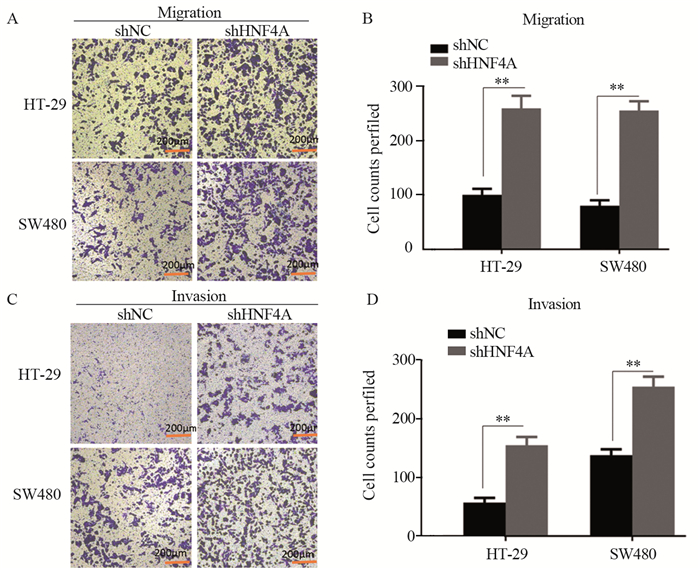
|
| **: P < 0.001; A, C: ×50. 图 5 敲降HNF4A增强结直肠癌细胞的迁移侵袭能力 Figure 5 Knockdown of HNF4A enhanced migration and invasion of colorectal cancer cells |
R2基因分析平台分析结果显示:HNF4A水平与MMP2及MMP9存在密切相关性(均P < 0.001),见图 6A~B。为进一步验证该相关性,在HNF4A高表达和HNF4A敲降的细胞中进行Western blot检测,结果显示,过表达HNF4A抑制了MMP2和MMP9的表达,敲降HNF4A促进了MMP2与MMP9的表达,见图 6C~D。

|
| 图 6 HNF4A调节MMP2与MMP9的表达 Figure 6 HNF4A regulated expression of MMP2 and MMP9 |
结直肠癌依赖于复杂的细胞内信号调节网络实现持续的生长,侵袭和转移。HNF4A是一种核受体,可以参与调节细胞增殖、分化和代谢中的多种过程。HNF4A在多种肿瘤中的失调使其成为肿瘤有希望的治疗靶标[13]。目前有多种研究已经建立了HNF4A与大肠癌之间的联系,Kriegsmann通过对1 021例非小细胞肺癌的标本包括472例肺腺癌和82例结直肠癌的肺转移进行免疫组织化学染色,结果显示HNF4A在原发性肺腺癌中比结直肠癌肺转移灶中明显升高(19%:0%),提出HNF4A可以作为结直肠癌肺转移与肺腺癌的鉴别诊断指标[14],这项研究同样提示了HNF4A在转移灶中的低表达。除此之外,还有研究证明HNF4A在肿瘤组织中明显上调并且可以靶向调节参与调节活性氧的氧化还原酶相关基因中的新功能进而使癌细胞免受氧化还原的伤害[10]。正是其作为抑癌基因或原癌基因的作用的争议削弱了其在癌症治疗中的潜在应用,这也是目前研究尚待解决的问题。
本研究证明了HNF4A在大肠癌中充当抑癌基因来抑制肿瘤的进展,证明了HNF4A抑制了结直肠肿瘤细胞的克隆、增殖及迁移侵袭能力,并进一步探讨了HNF4A通过下调MMP2及MMP9的表达调节结直肠肿瘤细胞的迁移和侵袭,在HNF4A敲降的细胞中得出了同样的结论。
增强的肿瘤细胞干性、较强的迁移与侵袭能力是肿瘤局部进展与远处转移的关键因素,所以干预上述过程有可能成为肿瘤的治疗策略,本研究表明HNF4A调节了结直肠肿瘤细胞的干性进而增强了细胞的克隆与增殖能力,并影响了迁移和侵袭能力,提出了HNF4A调节结直肠肿瘤进展的新机制,为结直肠肿瘤治疗提供新的可能靶点。
作者贡献
邵胜利:实验操作及论文撰写
杨熹:实验设计及指导
| [1] |
Bray F, Ferlay J, Soerjomataram I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6): 394-424. |
| [2] |
Hanahan D, Weinberg RA. The hallmarks of cancer[J]. Cell, 2000, 100(1): 57-70. DOI:10.1016/S0092-8674(00)81683-9 |
| [3] |
Pearson ER, Boj SF, Steele AM, et al. Macrosomia and hyperinsulinaemic hypoglycaemia in patients with heterozygous mutations in the HNF4A gene[J]. PLoS Med, 2007, 4(4): e118. DOI:10.1371/journal.pmed.0040118 |
| [4] |
Chen L, Vasoya RP, Toke NH, et al. HNF4 Regulates Fatty Acid Oxidation and is Required for Renewal of Intestinal Stem Cells in Mice[J]. Gastroenterology, 2019, 158(4): 985-999. |
| [5] |
Aydin Y, Kurt R, Song K, et al. Hepatic Stress Response in HCV Infection Promotes STAT3-Mediated Inhibition of HNF4A-miR-122 Feedback Loop in Liver Fibrosis and Cancer Progression[J]. Cancers (Basel), 2019, 11(10): pii: E1407.
|
| [6] |
Yang M, Li SN, Anjum KM, et al. A double-negative feedback loop between Wnt-beta-catenin signaling and HNF4alpha regulates epithelial-mesenchymal transition in hepatocellular carcinoma[J]. J Cell Sci, 2013, 126(Pt 24): 5692-5703. |
| [7] |
Rogerson C, Britton E, Withey S, et al. Identification of a primitive intestinal transcription factor network shared between esophageal adenocarcinoma and its precancerous precursor state[J]. Genome Res, 2019, 29(5): 723-736. DOI:10.1101/gr.243345.118 |
| [8] |
Li Z, Chen H. miR-34a inhibits proliferation, migration and invasion of paediatric neuroblastoma cells via targeting HNF4α[J]. Artif Cells Nanomed Biotechnol, 2019, 47(1): 3072-3078. DOI:10.1080/21691401.2019.1637886 |
| [9] |
Shi J, Dai R, Chen Y, et al. LncRNA LINP1 regulates acute myeloid leukemia progression via HNF4alpha/AMPK/WNT5A signaling pathway[J]. Hematol Oncol, 2019, 37(4): 474-482. DOI:10.1002/hon.2651 |
| [10] |
Darsigny M, Babeu JP, Seidman EG, et al. Hepatocyte nuclear factor-4alpha promotes gut neoplasia in mice and protects against the production of reactive oxygen species[J]. Cancer Res, 2010, 70(22): 9423-9433. DOI:10.1158/0008-5472.CAN-10-1697 |
| [11] |
Saandi T, Baraille F, Derbal-Wolfrom L, et al. Regulation of the tumor suppressor homeogene Cdx2 by HNF4alpha in intestinal cancer[J]. Oncogene, 2013, 32(32): 3782-3788. DOI:10.1038/onc.2012.401 |
| [12] |
Wang G, Yang X, Li C, et al. PIK3R3 Induces Epithelial-to-Mesenchymal Transition and Promotes Metastasis in Colorectal Cancer[J]. Mol Cancer Ther, 2014, 13(7): 1837-1847. DOI:10.1158/1535-7163.MCT-14-0049 |
| [13] |
Babeu JP, Jones C, Geha S, et al. P1 promoter-driven HNF4α isoforms are specifically repressed by β-catenin signaling in colorectal cancer cells[J]. J Cell Sci, 2018, 131(13): pii: jcs214734.
|
| [14] |
Kriegsmann M, Harms A, Longuespee R, et al. Role of conventional immunomarkers, HNF4-alpha and SATB2, in the differential diagnosis of pulmonary and colorectal adenocarcinomas[J]. Histopathology, 2018, 72(6): 997-1006. DOI:10.1111/his.13455 |
 2020, Vol. 47
2020, Vol. 47


