文章信息
- LncRNA和其他表观遗传学在三阴性乳腺癌中的研究进展
- Research Progress of LncRNA and Other Epigenetics in Triple-negative Breast Cancer
- 肿瘤防治研究, 2020, 47(9): 708-711
- Cancer Research on Prevention and Treatment, 2020, 47(9): 708-711
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2020.19.1390
- 收稿日期: 2019-11-07
- 修回日期: 2020-06-18
Competing interests: The authors declare that they have no competing interests.
乳腺癌(breast cancer, BC)是目前女性患病率最高的恶性肿瘤之一,死亡人数在前十名的恶性肿瘤中占14%,仅次于肺癌[1]。乳腺癌有很强的异质性,这种异质性超出了肿瘤之间的多样性[2]。三阴性乳腺癌(triple negative breast cancer, TNBC)占乳腺癌发病的15%~20%,是一种具有侵袭性强、预后差、较早出现淋巴结转移等特点的乳腺癌分子亚型,因缺乏激素受体(hormone receptor, HR)及表皮生长因子受体(human epidermal growth factor receptor 2, HER-2)而缺少相应的治疗靶点,化学药物治疗是目前的主导治疗方式,然而,耐药性成为其治疗的主要障碍[3]。分型不同,即使同为TNBC患者,相似的化疗方案也会产生不同的结果[4]。
TNBC的无病生存期(DFS)、总生存期(OS)、5年生存率均低于其他亚型,而且在发生远处转移后生存期仅为12~18月[5-6]。目前TNBC缺乏标准的分子靶向治疗药物,因此我们必须寻找潜在的生物标志物和治疗靶点以更精确地预测和治疗TNBC。本文概述了目前与BC,尤其是与TNBC表达相关的LncRNA及其表观遗传的研究。
1 TNBC的表观遗传改变表观遗传学研究基因表达的可遗传的变化,而不涉及核苷酸序列的任何改变。它可以通过组蛋白修饰、染色质重塑、DNA甲基化、基因沉默等经典的表观遗传机制,以及最近发现的miRNA、LncRNA等非编码RNA的表达来调控基因表达。分析TNBC生物标志物的一种方法是分析其表观遗传变化。但关于表观遗传控制所涉及的机制仍存在许多问题,这使人们对表观遗传的兴趣日益增加。
1.1 LncRNA全基因组序列的出现使人们发现了多种新的基因。在对新基因的探索中发现了多种LncRNA,却没有发现更多的蛋白质基因。人类基因组中大约80%的基因被转录,然而只有不到2%的转录基因组编码蛋白质,剩余的基因组则由非编码RNA(ncRNA)组成。它不参与蛋白质的翻译过程,但转录后可以控制基因的表达。并且LncRNA在发育复杂的生物基因组中种类数量较多,表明基于RNA的控制水平在多细胞生物进化中具有重要意义[7]。LncRNA几乎与人类所有的生物过程相关,其中包括基因印记、转录调控、转录干预、端粒维持、表观遗传机制调控、细胞分化、胚胎发育和病理功能障碍[7]等。
1.2 癌症中的LncRNA表达癌症基本上是一种遗传性疾病,改变细胞信息传递可以改变细胞稳态并促进肿瘤生长[8]。调节DNA突变可通过改变增强子和启动子活性或染色质状态而影响转录过程,从而导致癌症中LncRNA表达差异。非编码基因组内的单基因核苷酸多态性(SNP)和拷贝数的改变或突变可以显著改变LncRNA的转录[8]。例如,与血清PSA检测相比,PCA3作为尿液中前列腺癌的生物标志物,已成为前列腺癌有用的非侵入性实验指标,具有提高特异性、阳性预测值、阴性预测值的特点[9]。目前各种人类肿瘤(包括结肠癌、卵巢癌、甲状腺癌和乳腺癌)中都发现了LncRNA不同表达,表明LncRNA可能作为早期肿瘤检测或预后判断的生物标志物[10-12]。
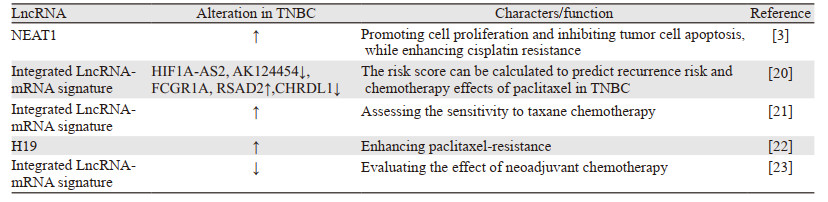
1.3 LncRNA在三阴性乳腺癌中的表达过去十余年,已有多项研究明确了与TNBC相关的LncRNA的变化。最近发现与TNBC侵袭和转移、化疗效果、发生、发展及预后相关的主要LncRNA,见表 1~3,这些研究结果对TNBC生物学各个方面的影响存在很大的不一致性,说明在将LncRNA作为生物标志物的分析中需要进行更好地验证,以提高其可靠性。同时有研究表明一些LncRNA表达与HR状态相关,靶向这些受体的LncRNA表达可能是TNBC中受体表达缺乏的原因。因此我们将进一步探索TNBC中可能涉及的LncRNA。

|
|

|
DNA甲基化,是正常哺乳动物发育中调节基因功能的可遗传的表观遗传过程[31]。根据Brinkman等[32]的研究,通过全基因组甲基化测序(WGBS)分析了30个原发性BC的DNA甲基化谱,发现在原发性乳腺癌样本中,部分甲基化结构域(PMD)的甲基化水平和基因组大小在肿瘤之间差异很大,强调了甲基化丧失的高度变异。BC表观遗传组的主要特征是,PMD甲基化的广泛丧失及其高变性,与此直接相关的是,CpG岛高甲基化是大多数肿瘤的一般标志。
2.1 TNBC中的DNA甲基化Stirzaker等为鉴定TNBC患者的DNA甲基化标记,使用癌症基因组图谱数据(TCGA),根据疾病结果(预后差,预后中等和预后良好)将患者分为三组。研究发现,基因标记中肿瘤DNA甲基化水平低的TNBC患者预后最好,甲基化水平高者预后中等,肿瘤甲基化水平中等的患者预后最差[33]。TET1蛋白是DNA去甲基化过程中的重要酶类,TET1具有脱甲基酶活性,在40%的TNBC中过表达,与CpG岛低甲基化及TNBC不良总生存相关[34]。BRCA1和BRCA2基因是与乳腺癌遗传高风险有关的主要基因,Prajzendanc等发现外周血中BRCA1启动子甲基化将TNBC的患病风险提高了5倍,通过甲基化突变或基因沉默使BRCA1基因体细胞失活是BRCA1基因功能损伤导致癌症的一种通路[35]。同时赖氨酸甲基转移酶(SMYD2)作为癌基因,可以通过甲基化以及抑制STAT3和NF-κB的P65亚基的活性来促进TNBC的进展[36]。SYNPO2作为肿瘤抑制基因,通过抑制YAP/TAZ活性抑制TNBC转移,其启动子甲基化可使Synaptopodin-2下调,TNBC5年复发增加; 相反,沉默SYNPO2可增强肿瘤转移。
2.2 LncRNA与其他表观遗传学的相互作用已有研究表明基因失活的表观遗传机制可以通过其他表观遗传过程来控制,LncRNA可以靶向DNA甲基转移酶(DNMT)并影响DNA甲基化过程,如Linc00152可激活DNMT,使肿瘤抑制基因BRCA1和PTEN基因失活,从而促进TNBC的发生[28];人内源性反转录病毒(HERVS)衍生的HROJAN(LncRNA AK124454序列)通过ZMYND8降解来促进TNBC进展,TROJAN可在多种细胞系中上调转移相关基因,而靶向TROJAN可明显抑制TNBC进展,TROJAN与转移抑制因子(ZMYND8)结合,使其作用丧失,从而增强TNBC增殖和侵袭,导致愈后不良[27]。LINC01416在不同分子亚型的乳腺癌组织中表达水平也不同,在HR阳性、HER2阳性乳腺癌中表达最高,在TNBC中低表达,可作为乳腺癌不良愈后的生物标志物,受TGFβ和黏着斑激酶(FKA)信号调节[37]。烟酰胺磷酸核糖基转移酶反义转录物可通过两种不同的方式调节NAMPT的表达,其中一种是募集POU2F2(NAMPT-AS的结合蛋白,也可以作为与NAMPT启动子区域结合的转录因子)来激活NAMPT的转录; 另一种是充当竞争性内源RNA来抑制NAMPT在miR-548b-3p的降解[19]。据报道,在TNBC中明显增强的DNA损伤特异性组蛋白伴侣蛋白紫杉醇PNK样因子(APLF)是必需的TNBC生物标志物和治疗靶标。APLF的下调会上调E-钙黏蛋白(CDH1)的表达,并抑制上皮间质转化(EMT)相关基因,从而抑制MDA-MB-231细胞的侵袭和转移[38]。基于组蛋白修饰图谱分析,一些研究旨在重建表观遗传失调基因的正常表达。因此,转移性TNBC细胞已用于评估特定的组蛋白修饰抑制剂诱导或减少TNBC转移的能力,还分析了上皮和细胞黏附标记的变化,以了解组蛋白修饰对EMT的作用,并最终确定潜在的诊断和预后生物标记。
3 结论由于TNBC的异质性,TNBC的治疗已进入个性化治疗时代,以期降低疾病进展及复发风险,而个性化治疗应该了解疾病发生发展的各个阶段,如疾病通路、遗传学、蛋白组学水平的生物信息及生物标志物等,发现更具针对性及毒性更低的治疗方法,因此我们必须加深对LncRNA发生及与之相关表观遗传变化的理解。本综述总结了一定的证据,证明LncRNA可作为肿瘤诊断的生物标记及治疗靶点,概括了不同表观遗传变化在TNBC中的联系。从分子水平入手,提高对生物过程的理解,更好地了解疾病过程并确定稳定有效的生物标志物。然而,LncRNA在BC不同亚型中的相对表达水平特别是TNBC亚型中的表达仍有待确定。
作者贡献
孙含笑:稿件的构思及写作
张风华:指导稿件书写及审核
| [1] |
Siegel RL, Miller KD, Jemal A. Cancer statistics, 2018[J]. CA Cancer J Clin, 2018, 68(1): 7-30. DOI:10.3322/caac.21442 |
| [2] |
Ng CKY, Schultheis AM, Bidard F, et al. Breast cancer genomics from microarrays to massively parallel sequencing: paradigms and new insights[J]. J Natl Cancer Inst, 2015, 107(5): djv015. |
| [3] |
Shin VY, Chen JW, Cheuk IWY, et al. Long non-coding RNA NEAT1 confers oncogenic role in triple-negative breast cancer through modulating chemoresistance and cancer stemness[J]. Cell Death Dis, 2019, 10(4): 270. DOI:10.1038/s41419-019-1513-5 |
| [4] |
Wong-Brown MW, Meldrum C, Carpenter JE, et al. Prevalence of BRCA1 and BRCA2 germline mutations in patients with triple-negative breast cancer[J]. Breast Cancer Res Treat, 2015, 150(1): 71-80. |
| [5] |
Carey L, Winer E, Viale G, et al. Triple-negative breast cancer: disease entity or title of convenience?[J]. Nat Rev Clin Oncol, 2010, 7(12): 683-692. DOI:10.1038/nrclinonc.2010.154 |
| [6] |
Dent R, Trudeau M, Pritchard KI, et al. Triple-negative breast cancer: clinical features and patterns of recurrence[J]. Clin Cancer Res, 2007, 13(15 Pt 1): 4429-4434. |
| [7] |
Fatica A, Bozzoni I. Long Non-coding RNAs: new players in cell differentiation and development[J]. Nat Rev Genet, 2014, 15(1): 7-21. |
| [8] |
Schmitt AM, Chang HY. Long Noncoding RNAs in Cancer Pathways[J]. Cancer Cell, 2016, 29(4): 452-463. DOI:10.1016/j.ccell.2016.03.010 |
| [9] |
Wei JT, Feng Z, Partin AW, et al. Can urinary PCA3 supplement PSA in the early detection of prostate cancer?[J]. J Clin Oncol, 2014, 32(36): 4066-4072. DOI:10.1200/JCO.2013.52.8505 |
| [10] |
Bo H, Fan L, Li J, et al. High Expression of lncRNA AFAP1-AS1 Promotes the Progression of Colon Cancer and Predicts Poor Prognosis[J]. J Cancer, 2018, 9(24): 4677-4683. DOI:10.7150/jca.26461 |
| [11] |
Chen Y, Bi F, An Y, et al. Identification of pathological grade and prognosis-associated lncRNA for ovarian cancer[J]. J Cell Biochem, 2019, 120(9): 14444-14454. DOI:10.1002/jcb.28704 |
| [12] |
Sui F, Ji M, Hou P. Long non-coding RNAs in thyroid cancer: Biological functions and clinical significance[J]. Mol Cell Endocrinol, 2018, 469: 11-22. DOI:10.1016/j.mce.2017.07.020 |
| [13] |
Bamodu OA, Huang WC, Lee WH, et al. Aberrant KDM5B expression promotes aggressive breast cancer through MALAT1 overexpression and downregulation of hsa-miR-448[J]. BMC Cancer, 2016, 16: 160. DOI:10.1186/s12885-016-2108-5 |
| [14] |
Zuo Y, Li Y, Zhou Z, et al. Long non-coding RNA MALAT1 promotes proliferation and invasion via targeting miR-129-5p in triple-negative breast cancer[J]. Biomed Pharmacother, 2017, 95: 922-928. DOI:10.1016/j.biopha.2017.09.005 |
| [15] |
Wang S, Ke H, Zhang H, et al. LncRNA MIR100HG promotes cell proliferation in triple-negative breast cancer through triplex formation with p27 loci[J]. Cell Death Dis, 2018, 9(8): 805. DOI:10.1038/s41419-018-0869-2 |
| [16] |
Wang L, Liu D, Wu X, et al. Long non-coding RNA (LncRNA) RMST in triple-negative breast cancer (TNBC): Expression analysis and biological roles research[J]. J Cell Physiol, 2018, 233(10): 6603-6612. DOI:10.1002/jcp.26311 |
| [17] |
Youness RA, Assal RA, Motaal AA, et al. A novel role of sONE/NOS3/NO signaling cascade in mediating hydrogen sulphide bilateral effects on triple negative breast cancer progression[J]. Nitric Oxide, 2018, 80: 12-23. DOI:10.1016/j.niox.2018.07.004 |
| [18] |
Youness RA, Hafez H, Khallaf E, et al. The long noncoding RNA sONE represses triple-negative breast cancer aggressiveness through inducing the expression of miR-34a, miR-15a, miR-16, and let-7a[J]. J Cell Physiol, 2019, 234(11): 20286-20297. DOI:10.1002/jcp.28629 |
| [19] |
Zhang H, Zhang N, Liu Y, et al. Epigenetic regulation of NAMPT by NAMPT-AS drives metastatic progression in triple-negative breast cancer[J]. Cancer Res, 2019, 79(13): 3347-3359. DOI:10.1158/0008-5472.CAN-18-3418 |
| [20] |
Jiang YZ, Liu YR, Xu XE, et al. Transcriptome Analysis of Triple-Negative Breast Cancer Reveals an Integrated mRNA-lncRNA Signature with Predictive and Prognostic Value[J]. Cancer Res, 2016, 76(8): 2105-2114. DOI:10.1158/0008-5472.CAN-15-3284 |
| [21] |
Liu YR, Jiang YZ, Xu XE, et al. Comprehensive Transcriptome Profiling Reveals Multigene Signatures in Triple-Negative Breast Cancer[J]. Clin Cancer Res, 2016, 22(7): 1653-1662. DOI:10.1158/1078-0432.CCR-15-1555 |
| [22] |
Han J, Han B, Wu X, et al. Knockdown of lncRNA H19 restores chemo-sensitivity in paclitaxel-resistant triple-negative breast cancer through triggering apoptosis and regulating Akt signaling pathway[J]. Toxicol Appl Pharmacol, 2018, 359: 55-61. DOI:10.1016/j.taap.2018.09.018 |
| [23] |
Wang Q, Gao S, Li H, et al. Long noncoding RNAs (lncRNAs) in triple negative breast cancer[J]. J Cell Physiol, 2017, 232(12): 3226-3233. DOI:10.1002/jcp.25830 |
| [24] |
Tang J, Li Y, Sang Y, et al. LncRNA PVT1 regulates triple-negative breast cancer through KLF5/beta-catenin signaling[J]. Oncogene, 2018, 37(34): 4723-4734. DOI:10.1038/s41388-018-0310-4 |
| [25] |
Tang J, Zhong G, Zhang H, et al. LncRNA DANCR upregulates PI3K/AKT signaling through activating serine phosphorylation of RXRA[J]. Cell Death Dis, 2018, 9(12): 1167. DOI:10.1038/s41419-018-1220-7 |
| [26] |
Yang F, Shen Y, Zhang W, et al. An androgen receptor negatively induced long non-coding RNA ARNILA binding to miR-204 promotes the invasion and metastasis of triple-negative breast cancer[J]. Cell Death Differ, 2018, 25(12): 2209-2220. DOI:10.1038/s41418-018-0123-6 |
| [27] |
Jin X, Xu X, Jiang Y, et al. The endogenous retrovirus-derived long noncoding RNA TROJAN promotes triple-negative breast cancer progression via ZMYND8 degradation[J]. Sci Adv, 2019, 5(3): eaat9820. DOI:10.1126/sciadv.aat9820 |
| [28] |
Wu J, Shang Z, Zhao J, et al. Linc00152 promotes tumorigenesis by regulating DNMTs in triple-negative breast cancer[J]. Biomed Pharmacother, 2018, 97: 1275-1281. DOI:10.1016/j.biopha.2017.11.055 |
| [29] |
Wang PS, Chou CH, Lin CH, et al. A novel long non-coding RNA linc-ZNF469-3 promotes lung metastasis through miR-574-5p-ZEB1 axis in triple negative breast cancer[J]. Oncogene, 2018, 37(34): 4662-4678. DOI:10.1038/s41388-018-0293-1 |
| [30] |
Li S, Zhou J, Wang Z, et al. Long noncoding RNA GAS5 suppresses triple negative breast cancer progression through inhibition of proliferation and invasion by competitively binding miR-196a-5p[J]. Biomed Pharmacother, 2018, 104: 451-457. DOI:10.1016/j.biopha.2018.05.056 |
| [31] |
Yu J, Qin B, Moyer AM, et al. DNA methyltransferase expression in triple-negative breast cancer predicts sensitivity to decitabine[J]. J Clin Invest, 2018, 128(6): 2376-2388. DOI:10.1172/JCI97924 |
| [32] |
Brinkman AB, Nik-Zainal S, Simmer F, et al. Partially methylated domains are hypervariable in breast cancer and fuel widespread CpG island hypermethylation[J]. Nat Commun, 2019, 10(1): 1749. |
| [33] |
Stirzaker C, Zotenko E, Song JZ, et al. Methylome sequencing in triple-negative breast cancer reveals distinct methylation clusters with prognostic value[J]. Nat Commun, 2015, 6: 5899. DOI:10.1038/ncomms6899 |
| [34] |
Good CR, Panjarian S, Kelly AD, et al. TET1-Mediated Hypomethylation Activates Oncogenic Signaling in Triple-Negative Breast Cancer[J]. Cancer Res, 2018, 78(15): 4126-4137. DOI:10.1158/0008-5472.CAN-17-2082 |
| [35] |
Prajzendanc K, Domagała P, Hybiak J, et al. BRCA1 promoter methylation in peripheral blood is associated with the risk of triple-negative breast cancer[J]. Int J Cancer, 2020, 146(5): 1293-1298. DOI:10.1002/ijc.32655 |
| [36] |
Li XY, Zhou X, Calvet JP, et al. Lysine methyltransferase SMYD2 promotes triple negative breast cancer progression[J]. Cell Death Dis, 2018, 9(3): 326. DOI:10.1038/s41419-018-0347-x |
| [37] |
Vishnubalaji R, Shaath H, Elkord E, et al. Long non-coding RNA (lncRNA) transcriptional landscape in breast cancer identifies LINC01614 as non-favorable prognostic biomarker regulated by TGFβ and focal adhesion kinase (FAK) signaling[J]. Cell Death Discov, 2019, 5: 109. DOI:10.1038/s41420-019-0190-6 |
| [38] |
Majumder A, Syed KM, Mukherjee A, et al. Enhanced expression of histone chaperone APLF associate with breast cancer[J]. Mol Cancer, 2018, 17(1): 76. |
 2020, Vol. 47
2020, Vol. 47


