文章信息
- MYEOV对人胰腺癌细胞增殖和迁移的影响
- Effect of MYEOV on Proliferation and Migration of Human Pancreatic Cancer Cells
- 肿瘤防治研究, 2020, 47(5): 324-329
- Cancer Research on Prevention and Treatment, 2020, 47(5): 324-329
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2020.19.1387
- 收稿日期: 2019-11-06
- 修回日期: 2020-02-24
2. 730000 兰州,兰州大学第一医院普外二科
2. Second Department of General Surgery, The First Hospital of Lanzhou University, Lanzhou 730000, China
在我国,胰腺癌发病率位居恶性肿瘤的第10位,新发病例占恶性肿瘤的2.42%;而死亡病例高居肿瘤相关死亡的第6位[1]。历年来胰腺癌的死亡人数与发病人数相当[2-3],发病隐匿,进展迅速,病死率高,预后差[4]。因此寻找胰腺癌早期筛查分子标志物和治疗靶点是当前研究的热点[5],孤儿基因是指在系统地层学中被限制在特定谱系、与其他谱系没有相似序列的基因[6],其表达多具有时间特异性和组织特异性,与胚胎发育、组织分化、罕见疾病和肿瘤发生发展等多种生物过程相关[7],孤儿基因中的骨髓瘤过表达基因(myeloma overexpressed, MYEOV)与多种肿瘤增殖、侵袭、转移和临床预后相关,其在胰腺癌研究中尚无报道。因此,本实验通过检测MYEOV在胰腺细胞系中表达情况,并通过慢病毒转染下调MYEOV基因表达水平,探讨其对人胰腺癌细胞的体外增殖和迁移能力的影响及其机制。
1 材料与方法 1.1 细胞与试剂人胰腺细胞株AsPC-1、BxPC-3、Mia Paca-2、SW1990和HPDE6由兰州大学第二医院陈昊教授惠赠,CFPAC-1和PANC-1购自上海中科院。RPMI1640培养基、DMEM培养基、胎牛血清、胰蛋白酶购自以色列Biolnd公司;TRIzol试剂购自美国Invitrogen公司;qPCR引物序列购自上海生工生物股份有限公司;cDNA反转录反应试剂盒和SYBY Green PCR试剂盒购自日本TaKaRa公司;细胞裂解及蛋白抽提试剂、BCA蛋白浓度检测试剂盒、Western blot配胶试剂盒购自江苏碧云天生物技术有限公司;PVDF膜、ECL发光液购自美国Millipore公司;LV-MYEOV-RNAi慢病毒购自上海吉凯生物公司;CCK-8试剂购自日本同仁公司;兔抗人MYEOV抗体、鼠抗人GAPDH抗体购自美国Proteintech公司,HRP标记的山羊抗兔、鼠二抗购自美国SAB公司。
1.2 方法 1.2.1 细胞培养人胰腺癌细胞株AsPC-1、BxPC-3用含有10%胎牛血清的RPMI 1640培养基,CFPAC-1、PANC-1、SW1990、Mia Paca-2和HPDE6用含有10%胎牛血清的DMEM培养基,在37℃、5%CO2恒温培养箱中培养,用含0.25%的胰蛋白酶消化。
1.2.2 慢病毒的设计和细胞转染设计人MYEOV shRNA慢病毒MYEOV-sh,载体为GV248,元件顺序hU6-MCS-Ubiquitin-EGFP-IRES-puromycin,其中MYEOV-sh1序列为:5′-CCGGGCTGACTGTTGTGACTGTTCTCGAGAACAGTCACAACAGTCAGCCATTTTTG-3′,MYEOV-sh2序列为:5′-CCGGGGCTCTCCATGGAAATTATCTCGAGATAATTTCCATGGAGAGCCCATTTTTG-3′,空载体序列为:5′-TTCTCCGAACGTGTCACGT-3′。取对数生长期的SW1990细胞以1.5×105个每孔接种于6孔板中,12 h当细胞融合度达40%时按制造商的指南进行感染,MOI值为10。根据转染状态及转染载体不同分为4组:SW1990-NC组为没有任何干预的正常对照组(Normal control),SW1990-VC组为转染空白载体的阴性对照组(Vector control),SW1990-sh1和SW1990-sh2为实验组。转染12 h后换液,72 h观察转染效果,用2 μg/ml嘌呤霉素筛选10天获得混合稳定转染细胞株。
1.2.3 qPCR检测MYEOV及TGF-β通路中SMADs RNA水平表达将融合度在85%左右的各组细胞处理后使用TRIzol法提取总RNA,检测RNA浓度和质量,使用反转录试剂盒按说明将RNA反转录为cDNA,以GAPDH为内参照进行qPCR反应,基因特异性引物序列见表 1。qPCR扩增反应条件:95℃ 30 s,1个循环,预变性;95℃ 3 s,60℃ 30 s,40个循环,扩增;95℃ 15 s,60℃ 1 min,95℃ 15 s,1个循环,引物熔解曲线测定。使用2-Δ ΔCt相对定量法计算mRNA表达水平。
细胞融合度达到85%左右,使用含PMSF、PI的强RIPA裂解液提取细胞总蛋白,使用BCA浓度试剂盒检测蛋白浓度,加入5×SDS上样缓冲液和适量RIPA将蛋白浓度定量,在95℃ 5 min金属浴条件下蛋白变性。配制5%浓缩胶和10%分离胶,每孔上样50 μg总蛋白进行SDS-PAGE电泳(浓缩胶80 V 30 min,分离胶120 V 1.5 h,恒压),200 mA恒流湿转2 h将蛋白转移到PVDF膜上,使用2%BSA室温摇床封闭1 h,一抗(稀释比GAPDH 1:2 000,MYEOV 1:500)4℃孵育14 h,TBST室温摇床洗膜3次(每次20 ml, 5 min),对应的二抗(稀释比1:2 000)室温摇床孵育1 h,TBST再次洗膜3次后ECL化学发光法显影。使用Image J软件对蛋白条带进行分析。
1.2.5 CCK-8法测定细胞体外增殖能力将处于对数生长期的SW1990对照组和实验组细胞消化后重悬至1×104个每毫升,接种于96孔板中,每孔100 μl,每组设5个复孔,待3 h细胞贴壁后弃原培养基,每孔加入100 μl含10%CCK-8溶液的基础培养基,孵箱温育2 h,酶标仪测定450 nm处吸光度。间隔24 h更换96孔板中培养基,分别于24、48、72和96 h使用CCK-8法测定细胞吸光度,使用GraphPad软件绘制细胞增殖曲线。
1.2.6 细胞划痕实验检测细胞迁移能力使用胰蛋白酶消化处于对数生长期的各组细胞,调整细胞密度为1×106个每毫升,接种于6孔板,铺板均匀后恒温培养箱中培养,待细胞融合度达90%左右时,使用10 μl枪头垂直划痕,PBS洗3次后加入2 ml无血清培养基,分别于0 h和24 h在倒置显微镜下拍照。用Image J软件对各组细胞迁移面积进行分析。划痕愈合率=(划痕面积0 h–划痕面积24 h)/划痕面积0 h×100%。
1.3 统计学方法采用GraphPad Prism 6.0和SPSS18.0对实验数据进行统计学分析,计量资料采用均数±标准差(x±s)表示,所得数据进行正态分布检验,组间比较采用t检验与单因素方差分析,P < 0.05为差异有统计学意义。
2 结果 2.1 胰腺细胞系中MYEOV的表达水平通过qPCR和Western blot检测胰腺细胞系中MYEOV mRNA与蛋白的表达水平。MYEOV mRNA在正常胰腺上皮细胞HPDE6中表达水平(1.01±0.09)显著低于胰腺癌细胞,在PANC-1和SW1990中表达水平高(1024.00±20.49和1691.00±84.51),差异有统计学意义(均P < 0.01),其中SW1990细胞中mRNA表达最高,见图 1A。而在蛋白表达水平上,在PANC-1和SW1990细胞中检测到MYEOV蛋白表达,见图 1B。因此选择SW1990细胞进行慢病毒转染。

|
| **: P < 0.01, ***: P < 0.001, compared with HPDE6 cells. 图 1 人胰腺导管上皮细胞及6种胰腺癌细胞系中MYEOV mRNA(A)和蛋白(B)的表达水平 Figure 1 Expression levels of MYEOV mRNA(A) and protein(B) in pancreatic ductal epithelium cell line and six pancreatic cancer cell lines |
MYEOV-shRNA转染SW1990细胞后用嘌呤霉素筛选获得混合稳定转染细胞株,提取各组细胞RNA和蛋白。SW1990-NC与SW1990-VC组比较,MYEOV mRNA和蛋白水平表达变化差异无统计学意义(P=0.100和P=0.195);在mRNA水平上,与SW1990-VC组(1.001±0.020)比较,SW1990-sh1组(0.131±0.014)和SW1990-sh2组(0.091±0.001)中MYEOV mRNA表达明显下降,差异有统计学意义(P < 0.001和P=0.0005);在蛋白表达水平上,与SW1990-NC组(1.056±0.065)和SW1990-VC组(0.918±0.061)比较,SW1990-sh1组(0.558±0.046)和SW1990-sh2组(0.537±0.025)中MYEOV表达均下调,差异有统计学意义(P < 0.001和P < 0.01),见图 2。
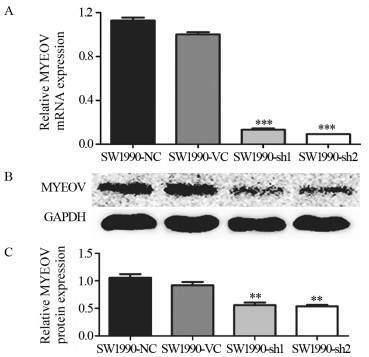
|
| **: P < 0.01, ***: P < 0.001, compared with SW1990-VC group. 图 2 MYEOV-shRNA转染后SW1990细胞中MYEOV mRNA(A)和蛋白(B, C)的表达水平 Figure 2 Expression levels of MYEOV mRNA(A) and protein(B, C) in SW1990 cells after MYEOV-shRNA lentivirus infection |
CCK-8法分别比较24、48、72和96 h时4组细胞增殖速度,双因素方差分析,F细胞组=1148.8, P < 0.001; F时间=360, P < 0.001; F细胞组×时间=31.8, P < 0.001,差异有统计学意义。分别比较各时间点4组细胞增殖速度,与SW1990-NC组对比,SW1990-VC组细胞增殖速度下降,差异有统计学意义(24 h时P=0.018,余P < 0.01);与SW1990-VC组比较,SW1990-sh1和SW1990-sh2组细胞增殖速度均下降,差异有统计学意义(SW1990-sh1组P < 0.01,SW1990-sh2组P < 0.001);与SW1990-sh1组相比,SW1990-sh2组细胞增殖速度更慢,差异有统计学意义(P < 0.01),见图 3。
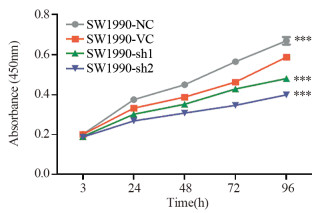
|
| ***: P < 0.001, compared with SW1990-VC group. 图 3 MYEOV敲低对SW1990细胞增殖能力的影响 Figure 3 Effect of MYEOV knockdown on proliferation of SW1990 cells |
细胞划痕实验检测SW1990细胞迁移能力变化,单因素方差分析划痕愈合率,差异有统计学意义(F=272.2, P < 0.001)。与SW1990-NC组(90.95±2.515)%和SW1990-VC组(76.21±0.532)%相比,SW1990-sh1组(45.38±1.985)%和SW1990-sh2组(20.82±2.808)%的划痕愈合率均降低,差异有统计学意义(P < 0.001);与SW1990-sh1组相比,SW1990-sh2组划痕愈合率更低,差异有统计学意义(P=0.0009),见图 4。
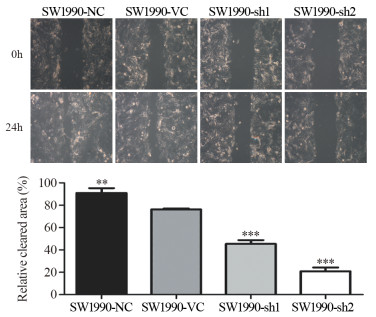
|
| **: P < 0.01, ***: P < 0.001, compared with SW1990-VC group. 图 4 MYEOV敲低对SW1990细胞迁移能力的影响 Figure 4 Effect of MYEOV knockdown on migration of SW1990 cells |
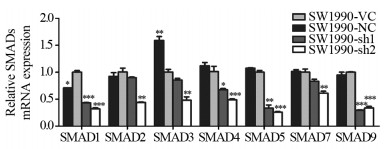
使用qPCR检测4组细胞中SMADs mRNA的表达水平变化。与SW1990-VC组相比,SMAD1、SMAD5和SMAD9 mRNA在SW1990-sh1组和SW1990-sh2组中表达明显下调,差异有统计学意义(P < 0.001);SMAD4 mRNA在两实验组中表达下调,差异有统计学意义(P=0.031和P=0.007);而SMAD2、SMAD3和SMAD7 mRNA仅在SW1990-sh2中表达下调,差异有统计学意义(P < 0.01),但在SW1990-sh1组中的表达差异无统计学意义(P > 0.05),见图 5。
|
| *: P < 0.05, **: P < 0.01, ***: P < 0.001, compared with SW1990-VC group. 图 5 MYEOV敲低对SW1990细胞中SMADs mRNA表达水平影响 Figure 5 Effect of MYEOV knockdown on expression levels of SMADs mRNA in SW1990 cells |
骨髓瘤过表达基因(MYEOV)是人类孤儿基因中第一个被确定的致癌性基因[8-9],分布在染色体11q13上,虽然在猩猩、猴等其他类人猿物种中也存在MYEOV基因但并未发现其转录和翻译,而非人灵长类动物的癌症发病率非常低,因此MYEOV对于研究人类恶性肿瘤高发病率具有重要意义[10]。MYEOV在食管癌[11]、胃癌[12]、乳腺癌[13]、结直肠癌[14-15]、非小细胞肺癌[16-17]、多发性骨髓瘤[18-19]等肿瘤中高表达,参与调节肿瘤的增殖、侵袭等过程,与患者预后关系密切。通过GEPIA数据库发现MYEOV在胰腺癌中高表达,与胰腺癌预后相关,提示MYEOV可能参与胰腺的恶性生物过程[20]。
本研究发现,相比于正常胰腺上皮细胞HPDE6,MYEOV mRNA在胰腺癌细胞系中高表达;胰腺癌细胞系中,在PANC-1和SW1990两株细胞中检测出MYEOV蛋白表达,提示MYEOV蛋白的翻译过程可能受到其过长的5'-UTR高度抑制[21],MYEOV蛋白的表达可能是通过内部核糖体进入位点的非帽依赖性机制进行的[22-23]。MYEOV mRNA和蛋白表达差异的现象也存在于食管癌[11]、胃癌[12]、结直肠癌[14]、非小细胞肺癌[17]中,因此MYEOV的翻译机制及真核生物的非帽依赖性翻译机制仍需进一步研究。
为探讨MYEOV在胰腺癌细胞增殖、迁移中的作用及可能机制,本实验建立了稳定的MYEOV表达下调细胞系SW1990,通过qPCR和Western blot检测MYEOV表达下调后,SW1990细胞增殖和迁移能力明显下降,而且在SW1990-sh1和SW1990-sh2两实验组中,细胞增殖和迁移能力之间差异有统计学意义。本研究检测了TGF-β/SMAD信号通路中部分基因的mRNA表达情况,与对照组相比,实验组中SMAD1、SMAD5和SMAD9表达明显下调,而SMAD2、SMAD3和SMAD7仅在SW1990-sh2组中明显下调。在TGF-β通路中,SMAD2、SMAD3在细胞外基质形成、血管生成等过程起重要作用[25],其表达及其磷酸化形成RSMAD/coSMAD复合物进入核是细胞信号转导的关键环节。SMAD2、SMAD3在两实验组的表达差异可能是两组细胞增殖和迁移能力差异的原因。
MYEOV在恶性肿瘤中的作用机制尚不清楚,有研究表明在非小细胞肺癌中MYEOV mRNA可直接作为ceRNA通过TGF-β通路调节肺癌细胞增殖、侵袭和小鼠体内成瘤过程[17]。TGF-β信号通路具有抑制肿瘤发生和促进肿瘤进展的双重拮抗性效应,更与肿瘤上皮间质转化息息相关[24-25]。TGF-β抑制剂是当前研究热点之一,一些TGF-β抑制剂在临床试验中使部分晚期肿瘤获益[26],因此对MYEOV的研究可能为寻找新的TGF-β抑制剂提供思路。
综上所述,MYEOV mRNA在胰腺癌细胞系中高表达,下调MYEOV表达可抑制胰腺癌细胞的增殖和迁移能力,其调节机制可能与TGF-β通路中SMADs表达下调有关。然而,MYEOV在肿瘤中的翻译机制以及相关的调控机制目前仍不完全清楚,因此全面深入了解MYEOV在胰腺癌发生发展中的分子机制,对于寻找胰腺癌早诊及治疗的新靶点有重要意义。
作者贡献
吕根:设计与实施实验、收集与分析数据、撰写论文
严俊:分析核对数据
任龙飞:指导实验技术及论文写作
周文策:指导实验设计、修改和审核论文
| [1] |
Siegel RL, Miller KD, Jemal A. Cancer statistics, 2019[J]. CA Cancer J Clin, 2019, 69(1): 7-34. DOI:10.3322/caac.21551 |
| [2] |
Chen W, Zheng R, Baade PD, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin, 2016, 66(2): 115-132. DOI:10.3322/caac.21338 |
| [3] |
Patel N, Khorolsky C, Benipal B. Incidence of Pancreatic Adenocarcinoma in the United States from 2001 to 2015: A United States Cancer Statistics Analysis of 50 States[J]. Cureus, 2018, 10(12): e3796. |
| [4] |
张驰, 任庆兰. 外泌体在胰腺癌诊治中的研究进展[J]. 肿瘤防治研究, 2019, 46(8): 737-740. [Zhang C, Ren QL. Advances of exosomes in diagnosis and treatment of pancreatic cancer[J]. Zhong Liu Fang Zhi Yan Jiu, 2019, 46(8): 737-740. DOI:10.3971/j.issn.1000-8578.2019.19.0046] |
| [5] |
Yoneyama T, Ohtsuki S, Honda K, et al. Identification of IGFBP2 and IGFBP3 As Compensatory Biomarkers for CA19-9 in Early-Stage Pancreatic Cancer Using a Combination of Antibody-Based and LC-MS/MS-Based Proteomics[J]. PLoS One, 2016, 11(8): e0161009. DOI:10.1371/journal.pone.0161009 |
| [6] |
张焕萍, 尹佟明. 谱系特有基因研究进展[J]. 遗传, 2015, 37(6): 544-553. [Zhang HP, Yin TM. Advances in lineage-specific genes[J]. Yi Chuan, 2015, 37(6): 544-553.] |
| [7] |
McLysaght A, Hurst LD. Open questions in the study of de novo genes: what, how and why[J]. Nat Rev Genet, 2016, 17(9): 567-578. DOI:10.1038/nrg.2016.78 |
| [8] |
Xie C, Zhang YE, Chen JY, et al. Hominoid-specific de novo protein-coding genes originating from long non-coding RNAs[J]. PLoS Genet, 2012, 8(9): e1002942. DOI:10.1371/journal.pgen.1002942 |
| [9] |
Papamichos SI, Margaritis D, Kotsianidis I. Adaptive Evolution Coupled with Retrotransposon Exaptation Allowed for the Generation of a Human-Protein-Specific Coding Gene That Promotes Cancer Cell Proliferation and Metastasis in Both Haematological Malignancies and Solid Tumours: The Extraordinary Case of MYEOV Gene[J]. Scientifica(Cairo), 2015, 2015: 984706. |
| [10] |
Puente XS, Velasco G, Gutiérrez-Fernández A, et al. Comparative analysis of cancer genes in the human and chimpanzee genomes[J]. Bmc Genomics, 2006, 7: 15. DOI:10.1186/1471-2164-7-15 |
| [11] |
Janssen JW, Imoto I, Inoue J, et al. MYEOV, a gene at 11q13, is coamplified with CCND1, but epigenetically inactivated in a subset of esophageal squamous cell carcinomas[J]. J Hum Genet, 2002, 47(9): 460-464. DOI:10.1007/s100380200065 |
| [12] |
Leyden J, Murray D, Moss A, et al. Net1 and Myeov: computationally identified mediators of gastric cancer[J]. Br J Cancer, 2006, 94(8): 1204-1212. DOI:10.1038/sj.bjc.6603054 |
| [13] |
Janssen JW, Cuny M, Orsetti B, et al. MYEOV: a candidate gene for DNA amplification events occurring centromeric to CCND1 in breast cancer[J]. Int J Cancer, 2002, 102(6): 608-614. DOI:10.1002/ijc.10765 |
| [14] |
Moss AC, Lawlor G, Murray D, et al. ETV4 and Myeov knockdown impairs colon cancer cell line proliferation and invasion[J]. Biochem Biophys Res commun, 2006, 345(1): 216-221. DOI:10.1016/j.bbrc.2006.04.094 |
| [15] |
Lawlor G, Doran PP, MacMathuna P, et al. MYEOV (myeloma overexpressed gene) drives colon cancer cell migration and is regulated by PGE2[J]. J Exp Clin Cancer Res, 2010, 29: 81. DOI:10.1186/1756-9966-29-81 |
| [16] |
Horie M, Kaczkowski B, Ohshima M, et al. Integrative CAGE and DNA Methylation Profiling Identify Epigenetically Regulated Genes in NSCLC[J]. Mol Cancer Res, 2017, 15(10): 1354-1365. DOI:10.1158/1541-7786.MCR-17-0191 |
| [17] |
Fang L, Wu S, Zhu X, et al. MYEOV functions as an amplified competing endogenous RNA in promoting metastasis by activating TGF-beta pathway in NSCLC[J]. Oncogene, 2019, 38(6): 896-912. DOI:10.1038/s41388-018-0484-9 |
| [18] |
Janssen JW, Vaandrager JW, Heuser T, et al. Concurrent activation of a novel putative transforming gene, myeov, and cyclin D1 in a subset of multiple myeloma cell lines with t(11;14)(q13;q32)[J]. Blood, 2000, 95(8): 2691-2698. |
| [19] |
Moreaux J, Hose D, Bonnefond A, et al. MYEOV is a prognostic factor in multiple myeloma[J]. Exp Hematol, 2010, 38(12): 1189-1198. DOI:10.1016/j.exphem.2010.09.002 |
| [20] |
Tang Z, Li C, Kang B, et al. GEPIA: a web server for cancer and normal gene expression profiling and interactive analyses[J]. Nucleic Acids Res, 2017, 45(W1): W98-W102. DOI:10.1093/nar/gkx247 |
| [21] |
de Almeida RA, Heuser T, Blaschke R, et al. Control of MYEOV protein synthesis by upstream open reading frames[J]. J Biol Chem, 2006, 281(2): 695-704. DOI:10.1074/jbc.M511467200 |
| [22] |
Weingarten-Gabbay S, Elias-Kirma S, Nir R, et al. Comparative genetics. Systematic discovery of cap-independent translation sequences in human and viral genomes[J]. Science, 2016, 351(6270): pii: aad4939. DOI:10.1126/science.aad4939 |
| [23] |
Leppek K, Das R, Barna M. Functional 5'-UTR mRNA structures in eukaryotic translation regulation and how to find them[J]. Nat Rev Mol Cell Biol, 2018, 19(3): 158-174. DOI:10.1038/nrm.2017.103 |
| [24] |
Massagué J. TGFbeta in Cancer[J]. Cell, 2008, 134(2): 215-230. DOI:10.1016/j.cell.2008.07.001 |
| [25] |
Ikushima H, Miyazono K. TGFbeta signalling: a complex web in cancer progression[J]. Nat Rev Cancer, 2010, 10(6): 415-424. DOI:10.1038/nrc2853 |
| [26] |
Neuzillet C, Tijeras-Raballand A, Cohen R, et al. Targeting the TGFβ pathway for cancer therapy[J]. Pharmacol Ther, 2015, 147: 22-31. DOI:10.1016/j.pharmthera.2014.11.001 |
 2020, Vol. 47
2020, Vol. 47