文章信息
- 核糖体生物合成与肿瘤的研究进展
- Research Progress of Ribosome Biogenesis and Cancer
- 肿瘤防治研究, 2020, 47(5): 393-397
- Cancer Research on Prevention and Treatment, 2020, 47(5): 393-397
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2020.19.1138
- 收稿日期: 2019-09-09
- 修回日期: 2020-02-10
核糖体是细胞内蛋白质合成重要的细胞器,影响细胞的正常功能。合成新核糖体的过程被定义为核糖体生物合成。核糖体生物合成是一个高度复杂和保守的过程,主要发生在核仁中[1]。核糖体生物合成包括核糖体RNA(ribosomal RNA, rRNA)转录、rRNA前体加工和核糖体亚基组装的过程。在生物体中,除了rRNA和核糖体蛋白(ribosomal protein),还有超过300种非核糖体反式作用因子在核糖体生物合成的过程中发挥作用,它们也被称为核糖体组装因子[2]。
早期研究认为,为了满足肿瘤细胞持续生长,通过核糖体生物合成的增加来维持高效的蛋白质合成效率是必需的,但是越来越多的研究表明核糖体生物合成的异常,包括核糖体数量的增加和修饰的改变可能会影响肿瘤的发生[3]。因此,核糖体生物合成与肿瘤发生发展息息相关。本文中我们将综述核糖体生物合成过程及其与肿瘤发生发展的关系。
1 核糖体生物合成过程作为细胞内合成蛋白质的重要细胞器,真核生物的核糖体由大小亚基组成,按照沉降系数,称为60S大亚基和40S小亚基。核糖体的大小亚基均由rRNA与核糖体蛋白构成。小亚基包含了18S rRNA和33种核糖体蛋白。而大亚基包含了5S rRNA、5.8S rRNA、28S rRNA以及47种核糖体蛋白。
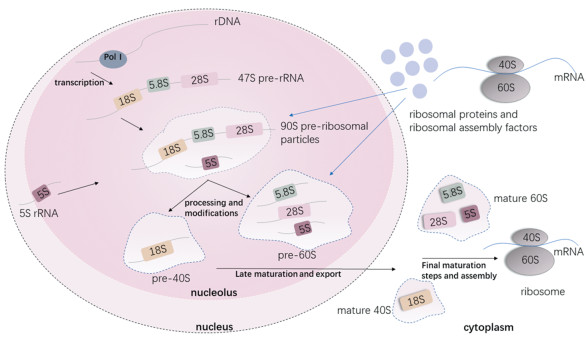
核糖体生物合成过程见图 1(自绘)。在RNA聚合酶Ⅰ、选择性因子1(selectivity factor 1, SL1)、上游结合因子(upstream binding factor, UBF)、转录起始因子1(transcription initiation factor 1, TIF-1A)和转录终止因子1(transcription termination factor 1, TTF-1)作用下,核糖体DNA(ribosomal DNA, rDNA)转录成47S pre-rRNA。47S pre-rRNA是由18S rRNA、5.8S rRNA、28S rRNA串联组成的rRNA前体,这三种rRNA由转录间隔序列分隔开。rRNA转录的速率由RNA聚合酶Ⅰ的活性决定,并且rRNA的转录是核糖体生物合成的限速步骤[1]。而5S rRNA则是在核浆中由RNA聚合酶Ⅲ作用下转录得到。在这个过程中,转录因子TFⅢC和TFⅢB是必需的。
|
| 图 1 核糖体生物合成的过程 |
Pre-rRNA转录本的成熟包括:剪切步骤和化学修饰。47S pre-rRNA需要经过剪切,切除多余的转录间隔序列。并且在小核仁RNA(small nucleolar RNA, snoRNA)和核糖核蛋白(ribonucleoprotein, RNP)形成的复合物作用下,rRNA发生特定修饰,包括甲基化和假尿苷化修饰[4],从而得到成熟的28S、18S和5.8S rRNA。这种rRNA的特定修饰,有利于rRNA的正确折叠以及与核糖体蛋白的结合[5]。
在pre-rRNA的加工过程中,核糖体组装因子和核糖体蛋白会以协同的方式组装到pre-rRNA上,形成前核糖体颗粒(pre-ribosomal particles)。28S、5.8S及5S rRNA与核糖体蛋白组成pre-60S亚基,18S rRNA与核糖体蛋白组成pre-40S亚基。核糖体组装因子和核糖体蛋白是在细胞质中生成,并通过核孔复合物被运输到细胞核中参与核糖体的组装[6]。
前核糖体颗粒形成后,通过出核转运蛋白被转运到细胞质中。当到达细胞质后,前核糖体颗粒经历最终成熟过程,包括pre-rRNA的进一步加工、剩余核糖体蛋白的组装以及组装和转运因子的释放[6],形成具有翻译能力的核糖体。
核糖体生物合成是一个非常复杂的过程,关于人类核糖体生物合成机制的研究尚未完善。近来对核糖体生物合成的深入研究有助于我们探讨核糖体生物合成与肿瘤之间的关系。
2 肿瘤中核糖体生物合成的异常由于肿瘤细胞代谢旺盛,生长和增殖能力增强,在肿瘤中核糖体生物合成异常频繁发生,具体描述如下。
2.1 rDNA的不稳定人类rDNA在基因组中存在数百个拷贝,它们组成头尾相连的串联重复序列,形成基因簇。这些重复序列很不稳定,彼此之间容易发生重组。转录产生47S pre-rRNA的47S rDNA基因簇位于5对人类常染色体上,转录产生5S rRNA的5S rDNA基因簇位于1对人类常染色体上。在超过一半的肺癌和结直肠癌样本中,检测到rDNA基因簇的结构改变,使这种基因组重排成为在成人实体肿瘤中最常见的特殊染色体改变之一[7]。对肿瘤和配对正常组织样本的全基因组数据分析发现,肿瘤中rDNA的拷贝数变异超过正常组织的十倍,并且大多数肿瘤都发生了47S rDNA拷贝数缺失和5S rDNA拷贝数扩增[8]。在一项前瞻性队列研究中发现,rDNA拷贝数的变化与肺癌风险有关[9]。
2.2 rRNA合成异常rRNA合成异常包括rRNA合成增加和rRNA修饰异常。在原发性结直肠癌组织中47S pre-rRNA的表达量明显高于正常结直肠黏膜组织中47S pre-rRNA的表达量[10]。在大多数宫颈鳞状上皮内病变组织中发现rRNA表达水平高于正常组织,在大多数前列腺癌组织中也发现rRNA表达水平升高[11]。目前在肿瘤中rRNA修饰的研究还不是很多,但已有研究发现在侵袭性的乳腺癌细胞系中,rRNA特定位点的甲基化修饰增强。同时,参与rRNA修饰的修饰因子在多种肿瘤中的突变提示在肿瘤中rRNA修饰的异常[12]。
2.3 核糖体蛋白基因发生突变已有研究报道,核糖体蛋白基因突变直接导致的核糖体疾病[13]会导致癌症易感性综合征:人类疾病戴-布二氏贫血(Diamond Blackfan Anemia)与核糖体蛋白基因RPS19、RPS17、RPS24、RPL35a、RPS7、RPL5和RPL11的突变有关,它的重要特征是再生障碍性贫血和易患白血病;RPS14的突变与急性髓系白血病的易感性有关[14]。近来一系列应用全外显子测序或者全基因组测序的研究[15],在多种肿瘤中都发现核糖体蛋白基因的突变相对频繁,例如,子宫内膜癌中RPL22的突变、在急性T细胞型淋巴细胞白血病中RPL10、RPL5、RPL11的突变、结直肠癌中RPS20的突变以及胶质瘤中RPL5的突变。
2.4 核糖体蛋白表达的失调在多种肿瘤中发现特定核糖体蛋白表达量上调,例如,RPL5、RPS3、RPS6、RPS8、RPS12在结直肠癌中表达量高于正常结直肠黏膜组织。在胃癌组织和细胞系中RPL15的表达量上调。在人类肝癌组织和细胞系中检测到RPS8、RPL12、RPL23a、RPL27、RPL30 mRNA水平升高[16]。使用TCGA数据分析也发现,与正常组织相比,肿瘤组织中核糖体蛋白表达水平的中位数增加了30%。并且许多核糖体蛋白仅在特定肿瘤中表现出明显的失调,例如:RPL21L1和RPS27L在乳腺癌和甲状腺癌中表达上调,而RPL21表达下调[17]。
以上是肿瘤中核糖体生物合成的异常情况。值得一提的是,在正常组织干细胞中也具有较高的rRNA合成速率,意味着干细胞与肿瘤细胞都具有活跃的核糖体生物合成。并且与肿瘤细胞类似地,干细胞中的核糖体生物合成会促使核糖体选择性翻译mRNA从而调控特定基因的表达[18]。然而,干细胞与肿瘤细胞之间核糖体生物合成存在差异性,表现为:在肿瘤细胞中,核糖体生物合成异常频繁发生,这种失调会打破蛋白质合成稳态;而在干细胞中,核糖体生物合成受到严格调控,并且在干细胞的分化过程中,rRNA合成速率降低。核糖体生物合成受到严格调控从而维持干细胞稳态,而它在肿瘤中的异常促进了肿瘤发展,具体作用机制还有待进一步研究。
3 肿瘤细胞中核糖体生物合成的调控核糖体生物合成在肿瘤中异常,取决于多种因素,包括原癌基因和抑癌基因的改变以及细胞内特定信号通路的激活。核糖体生物合成受到许多关键的细胞生长和增殖信号通路的严格调控[19],主要包括:RAS/RAF/MEK/ERK、MYC和PI3K/AKT/mTOR。这些通路形成了一个复杂的网络,共同调节细胞生长和增殖。
3.1 转录因子MYC转录因子MYC(c-MYC癌基因产物)是最常见的激活癌蛋白之一,在约50%的癌症中过度表达,它能结合和调控大量基因,包括许多编码核糖体蛋白、rRNA加工因子和翻译因子的基因。MYC被认为是核糖体生物合成的主要调节因子,能够调控三种RNA聚合酶[20]。在MYC驱动的肿瘤中,MYC会调节很多RNA聚合酶Ⅱ转录的基因,包括核糖体蛋白基因。MYC也会调节RNA聚合酶Ⅲ介导的转录,增强tRNA、5SRNA以及其他小RNA基因的转录。同时,研究发现MYC与SL1结合促进SL1被招募到rDNA启动子上,直接促进RNA聚合酶Ⅰ介导rRNA的转录。MYC刺激核糖体生物合成可能是促进细胞生长和肿瘤发生的关键途径。
3.2 RAS/RAF/MEK/ERK信号通路细胞外信号调节激酶ERK通过信号转导级联被激活,包括RAS、RAF和ERK激酶MEK。ERK会调节RNA聚合酶Ⅲ和RNA聚合酶Ⅰ介导的转录。ERK磷酸化并激活UBF和TIF-1A,从而刺激RNA聚合酶Ⅰ合成rRNA。ERK也直接激活一般转录因子TFⅢB,以增加RNA聚合酶Ⅲ对编码tRNA和5S rRNA的基因的转录。可以认为,MYC增加了促进核糖体生物合成的转录因子的数量,而ERK则增加了它们的活性[21]。
3.3 PI3K/AKT/mTOR信号通路PI3K激活后,磷酸化AKT使其激活,信号通过AKT传递到下游靶点。mTOR存在两种蛋白复合物,即mTORC1和mTORC2,主要调控核糖体生物合成的是mTORC1。mTORC1主要通过核糖体蛋白S6激酶(S6K1/2)和翻译起始因子结合蛋白(4E-BP1/2/3)调控核糖体蛋白的合成和rRNA的合成[22]。mTORC1一方面增强5’-TOP mRNA的翻译从而促进核糖体蛋白的翻译。另一方面mTORC1上调UBF的表达和增加其羧基端激活结构域的磷酸化,磷酸化的UBF与SL1相互作用明显增强,促进RNA聚合酶Ⅰ对rDNA的转录[23]。此外,激酶AKT在多个水平上调节rRNA的转录,包括转录的起始和延伸,促进rRNA的合成,并且RNA聚合酶Ⅰ的转录延长需要持续的依赖于AKT的信号。AKT的这种促进核糖体生物合成的作用独立于其激活mTORC1产生的作用。并且AKT还会与MYC协同作用,在MYC驱动的淋巴瘤生成过程中,AKT信号对核糖体生物合成和细胞存活是必需的。
众所周知,RAS/RAF/MEK/ERK、MYC和PI3K/AKT/mTOR信号网络调控异常已被证明在肿瘤的发生和发展中起着关键作用。考虑到这个信号网络对核糖体生物合成的关键调控作用,可以认为,核糖体生物合成是RAS/RAF/MEK/ERK、MYC和PI3K/AKT/mTOR通路驱动恶性肿瘤的重要环节。
因此,核糖体生物合成异常不只是肿瘤发生的结果,它可能会直接促进恶性转化和肿瘤的发生,并且也逐渐被认为是多种肿瘤的一个可行的治疗靶点。
4 核糖体生物合成作为肿瘤治疗的有效靶点使用药物抑制核糖体生物合成可以激活核仁应激反应[24]。具体来说,当核糖体生物合成受到抑制时,多余的核糖体蛋白聚集起来,激活了RP-MDM2-P53信号通路:P53的表达和活性主要受MDM2调控,MDM2是一种E3泛素连接酶,通过与P53结合抑制其转录活性,并促进其蛋白酶体的降解。部分核糖体蛋白如RPL5、RPL11、RPL23会与MDM2相互作用,从而抑制其活性,稳定P53。P53作为一种抑癌基因,导致细胞周期阻滞和细胞凋亡。
除了依赖于P53途径的抑癌作用,药物靶向核糖体生物合成治疗肿瘤的理论依据是相比正常细胞,肿瘤细胞产生更多的核糖体,一旦核糖体生物合成被抑制,肿瘤细胞比正常细胞更容易受到影响。这里药物设计的基本思想是针对核糖体生物合成的具体几个步骤,包括rRNA转录、Pre-RNA加工、核糖体组装等。事实上,有许多经临床批准的药物,其治疗作用至少部分是通过破坏核糖体生物合成来实现的,例如,顺铂、奥沙利铂、阿霉素、米托蒽醌和丝裂霉素C均在rRNA转录水平上抑制核糖体的合成,而5-氟尿嘧啶、喜树碱在rRNA加工水平上抑制核糖体的合成[19, 25-26]。
5-氟尿嘧啶可以抑制胸苷酸合酶,与47S pre-rRNA结合,抑制rRNA后期加工,导致细胞凋亡。5-氟尿嘧啶的细胞毒性依赖于P53,且依赖于HDM2的活性。顺铂能诱导DNA链内交联,抑制RNA聚合酶Ⅰ转录。低浓度顺铂可特异性抑制RNA聚合酶Ⅰ,而不抑制RNA聚合酶Ⅱ的转录,从而在rRNA转录水平上抑制核糖体生物合成。考虑到顺铂的抗肿瘤作用也与其DNA损伤作用有关,有研究指出,顺铂的类似物奥沙利铂不是通过DNA损伤反应杀死细胞,它主要通过抑制核糖体生物合成引发核仁应激发挥抗癌作用[27]。其他抑制了核糖体生物合成的抗肿瘤药物见表 1[19, 26, 28]。
为了特异性抑制核糖体生物合成而不产生遗传毒性,科学家们正在努力开发新的药物,现在研究最多的是抑制rRNA转录的化合物,比如CX-5461,它可以选择性抑制RNA聚合酶Ⅰ的功能从而实现抑制核糖体生物合成。到目前为止,CX-5461已被广泛研究,在多种肿瘤包括白血病、淋巴瘤、骨髓瘤、前列腺癌、骨肉瘤、卵巢癌和神经母细胞瘤中具有抗癌作用。具有相似作用的化合物还有CX-3543、BMH-21等。
但是这些化合物存在一些局限性:首先,并不是所有的细胞系对RNA聚合酶Ⅰ抑制剂都敏感;其次,两个CX-衍生物和BMH-21是一种DNA结合化合物,能够直接与任何富含GC或含有G4的DNA序列相互作用,表明它们可以与其他基因组DNA结合;再次,已有案例表明CX-5461能够产生获得性耐药[29]。
5 展望核糖体生物合成逐渐被认为是治疗肿瘤的有效靶点,越来越多抗癌新药的开发都把方向对准核糖体生物合成,例如Scull等[30]利用高通量筛选器对核糖体生物合成抑制剂进行鉴定,开发了有效的rRNA合成抑制剂。当然,设计药物的方向还可以针对核糖体生物合成的其他步骤,例如针对核糖体的组装步骤,或者针对特定核糖体组分的化学修饰。此外,联合靶向核糖体生物合成的药物与核糖体生物合成相关信号通路可能会产生更好的治疗效果,例如联合应用rDNA转录抑制剂(CX-5461)与mTORC1抑制剂依维莫司治疗淋巴瘤[31],或者将CX-5461与pan-PIM激酶抑制剂CX-6258联用治疗前列腺癌[32]。总之,针对核糖体生物合成开发抗癌新药将具有广阔的应用前景。
作者贡献
钱丽丽:资料收集、文章撰写
张红河:研究指导、文章修改
| [1] |
Derenzini M, Montanaro L, Trerè D. Ribosome biogenesis and cancer[J]. Acta Histochem, 2017, 119(3): 190-197. DOI:10.1016/j.acthis.2017.01.009 |
| [2] |
De La Cruz J, Gomez-Herreros F, Rodriguez-Galan O, Reviews. Feedback regulation of ribosome assembly[J]. Curr Genet, 2018, 64(2): 393-404. |
| [3] |
Pelletier J, Thomas G, Volarević S. Corrigendum: Ribosome biogenesis in cancer: new players and therapeutic avenues[J]. Nat Rev Cancer, 2018, 18(2): 134. DOI:10.1038/nrc.2018.3 |
| [4] |
Bustelo X R, Dosil M. Ribosome biogenesis and cancer: basic and translational challenges[J]. Curr Opin Genet Dev, 2018, 48: 22-29. DOI:10.1016/j.gde.2017.10.003 |
| [5] |
Sharma S, Lafontaine DLJ. 'View from a bridge': a new perspective oneukaryotic rRNA base modification[J]. Trends Biochem Sci, 2015, 40(10): 560-575. DOI:10.1016/j.tibs.2015.07.008 |
| [6] |
Peña C, Hurt E, Panse VG. Eukaryotic ribosome assembly, transport and quality control[J]. Nat Struct Mol Biol, 2017, 24(9): 689-699. DOI:10.1038/nsmb.3454 |
| [7] |
Stults DM, Killen MW, Williamson EP, Reviews. Human rRNA gene clusters are recombinational hotspots in cancer[J]. Cancer Res, 2009, 69(23): 9096-9104. DOI:10.1158/0008-5472.CAN-09-2680 |
| [8] |
Wang M, Lemos B. Ribosomal DNA copy number amplification and loss in human cancers is linked to tumor genetic context, nucleolus activity, and proliferation[J]. PLoS Genet, 2017, 13(9): e1006994. DOI:10.1371/journal.pgen.1006994 |
| [9] |
Hosgood HD, Hu W, Rothman N, Reviews. Variation in ribosomal DNA copy number is associated with lung cancer risk in a prospective cohort study[J]. Carcinogenesis, 2019, 40(8): 975-978. DOI:10.1093/carcin/bgz052 |
| [10] |
Tsoi H, Lam KC, Dong Y, Reviews. Pre-45s rRNA promotes colon cancer and is associated with poor survival of CRC patients[J]. Oncogene, 2017, 36(44): 6109-6118. DOI:10.1038/onc.2017.86 |
| [11] |
Zhou H, Wang Y, Lv Q, Reviews. Overexpression of Ribosomal RNA in the Development of Human Cervical Cancer Is Associated with rDNA Promoter Hypomethylation[J]. PLoS One, 2016, 11(10): e0163340. DOI:10.1371/journal.pone.0163340 |
| [12] |
Penzo M, Galbiati A, Treré D, Reviews. The importance of being (slightly) modified: The role of rRNA editing on gene expression control and its connections with cancer[J]. Biochim Biophys Acta, 2016, 1866(2): 330-338. |
| [13] |
Aspesi A, Ellis SR. Rare ribosomopathies: insights into mechanisms of cancer[J]. Nat Rev Cancer, 2019, 19(4): 228-238. DOI:10.1038/s41568-019-0105-0 |
| [14] |
Vlachos A. Acquired ribosomopathies in leukemia and solid tumors[J]. Hematology Am Soc Hematol Educ Program, 2017, 2017(1): 716-719. DOI:10.1182/asheducation-2017.1.716 |
| [15] |
Goudarzi KM, Lindström MS. Role of ribosomal protein mutations in tumor development (Review)[J]. Int J Oncol, 2016, 48(4): 1313-1324. |
| [16] |
Wang W, Nag S, Zhang X, Reviews. Ribosomal proteins and human diseases: pathogenesis, molecular mechanisms, and therapeutic implications[J]. Med Res Rev, 2015, 35(2): 225-285. |
| [17] |
Guimaraes JC, Zavolan M. Patterns of ribosomal protein expression specify normal and malignant human cells[J]. Genome Biol, 2016, 17(1): 236. |
| [18] |
Brombin A, Joly JS, Jamen F. New tricks for an old dog: ribosome biogenesis contributes to stem cell homeostasis[J]. Curr Opin Genet Dev, 2015, 34: 61-70. DOI:10.1016/j.gde.2015.07.006 |
| [19] |
Woods SJ, Hannan KM, Pearson RB, Reviews. The nucleolus as a fundamental regulator of the p53 response and a new target for cancer therapy[J]. Biochim Biophys Acta, 2015, 1849(7): 821-829. DOI:10.1016/j.bbagrm.2014.10.007 |
| [20] |
Quin JE, Devlin JR, Cameron D, Reviews. Targeting the nucleolus for cancer intervention[J]. Biochim Biophys Acta, 2014, 1842(6): 802-816. DOI:10.1016/j.bbadis.2013.12.009 |
| [21] |
Hannan KM, Sanij E, Hein N, Reviews. Signaling to the ribosome in cancer-It is more than just mTORC1[J]. IUBMB Life, 2011, 63(2): 79-85. DOI:10.1002/iub.428 |
| [22] |
Gentilella A, Kozma SC, Thomas G. A liaison between mTOR signaling, ribosome biogenesis and cancer[J]. Biochim Biophys Acta, 2015, 1849(7): 812-820. DOI:10.1016/j.bbagrm.2015.02.005 |
| [23] |
Iadevaia V, Liu R, Proud CG. mTORC1 signaling controls multiple steps in ribosome biogenesis[J]. Semin Cell Dev Biol, 2014, 36: 113-120. DOI:10.1016/j.semcdb.2014.08.004 |
| [24] |
Deisenroth C, Franklin DA, Zhang Y. The Evolution of the Ribosomal Protein-MDM2-p53 Pathway[J]. Cold Spring Harb Perspect Med, 2016, 6(12): a026138. DOI:10.1101/cshperspect.a026138 |
| [25] |
Derenzini E, Rossi A, Treré D. Treating hematological malignancies with drugs inhibiting ribosome biogenesis: when and why[J]. J Hematol Oncol, 2018, 11(1): 75. |
| [26] |
Burger K, Eick D. Functional ribosome biogenesis is a prerequisite for p53 destabilization: impact of chemotherapy on nucleolar functions and RNA metabolism[J]. Biol Chem, 2013, 394(9): 1133-1143. DOI:10.1515/hsz-2013-0153 |
| [27] |
Bruno PM, Liu Y, Park GY, Reviews. A subset of platinum-containing chemotherapeutic agents kills cells by inducing ribosome biogenesis stress[J]. Nat Med, 2017, 23(4): 461-471. DOI:10.1038/nm.4291 |
| [28] |
Hein N, Hannan KM, George AJ, Reviews. The nucleolus: an emerging target for cancer therapy[J]. Trends Mol Med, 2013, 19(11): 643-654. DOI:10.1016/j.molmed.2013.07.005 |
| [29] |
Catez F, Dalla Venezia N, Marcel V, Reviews. Ribosome biogenesis: An emerging druggable pathway for cancer therapeutics[J]. Biochem Pharmacol, 2019, 159: 74-81. DOI:10.1016/j.bcp.2018.11.014 |
| [30] |
Scull CE, Zhang Y, Tower N, Reviews. Discovery of novel inhibitors of ribosome biogenesis by innovative high throughput screening strategies[J]. Biochem J, 2019, 476(15): 2209-2219. DOI:10.1042/BCJ20190207 |
| [31] |
Devlin JR, Hannan KM, Hein N, Reviews. Combination Therapy Targeting Ribosome Biogenesis and mRNA Translation Synergistically Extends Survival in MYC-Driven Lymphoma[J]. Cancer Discov, 2016, 6(1): 59-70. DOI:10.1158/2159-8290.CD-14-0673 |
| [32] |
Lawrence MG, Obinata D, Sandhu S, Reviews. Patient-derived models of abiraterone- and enzalutamide-resistant prostate cancer reveal sensitivity to ribosome-directed therapy[J]. Eur Urol, 2018, 74(5): 562-572. |
 2020, Vol. 47
2020, Vol. 47