文章信息
- 福建地区汉族人群中CLDN23基因SNPs多态性与胃癌遗传易感性及预后的相关性
- Correlation of CLDN23 SNP Polymorphisms with Hereditary Susceptibility and Prognosis of Gastric Carcinoma in Han Nationality in Fujian
- 肿瘤防治研究, 2020, 47(5): 353-357
- Cancer Research on Prevention and Treatment, 2020, 47(5): 353-357
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2020.19.1063
- 收稿日期: 2019-08-21
- 修回日期: 2020-03-24
2. 362000 泉州,福建医科大学附属第二医院病理科;
3. 100083 北京,北京大学医学部病理学系
2. Department of Pathology, Affiliated Second Hospital of Fujian Medical University, Quanzhou 362000, China;
3. Department of Pathology, Peking University Health Science Center, Beijing 100083, China
单核苷酸多态性(single nucleotide polymorphism, SNP)是人类遗传变异中最常见的一种,占所有已知基因多态性的90%以上。存在于编码区及其紧邻上下游序列的SNPs可能改变基因的表达水平和表达产物,从而使蛋白质的结构和功能发生变化,引起不同个体对环境因素的敏感度不同,对肿瘤等疾病也存在易感差异。在胃癌人群和健康对照人群中进行SNPs对比研究,可以确定SNPs和(或)其邻近变异与胃癌的关联。
有研究发现恶性肿瘤的侵袭转移与上皮间质转化(epithelial-mesenchymal transitions, EMT)密切相关。紧密连接结构与功能的改变参与EMT过程,可调控肿瘤的侵袭转移[1-4]。Claudin蛋白是构成紧密连接的骨架蛋白,也是紧密连接最主要的功能分子[5],其编码基因Claudin基因家族包括24个成员[6]。研究发现肠型胃癌的CLDN23基因下调[7],可作为一种候选胃癌抑制基因,但其对胃癌的易感性及预后的影响尚未见报道。本研究通过应用多重PCR-LDR方法检测Claudin-23基因CLDN23的SNPs多态性在福建地区汉族人群原发性胃癌发生发展中的意义。
1 资料与方法 1.1 CLDN23 SNPs多态性研究的对象对照组:福建地区无血缘关系健康体检者197例,其中男125例,女72例,中位年龄52岁,询问病史及健康体检排除其他肿瘤病史,取外周血样提取基因组DNA。病例组:福建医科大学附属第一医院1993—2007年间手术切除的原发性胃癌存档蜡块215例,其中男163例、女52例,中位年龄58岁,所有病例均经病理证实。取非肿瘤性组织蜡块提取基因组DNA。
按WHO肿瘤病理学及遗传学分类(2010年消化系统肿瘤分册)对所有病例进行组织学分型及TNM分期。215例原发性胃癌中,早期胃癌(pT1)31例,进展期胃癌184例。进展期胃癌中高分化14例,中分化39例,低分化及未分化癌90例,黏液腺癌和印戒细胞癌41例; pT分期为T2者38例、T3者58例,T4者88例; 区域淋巴结转移者162例,未发生转移者22例。对上述病例进行随访,获回访资料108例,其中死亡病例107例,存活病例1例,随访时间为1~78月。
1.2 试剂总DNA抽提试剂盒TaKaRa DEXPATTM试剂(日本TaKaRa公司); AxyPrep-96全血基因组DNA试剂盒(美国AXYGEN公司); Qiagen HotStarTaq酶体系(酶5 u/μl,buffer,Q-solution, Mg2+等)(Qiagen德国生物技术有限公司)。
1.3 PCR引物及LDR探针合成PCR引物及LDR探针合成由上海翼和生物应用有限公司设计并合成。引物序列、扩增片段长度及LDR探针见表 1、表 2。

|

|
石蜡包埋组织基因组DNA和对照组全血基因组DNA的提取按DEXPATTM试剂盒说明进行。0.8%琼脂糖凝胶电泳检测基因组DNA多重PCR反应产物的完整性。PCR扩增SNP位点所在片段:95℃ 15 min,94℃ 30 s,59℃ 1 min,72℃ 1 min,72℃ 7 min,35个循环,3.0%琼脂糖凝胶,0.5×TBE电泳分析。PCR产物的连接酶检测反应(LDR):取1 μl LDR连接产物与1 μl ABI GS-500 ROX荧光标记分子和去离子甲酰胺上样液混合,95℃加热变性2 min,冰中骤冷,于5%聚丙烯酰胺和5 mol/L尿素中3 000 V电泳2.5 h,应用GENESCANTM672软件进行数据收集、泳道线校正、迁移片段大小测量和校正内在分子量标准; 应用Gene Mapper软件进行数据分析和基因分型。测序仪测序检验PCR-LDR的结果。
1.5 统计学方法应用SPSS16.0软件包,对胃癌病例组与健康对照组各等位基因频率和基因型频率差异及胃癌病例组各基因型分布与临床病理因素差异作χ2及精确概率分析,P < 0.05为差异有统计学意义。应用SHEsis软件包(http://analysis.bio-x.cn)进行Hardy-Weinberg平衡检验分析,P > 0.05为符合Hardy-Weinberg平衡规律。
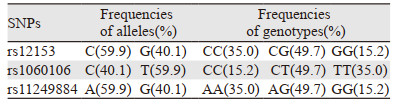
2 结果 2.1 CLDN23 3个位点的SNP多态性在福建汉族人群中的分布经PCR-LDR检测,197例中国福建地区汉族人群的CLDN23 rs12153、rs1060106和rs11249884的3个SNP位点,各等位基因频率和基因型分布频率见表 3。CLDN23 rs12153位点为CC、CG和GG三种基因型,rs1060106位点为CC、CT和TT三种基因型,rs11249884位点为AA、AG和GG三种基因型。各位点SNP基因型经PCR产物测序证实。
|
福建汉族人群中CLDN23 SNP位点rs12153、rs1060106及rs11249884的基因型及等位基因频率在胃癌患者和健康者之间差异均无统计学意义(均P > 0.05)。
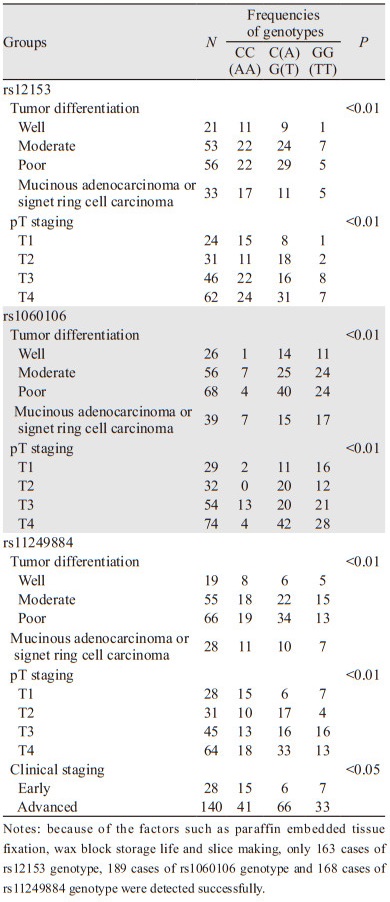
2.3 CLDN23 SNP位点rs12153、rs1060106和rs11249884的基因型分布与胃癌临床病理特征之间的关系CLDN23 3个SNPs位点的基因型分布均与胃癌分化程度、pT分期显著相关(均P < 0.01); rs11249884基因型分布与胃癌临床分期显著相关(P < 0.05),见表 4; rs12153和rs1060106基因型分布均与胃癌临床分期无显著相关性(均P > 0.05)。3个位点的基因型分布与胃癌患者性别、年龄及有无淋巴结转移差异均无统计学意义(P > 0.05)。
|
rs12153、rs1060106和rs11249884位点相互邻近。rs12153和rs1060106、rs12153和rs11249884遗传连锁不平衡分析结果分别为:D’=0.955、r2=0.907和D’=0.972、r2=0.864,两个位点之间无明显的遗传连锁不平衡性; rs1060106和rs11249884遗传连锁不平衡分析结果为:D’=0.967、r2=0.840,两个位点之间无明显遗传连锁不平衡性。
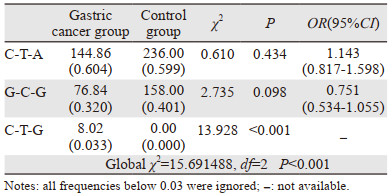
2.5 CLDN23基因SNP位点rs12153、rs1060106和rs11249884单体基因型分析CLDN23 SNP位点rs12153、rs1060106和rs11249884组合的基因单体型有C-T-A、G-C-G、C-C-G、C-T-G、G-C-A、G-T-A和G-T-G 7种。结果显示C-T-G单体型在胃癌病例组与健康对照组之间差异有统计意义(χ2=15.691488, P < 0.001),见表 5。
|
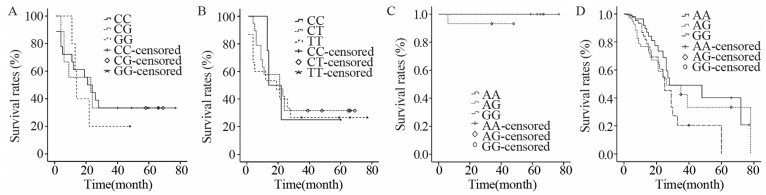
将有完整随访资料的108例原发性胃癌患者的生存时间分别和rs12153、rs1060106和rs11249884的各基因型分布及3个SNP位点构建的各单体基因型分布进行统计分析。结果显示rs12153位点CC基因型患者术后生存率高于CG基因型患者(P < 0.01); 而CC基因型患者与GG基因型患者之间、CG基因型患者与GG基因型患者之间术后生存率差异均无统计学意义(P > 0.05),见图 1A。rs1060106位点TT基因型患者和CT基因型患者术后生存率均高于CC基因型患者,差异有统计学意义(均P < 0.01); 而TT基因型患者与CT基因型患者之间术后生存率差异无统计学意义(P > 0.05),见图 1B。3个SNP位点中,只有rs11249884位点的基因型分布与临床分期高度相关,而临床分期可显著影响生存率。进一步分组统计显示,无论是早期胃癌组还是进展期胃癌组,rs11249884位点AA、AG与GG基因型患者之间术后生存率差异均无统计学意义(均P > 0.05),见图 1C、D。另外,3个位点构建的GG-CC-GG、CG-CT-AG、CC-TT-AA单体基因型患者术后生存率差异无统计学意义(P > 0.05)。
|
| A: different CLDN23 rs12153 genotypes; B: different CLDN23 rs1060106 genotypes; C: early gastric carcinoma patients with different CLDN23 rs11249884 genotypes; D: advanced gastric carcinoma patients with different CLDN23 rs11249884 genotypes. 图 1 CLDN23基因3个SNPs位点各基因型胃癌患者的Kaplan-Meier生存曲线 Figure 1 Kaplan-Meier survival curves of gastric carcinoma patients with different genotypes of CLDN23 SNP loci |
Claudin基因家族共24个成员,其序列的一致性为12.5%~69.7%,表明它们在进化上呈功能高度保守性。人类CLDN23基因位于8q23.1,包含有多个多态性位点,其编码的蛋白质含有292个氨基酸。位于编码区的SNP位点可能改变基因的表达水平和表达产物,从而使蛋白质的结构和功能发生变化,直接导致肿瘤的易感性。本研究通过检测CLDN23基因3个外显子SNP位点的多态性,探讨其与胃癌发生发展的关系。
对于Claudin基因多态性的研究,Liao等通过基因测序研究发现CLDN10基因SNP位点的多态性与乳腺癌的患癌风险相关[8]; Perdomo-Ramirez等发现CLDN19基因的多态性与低镁血症相关[9]; 而CLDN1基因SNP位点的多态性则与血管性痴呆及三阴性乳腺癌相关[10-11]。这些均提示Claudin基因的多态性可影响Claudin蛋白功能和表达,从而与多种疾病的发生发展相关。
本研究应用PCR-LDR法检测CLDN23基因3个SNP位点rs12153、rs1060106和rs11249884在原发性胃癌病例组和健康对照组中的基因型,发现这3个SNP位点的多态性与胃癌遗传易感性无关,但与胃癌组织学分化程度、pT分期相关; rs12153、rs1060106的多态性与胃癌患者的预后有关; 3个SNP位点之间无连锁不平衡,且存在有统计学意义的基因单体型分布。
紧密连接蛋白的表达异常或者结构功能异常是EMT过程中的关键,也是参与腺癌进展的重要病理变化[12]。Claudin分子在维持紧密连接结构和功能中起重要作用[13],其异常表达可以促进癌细胞的运动、侵袭能力和生存[14-15]。如前所述,研究发现在肠型胃癌中CLDN23基因下调,可作为一种候选胃癌抑制基因。本研究结果也显示,胃癌患者中CLDN23基因rs12153、rs1060106和rs11249884的多态性与组织学分化、pT分期有关,提示SNPs可能对Claudin-23的差异表达起调节作用。CLDN23基因表达调节的机制鲜有报道,有研究显示组蛋白甲基化水平,包括H3K27me3(H3 lysine 27 trimethylation)和H3K4me3(H3 lysine 4 trimethylation)的相互平衡是调节CLDN23基因表达的基础; 并且在大肠癌中EZH2(enhancer of zeste homolog 2)诱导下的H3K27me3过表达会导致CLDN23基因表达下调[16]; 此外转录因子Tcf7l2/Hnf4a可上调CLDN23的基因表达[17]。CLDN23的SNP是否通过上述分子发挥对其表达的调节作用还未知。
rs12153、rs1060106和rs11249884在CLDN23基因上邻近,本研究发现C-T-G单体型在胃癌病例组与健康对照组之间差异有统计学意义,结果提示C-T-G单体型与胃癌易感性相关,还需要更多的标签SNPs位点或扩大样本数量分析加以验证。有研究[18-19]认为肿瘤相关基因中,包含某些SNP位点的单体型可影响该基因的转录,引起转录后蛋白的空间结构或表达水平的改变,影响蛋白的功能,从而与肿瘤易感性相关。我们推测C-T-G单体型或影响CLDN23基因转录,导致Claudin-23表达下调并引起细胞间紧密连接异常,与胃癌的发生相关。
胃癌发生发展是多基因综合作用的结果。本研究发现,CLDN23基因rs12153、rs1060106的多态性与胃癌术后生存率具有明显相关性,这可能为评估胃癌患者的预后提供新的参考指标。
综上,我们首次研究了CLDN23基因SNPs位点多态性与中国人群胃癌发病风险之间的关系,认为CLDN23基因SNPs多态性可能与胃癌的进展相关。尽管本研究病例组及对照组的基因频率分布均符合Hardy-Weinberg平衡,说明选择的样本具有群体代表性,但样本数量较小、选取位点数较少,未来研究需要增加样本数量及SNP位点数,以得到更多CLDN23基因SNP多态性与胃癌之间关系的研究证据。
作者贡献
陈奕昭:实验项目负责人及文稿撰写
吴春林:指导并参与实验方案设计和实施
贺慧颖:指导实验结果分析和文章修改
| [1] |
Wang K, Xu C, Li W, et al. Emerging clinical significance of claudin-7 in colorectal cancer: a review[J]. Cancer Manag Res, 2018, 10: 3741-3752. DOI:10.2147/CMAR.S175383 |
| [2] |
Blanchard AA, Ma X, Wang N, et al. Claudin 1 Is Highly Upregulated by PKC in MCF7 Human Breast Cancer Cells and Correlates Positively with PKCε in Patient Biopsies[J]. Transl Oncol, 2019, 12(3): 561-575. DOI:10.1016/j.tranon.2018.12.011 |
| [3] |
Katayama A, Handa T, Komatsu K, et al. Expression patterns of claudins in patients with triple-negative breast cancer are associated with nodal metastasis and worse outcome[J]. Pathol Int, 2017, 67(8): 404-413. DOI:10.1111/pin.12560 |
| [4] |
Nilchian A, Johansson J, Ghalali A, et al. CXADR-Mediated Formation of an AKT Inhibitory Signalosome at Tight Junctions Controls Epithelial-Mesenchymal Plasticity in Breast Cancer[J]. Cancer Res, 2019, 79(1): 47-60. DOI:10.1158/0008-5472.CAN-18-1742 |
| [5] |
Heiskala M, Peterson PA, Yang Y. The roles of claudin superfamily proteins in paracellular transport[J]. Traffic, 2001, 2(2): 93-98. |
| [6] |
Tsukita S, Furuse M, Itoh M. Multifunctional strands in tight junctions[J]. Nat Rev Mol Cell Biol, 2001, 2(4): 285-293. DOI:10.1038/35067088 |
| [7] |
Katoh M, Katoh M. CLDN23 gene, frequently down-regulated in intestinal-type gastric cancer, is a novel member of CLAUDIN gene family[J]. Int J Med, 2003, 11(6): 683-689. |
| [8] |
Liao J, Li J, Cheng H, et al. CLDN10 single nucleotide polymorphism rs1325774 alters the risk of breast cancer in south chinese women[J]. Medicine (Baltimore), 2018, 97(49): e13187. DOI:10.1097/MD.0000000000013187 |
| [9] |
Perdomo-Ramirez A, Aguirre M, Davitaia T, et al. Characterization of two novel mutations in the claudin-16 and claudin-19 genes that cause familial hypomagnesemia with hypercalciuria and nephrocalcinosis[J]. Gene, 2019, 689: 227-234. DOI:10.1016/j.gene.2018.12.024 |
| [10] |
Srinivasan V, Braidy N, Xu YH, et al. Association of genetic polymorphisms of claudin-1 with small vessel vascular dementia[J]. Clin Exp Pharmacol Physiol, 2017, 44(6): 623-630. DOI:10.1111/1440-1681.12747 |
| [11] |
Hu A, Li J, Ruan S, et al. Polymorphisms in CLDN1 are associated with age and differentiation of triple-negative breast cancer patients[J]. Biosci Rep, 2019, 39(4): pii: BSR20181952. DOI:10.1042/BSR20181952 |
| [12] |
Grände M, Franzen A, Karlsson JO, et al. Transforming growth factor-beta and epidermal growth factor synergistically stimulate epithelial to mesenchymal transition (EMT) through a MEK-dependent mechanism in primary cultured pig thyrocytes[J]. J Cell Sci, 2002, 115(Pt 22): 4227-4236. |
| [13] |
Tsukita S, Tanaka H, Tamura A. The Claudins: From Tight Junctions to Biological Systems[J]. Trends Biochem Sci, 2019, 44(2): 141-152. DOI:10.1016/j.tibs.2018.09.008 |
| [14] |
Agarwal R, D Souza T, Morln PJ. Claudin-3 and claudln-4 expression in ovarian epithelial cells enhances invasion and is associated with increased matrix metalloproteinase-2 activity[J]. Cancer Res, 2005, 65(16): 7378-7385. DOI:10.1158/0008-5472.CAN-05-1036 |
| [15] |
Kim DH, Lu Q, Chen YH. Claudin-7 modulates cell-matrix adhesion that controls cell migration, invasion and attachment of human HCC827 lung cancer cells[J]. Oncol Lett, 2019, 17(3): 2890-2896. |
| [16] |
Maryan N, Statkiewicz M, Mikula M, et al. Regulation of the expression of claudin 23 by the enhancer of zeste 2 polycomb group protein in colorectal cancer[J]. Mol Med Rep, 2015, 12(1): 728-736. DOI:10.3892/mmr.2015.3378 |
| [17] |
Lili LN, Farkas AE, Gerner-Smidt C, et al. Claudin-based barrier differentiation in the colonic epithelial crypt niche involves Hopx/Klf4 and Tcf7l2/Hnf4-α cascades[J]. Tissue Barriers, 2016, 4(3): e1214038. DOI:10.1080/21688370.2016.1214038 |
| [18] |
Freedman ND, Ahn J, Hou L, et al. Polymorphisms in estrogen- and androgen-metabolizing genes and the risk of gastric cancer[J]. Carcinogenesis, 2009, 30(1): 71-77. DOI:10.1093/carcin/bgn258 |
| [19] |
Jiang JH, Jia WH, Chen HK, et al. Genetic polymorphisms of CYP2A13 and its relationship to nasopharyngeal carcinoma in the Cantonese population[J]. J Transl Med, 2004, 29, 2(1): 24.
|
 2020, Vol. 47
2020, Vol. 47


