文章信息
- 胰腺癌诊断和预后关键生物标志物的筛选鉴定和综合分析
- Identification and Integrated Analysis of Key Biomarkers for Diagnosis and Prognosis of Pancreatic Adenocarcinoma
- 肿瘤防治研究, 2020, 47(1): 46-51
- Cancer Research on Prevention and Treatment, 2020, 47(1): 46-51
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2020.19.0743
- 收稿日期: 2019-06-05
- 修回日期: 2019-09-26
2. 121000 锦州, 锦州医科大学附属第一医院药学部
2. Department of Pharmacy, The First Af filiated Hospital of Jinzhou Medical University, Jinzhou 121000, China
胰腺癌是一种恶性程度高, 诊断和治疗都很困难, 且预后极差的消化道恶性肿瘤[1], 但肿瘤发生的具体分子网络机制尚未完全阐明。研究肿瘤发生的潜在机制可能是延长患者生存时间的关键。近年来, 基于高通量平台的基因分析已成为一种具有广泛临床应用前景的工具, 如肿瘤的分子诊断和分类、肿瘤反应和患者预后的预测等。微阵列技术探索与胰腺癌发生有关的基因表达产物, 揭示了数百个参与肿瘤发生过程的差异表达基因(DEGs), 在识别新的治疗靶点方面具有潜在的作用。本研究应用生物信息学分析和实验方法, 对胰腺癌进展过程中的DEGs进行了鉴定和验证, 进行了包括细胞成分(CC), 生物过程(BP)和分子功能(MF)的GO分析、KEGG通路分析并且进行了蛋白-蛋白质相互作用(PPI)网络分析, 探讨了可能在胰腺癌发生发展中起关键作用的Hub基因, 并在一些胰腺癌组织中用反转录定量聚合酶链反应(RT-qPCR)证实这些Hub基因的mRNA水平在胰腺癌组织中有差异表达。
1 资料与方法 1.1 微阵列数据集GSE15471、GSE16515、GSE28735、GSE62165是从NCBI GEO数据库下载的胰腺癌基因芯片数据集。
1.2 DEGs的筛选使用GEO2R交互式网络工具识别在不同实验条件下差异表达的基因, 并用LIMMA软件包识别数据集中的DEGs, 这些数据已经处理、归一化和转化。只有log|FC|>1且adj.P.Value < 0.05的基因被认为是有统计学意义的DEGs。此外, 选择性的将DEGs分为两组:上调差异表达基因(uDEGs)和下调的差异表达基因(dDEGs), 使用韦恩图工具(http://bioinfogp.cnb.csic.es/tools/venny/index.html)分别得到了所识别的uDEGs和dDEGs的共同交集。
1.3 DEGs的GO分析和KEGG分析本研究使用DAVID对两组DEGs进行了显著性富集分析, 以P < 0.05为判定标准。
1.4 构建生物网络为了评估被识别的DEGs之间的相互作用, 我们将它们映射到STRING数据库并构建了PPI网络。将PPI网络导入Cytoscape, 使用Cytoscape插件cytohubba和mcode对网络中的重要节点和子网络进行预测和探索。
1.5 Hub基因的生存分析GEPIA是一种交互式探索生存相关性的工具, 包含多项癌症研究的生存数据。采用Kaplan Meier图和Log rank P值评估两组患者的总生存率, Log rank P < 0.05为截止标准, 对Hub基因进行了生存分析, 以验证结果。
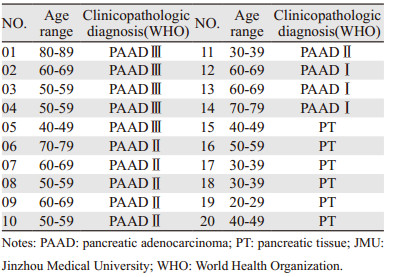
1.6 患者样本2019年1月-2019年6月在锦州医科大学附属第一医院采集14例胰腺癌标本; 此外, 收集了6例严重胰腺损伤并需要手术治疗的正常成人胰腺组织标本, 用于研究的人群特征见表 1。在应用组织样本之前, 所有患者均获得知情同意。本研究符合"赫尔辛基宣言"的规定, 并得到了锦州医科大学医学伦理委员会的批准。标本由临床病理学家根据消化系统肿瘤WHO分类(WHO Classification of Tumors of the Digestive System)2012第四版进行组织学分级。每个活检标本分为两个部分, 一部分用于常规组织学诊断, 另一部分用于本研究。
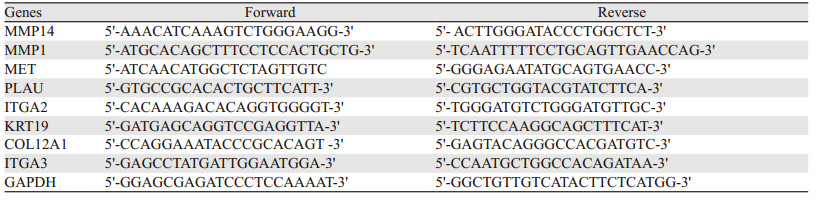
首先用TRIzol®试剂(InvitrogenTM)从组织中提取总RNA, 而后PrimeScriptTM RT-PCR试剂盒(TakaraBio)合成mRNA cDNA。反转录聚合酶链反应(RT-qPCR)采用7500实时荧光PCR系统(Thermo Fisher)。先在95˚C下变性10 min, 而后在95˚C下进行40个循环15 s, 然后在60˚C下用SYBR®绿色混合物进行1 min的变性, 以GAPDH作为内源性对照。所用到的引物序列参见表 2。
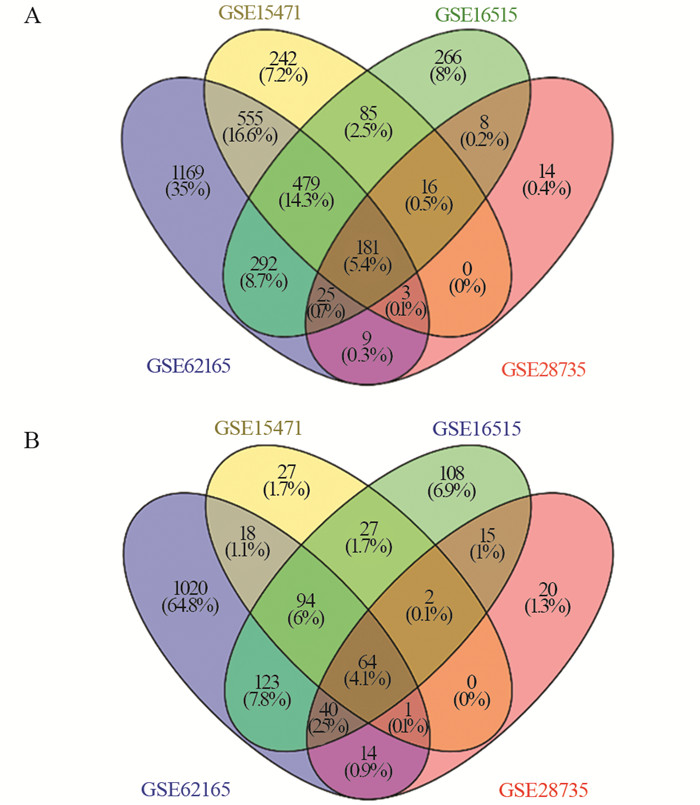
共有245个基因被鉴定为GSE15471、GSE16515、GSE28735、GSE62165之间的DEGs, 其中181个基因表达上调, 64个基因表达下调, 见图 1。
|
| uDEGs: up-regulated differentially expressed genes; dDEGs: down-regulated differentially expressed genes. 图 1 四个mRNA表达谱(GSE15471、GSE16515、GSE28735、GSE62165)中uDEGs(A)和dDEGs(B)的Venn图 Figure 1 Venn diagram of uDEGs(A) and dDEGs(B) in four mRNA expression profiles (GSE15471, GSE16515, GSE28735 and GSE62165) |
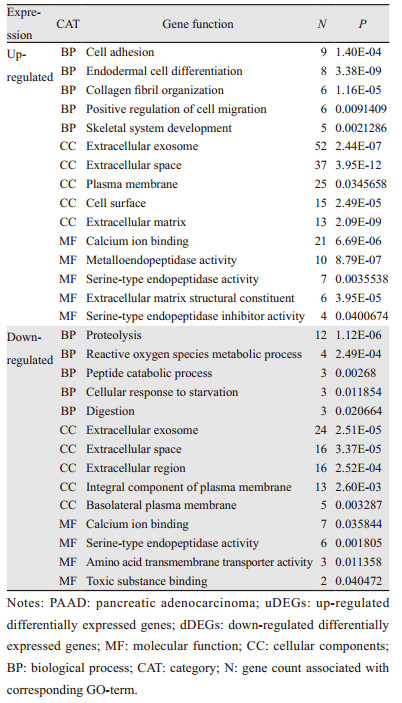
GO分析表明uDEGs和dDEGs在生物过程、细胞成分、分子功能中分别富集, 结果见表 3。
|
KEGG分析结果表示, uDEGs与ECM-receptor interaction、Focal adhesion、Amoebiasis、PI3K-Akt signaling pathway信号通路密切相关。而dDEGs与Pancreatic secretion、Protein digestion and absorption、Complement and coagulation cascades、ErbB signaling pathway信号通路密切相关。
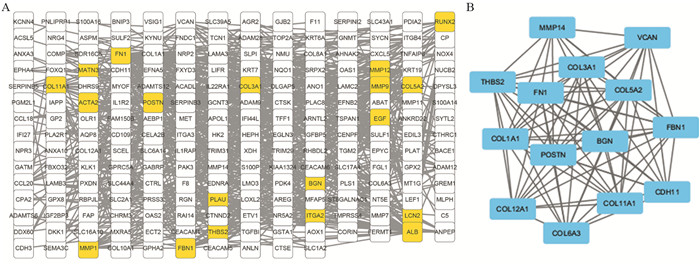
2.4 PPI网络构建用筛选出的DEGs构建PPI网络, 结果见图 2A。在STRING数据库构建的PPI网络中, 可见网络中共有206个节点和747条边。用mcode插件筛选出的结果见图 2B。用cytohubba按网络特征对节点进行排序, 从DEGs中筛选出Top 25的差异基因包括FN1、ALB、EGF、MMP9、COL1A1、POSTN、COL3A1、FBN1、BGN、VCAN、MMP14、MMP1、MET、MMP7、RUNX2、PLAU、ITGA2、THBS2、MMP12、KRT19、LCN2、COL5A2、COL12A1、COMP、ITGA3。
|
| Yellow dots: degree >10. 图 2 使用String数据库构建DEGs的蛋白质相互作用网络图(A); 蛋白质相互作用网络进行模块分析后得到的重要模块(B) Figure 2 Protein-protein interaction (PPI) network diagram of DEGs(A) constructed using STRING database and important module(B) obtained using PPI network |
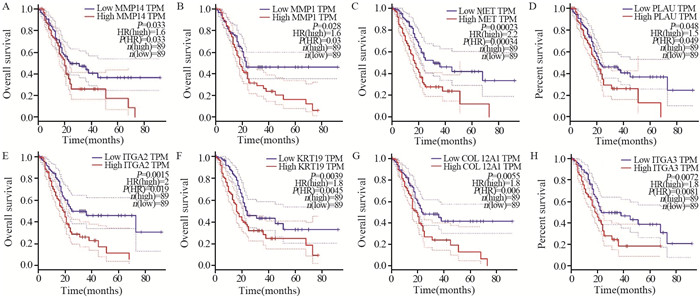
在Top 25的基因中用GEPIA预测, 其中MMP14、MMP1、MET、PLAU、ITGA2、KRT19、COL12A1、ITGA3的过表达可能与胰腺癌患者生存不良有关(P < 0.05), 见图 3。
|
| 图 3 Kaplan-Meier曲线评估Hub基因对PAAD患者的预后意义 Figure 3 Prognostic significance of Hub gene interaction in PAAD patients evaluated by Kaplan-Meier curves |
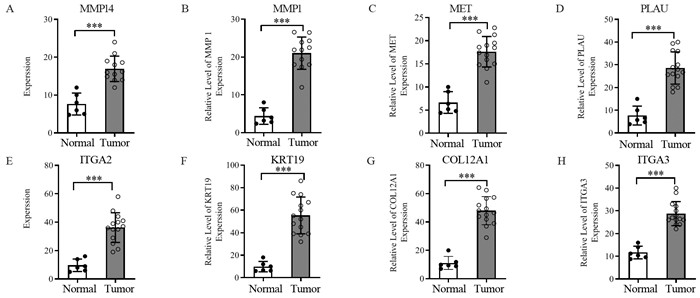
本研究旨在确定基因芯片分析中识别的DEGs是否可以非选择性地应用于临床胰腺癌患者。RT-qPCR实验结果表明, MMP14、MMP1、MET、PLAU、ITGA2、KRT19、COL12A1、ITGA3基因表达明显上调, 与基因芯片分析结果一致。RT-qPCR结果验证了这些Hub基因在人胰腺癌组织中的mRNA水平高于对照组, 提示这8个Hub基因可能是胰腺癌患者的新的基因特征, 见图 4。
|
| ***: P < 0.001. 图 4 RT-qPCR法对临床样本MMP14、MMP1、MET、PLAU、ITGA2、KRT19、COL12A1、ITGA3基因的mRNA水平进行定量分析 Figure 4 Quantitative analysis of mRNA level of MMP14, MMP1, MET, PLAU, ITGA2, KRT19, COL12A1 and ITGA3 gene in clinical samples by RT-qPCR |
尽管目前胰腺癌的治疗方法取得了进展, 但胰腺癌仍然是一种顽固性癌症, 揭示胰腺癌的发病和分子机制对于预防、诊断和治疗胰腺癌具有重要意义。目前, 随着DNA芯片和高通量测序技术的迅速发展, 使在基因水平上对包括癌症在内的疾病进行研究成为可能。微阵列基因表达谱已被广泛应用于探索与肿瘤发生、诊断和治疗有关的DEGs。
肿瘤进展过程中的恶性转化是由一系列的基因改变引起的, 在这项研究中, 我们从GSE15471、GSE16515、GSE28735、GSE62165中提取数据, 并利用生物信息学分析方法在胰腺癌和正常组织之间鉴定了181个uDEGs和64个dDEGs。
结果表明, 由uDEGs和dDEGs主要参与细胞黏附、内皮细胞分化、胶原原纤维组织、细胞迁移的正向调控、蛋白水解作用、活性氧代谢等过程都是肿瘤发生、发展和化疗抵抗发生的重要机制, 这同部分研究是一致的[2-4]。此外, uDEGs富集的KEGG通路包括ECM-receptor interaction、Focal adhesion、Amoebiasis, 这些通路在患者预后[5]和癌细胞增殖[6]中起着重要的作用。dDEGs参与的Pancreatic secretion、Protein digestion and absorption、Complement and coagulation cascades通路, 它们在胰腺癌的发生和生长中起着重要作用[7]。
从DEGs构建的PPI网络中, 按连通性高低鉴别出Top 25的关键基因。在这25个候选基因中只有ALB、EGF表现为下调, 其余的基因都表现为上调。
利用GEPIA进行了胰腺癌患者生存分析, 过滤得到了8个基因与胰腺癌的生存分析曲线相关:MMP14、MMP1、MET、PLAU、ITGA2、KRT19、COL12A1、ITGA3。
MMP14(Matrix Metallopeptidase 14)被认为是在细胞事件中受到广泛调控并具有最高连通性的枢纽基因。MMP14过表达与鼻咽癌的临床分期、N分期和远处转移呈正相关。MMP14过表达是鼻咽癌患者潜在的不良预后因素[8]。MMP14在HeLa细胞侵袭中起重要作用, MMP14基因敲除有助于减轻宫颈癌细胞恶性表型[9]。MMP1在肿瘤组织中的过度表达已被认为与肿瘤的侵袭和转移有关。有研究表明, MMP1在大多数食管鳞癌组织中的表达明显高于非肿瘤组织, 并且MMP1的过表达与淋巴结转移、微血管密度和晚期TNM分期密切相关[10]。MET(MET Proto-Oncogene, Receptor Tyrosine Kinase)为编码受体酪氨酸激酶家族蛋白的一个成员也是原癌基因的产物。恶性黑色素瘤具有通过细胞固有自主机制发生表型转变的能力, 这种机制与MET的过表达相关[11]。通过抑制MET而控制肿瘤生长转移成为靶向药物研发的方向。PLAU(Plasminogen Activator)为一种编码纤溶酶原激活物的蛋白编码基因。研究表明, 与PLAU相关的疾病包乳腺癌[12]、胃癌[13]。ITGA2(Integrin Subunit Alpha 2)的差异表达在多种实体瘤的转移过程中起着重要的作用。ITGA2高表达不仅能抑制细胞迁移, 还能诱导胃癌细胞凋亡, ITGA2是胃癌潜在的治疗靶点[14]。另外, 还有研究表明, ITGA2在调节途径中通过焦点黏附途径相关蛋白如非受体酪氨酸激酶、蛋白酪氨酸激酶和AKT丝氨酸/苏氨酸激酶促进胰腺癌进展[15]。KRT19(Keratin 19)基因编码的蛋白质属于角蛋白家族。KRT19的阳性表达是胰导管腺癌患者独立的不良预后因素。KRT19过表达与胰导管腺癌的发生、发展和不良预后有关, 是诊断胰腺癌的有价值的生物标志物[16]。KRT19表达的调节可能与肿瘤干细胞重编程和药物敏感度有关, 这可能对癌症或肿瘤干细胞治疗具有临床意义[17]。COL12A1(Collagen Type XII Alpha 1 Chain)在肌上皮类型疾病发生中有统计学意义(PMID:27348394), 而肌上皮功能的丧失是一些肿瘤进展的关键步骤。COL12A1在卵巢癌上调超过20倍, 耐药与细胞外基质成分表达之间的关系密切[18]。ITGA3(Integrin Subunit Alpha 3)作为整合素蛋白的一员, 在结肠癌中存在异常表达的现象, 通过对肿瘤细胞的生物学行为产生影响, 促进肿瘤的发生发展。虽然单一的预后生物标志物已被广泛报道, 但其稳定性仍然是一个主要问题[19]。
我们进一步验证了8个Hub基因在胰腺癌诊断标记中的作用。综合分析表明它们是预测胰腺癌的一个有价值和稳健性的模型, 并且可以有效预测患者的预后生存状况。综上所述, 通过鉴定和综合分析后获得的Hub基因均深入参与了胰腺癌的发生或进展过程, 提示这些Hub基因可能作为该疾病的预后生物标志物或治疗靶点。揭示了一系列可能影响胰腺癌发生发展的靶点和途径, 为今后的研究提供了依据。
作者贡献
柳兴源:图表制作、数据整理、标本收集和文章撰写
李菁媛:数据分析和实验技术支持
杨静:文章总体构思,临床标本的诊断和鉴定
| [1] |
Dreyer SB, Chang DK, Bailey P, et al. Pancreatic cancer genomes:implications for clinical management and therapeutic development[J]. Clin Cancer Res, 2017, 23(7): 1638-1646. DOI:10.1158/1078-0432.CCR-16-2411 |
| [2] |
Alimbetov D, Askarova S, Umbayev B, et al. Pharmacological targeting of cell cycle, apoptotic and cell adhesion signaling pathways implicated in chemoresistance of cancer cells[J]. Int J Mol Sci, 2018, 19(6): E1690. DOI:10.3390/ijms19061690 |
| [3] |
Wang J, Boddupalli A, Koelbl J, et al. Degradation and remodeling of epitaxially grown collagen fibrils[J]. Cell Mol Bioeng, 2019, 12(1): 69-84. |
| [4] |
Lin Y, Jiang M, Chen W, et al. Cancer and ER stress:Mutual crosstalk between autophagy, oxidative stress and inflammatory response[J]. Biomed Pharmacother, 2019, 118: 109249. DOI:10.1016/j.biopha.2019.109249 |
| [5] |
Yeh MH, Tzeng YJ, Fu TY, et al. Extracellular matrix-receptor interaction signaling genes associated with inferior breast cancer survival[J]. Anticancer Res, 2018, 38(8): 4593-4605. DOI:10.21873/anticanres.12764 |
| [6] |
Rigiracciolo DC, Santolla MF, Lappano R, et al. Focal adhesion kinase (FAK) activation by estrogens involves GPER in triplenegative breast cancer cells[J]. J Exp Clin Cancer Res, 2019, 38(1): 58. DOI:10.1186/s13046-019-1056-8 |
| [7] |
Choi MH, Mejlaender-Andersen E, Manueldas S, et al. Mutation analysis by deep sequencing of pancreatic juice from patients with pancreatic ductal adenocarcinoma[J]. BMC cancer, 2019, 19(1): 11. DOI:10.1186/s12885-018-5195-7 |
| [8] |
Yan TH, Lin ZH, Jiang JH, et al. Matrix metalloproteinase 14 overexpression is correlated with the progression and poor prognosis of nasopharyngeal carcinoma[J]. Arch Med Res, 2015, 46(3): 186-192. DOI:10.1016/j.arcmed.2015.03.006 |
| [9] |
Zhang YH, Wang JJ, Li M, et al. Matrix metallopeptidase 14 plays an important role in regulating tumorigenic gene expression and invasion ability of hela cells[J]. Int J Gynecol Cancer, 2016, 26(3): 600-606. DOI:10.1097/IGC.0000000000000652 |
| [10] |
Liu M, Hu Y, Zhang MF, et al. MMP1 promotes tumor growth and metastasis in esophageal squamous cell carcinoma[J]. Cancer Lett, 2016, 377(1): 97-104. DOI:10.1016/j.canlet.2016.04.034 |
| [11] |
Adachi E, Sakai K, Nishiuchi T, et al. Different growth and metastatic phenotypes associated with a cell-intrinsic change of Met in metastatic melanoma[J]. Oncotarget, 2016, 7(43): 70779-70793. |
| [12] |
Doebar SC, Sieuwerts AM, De Weerd V, et al. Gene expression differences between ductal carcinoma in situ with and without progression to invasive breast cancer[J]. Am J Pathol, 2017, 187(7): 1648-1655. DOI:10.1016/j.ajpath.2017.03.012 |
| [13] |
Wang J, Gao P, Song Y, et al. Prognostic value of gastric cancerassociated gene signatures:Evidence based on a meta-analysis using integrated bioinformatics methods[J]. J Cell Mol Med, 2018, 22(11): 5743-5747. DOI:10.1111/jcmm.13823 |
| [14] |
Chuang YC, Wu HY, Lin YL, et al. Blockade of ITGA2 Induces Apoptosis and Inhibits Cell Migration in Gastric Cancer[J]. Biol Proced Online, 2018, 20: 10. DOI:10.1186/s12575-018-0073-x |
| [15] |
Gong J, Lu X, Xu J, et al. Coexpression of UCA1 and ITGA2 in pancreatic cancer cells target the expression of miR-107 through focal adhesion pathway[J]. J Cell Physiol, 2019, 234(8): 12884-12896. DOI:10.1002/jcp.27953 |
| [16] |
Yao H, Yang Z, Liu Z, et al. Glypican-3 and KRT19 are markers associating with metastasis and poor prognosis of pancreatic ductal adenocarcinoma[J]. Cancer Biomark, 2016, 17(4): 397-404. |
| [17] |
Saha SK, Kim K, Yang GM, et al. Cytokeratin 19(KRT19) has a role in the reprogramming of cancer stem cell-like cells to less aggressive and more drug-sensitive cells[J]. Int J Mol Sci, 2018, 19(5): E1423. DOI:10.3390/ijms19051423 |
| [18] |
Januchowski R, Zawierucha P, Rucinski M, et al. Extracellular matrix proteins expression profiling in chemoresistant variants of the A2780 ovarian cancer cell line[J]. Biomed Res Int, 2014, 2014: 365867. |
| [19] |
Linhares MM, Affonso RJ Jr, Viana Lde S, et al. Genetic and Immunohistochemical Expression of Integrins ITGAV, ITGA6, and ITGA3 As Prognostic Factor for Colorectal Cancer:Models for Global and Disease-Free Survival[J]. PLoS One, 2015, 10(12): e0144333. DOI:10.1371/journal.pone.0144333 |
 2020, Vol. 47
2020, Vol. 47